葡萄染色体制片技术优化及14个葡萄品种rDNA分布特征分析
2024-11-05于雪裴丹房经贵





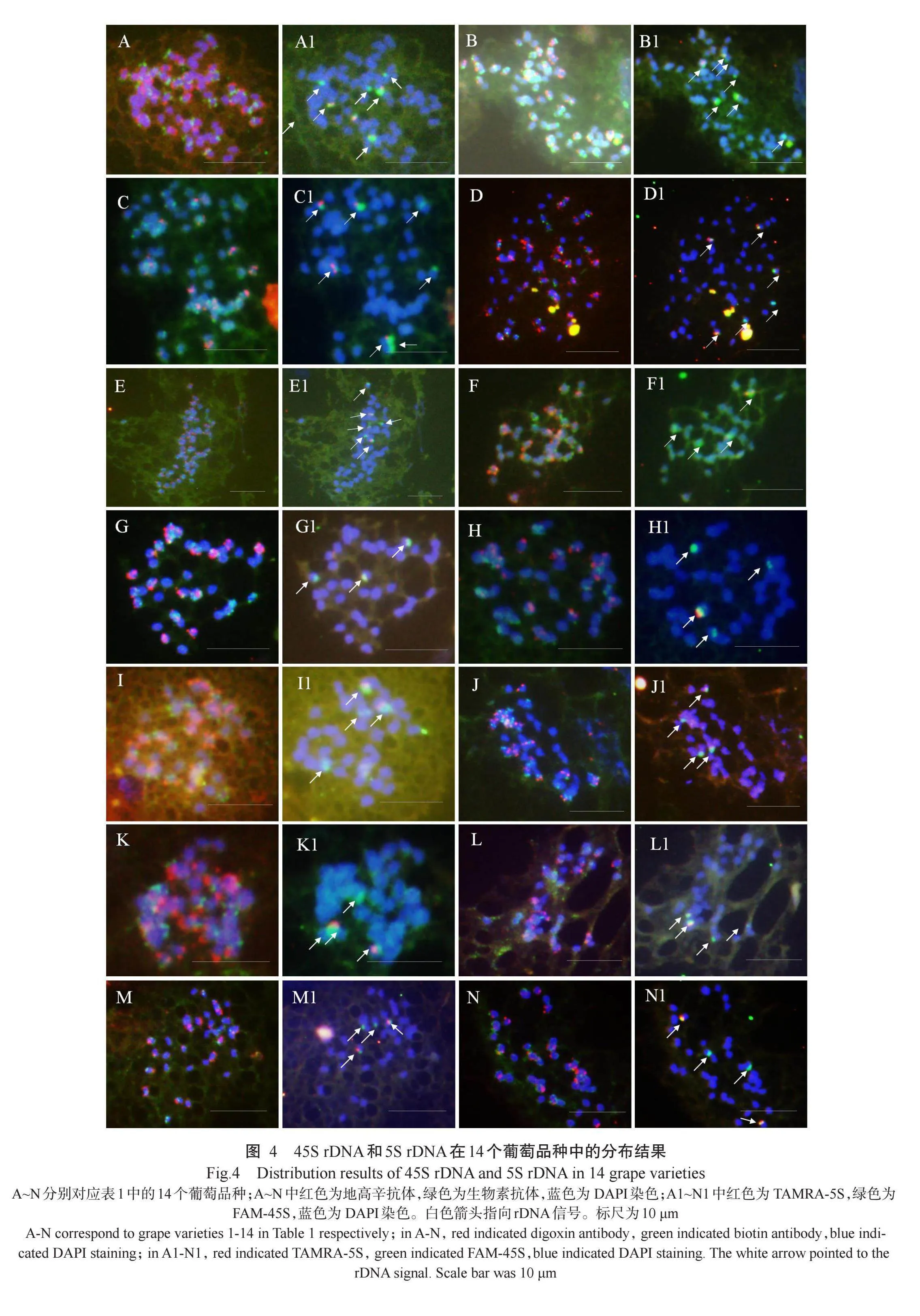
摘要:【目的】优化葡萄染色体制片技术并分析14个葡萄品种rDNA分布特征,为葡萄染色体鉴定、变异分析及葡萄品种间的细胞遗传学背景差异解析研究提供参考依据。【方法】以14个葡萄品种当年生半木质化枝条水培长出的根尖为试验材料,以冰水混合物处理根尖24 h后1 MPa笑气(N2O)处理30 min为对照,设0.2μmol/L的甲基氨草磷溶液(APM)浸泡根尖2 h后1 MPa N2O不同时长(0、30和60 min)处理,筛选染色体制片条件并计算各处理下形态良好的中期分裂相占比;比较酶解滴片法及火焰干燥法的染色体制片效果。以45S rDNA和5S rDNA为探针采用双色荧光原位杂交(FISH)技术分析rDNA在14个葡萄品种染色体上的分布特征。【结果】对照中葡萄根尖材料形态良好的中期分裂相占比仅为32.22%,FISH后易产生背景信号且信号模糊,采用APM处理2 h后1 MPa N2O处理30 min得到的染色体制片形态良好的中期分裂相占比最高,达80.00%,且染色体浓缩适当,FISH信号质量良好。采用火焰干燥法获得的制片细胞分散程度适中,中期分裂相多。45S rDNA和5S rDNA在葡萄染色体上始终呈连锁状态;5S rDNA信号与染色体组数目相关,在二、三、四倍体中的数量分别为2、3和4个。45S rDNA信号在二、三、四倍体中的数量分别为4、6和7个,在红香蕉葡萄中只有3个;5S rDNA信号位于17号染色体上,45S rDNA位于17和15号染色体。意大利和碧香无核葡萄例外,意大利葡萄2个未与5S rDNA连锁的45S rDNA位于15和16号染色体,碧香无核葡萄1个与55 rDNA连锁的45 rDNA位于15号染色体,1个未与55 rDNA连锁的45S rDNA位于17号染色体上。【结论】制作葡萄根尖中期染色体制片较优方法是APM预处理2 h后1 MPa N2O处理30 min,使用火焰干燥法制片。45S rDNA和5S rDNA在葡萄染色体上呈连锁排列,在二、三、四倍体中数量不同。14个葡萄品种的45S rDNA和5S rDNA在染色体上呈L形排列,5S rDNA位点数量比45S rDNA位点数量更稳定。
关键词:葡萄;染色体制片;5S rDNA;45S rDNA;荧光原位杂交
中图分类号:S663.1文献标志码:A文章编号:2095-1191(2024)08-2351-09
Optimization of grape chromosome preparation and rDNA distribution characteristics in 14 grape varieties
YU Xue,PEI Dan,FANG Jing-gui*
(College of Horticulture,Nanjing Agricultural University,Nanjing,Jiangsu 210095,China)
Abstract:【Objective】This study aimed to optimize grape chromosome preparation technique and analyze the rDNA distribution characteristics in 14 grape varieties,providing reference for grape chromosome identification,variation analysis,and analysis of cytogenetic background differentiation among grape varieties.【Method】The root tips of 14 grape varieties from hydroponically grown semi-lignified shoots were used as experimental materials.A control group treating root tips with an ice-water mixture for 24 h,followed by 1 MPa nitrous oxide(N2O)treatment for 30 min was set.The root tips were soaked in 0.2μmol/L amiprophos-methyl solution(APM)for 2 h,and treated with 1 MPa N2O for different durations(0,30,and 60 min).Chromosome preparation conditions were screened,and the proportion of meta-phase splitting phases in good shape under each treatment was calculated.The chromosome preparation effects of enzy-matic digestion and dropping method and the flame-drying method were compared.The distribution characteristics ofrDNA on chromosomes of 14 grape varieties were analyzed using dual-color fluorescence in situ hybridization(FISH)technique with 45S rDNA and 5S rDNA as probes.【Result】The proportion of metaphase splitting phases in good shape in grape root tip materials was only 32.22%in the control group.After FISH,background signals were easily generated and the signals were blurred.The highest proportion of metaphase splitting phases in good shape(80.00%)in chromosome slides was obtained by treating root tips with APM for 2 h and with 1 MPa N2O for 30 min.The chromosome concentra-tion was appropriate,and the quality of FISH signals was good.The flame-drying method resulted in moderate cell disper-sion and a high number of metaphase splitting phases.45S rDNA and 5S rDNA signals were consistently linked on grape chromosomes.The number of 5S rDNA signals was correlated with the ploidy level,with 3 and 4 signals observed in diploid,triploid and tetraploid grapes respectively.The number of 45S rDNA signals was 4,6 and 7 in diploid,triploid and tetraploid grapes respectively,except for Hongxiangjiao grape,which had only 3 signals.The 5S rDNA signals were located on chromosome 17,while the 45S rDNA signals were located on chromosomes 17 and 15.Exceptions were ob-served in Italy grape and Bixiang seedless grape with two 45S rDNA signals unlinked to 5S rDNA signals located on chro-mosomes 15 and 16 in Italy grape,and one 45S rDNA signal linked to 5S rDNA on chromosome 15 and one 45S rDNA signal unlinked to 5S rDNA on chromosome 17 in Bixiang seedless grape.【Conclusion】The optimal method for preparing metaphase chromosomes of grape root tips is pretreatment with APM for 2 h,and 1 MPa N2O for 30 min,using the flame-drying method for slide preparation.The 45S rDNA and 5S rDNA are linked on grape chromosomes,with varying num-bers in diploid,triploid,and tetraploid grapes.The 45S rDNA and 5S rDNA of 14 grape varieties exhibited an L-shape arrangement on the chromosomes,and the number of 5S rDNA loci is more stable than that of45S rDNA loci.
Key words:grape;chromosome preparation;5S rDNA;45S rDNA;fluorescence in situ hybridization(FISH)
Foundation items:National Natural Science Foundation of China(32272647);Shandong Key Research and Deve-lopment(Agricultural Breeding Engineering)Project(2022LZGCQY1018)
0引言
【研究意义】葡萄(Vitis vinifera)是营养价值和经济效益较高的果树,在世界各地均有种植(马龙等,2023)。葡萄染色体组有19个,基因组为500 Mb,是染色体最小的植株之一,因此难以识别体细胞中单个染色体。荧光原位杂交(Fluorescence in situ hybridization,FISH)技术能实现DNA序列在染色体上的物理定位,在植物分子细胞遗传学研究领域已得到广泛应用。45S rDNA和5S rDNA等重复DNA序列在植物基因组中含量较高,是FISH探针的重要来源(Mehrotra and Goyal,2014;Jiang,2019)。与葡萄相关的研究中,利用FISH技术进行细胞分子遗传学的研究较少。因此,优化葡萄染色体制片技术并分析其rDNA分布特征,对葡萄染色体鉴定、变异分析及葡萄品种间的细胞遗传学背景差异解析研究具有重要意义。【前人研究进展】FISH染色体制片过程分为染色体聚集、固定和制片。染色体聚集在根尖预处理阶段完成,不同植物的聚集方法有所差别,玉米及其亲缘物种经甲基氨草磷溶液(APM)浸泡和0.8~1.2 MPa笑气(N2O)处理0.5~1.5 h更容易获得分裂相较多且形态较好的染色体(姚乐沙,2021);纤毛鹅观草用APM处理2.0~2.5 h,0.8~1.0 MPa N2O处理1.5~2.0 h后效果较好,可进一步完善染色体鉴定体系(程梦豪等,2022);苹果则是使用冰水混合物和卡诺固定液后的染色体制片效果最佳(韩亚慧等,2024)。制片方法主要分为酶解滴片法、火焰干燥法、冰冻压片法和酶解去壁低渗压片法。FISH对制片的要求包括染色体分裂相多、形态好、分散和暴露等。Lou等(2014)研究发现采用冰冻压片法染色体易变性,采用酶解滴片法和火焰干燥法更利于染色体保持原始形态,且酶解可去除细胞壁,有利于染色体暴露。杜培(2017)以小麦为试验材料,使用火焰干燥法压片后进行FISH试验,开发并应用了新的寡核苷酸套。Liu等(2024)利用酶解滴片法制片,从基因组学和细胞遗传学角度研究了5份秋葵(Abel-moschusesculentus)种质中的卫星重复特征。染色体制片后需与探针杂交后才能进行FISH分析。FISH技术在植物基因组分析和改良、染色体组成和结构、杂种和种属鉴定等研究中被广泛应用,rDNA序列探针作为FISH探针的重要来源同样应用广泛。Ding等(2016)在对蔷薇的研究中采用45S rDNA探针进行FISH试验,观察rDNA位点数量的差异,结果发现rDNA位点在杂交事件后表现出不稳定性,推测导致rDNA变异的主要因素为染色体配对异常。He等(2021)利用rDNA序列设计寡核苷酸探针,开展8份菊花种质FISH试验,探究了菊花的进化历史和系统发育进化关系。Lin等(2022)研究发现甘蔗基因组复杂,传统育种方法很难在遗传水平上进行品种改良,而利用rDNA序列进行FISH能在甘蔗育种中快速确定野生甘蔗关系并进行倍性的有效标记。Mitrenina等(2023)开发并测试了45S rDNA和5S rDNA探针,对4个毛茛科品种进行了比较核型分析。Tomlekova等(2024)在菜豆研究中分析rDNA序列在染色体上的大小和位置,结果发现其核基因组大小发生变化,包括1个突变系基因组的增加。在有关葡萄的研究中,学者使用重复序列45SrDNA和5SrDNA探针进行FISH,结果发现二倍体葡萄品种中存在4个45S rDNA和2个5S rDNA位点(Pereira et al.,2005;Falistocco et al.,2007;Houel et al.,2010;Pereira et al.,2014)。【本研究切入点】FISH技术在果树研究领域的应用尚未完善,有关优化葡萄染色体制片技术及葡萄品种rDNA分布特征分析的研究也鲜有报道。【拟解决的关键问题】以葡萄属二、三和四倍性种质为试验材料,优化葡萄染色体制片技术,并通过FISH技术分析45S rDNA和5S rDNA在葡萄属植物染色体上的分布情况,为葡萄染色体鉴定、变异分析及葡萄品种间的细胞遗传学背景差异解析研究提供参考依据。
1材料与方法
1.1试验材料
供试葡萄品种及来源信息见表 其中13个品种引自中国农业科学院郑州果树研究所,1个品种引自沈阳农业大学。以14个葡萄品种当年生半木质化枝条水培长出的根尖为试验材料。
1.2试验方法
1.2.1探针设计与标记根据PN40024基因组数据设计并合成了4组共91280个寡核苷酸,包含位于葡萄基因组的40个特定染色体区域的单拷贝序列。寡核苷酸大量平行合成并标记为FISH探针,可用于识别葡萄全部19对染色体,本研究利用的是可识别9~19号染色体的寡核苷酸探针套(图1)。寡核苷酸文库由Arbor Biosciences(Ann Arbor,MI)合成。每个合成的文库含有200 ng DNA。将文库稀释至1 ng/μL后作为储备库于-80℃永久保存,再次稀释至0.07 ng/μL作为工作液,可短期存放于-20℃冰箱。工作液制备探针步骤为去泡沫PCR、产物纯化、离体转录、RNA纯化、反转录—探针标记和酶解法去除RNA酶。
在Rfam(http://rfam.xfam.org/)平台下载不同物种的45S rDNA(5.8S:FN597020.1;18S:FN597032.1;28S:FN597017.1)和5S rDNA(FN597042.1)序列;使用DNAMAN 9.0完成14个葡萄的同源多序列比对,参数选择Quick alignment;使用Oligo 7(Rychlik,2007)生成具有较高重复序列数的单元为探针,参数选择为Hybridization Probes;Search Stringency:High;Length:30~59 nt。探针由通用生物(安徽)股份有限公司修饰合成。
1.2.2预处理条件筛选及染色体制片与杂交葡萄根尖细胞中期染色体制片参照Pei等(2024)的方法并稍作改动。挑选长势旺盛的14个葡萄品种新根,剪掉其前端1.5~2.0 cm。一部分根系放入冰水混合物中处理24 h,在保持根系湿润的同时用1 MPa N2O处理30 min为对照。另一部分根系放入0.2μmol/L的APM(溶剂为丙酮)置于25℃培养箱浸泡2 h,之后将材料放入1.5 mL离心管中,在保持根系湿润的同时用1 MPa N2O处理0、30和60min。
火焰干燥法制片流程:葡萄新根于45%乙酸溶液中解离10min,用刀片切取根尖分生区组织,加入1滴45%乙酸溶液,盖上盖玻片后用镊子轻敲几下盖玻片使细胞分散开,利用酒精灯外焰烘烤制片,待载玻片上雾气散开后压片。相差显微镜(上海光学仪器厂)观察染色体制片,选取染色体形态好、分散均匀、数目完整的制片放入-80℃冰箱冷冻保存。制片冷冻过夜后揭去盖玻片,置于无水乙醇中脱水30~40 min,气干后即可用于原位杂交试验。
酶解滴片法制片流程:将预处理后的根在培养皿中使用ddH2O清洗1 min,取出并吸去多余水分,切取根尖分生区放入装有纤维素酶和果胶酶的酶解液离心管中,将离心管放入37℃保温箱中37 min,分别加入2次纯水和2次无水乙醇清洗干净残留酶,若期间根尖破碎,可再次离心,清洗后倒置晾干。最后向离心管中加入适量冰乙酸,用针将根尖打碎,滴加到保湿盒中事先标记好的玻片上,盖上保湿盒盖子,室温静置30min后进行后续试验。
杂交程序参照杜培(2017)的方法并稍作改动。制片杂交液配置:dFA 7.5μL、20×SSC 1.5μL、Salmon sperm DNA 1.0μL、Oligo Probes 1.0μL、50%Dex-tran Sulfate 2.0μL、Library 3&4探针各3.0μL。配置完成后,振荡混匀并离心2次,105℃金属浴加热13 min,加热结束后立即放入-20℃无水乙醇中静置10 min。载玻片上滴加70%dFA 50μL(配置方法:dFA 700μL+ddH2O 300μL),盖上盖玻片,于75℃的拷片机上变性70 s,随后在-20℃的70%、95%和100%的酒精中梯度脱水5min,吹干载玻片后将杂交液滴加在载玻片细胞区域,盖上盖玻片,置于湿盒中,37℃培养箱孵育,单拷贝探针杂交孵育至少24h,重复序列探针杂交孵育至少6h。
1.2.3洗片与镜检先进行单拷贝寡核苷酸探针库构建,观察照相完毕后将制片浸泡于1×磷酸盐缓冲液(PBS)中轻轻摇晃去除盖玻片,控干水分后在LED灯光下照射12h,放入100%无水乙醇中30~40 min。然后用45S rDNA和5S rDNA寡核苷酸探针进行第二次杂交。其他步骤参照裴丹(2023)的方法。应注意重复序列探针与寡核苷酸探针洗片流程不同,重复序列探针的洗片流程:揭掉盖玻片,在42℃水浴锅中使用2×SSC清洗2次,每次5 min,之后在ddH2O中清洗1 min,气干后滴加7μL封片胶[含4',6-二脒基-2-苯基吲哚(DAPI)],最后加盖玻片,轻轻按压后在Olympus BX60荧光显微镜下镜检,用SPOT CCD摄取图像。每材料至少观察5个细胞。染色体分散程度适中、浓缩适当即为形态良好。
1.3统计分析
试验数据采用Excel 2016进行计算分析,使用Adobe Photoshop 6.0进行图片处理。
2结果与分析
2.1不同预处理对葡萄根尖细胞染色体制片效果的影响
在葡萄根尖预处理试验中,每处理制备10张染色体制片,选择其中染色体数量多、分散程度好的3张制片进行统计分析。由表2可知,APM预处理2 h组(1 MPa N2O处理30、60 min)形态良好的中期分裂相占比高于冰水混合物预处理24 h;冰水混合物预处理24 h形态良好的中期分裂相占比仅32.22%。1 MPa N2O不同时间预处理组中,处理30 min形态良好的中期分裂相占比最高,达到80.00%。由图2可知,冰水混合物与APM预处理后的染色体形态相似,但冰水混合物处理的FISH后易产生背景信号且信号模糊。在APM预处理下,1 MPa N2O预处理0 h组,染色体较长且浓缩不充分,FISH信号质量差;1 MPa N2O预处理30 min组,染色体浓缩适当,FISH信号质量良好;1 MPaN2O预处理60min组,染色体浓缩适当,但FISH信号质量差。因此,综合考虑染色体分散程度、染色体形态及FISH信号质量,选择APM处理2 h后1 MPa N2O处理30 min为葡萄根尖预处理最适条件。
2.2 2种染色体制片方法效果比较结果
选用APM处理2h后1 MPa N2O处理30 min的根尖,染色体制片形态良好的中期分裂相占比高,且染色体浓缩适当开展后续试验。分别使用火焰干燥法和酶解滴片法制片,结果如图3可知,火焰干燥法获得的制片,细胞分散程度适中,中期分裂相多(图3-A);酶解滴片法获得的制片,细胞过于分散且中期分裂相少(图3-B)。
2.3 45S rDNA和5S rDNA在14个葡萄品种中的分布结果
由图4可知,采用优化后的制片技术进行FISH,45S rDNA和5S rDNA探针在二、三和四倍体葡萄染色体上均产生信号。杂交结果显示,45S rDNA与5S rDNA始终呈连锁状态,属于L形排列,分布在相同的染色体上,但二者在染色体上的数目与位置并不固定。3个四倍体品种均有4个5S rDNA信号,位于17号染色体;7个45S rDNA信号,位于17和15号染色体。2个三倍体品种均有3个5S rDNA信号,位于17号染色体;6个45S rDNA信号,位于17和15号染色体。二倍体品种中,北红、贵妃玫瑰、葡萄园皇后、沈阳玫瑰、郑州早玉、左山一号葡萄均有2个5S rDNA信号,位于17号染色体;4个45S rDNA信号,位于17和15号染色体。而二倍体品种红香蕉有2个5S rDNA信号,位于17号染色体;3个45S rDNA信号,位于17和15号染色体。以上葡萄品种与5S rDNA连锁的45S rDNA均位于17号染色体,未与5S rDNA连锁的45S rDNA位于15号染色体。意大利和碧香无核rDNA数量与其他二倍体相同,分布位置不同。意大利2个未与5S rDNA连锁的45S rDNA位于15和16号染色体,碧香无核1个与5S rDNA连锁的45S rDNA位于15号染色体,1个未与5S rDNA连锁的45S rDNA位于17号染色体。
3讨论
分子细胞遗传学研究的基础是获得高质量的染色体制片,制片要求染色体分散性好、形态良好、浓缩适当、分裂相无细胞质背景等,以便进行FISH试验(郭瑞红等,2021)。根尖预处理及制片方法是影响染色体制片的主要因素(杜培,2017)。冰水处理是一种将根尖细胞停滞在中期的处理方法,尽管冰水处理在中期积累同步细胞的有效性低,但其流程简单易操作,且在某些品种中积累效果显著。苹果、朱槿和蜀葵根尖染色体制片预处理第一步在冰水混合物中预处理效果显著(肖苏芯等,2021;Rachma et al.,2023;韩亚慧等,2024)。本研究中,冰水混合物预处理24 h,采用1 MPa笑气(N2O)处理30 min中形态良好的中期分裂相占比仅32.22%,FISH后易产生背景信号且信号模糊,与上述前人研究结果不符。本研究采用APM预处理2 h后1 MPa N2O处理30 min得到的染色体制片形态良好的中期分裂相占比最高,达到80%,且染色体浓缩适当、FISH信号质量良好,可用于进一步的染色体分析。与孔令娜等(2020)研究发现使用APM预处理2 h后进行后续FISH试验效果较好;郭瑞红等(2021)在绣球根尖染色体制片预处理第二步中发现绣球根尖预处理的最适条件为1 MPa N2O处理1 h的研究结果相一致。预处理后的葫芦科、茶树和小麦使用酶解滴片法进行染色体制片效果最佳(Evtushenko etal.,2019;刘昱希等,2020;喻雪莲等,2024),但葡萄染色体小,酶解滴片法容易丢失染色体,本研究发现使用火焰干燥法细胞分散程度适中,中期分裂相多,可以获得质量更高的染色体制片。
W6IibpsFOvExjunvR2eSFw==真核生物中存在两种类型的rDNA,即45SrDNA和5S rDNA,二者位置分为连锁(L形)和分离(S形),L形分布在同一条染色体上,S形则相反。在大多数动植物中,45S rDNA与5S rDNA呈S形排列,少数呈L形排列,如黄瓜(Zhang et al.,2016;Gar‐cia et al.,2017)呈L形排列。徐川梅等(2022)在比较12个竹种的45S rDNA和5S rDNA分布特性时发现,部分竹种45S rDNA和5S rDNA呈L形排列。本研究中所有葡萄品种的5S rDNA与45S rDNA均属于L形排列,分布于相同的染色体上。
5S rDNA位点的数量比45S rDNA位点的数量更稳定,后者有时变化很大,rDNA位点个体间的变异有可能被用作群体遗传学标记(Olanjet al.,2015)。在广泛的分类范围内,5S rRNA基因编码的序列高度保守,其DNA位点的数量与分布多与染色体的倍性联系在一起(Olanjet al.,2015)。本研究中二、三、四倍体中5S rDNA位点数量分别为2、3和4个,表明在部分葡萄品种中,可以将5S rDNA位点与染色体的倍性联系在一起。另外,5S rDNA位点数量稳定,随着染色体组数量的增加而增加,反映了整个基因组的加倍。在菜豆中分析rDNA序列在染色体上的大小及位置,发现其核基因组大小发生变化,包括1个突变系基因组的增加(TomLekova et al.,2024)。本研究中,二、三、四倍体葡萄上45S rDNA位点数分别为4(红香蕉为3个)、6和7个,与5S rDNA位点分布相比,45S rDNA分布模式彼此间存在较大差异。大量试验证实45S rDNA为脆性位点,脆性位点表现为在前中期和中期染色体上的染色质纤维上的非随机不完全断裂或缺口,目前为止,由易损位点引起的染色体损伤在人类中最为常见,植物染色体断裂明显较少,且几乎完全限制在45S rDNA位点上(Lan et al.,2016;黄敏等,2017)。红香蕉葡萄品种中缺失1个45S rDNA位点,意大利和碧香无核葡萄的1个45S rDNA位点分布在其他染色体上,推测是由染色体结构变异导致。植物基因组中,rDNA区域是遗传重组的热点区域,同源染色体间的重组或不等交换往往导致rDNA拷贝数产生差异(Tsang and Carr,2008)。此外,rDNA重复序列内部及邻近区域转座子的插入或删除进一步促进了rDNA位点的进化,使物种的核型不断变化(Pedrosa-Harand et al.,2006;Raskina et al.,2008)。
4结论
制作葡萄根尖中期染色体制片较优方法是APM预处理2h后1 MPa N2O处理30min,使用火焰干燥法制片。45S rDNA和5S rDNA在葡萄染色体上连锁排列,在二、三、四倍体中数量不同。14个葡萄品种的45S rDNA和5S rDNA在染色体上呈L形排列,5S rDNA位点数量比45S rDNA位点数量更稳定。
参考文献(References):
程梦豪,Miroslava Karafiátová,孙昊杰,Kateřina Holušová,Jaroslav Doležel,宋新颖,王海燕,王秀娥.2022.基于纤毛鹅观草特异的卫星重复序列开发寡核苷酸探针[J].南京农业大学学报,45(3):442-452.[Cheng MH,KarafiátováM,Sun H J,HolušováK,Doležel J,Song X Y,Wang H Y,Wang X E.2022.Development of oligonucleotide probes specific to Roegneria ciliaris chromosomes based on satellite repeats[J].Journal of Nanjing Agricultural Uni-versity,45(3):442-452.]doi:10.7685/jnau.202107035.
杜培.2017.小麦、百萨偃麦草和花生染色体荧光原位杂交寡核苷酸探针(套)开发与应用[D].南京:南京农业大学.[Du P.2017.Development and application of Wheat,Thi-nopyrumBessarabicum and Peanut oligonucleotide and multiplex probes for fluorescence in Situ hybridization[D].Nanjing:Nanjing Agricultural University.]doi:10.27244/d.cnki.gnjnu.2017.000137.
郭瑞红,邱帅,刘光欣,高凯,魏建芬,席梦丽.2021.绣球染色体制片技术优化及rDNA物理定位[J].林业科学,57(11):59-67.[Guo R H,Qiu S,Liu G X,Gao K,Wei J F,Xi M L.2021.Optimization of chromosome preparation and rDNA physical localization of Hydrangea macrophylla[J].Scientia SilvaeSinicae,57(11):59-67.]doi:10.11707/j.1001-7488.20211106.
韩亚慧,贺宇,张春芬,邓舒,肖蓉,曹秋芬.2024.嘎啦苹果花药培养再生植株根尖染色体制片体系的建立[J/OL].分子植物育种.https://kns.cnki.net/kcms2/detail/46.1068.S.20230710.1911.005.htm.[Han Y H,He Y,Zhang C F,Deng S,Xiao R,Cao Q F.2024.Establishment of chromo‐some production system for root tip of regenerated anther culture plantlets of Gala apple[J/OL].Molecular Plant Breeding.https://kns.cnki.net/kcms2/detail/46.1068.S.202 30710.1911.005.htm.]
黄敏,周玲,黄婧,徐安莉,王平.2017.45S rDNA基因的不稳定性及其与转录的相关性[J].激光生物学报,26(2):151-155.[Huang M,Zhou L,Huang J,Xu A L,Wang P.2017.Instability of 45S rDNA gene and its correlation with transcription[J].Acta Laser Biology Sinica,26(2):151-155.]doi:10.3969/j.issn.1007-7146.2017.02.010.
孔令娜,冯祎高,孙冰晓,张慧,刘晓雪,陈颖,张瑞奇.2020.基于改进的多色荧光原位杂交技术的染色体分析[J].中国农学通报,36(12):97-103.[Kong L N,Feng Y G,Sun B X,Zhang H,Liu X X,Chen Y,Zhang R Q.2020.Chro‐mosome analysis based on an improved multiplex fluores‐cence in situ hybridization(M-FISH)[J].Chinese Agricul‐tural Science Bulletin,36(12):97-103.]
刘昱希,韩兰英,李梦雪,赵勤政,陈劲枫,娄群峰.2020.葫芦科植物幼嫩子房壁制片技术的优化及其染色体倍性鉴定[J].西北植物学报,40(5):882-887.[Liu Y X,Han L Y,Li M X,Zhao Q Z,Chen J F,Lou Q F.2020.Optimiza‐tion of chromosome preparation method using young ovary wall of Cucurbitaceae plants and pioidyidentifica‐tion[J].Acta Botanica Boreali-Occidentalia Sinica,40(5):882-887.]doi:10.7606/j.issn.1000-4025.2020.05.0882.
马龙,徐美隆,范培格,乔改霞,刘玉娟,王荣,谢军,马东海.2023.干旱胁迫下砧木‘110R’对黑比诺'葡萄抗旱性的影响[J].中外葡萄与葡萄酒,(6):18-25.[Ma L,Xu M L,Fan P G,Qiao G X,Liu Y J,Wang R,Xie J,Ma D H.2023.Effects of rootstock‘110R’on drought resistance of'PinotNoir'grapevine under drought stress[J].Sino-Overseas Grapevine&Wine,(6):18-25.]doi:10.13414/j.cnki.zwpp.2023.06.003.
裴丹.2023.葡萄染色体荧光原位杂交寡核苷酸探针开发与利用[D].南京:南京农业大学.[Pei D.2023.Develop‐ment and application of oligonucleotide probes for fluore-sence in situ hybridization in Vitis chromosomes[D].Nan‐jing:Nanjing Agricultural University.]
肖苏芯,黄依然,高丽,贾桂霞,高雪.2021.蜀葵根尖染色体制片技术优化和核型分析[J].分子植物育种,19(17):5800-5807.[Xiao S X,Huang Y R,Gao L,Jia G X,Gao X.2021.Optimization of chromosome tableting techno-logy and karyotype analysis of Althaea rosea root tips[J].Molecular Plant Breeding,19(17):5800-5807.]doi:10.13271/j.mpb.019.005800.
徐川梅,王子晗,谢峰,金旭胤,吴心妍,陈荣,黄坚钦.2022.12个竹种的45S rDNA和5S rDNA分布特性比较[J].林业科学,58(7):93-102.[Xu C M,Wang Z H,Xie F,Jin X Y,Wu X Y,Chen R,Huang J Q.2022.Comparison of 45SrDNA and 5S rDNA distribution characteristics of 12 bam‐boo species[J].Scientia SilvaeSinicae,58(7):93-102.]doi:10.11707/j.1001-7488.20220710.
姚乐沙.2021.玉米及亲缘物种寡核苷酸探针开发与应用[D].南京:南京农业大学.[Yao L S.2021.Development and application of oligonucleotide probes for maize andrelated species[D].Nanjing:Nanjing Agricultural Univer‐sity.]doi:10.27244/d.cnki.gnjnu.2021.001356.
喻雪莲,李兴锋,张丽霞.2024.茶树染色体制片技术优化[J/OL].分子植物育种.https://link.cnki.net/urlid/46.1068.s.20231218.1911.024.[Yu X L,Li X F,Zhang L X.2024.Technical optimization of chromosome preparation in Camellia sinensis[J/OL].Molecular Plant Breeding.https://link.cnki.net/urlid/46.1068.s.20231218.1911.024.]
Ding X L,Xu T L,Wang J,Luo L,Yu C,Dong G M,Pan H T,Zhang Q X.2016.Distribution of 45S rDNA in modernrose cultivars(Rosa hybrida),Rosa rugosa,and theirinterspecific hybrids revealed by fluorescence in situ hybridization[J].Cytogenetic and Genome Research,149(3):226-235.doi:10.1159/000448063.
Evtushenko E V,Lipikhina YA,Stepochkin P I,Vershinin AV.2019.Cytogenetic and molecular characteristics of rye genome in octoploid triticale(×TriticosecaleWittmack)[J].Comp Cytogenet,13(4):423-434.doi:10.3897/Comp-Cytogen.v 13i4.39576.eCollection 2019.
Falistocco E,Passeri V,Marconi G.2007.Investigations of 5S rDNA of Vitis vinifera L.:Sequence analysis and physical mapping[J].Genome,50(10):927-938.doi:10.1139/g07-070.
Garcia S,KovaříkA,Leitch A R,Garnatje T.2017.Cytogenetic features of rRNA genes across land plants:Analysis of the plant rDNA database[J].The Plant Journal,89(5):1020-1030.doi:10.1111/tpj.13442.
He J,Lin S S,Yu Z Y,SongAP,Guan Z Y,Fang W M,Chen S M,Zhang F,Jiang J,Chen F D,Wang H B.2021.Identifi‐cation of 5S and 45S rDNA sites in Chrysanthemum spe‐cies by using oligonucleotide fluorescence in situ hybridi-zation(Oligo-FISH)[J].Molecular Biology Reports,48(1):21-31.doi:10.1007/s 11033-020-06102-1.
Houel C,Bounon R,Chaïb J,Guichard C,Peros J P,Bacilieri R,Dereeper A,Canaguier A,Lacombe T,N'Diaye A,Le Paslier M C,Vernerey M S,Coriton O,Brunel D,This P,Torregrosa L,Adam-Blondon A F.2010.Patterns of sequence polymorphism in the fleshless berry locus in cul‐tivated and wild Vitis vinifera accessions[J].BMC Plant Biology,10:284.doi:10.1186/1471-2229-10-284.
Jiang J J.2019.Fluorescence in situ hybridization in plants:Recent developments and future applications[J].Chromo‐some Reseach,27(3):153-165.doi:10.1007/s 10577-019-09607-z.
Lan H,Chen C L,Miao Y,Yu C X,Guo W W,Xu Q,Deng X X.2016.Fragile sites of‘Valencia’sweet orange(Citrus sinensis)chromosomes are related with active 45s rDNA[J].PLoS One,11(3):e0151512.doi:10.1371/journal.pone.0151512.
Lin P,Hu X,Xue L,Li X,Wang P,Zhao X,Zhang M,Deng Z,Yu F.2022.Identification of sugarcane S.spontaneum(Poaceae)germplasm:Evidence from rDNA-ITS andrDNA locus analyses[J].Agronomy,12(12):3167.doi:10.3390/agronomy 12123167.
Liu J R,Lin X Y,Wang X J,Feng L Q,Zhu S X,Tian R M,Fang J P,Tao A F,Fang P P,Qi J M,Zhang LW,Huang Y J,Xu J T.2024.Genomic and cytogenetic analyses reveal satellite repeat signature in allotetraploid okra(Abelmos-chusesculentus)[J].BMC Plant Biology,24(1):71.doi:10.1186/s 12870-024-04739-9.
Lou Q F,Zhang Y X,He Y H,Li J,Jia L,Cheng C Y,Guan W,Yang S Q,Chen J F.2014.Single-copy gene-based chro-mosome painting in cucumber and its application for chro-mosome rearrangement analysis in Cucumis[J]w7IFczOegYqxWee3aARxlw==.The Plant Journal,78(1):169-79.doi:10.1111/tpj.12453.
Mehrotra S,Goyal V.2014.Repetitive sequences in plant nuclear DNA:Types,distribution,evolution and function[J].Genomics Proteomics Bioinformatics,12(4):164-171.doi:10.1016/j.gpb.2014.07.003.
Mitrenina E Y,Alekseeva S S,Badaeva E D,Peruzzi L,Arte-mov G N,Krivenko D A,Pinzani L,AytaçZ,ÇeçenÖ,Baasanmunkh S,Choi H J,Mesterházy A,Tashev A N,BanchevaS,Lian L,Xiang K L,Wang W,Erst A S.2023.Karyotypes and physical mapping of ribosomal DNA with oligo-probes in Eranthissect.Eranthis(Ranunculaceae)[J].Plants,13(1):47.doi:10.3390/plants 13010047.
Olanj N,Garnatje T,Sonboli A,Vallès J,Garcia S.2015.The striking and unexpected cytogenetic diversity of genus Tanacetum L.(Asteraceae):A cytometric and fluorescent in situ hybridisation study of Iranian taxa[J].BMC Plant Biology,15:174.doi:10.1186/s 12870-015-0564-8.
Pedrosa-Harand A,de Almeida C C S,Mosiolek M,Blair M W,Schweizer D,Guerra M.2006.Extensive ribosomal DNA amplification during Andean common bean(Phaseolus vul-garis L.)evolution[J].Theoretical and Applied Genetics,112(5):924-933.doi:10.1007/s00122-005-0196-8.
Pei D,Yu X,Fu W H,Ma X H,Fang J G.2024.The evolution and formation of centromeric repeats analysis in Vitis vinifera[J].Planta,259(5):99.doi:10.1007/s00425-024-04374-6.
Pereira H S,Barão A,Delgado M,Morais-Cecílio L,Viegas W.2005.Genomic analysis of Grapevine Retrotransposon 1(Gret 1)in Vitis vinifera[J].Theoretical and Applied Gene-tics,111(5):871-878.doi:10.1007/s00122-005-0009-0.
Pereira H S,Delgado M,AvóA P,Barão A,Serrano I,Viegas W.2014.Pollen grain development is highly sensitive to temperature stress in Vitis vinifera[J].Australian Jouranl of Grape and Wine Research,20:474-484.doi:10.1111/ajgw.12105.
Rachma I,Dwiranti A,Salamah A.2023.Optimization of Hi-biscus rosa-sinensis L.chromosome pretreatment[C].AIP Conference Proceedings,0RQv1jl2laqgCppBzp4LukQ==20149.doi:10.1063/1.5064146.
Raskina O,Barber J C,Nevo E,Belyayev A.2008.Repetitive DNA and chromosomal rearraraskina:Speciation-related events in plant genomes[J].Cytogenetic and Genome Re-search,120(3-4):351-357.doi:10.1159/000121084.
Tomlekova N,Idziak-Helmcke D,Franke P,Rojek-Jelonek M,Kwasniewska J.2024.Phaseolus vulgaris mutants reveal variation in the nuclear genome[J].Front in Plant Science,14:1308830.doi:10.3389/fpls.2023.1308830.
Tsang E,Carr A M.2008.Replication fork arrest,recombina-tion and the maintenance of ribosomal DNA stability[J].DNA Repair,7(10):1613-1623.doi:10.1016/j.dnarep.2008.06.010.
Zhang Z T,Yang S Q,Li Z A,Zhang Y X,Wang Y Z,Cheng C Y,Li J,Chen J F,Lou Q F.2016.Comparative chromo-somal localization of 45S and 5S rDNAs and implications for genome evolution in Cucumis[J].Genome,59(7):449-457.doi:10.1139/gen-2015-0207.
(责任编辑李洪艳)
