基因沉默番木瓜环斑病毒复制酶基因(PRSV-Nib)获得抗病毒病番木瓜的研究
2024-06-09吴清铧贾瑞宗郭静远杨牧之胡玉娟郝志刚赵辉郭安平
吴清铧 贾瑞宗 郭静远 杨牧之 胡玉娟 郝志刚 赵辉 郭安平


关键词:番木瓜;番木瓜环斑病毒;Nib 基因;RNA 介导的病毒抗性
中图分类号:S436.67 文献标志码:A
番木瓜(Carica papaya L.)是热带和亚热带地区重要的经济水果之一,其富含丰富的营养价值与潜在的医疗价值,深受大众喜爱。番木瓜最主要的病害是番木瓜环斑病毒病(Papaya ringspotvirus, PRSV)[1-2],对番木瓜产业造成重大打击。传统的病毒防治措施如交叉保护[3-4],田间综合管理[5],传播媒介防治[6-7],以及抗病育种[8-9]在一定程度上阻止了病毒的传播,但这并不足以避免农业生产上的损失[10]。通过基因工程提高番木瓜的抗病能力,使得转基因番木瓜成为早期商业化种植的成功案例之一。
根据“致病菌衍生的抗病性(pathogen-derivedresistance, PDR)”原理,第一例夏威夷抗病毒的转基因番木瓜问世,PDR 的抗病育种策略是转录后基因沉默(post-transcriptional gene silencing,PTGS)[11-12]。RNA 干扰(RNAi)是表现在转录水平上的基因沉默[13],已被证明在病毒防御反应中发挥作用,并已成功地作为生物技术工具用于转基因植物的病毒抗性。RNAi 是一种由小RNA介导的基因表达调控机制。RNAi 过程涉及3 个基本特征:双链RNA(dsRNA)诱导,dsRNA被切割成21~25 个核苷酸大小的小干扰RNA(siRNA)[14-15],以及siRNA 被整合到RNA 诱导沉默复合物(RNA-induced silencing complex,RISC)中进行序列特异性降解[16]。通过使用RNAi技术,针对目标mRNA 的外源性双链RNA,抑制了靶基因的表达水平。由于外源序列并未表达产物,RNA 介导的病毒抗性具有生物安全优势,且降低了引入外源基因植株带来的风险[17]。
通过转病毒外壳蛋白(coat protein, CP)基因在植物中表达而产生抗病性[18-20]。然而,外壳蛋白基因可能与自然界中的其他病毒或致病因子重组成新的致病因子产生可传播和流行的危险病毒[21-22]。复制酶(nuclear inclusion b, Nib)基因是一种依赖于病毒RNA 的RNA 聚合酶(RNAdependentRNA polymerase, RdRp)[23],用病毒复制酶转化植物已被证明可以产生稳定高效的抗性,通过与复制酶mRNA 结合[24],阻断病毒mRNA 的复制或使病毒mRNA 通过RNAi 衰减直接影响病毒RNA 的复制[25],是获得病毒抗性植物有效的方法。目前通过转病毒Nib 基因使作物获取抗性的方法被广泛运用于大豆[26]、小麦[27-28]、豌豆[29]、烟草[24, 30]、芥菜[25]、大白菜[31]等作物。
前期工作发现,海南番木瓜环斑病毒遗传多样性差异较大[32],大规模种植单一抗性的转基因番木瓜很容易促进靶标病毒的突变,导致抗性丧失。本团队前期已经获得1 个基于番木瓜外壳蛋白基因沉默番木瓜株系的培育并验证具有一定抗性[33-34],建立基于多目标基因的沉默(CP+Nib)来提高番木瓜的广谱抗性,以期应对靶标病毒的突变以及遗传多样性。因此本研究在前期工作的基础上,采取了对Nib 基因沉默的策略创制番木瓜新品系,并验证其抗病性,积累抗病新资源。
1 材料与方法
1.1 材料
受体材料选择番木瓜品种穗中红48,由广州市果树研究所惠赠。遗传转化方法及再生苗由本实验室提供[32-34]。大肠杆菌DH5α 购自Sangon 公司(B528413),农杆菌GV3101 由本实验室保存。载体采用新霉素磷酸转移Ⅱ 基因( NeomycinphosphotransferaseⅡ,NPTⅡ)作为标记基因,抗生素为卡那霉素(50 mg/L)。载体构建如图1。
试剂与仪器:植物总RNA 抽提纯化试剂盒(TIANGEN,DP441-H)、多糖多酚植物基因组DNA 提取试剂盒(TIANGEN,DP360)、FastKingRT Kit(TIANGEN,KR116)用于核酸提取试验;琼脂糖凝胶DNA 回收试剂盒(Sangon,B511139)、质粒小提试剂盒(Omega,D6943-100T)、pUCm-Tvector(Sangon,B620435)、IPTG 溶液(Sangon,B541007)、X-Gal 溶液(Sangon,B541006)用于hiTAIL-PCR 验证转化插入的位点实验,其他常用试剂为国产分析纯。超微紫外分光光度计(倍辉,DS-11)、冷冻型离心机(Therom Fisher,Micro21R)用于核酸提取、质粒提取;PCR 仪(AnalytikJena AG,TAdvanced 96SG)用于阳性株系的筛选;电泳仪( 北京君意东方电泳设备有限公司,JY600E)用于PCR 检验;化学发光成像系统用于凝胶回收(Azure,C300);控温摇床用于大肠杆菌转化(IKA,4000i);电热恒温水浴箱用于大肠杆菌转化(上海一恒,DK-8D)。
1.2 方法
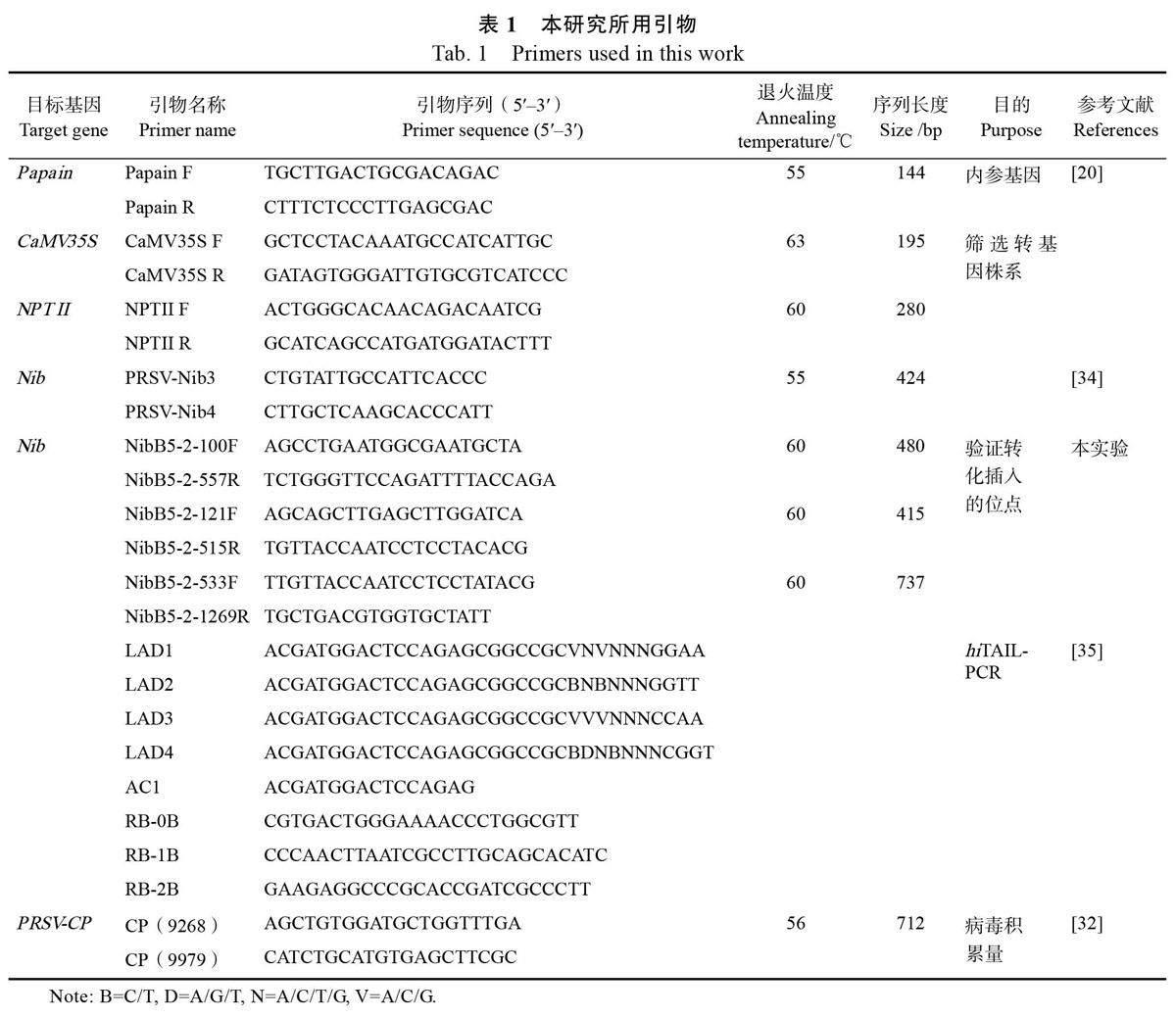
1.2.1 引物合成 引物设计使用NCBI 在线引物设计工具(https://www.ncbi.nlm.nih.gov/tools/primerblast/)在插入位点上游286 bp 处设计验证引物NibB5-2-533F,在插入位置的下游451 bp 处设计验证引物Nibb5-2-1269R;在插入位点上游和载体上设计2 对嵌合引物NibB5-2-100F/NibB5-2-557R和NibB5-2-121F/NibB5-2-515R,用于插入位点的验证。引物及探针序列见表1,引物合成以及DNA测序均由北京六合华大基因科技有限公司完成。
1.2.2 核酸提取方法(DNA/RNA) 将0.2 g 新鲜番木瓜叶片在液氮中充分研磨后,使用植物基因组提取试剂盒提取植物总DNA,用于转化特异性PCR 进行阳性转化株的筛选,通过1.0%琼脂糖凝胶电泳检测DNA 的完整性,并利用紫外分光光度计测定DNA 浓度和纯度,–20 ℃保存备用。将50 mg 的番木瓜叶片在液氮充分研磨后使用植物总RNA抽提纯化试剂盒提取植物總RNA,通过1.0%琼脂糖凝胶电泳检测RNA 的完整性,利用紫外分光光度计测定RNA 浓度和纯度,–80 ℃保存,用于后续研究叶片中PRSV 的病毒积累量。
1.2.3 转基因植株转化事件特异性PCR 筛选阳性株系 用转化事件特异PCR 进行阳性转化苗的筛选。引物分别是目标基因Nib 引物(PRSV-Nib3/PRSV-Nib4)、转化事件特异性引物(CaMV35S F/Nib4)和内参引物(Papain F/PapainR)(表1),以阳性质粒为阳性对照(CK+),水为阴性对照(CK–)。PCR 扩增反应体系为:12.5 μL2×Taq master Mix、1.0 μL 上游引物(100 μmoL/L)、1.0 μL 下游引物(100 μmoL/L)、1.0 μL 番木瓜基因组DNA 模板、9.5 μL Nuclease-free water。PCR扩增条件:95 ℃预变性 5 min;95 ℃变性30 s,55 ℃退火30 s,72 ℃延伸30 s,共35 个循环;72 ℃再延伸10 min。反应完毕后,通过1.0%琼脂糖凝胶电泳后EB 染色,拍照记录。
1.2.4 hiTAIL-PCR 方法确定转化株系的插入位点位置 采用高效热不对称交互式PCR( highefficiencyTAIL-PCR, hiTAIL-PCR)[35]方法检测阳性品系插入位点的侧翼序列。根据已有报道的研究方法[36]进行PCR 扩增,反应结束后将PCR 产物切胶回收,将回收的目标条带DNA 与pUCm-Tvector 连接进行克隆载体的转化,最后通过菌液PCR 挑选每个转化的3 个重复阳性菌送往北京六合华大基因科技有限公司进行测序。
将获得的DNA 序列在Phytozome v13.0(https://phytozome.jgi.doe.gov)在线软件与番木瓜基因组(C. papaya, ASGPBv0.4)进行Blast 比对,以获得侧翼序列确定插入的位点信息。
1.2.5 插入位点验证 利用引物NibB5-2-533F/Nibb5-2-1269R 验证插入位点的准确性。使用2 对嵌合引物NibB5-2-100F/NibB5-2-557R 和NibB5-2-121F/NibB5-2-515R 进一步验证插入位点。PCR 扩增反应体系为:12.5 μL 2×Taq masterMix,1.0 μL 上游引物(100 μmoL/L),1.0 μL 下游引物(100 μmoL/L),1.0 μL DNA 模板,9.5 μLNuclease-free water。PCR 扩增条件:95 ℃预变性5 min;95 ℃变性30 s,60 ℃退火30 s,72 ℃延伸30 s,共35 个循环;72 ℃再延伸10 min。扩增结束后通过1.0%琼脂糖凝胶电泳后EB 染色,拍照记录。
1.2.6 不同转化品系T0 代田间自然抗病评估将转Nib 基因的T0 代幼苗以及非转基因对照移栽到田间中国热带农业科学院文昌试验基地(19°32?14.521?N,110°44?51.652?E,具有隔离条件),进行抗病性筛选。分别在移栽3 个月后第90、100、110、120、130、140、150、180、210、240 天观测和采样。
1.2.7 T1代植株病毒接种试验 海南PRSV 病毒活体植株保存于本实验室。参照黄静等[33] 的PRSV 制备病毒接种液和接种方法,具体如下:
(1)取含有PRSV 病毒的番木瓜叶样适量,将感染病毒的叶片和磷酸缓冲液(1×PBS)按照1∶10 的比例(即1 g 叶片,10 mL PBS)混匀后充分研磨至匀浆状,5000 r/min 离心10 min 后,吸取上清液作为接种液,4 ℃保存备用。
(2)在茎尖向下数第三叶,人工接种PRSV。首先在叶片表面上洒上少许石英砂,用碾磨棒或者指腹由叶脉向叶尖轻轻摩擦,而后滴加10 μL接种液并将其在叶片表面涂抹均匀。
(3)转基因株系(NibB5-2 T1 代)为实验组,非转基因株系(NibB5-5 T1代)为对照组。分别进行病毒接种试验,只接种缓冲液(不含病毒)的对照试验,和不接种对照试验。每种处理设3 个生物学重复。接种病毒后第0、4、8、12、16、20、24、28、32 天观测发病情况,并取相邻叶片(茎尖向下数第二叶片)检测PRSV 病毒积累量。
1.2.8 PRSV 病毒积累量检测 番木瓜总RNA的提取参见1.2.2,利用FastKing cDNA 第一链合成试剂盒以番木瓜总RNA 为模板合成cDNA 第一链。反应体系和程序为:2 μL 5×g DNA Buffer、2μL Total RNA、6 μL Nuclease-free water,PCR 仪中42 ℃ 3 min 后,置于冰水混合物中放置3 min。然后加入2 μL 10×King RT Buffer、1 μL FastKingRT Enzyme Mix、2 μL FQ-RT Primer Mix、5 μLNuclease-free water,于42 ℃ 15 min,95 ℃ 3 min,于–20 ℃保存。
使用PRSV-CP 特异性引物进行PCR 检验,以病毒接种液作为阳性对照(CK+),无核酸酶水作为阴性对照(CK–)。PCR 扩增反应体系为:12.5 μL2×Taq master Mix、1.0 μL CP(9268)、1.0 μL CP(9979)、1.0 μL cDNA 模板、9.5 μL Nuclease-freewater。PCR 扩增条件:95 ℃预变性5 min;95 ℃变性30 s,56 ℃退火30 s,72 ℃延伸30 s,共35个循环;72 ℃延伸10 min。反應完毕后,通过1.0%琼脂糖凝胶电泳后EB 染色,拍照记录。
2 结果与分析
2.1 PCR 筛选阳性转化株系
本研究共计获得52 株已经过抗性筛选的再生苗,用转化事件特异PCR 对再生苗进行筛选,一共筛选出24 株阳性株系,图2 为部分筛选结果,选取在田间表现良好、栽培后能正常开花结果、抗性表现较好的T0 代阳性转化株系代表Nib5-2进行分子特征检测。
2.2 hiTAIL-PCR 验证转化体在番木瓜基因组上的插入位点
NibB5-2 样品的hiTAIL-PCR 电泳结果如图3,将测序结果经过Blast 比对,发现插入位点位于2号染色体的supercontig_30.113 的1976766 处,比对的详细结果为Score=675 bits(748),Expect=0.0Identities=376/377(99%)(图3)。插入位点上、下游最近的基因距离为3.6 kb,插入位点上游10 kb内有2 个Ankyrin Repeat 家族蛋白(evm.TU.supercontig_30.153、evm.TU.supercontig_30.149),下游10 kb 内有2 个Ankyrin Repeat 家族蛋白(evm.TU.supercontig_30.154、evm.TU.supercontig_30.155)、1 个反转录转座子闭合蛋白(PF03732,evm.TU.supercontig_30.156)和1 个糖基转移酶(evm.TU.supercontig_30.157)。
插入位点的上游引物NibB5-2-533F 与下游引物Nibb5-2-1269R 在非转基因番木瓜DNA 中扩增得到预期约737 bp 的产物;而在转基因株系中,由于外源载体的插入,PCR 产物预计大小为4.2 kb+737 bp(图4)。将插入位点上游引物NibB5-2-100F/NibB5-2-121F 和载体上的引物NibB5-2-557R/NibB5-2-515R 匹配,扩增番木瓜基因组和载体序列的嵌合产物,在转基因番木瓜中扩增得到预期大小条带(480 bp 和415 bp),而在非转基因番木瓜中未得到扩增产物(图4)。
2.3 不同转化株系T0代田间自然发病评估
通过对转基因和非转基因株系的田间发病观测发现,转Nib 番木瓜株系和非转基因株系在田间的抗病性差异明显。非转基因株系均感病,具体表现为在移苗后第5 个月时表现出病症,随着时间的延长,病症日益明显,在第6 个月时出现典型病症如果实上出现环斑(图5C);转Nib 番木瓜株系移苗后,植株整体表现健康,一直至第12 个月无明显症状。通过对3~6 个月番木瓜样品进行病毒积累量的PCR 检测,NibB5-1 在第151 天开始出现病毒积累,NibB5-2 在第151 天开始出现病毒积累,NibB5~9 在第112 天出现病毒积累,非转基因对照NibB5-5 在第112 天出现病毒积累(图5A)。
2.4 转基因番木瓜T1代抗性评价
转基因番木瓜NibB5-2 T1代株系和非转基因对照在接种病毒后32 d 内抗病性差异明显。3 株接种病毒的对照植株表现一致,在第8 天时表现出病症,随着时间的延长,病症日益明显,在第24 天时出现病症典型;同时接种病毒的3 株NibB5-2 T1植株在32 d 内,部分有病毒积累,但表型都正常。9 个采集时间点的植物样品进行PCR检测病毒积累量的检测结果如图6A。在相同模板量下,对照植株的凝胶检测灰度呈指数增长趋势,即随着时间的延长感染病毒量逐渐递增,NibB5-2T1代植株基本无变化(图6B)。
3 讨论
PRSV 是海南番木瓜生产的主要制约因素。虽然基于siRNA 和植物人工微RNA(amiRNA)的短发夹RNA(shRNA)可以有效地减少病毒复制,但广泛的病毒遗传变异可能会使病毒逃逸[37],PRSV 存在地理分化,各地的病毒株系差异大,在海南不同地区分布的PRSV 病毒株系差异大[32, 38],所以抗PRSV 转基因番木瓜品种的种植仅限于某些地理区域[39]。RNA 介导的病毒抗性受到入侵病毒与目标转基因之间序列同源性的影响[40],若siRNA 与其靶标之间存在不匹配,会影响靶标RNA 的识别切割,干扰效应降低,甚至使siRNA失活[41-42]。此外,有研究表明,抗病毒效果可能在于小靶区的同源性而并非全序列的同源性[43-44]。课题组前期通过海南全境内PRSV 遗传多样性进行研究发现,不同群体(HainanⅠ,Ⅱ,Ⅲ)[32]还根据RNAi 原理利用海南PRSV-CP 基因保守序列将抗海南番木瓜环斑病毒植物表达载体pCAMBIA2300-35S-CP-RNAi-OCS 转入番木瓜中,对转CP 基因番木瓜进行分子特征及抗病毒试验分析,获得了具有较好的PRSV 抗性的转基因株系[33-34]。
在番木瓜中引入1 个发夹结构来靶向PRSVNib,理论上,病毒的RNA 与转基因的RNA 的靶向方式相同,从而使植物能够抵抗感染。在本研究中,描述了通过转化苗进行阳性株系筛选、hiTAIL-PCR 验证特异插入位点以及转基因株系抗性试验,鉴定了1 个抗病毒转基因番木瓜NibB5-2 的基因组插入和侧翼区域,这在转基因作物的安全性评估和追踪单个转基因事件中非常重要[45]。转基因植株NibB5-2 外源基因未插入到番木瓜编码基因中,hiTAIL-PCR 显示插入位点在第2 号染色体的supercontig_30 的1976766 位置,设计特异引物验证插入位点时发现转基因植株NibB5-2 为杂合子。转基因植株NibB5-2 和非转基因植株NibB5-5 通过田间自然发病试验,对症状进行视觉评估,并使用PRSV(CP)外壳蛋白引物,通过RT-PCR 检测病毒的存在,结果表明植株NibB5-2 对由病虫传播的PRSV 产生了抗性。这是由于转基因番木瓜植株NibB5-2 体内含有Nib 发夾插入结构,转入Nib 基因反向重复载体转录形成了dsRNA,含有dsRNA 的发夹结构被dicer 酶识别,诱导RNAi 机制的启动[46],从而有效抑制了在田间PRSV 对番木瓜的危害。在转基因植株NibB5-2 T1 代和非转基因植株NibB5-5 T1代植株中进行摩擦接种实验,在接种后,NibB5-2T1 代植株表现出抗性,T1 代植株中含有Nib 发夹插入,并在大多数情况下表达了该基因,假设RNAi 可以诱导植物的病毒防御,转基因表达水平足够强,足以引起影响。由双链反向重复序列茎和单链内含子环组成了发夹RNA(hpRNA),双链茎决定了反应的特异性是RNA 沉默的关键因素[47],内含子与RNA 沉默虽然没有直接关系但内含子有利于hpRNA 的形成,提高其稳定性,诱导有效的基因沉默[48]。当茎长为200~800 bp 时,开始出现明显的沉默, 沉默效率高达60%~80%[49]。
采用RNAi 策略获得PRSV 抗性,目标基因的小片段可以最大限度地降低重组、转化、协同作用和互补的风险,且还可避免田间释放和商业化的潜在风险[50]。含PRSV-Nib 的转基因番木瓜已遗传到T1 代,对PRSV 仍具有抗性,可进一步用于抗病育种。为获得广谱抗 PRSV 的转基因植株,还需要通过多代选择,以此产生更稳定的品系,未来的工作还需要确定这些转基因植株是否在田间条件和较高温度下保持表达并提供抗性。
