微生物次级代谢产物多样性发掘方法
2024-05-18方岫琴王文璟李华东张晓婷朱天骄车茜李德海张国建
方岫琴 王文璟 李华东 张晓婷 朱天骄 车茜 李德海 张国建

摘要:微生物次級代谢产物是微生物在生长过程中产生的非必需性代谢产物,其往往具有独特的生物活性。随着对微生物次级代谢产物多样性研究的不断深入开展,研究者逐渐发现在实验室的常规培养条件下微生物的代谢潜能不能完全被激发,从中可获得的天然产物结构类型远远少于其代谢潜能所决定的数量。近年来,随着基因组技术、分子生物学技术特别是合成生物学相关技术的发展,研究人员开发了如OSMAC、表观遗传、异源表达等多种方法,从多个角度多层次地发掘次级代谢产物多样性。这些方法有效激活潜在的生物合成基因簇、提高了微生物次级代谢产物多样性,加之与高通量筛选方法联合应用,能够显著改善了微生物活性天然产物的发现效率。本文综述了微生物次级代谢潜能的激活以及代谢产物高效筛选的方法和技术,为提高微生物次级代谢产物多样性发掘,加快先导化合物的发现提供参考。
关键词:微生物次级代谢产物;沉默基因簇;代谢潜能;激活;结构多样性
中图分类号:R978.1文献标志码:A
Methods for exploring the chemical diversity of microbial secondary metabolites
Fang Xiuqin1, Wang Wenjing1, Li Huadong1, Zhang Xiaoting1, Zhu Tianjiao1, Che Qian1, Li Dehai1, 2, and Zhang Guojian1, 2, 3
(1 Key Laboratory of Marine Drugs, Ministry of Education, School of Medicine and Pharmacy, Ocean University of China, Qingdao 266003; 2 Laboratory for Marine Drugs and Bioproducts of Qingdao National Laboratory for Marine Science and Technology, Qingdao 266237; 3 Marine Biomedical Research Institute of Qingdao, Qingdao 266100)
Abstract Microbial secondary metabolites are non-essential metabolites created by microorganisms throughout the development process, but they frequently exhibit unique biological activities. As research on microbial natural products went on, researchers gradually found that the metabolic capacity of microorganisms could not be completely activated under normal laboratory growth conditions, which led to the fact that the skeletal diversities of natural products obtained from some microbial strains were significantly less than the amount indicated by their potential metabolic capacity. With recent years' development in genomic technology and molecular biology technology, especially synthetic biology-related technologies, several advanced approaches, including OSMAC, epigenetic expression and heterologous expression, have been applied to investigate the diversity of secondary metabolites at the multi-angle and multi-level. Combined with proper screening techniques, those above methods showed increased efficiency in activating silent biosynthetic gene clusters and obtaining diversified secondary metabolites from microbial strains. This article reviewed practical methodologies and strategies for tapping up microbial metabolic potential and screening unique metabolites to provide references for efficiently exploring the variety of microbial natural products and speeding up the identification of drug lead compounds.
Key words Microbial secondary metabolites; Silent gene clusters; Metabolic potential; Activation; Structural diversity
微生物来源广泛、成本低廉、基因组较小、次级代谢产物丰富,是重要的药用资源。微生物次级代谢产物是微生物在特定条件下以初级代谢产物为基础,经过不同生源途径合成的生长非必需的代谢产物,其具有结构多样复杂、生物活性广泛等特点[1]。自1928年Fleming从真菌中发现青霉素以来,微生物次级代谢产物研究一直作为药物研发的热点领域为人们所关注。
研究发现,在微生物体内存在着在常规培养中未激活的次级代谢产物生物合成途径,而与之相关的系列基因则称为沉默基因簇(silent biosyntheic gene clusters)[2-3]。例如,通过对青霉菌Penicillium基因组测序分析结果表明,从24个青霉菌的基因组中发现的1317个生物合成基因簇中有61%(798个)编码PKS、NRPS或PKS-NRPS杂交途径,其中仅有16%的PKS和NRPS基因簇可以确定生产某类天然产物[4];同样,关于维吉芽胞杆菌Virgibacillus的基因组测序分析结果表明,在已发现的215个合成基因簇中,仅有少数被发现与次级代谢产物相关,其中约79%的合成基因簇均为未知基因簇[5]。上述实例说明在实验室常规培养条件下,微生物體内存在着大量处于沉默的基因簇尚未激活。这些沉默的基因资源中蕴含着巨大的次级代谢产物生产潜能,成为微生物药用资源亟待开发的“暗物质”。因此,如何激活沉默基因簇,点亮这些“暗物质”并获得新颖的活性先导化合物结构,是进一步探寻微生物次级代谢产物多样性的重要的途径。
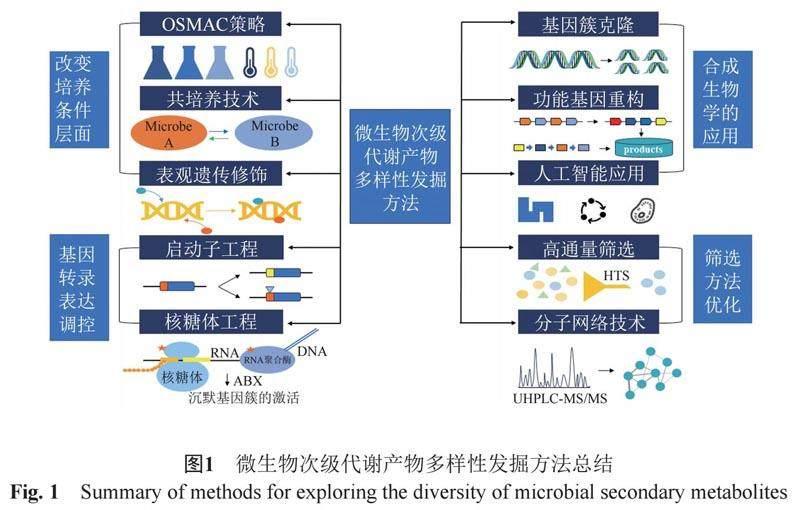
围绕着“如何激活沉默基因簇”这一问题,世界各国的微生物学家、化学家近年来发展了诸多新颖的技术和策略,如:OSMAC(one strain many compounds)策略、共培养技术、表观遗传修饰、启动子工程、核糖体工程、合成生物学技术等。上述方法的应用,进一步揭示了微生物次级代谢产物的多样性。同时,这些技术和策略结合下游的分子网络、高通量筛选等技术,大大提高了微生物次级代谢产物的发现效率。如图1所示,本文将从培养条件的优化、基因转录与表达的调控、合成生物学的应用和筛选方法的优化4个角度概括微生物次级代谢产物开发技术的进展及其应用,旨在为未来的微生物次级代谢产物多样性发掘提供思路。
1 培养条件的改变
1.1 OSMAC策略
1999年,Zeeck等[6]提出了OSMAC策略,该策略通过系统地改变培养条件(如:培养基成分、温度、通气量等)来发掘微生物次级代谢产物的多样性。OSMAC策略可能激活微生物体内的沉默基因簇表达,进而产生更多样的次级代谢产物,该方法操作简便,效果显著,已被广泛应用。
Chen等[7]通过向培养基中添加3% NaBr或3% KI从红树林真菌Phomopsis sp. QYM-13中分离得到12个新的细胞松弛素phomopchalasins D-O (1-3,5-12,14),包括1种溴化和2种碘化的细胞松弛素。其中,溴化细胞松弛素对人癌细胞系MDAMB-435具有选择性细胞毒性(IC50=7.4 μmol/L)。
Lei等[8]通过向大米培养基中添加3%人工海盐,从海绵来源真菌异角疫霉Pestaltiopsis heterocornis XWS03F09中分离出9个新化合物,包括8个新的聚酮衍生物heterocornols Q-X和1个新的神经酰胺ceramide,其中heterocornols W-X可以显著抑制LPS诱导的NO的产生,其活性与抗炎药物地塞米松相当。
通过OSMAC策略发掘微生物次级代谢产物多样性的例子多有报道,但其背后的调控机制仍不甚明了。随着相关菌株生理生化功能的不断揭示,OSMAC策略对沉默基因表达调控的影响机制会越来越清晰,这将为培养条件的优化提供更为清晰的指导。
1.2 共培养技术
近几十年来,研究者们逐渐意识到一些生化过程需要多种微生物的共同参与才能进行,并且不同菌株在同一生境中相互作用、相互影响,对复杂次级代谢产物的产生有促进作用。在此基础上,共培养技术(coculture)应运而生。共培养技术是指在相对封闭的环境中培养2种或2种以上微生物,通过对其原始生存环境的模拟,达到改变相应微生物次级代谢产物的效果[9-11]。共培养技术可以看作OSMAC策略的延伸,该技术已然成为了发掘微生物次级代谢产物多样性的有效途径之一。例如,Thissera等[12]将小麦植物根系相关细菌Pantoea aggolomerans和枣棕榈叶衍生真菌Penicillium citrinum(PC)使用PDA培养基进行共培养,发现了2个新的普利西汀衍生物pulicatin H、I和6个已知化合物,其中pulicatin H对PC具有较强的抗菌活性。Li等[13]运用多代谢物交叉喂养(multi-metabolite cross-feeding,MMCF)的方法,通过对氨基酸合成代谢和能量代谢进行调控,成功构建了稳定的微生物共培养体系,并且在体系中引入代谢物响应性的生物传感器,进一步提高了体系的可调节性。将该体系运用于3菌株的共培养体系中,最终实现了水飞蓟宾(silybin)和异水飞蓟宾(isosilybin)的合成。
虽然共培养技术应用广泛,但是微生物生态关系复杂,且易受环境影响,故深入研究菌株之间的相互作用机制,对进一步拓展共培养技术在次级代谢产物挖掘中具有重要意义。目前,杨志超等[14]总结了群体感应现象对乳酸菌产细菌素的调控机制,并解析了不同因素对其产细菌素的促进机制。李洪涛等[15]总结了微生物共培养互作机制研究的方法及进展。徐德阳等[9]也对微生物共培养中存在的协同代谢作用、诱导作用和基因转移现象进行了分析与总结。然而,微生物间的相互作用机制十分复杂,尚未完全解析,仍待进一步研究阐明。
1.3 表观遗传修饰剂的添加
自2007年首次报道微生物次级代谢的调控与表观遗传修饰具有相关性开始,表观遗传修饰方法成为了进一步发掘微生物次级代谢产物多样性的新方法[16]。表观遗传修饰是指在DNA的序列不发生改变的条件下,基因的表达发生稳定且可遗传变化的现象。其主要作用机制包含2个方面:①针对DNA的甲基化修饰和各种组蛋白的修饰;②非编码的RNA分子参与影响翻译后修饰过程[17-21]。其中,组蛋白的修饰部分因操作简便、对菌株次级代谢行为影响明显,在实验室研究中具有广泛的应用。向培养基中添加表观遗传化学调控剂是该方法最直接的应用。用于添加的表观遗传化学修饰抑制剂主要包括2种:DNA甲基转移酶抑制剂(DNA methyltransferase inhibitors,DNMTi)和组蛋白去乙酰化酶抑制剂(histone deacetylase inhibitors,HDACi)。
朱静轩等[22]通过向培养基中添加化学表观遗传调控剂——组蛋白去乙酰化抑制剂(C646)对海洋来源杂色曲霉(Aspergillus versicolo)进行化学表观遗传调控,最终得到3个新化合物——弯孢霉菌素(curvularin)、环形(L-色氨酸-L-苯丙氨酸)[cyclo-(L-Trp-L-Phe)]和二苯二酚(diorcinol);Wu等[23-24]通过向培养基中添加表观遗传化学调控剂—组蛋白去乙酰化酶抑制剂辛二酸苯胺异羟肟酸(SAHA),使1株海兔来源的土曲霉(Aspergillus terreus)RA2905的代谢谱发生变化,最终分离得到两个新的阿斯呋喃酮(asperfuranone)对映体及两个新的阿斯呋喃酮化合物asperfuranone A和B,这是首次报道的从天然产物中发现含有苄基呋喃酮和苄基吡喃酮骨架的结构。
2 基因转录和表达的调控
通过改变微生物的培养状态激活沉默基因簇的表达,其最适培养条件的确定需要重复大量的筛选工作,且受菌株内在转录过程变化的影响,其不同培养批次的代谢产物图谱依然存在重现性差的问题。随着分子生物学技术的不断更新,从基因层面对菌株进行定向改造越来越受到重视,尤其是转录调控、启动子工程和核糖体工程等方法的应用,可以更为精准地调控代谢过程,获得多样化的次级代谢产物(图2)。
2.1 转录调控
直接添加表观遗传化学调控剂的方法无须了解菌株的遗传背景,应用简便,但该方法缺乏深入的机制解析,使用存在不确定性,且目前能选择的表观遗传修饰剂种类较少[25-26]。因此,直接从基因或蛋白层面进行定向改造可以更好地提高微生物次级代谢产物的多样性。DNA甲基转移酶(DNA methyltransferase,DNMT,如保守调控因子laeA、llm1等),与组蛋白脱乙酰酶(histone deacetylase,HDAC)功能的解析在分子酶学层面为实现表观遗传调控提供了可能。
早在2004年,Keller等[27]首次报道了laeA基因作为丝状真菌次级代谢的转录调控因子,可以调节多种次级代谢产物基因簇的表达,从而影响菌株的次级代谢图谱。近年来,本课题组还应用过表达转录调控因子laeA的方法对真菌次级代谢产物进行发掘,并获得了许多结构多样的活性代谢产物。2019年,本课题组通过高表达1株双齿青霉菌(Penicillium dipodomyis) YJ-11自身的laeA基因,丰富了其中sorbicillinoid类化合物的代谢图谱,从中分离得到了2个该类新化合物10,11-dihydrobislongiquinolide和10,11,16,17-tetrahydrobislongiquinolide[28];2020年,通过对laeA基因的过表达,从1株溴化青霉(Penicillium brocae) HDN-12-143中分离得到1个新的烟熏氯醇(fumigatin chlorohydrin)和1个新的异烟熏氯醇(iso-fumigatin chlorohydrin),这两个化合物均对HL-60癌细胞系均表现出一定的细胞毒作用[29];2022年,同时对laeA基因和生物合成剪切酶MpaB基因进行过表达,从1株海洋来源链格孢菌(Alternaria alternata) JJY-32分离得到4种新的ACTG-毒素-硫代萜类三环交链芳烃O-R(meroterpenoids tricycloalternarenes O-R),其对TLR4转染的巨噬细胞(RAW264.7)均表现出较强的抗炎活性[30]。
llm1基因与laeA基因具有类似的结构域,故调控llm1基因表达量也可能促进微生物次级代谢产物多样性的产生。王云胜[31]构建了llm1基因过表达的冠突散囊菌(Eurotium cristatum)突变体,在PDB培养基中进行培养,最终通过HPLC检测发现llm1过表达的突变体相比于野生型产生了3个新的色谱峰,并且通过antiSMASH数据库对转录组数据进行分析,结果表明过表达llm1基因能激活冠突散囊菌次级代谢基因簇的表达。
2.2 启动子工程
对于微生物来说,影响基因表达水平的最重要因素是转录水平,基因的启动子序列在其中发挥着重要作用[32]。目前,启动子工程采用了许多策略,如:内源性启动子的改造和替换、启动子文库的筛选和构建等。
启动子改造和替换是激活基因簇的有力手段,前者直接对内源启动子进行突变改造或用特定的转录因子与之结合来改变启动子强度[33];后者将原有启动子替换,彻底改变受控基因的表达谱,在转录水平上实现对关键基因的精准控制。
Wu等[34]在枯草芽胞杆菌(Bacillus subtilis)中将启动子PgamA中转录因子GamR的结合位点gamO2插入到Pveg启动子序列中,实现了对转录因子结合位点(transcription factor binding site,TFBS)的优化,成功构建了可以响应胞内6-磷酸氨基葡萄糖(glucosamine-6-phosphate,GlcN6P)的合成啟动子,成功激活了GlcN6P的沉默生物合成基因簇。Wang等[35]将包含两个广泛应用的人工启动子kasOp*的片段插入TiaP2和聚酮骨架的编码基因,首次在链霉菌Streptomycetaceae宿主中成功异源表达了非达霉素(fidaxomicin)生物合成基因簇,获得了6个苷元,其中4个为新化合物,补充和拓展了非达霉素苷元的合成途径。
启动子文库的筛选和构建也是启动子工程常用的方法。但是传统启动子文库的构建耗时费力,且难以获得理想的启动子。因此,利用生物信息学和计算机科学工具,预测、设计、优选和改造启动子[36]等方法正受到越来越多的关注和研究。
非保守区随机突变(non conservative random mutation,NCR)可用于快速生成不同强度的大规模启动子库,并被广泛应用于代谢途径的通量优化。Wei等[37]设计了一个基于谷氨酰胺-10(NNTANANT)和-35(NNGNCN)共识区、保守RBS(AAAGGA)元件和60个随机核苷酸的启动子库,其所获得的启动子可以有效地调控基因的表达,并在很大范围内表现出不同的强度。在此基础上,该团队还开发了一种全新的基于启动子库的多模块组合(promoter library-based module combination,PLMC)技术,以有效地优化谷氨酰胺中基因的表达。
启动子工程也常与其他技术联用,以提高启动子工程的效率。Wei等[38]建立了一种基于流式细胞术、在单细胞分辨率下检测超折叠绿色荧光蛋白的丝状真菌启动子的定量评估方法,其中所鉴定的活性最强的启动子PzipA和PsltA分别比常用的组成型启动子PgpdA高2.9倍和1.5倍,并应用这两个启动子激活原生宿主烟曲霉(Aspergillus fumigatus)及异源宿主构巢曲霉(Aspergillus nidulans)中的沉默非核糖体肽合成酶基因Afpes1,并最终在其代谢产物中发现了多种新的环状四肽衍生物,为真菌中的天然产物发现提供了一种创新策略。
人工智能在启动子工程方面展现出良好前景,Lafleur等[39]通过大规模并行分析,将生物物理学和机器学习相结合,开发出一个可预测任何σ70启动子序列的特异性转录起始率的346参数模型。同时,利用机器学习进行人工启动子的设计、创制和优化的研究已经蓬勃兴起,并取得了一系列突破性的成果。Wang等[40]以大肠埃希菌中天然启动子序列为基础,通过研究不同位置核苷酸的互作关系,利用计算机从头设计启动子,将预测模型与深度生成模型相结合,经过两轮优化后发现超过70%的启动子具有功能性,且与大肠埃希菌的基因组没有明显的序列相似性,从而建立了启动子设计“从端到端”的方法,最终成功构建了基于人工智能从头设计启动子的框架;Van Brempt等[41]通过使用来自荧光激活细胞分类启动子库的高通量DNA测序数据来训练卷积神经网络,从而实现对启动子转录起始频率和σ因子特异性启动子正交性的预测,并以此作为在线启动子设计工具(ProD)的基础,为定制的遗传系统提供定制的启动子。
2.3 核糖体工程
“核糖体工程(ribosome engineering)”这一概念由日本国家食品研究所的Ochi教授首先提出[42]。该技术基于微生物严谨反应的激活,通过引入抗生素选择压力,其核糖体蛋白或RNA聚合酶发生突变,使突变后菌株对环境胁迫的耐受性增强,并伴随次级代谢产物多样性的产生,以此筛选耐药菌株。核糖体工程在天然产物发现方面的应用潜力受到研究者持续的关注。
在抗生素压力的选择上,链霉素、利福平和庆大霉素的研究开展较多,且筛选效果相对较好。Hosaka等[43]使用上述3种抗生素对1068株土壤放线菌进行了大规模的筛选,他们发现在这种压力下,土壤中43%的链霉菌和6%的非链霉菌放线原本具备不生产抗生素的菌株最终都能够明显进化出生产抗生素的能力,最后从1株放线菌突变株中分离得到一系列新型的大环内酰胺类抗生素piperidamycin。深入研究发现其突变体生产抗生素的能力得到显著提高是由于其RNA聚合酶的突变,或者核糖体蛋白基因s12的突变而引起二者亲和力的增加所导致。
核糖体工程的应用还可以激活沉默的BGC,并在其他细菌或真菌中发现新的活性化合物。Wu等[44]用新霉素和DMSO对海洋产紫青霉(Penicillium purpurogenum) G59进行抗性筛选,与之前培养结果相比,发现了5种新的次级代谢产物:curvularin, citrinin, penicitrinone A, erythro-23-O-methylneocyclocitrinol和22E-7α-methoxy-5α,6α-epoxyergosta-8(14),22-dien-3β-ol,并且这5个化合物对人癌细胞系K562、HL-60、HeLa和BGC-823均有不同程度的抑制。
目前核糖体工程与代谢工程联用的例子较多,其主要的应用方向为微生物次级代谢产物产量的提升,但是其对于微生物次级代谢产物多样性的发掘也具有广阔的前景。
3 合成生物学和异源表达技术的应用
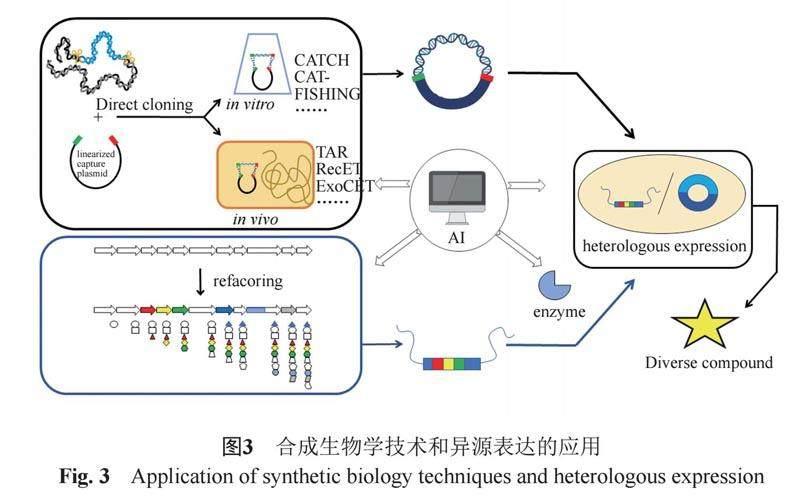
合成生物学是将工程化的思维逻辑应用到生物合成中来,学习自然的生物合成机制,在此基础上设计和构建全新的生物元件、装置和系统,或改良和优化已有的天然生物合成系统。其中,微生物的生物合成基因簇是应用基础。合成生物学通过对合成基因簇的改造及表达调控,借助不同的底盘细胞,如酵母菌、大肠埃希菌等,实现高效异源表达所需化合物的目的,该方法也是目前研究的热点之一(图3)。如何合理设计和构建生物元件、高效获取目的基因片段、构建良好的异源表达体系等问题是该技术所涉及的关键环节。
3.1 基因簇直接克隆与异源表达
随着生物信息学技术的发展,微生物的合成基因簇得到了快速的解析,在此基础上利用合成生物学和异源表達的方法,研究者可以高效获得结构新颖的天然产物。但是,目标基因的获取,尤其是大片段目标基因能否成功获取直接决定了异源表达能否开展实施。
传统方法通常通过构建基因组文库来进行筛选,该方法不仅资源投入大,还受到片段长度的限制,无法对大片段的目的基因进行有效提取。基于此,越来越多的基因簇克隆技术不断涌现,为微生物异源表达及微生物次级代谢产物多样性的发掘注入了新的活力。
Fu等[45]提出了RecET直接克隆技术,即利用大肠埃希菌全长Rac前噬菌体蛋白RecE和其伴侣RecT重组蛋白将目的基因直接克隆到表达载体中,运用该技术对发光杆菌(Photorhabdus luminescens) TT01的基因组进行挖掘,成功克隆出10个未知PKS-NRPS基因簇中的9个,并异源表达出了其中2个基因簇,最终鉴定得到了3个新的次级代谢产物——发光霉素A(luminmycin A)和发光酰胺A/B(luminmide A/B)。但RecET直接克隆技术不能克隆大于50 kb的DNA片段。对此,Wang等[46-47]将RecET系统和Redαβ DNA修饰系统整合到一个大肠埃希菌宿主中,实现了对基因组克隆、转移和异源表达的无缝对接,并将该技术命名为Red/ET重组技术;同时,该团队利用核酸外切酶介导的体外多分子组装和RecET直接克隆技术实现了将克隆载体和目的基因导入同一个宿主的同时,完成对大于100 kb的细菌基因组DNA进行直接克隆,并将该技术命名为ExoCET技术。在此基础上,Song等[48]利用Redαβ介导的线环重组、CcdB介导的反向筛选和核酸外切酶介导的体外同源重组,实现了对大型复杂基因簇中的基因编辑,并且通过该方法完成了对多杀菌素的基因簇的改造,获得了新颖的丁烯基多杀菌素A(butenyl-spinosyn A),并将该技术命名为RedEx技术。
TAR(Transformation associated recombination)克隆技术,即转化偶联重组技术,是1种基于酵母的同源重组系统、从复杂基因组中选择性地快速克隆目的DNA片段的方法,该方法也是目前已报道的唯一能选择性克隆300 kb及以上大片段的方法。例如,Yamanaka等[49]利用TAR克隆技术对海洋放线菌Saccharomonospora sp. CNQ-490进行产物多样性发掘,表达出一个67 kb的非核糖体肽合成酶生物合成基因簇并在模型宿主细胞Streptomyces coelicolor中生产出一种新的抗生素taromycin A,成功激活沉默的生物合成途径,体现了TAR直接克隆技术对生物合成流水线的转移、激活,以及多样化次级代谢物生产方面的便捷和高效。
与此同时,基因编辑与基因簇克隆的结合也催生出许多新方法。Jiang等[50]利用CRISPR/Cas9和Gibson组装开发了1种在体外将细菌基因组中特定的基因簇直接克隆的方法,称为CATCH技术(Cas9-assisted targeting of chromosome segments),该技术可在1个步骤内,完成对几乎任意的、长达100 kb的细菌基因组序列的有效靶向克隆。Liang等[51]开发了一种CRISPR-Cas12a介导的快速直接生物合成基因簇克隆法,称为CAT-FISHING,用于直接捕获大型BGCs。
3.2 功能基因的整合重构及异源表达
天然产物生物合成途径的异源表达不仅可以结合基因组挖掘技术,将隐性生物合成基因簇在异源宿主细胞中进行功能性表达,还可以结合生物合成工程,将不同来源的基因进行组合和重新排列来产生一系列新的类似物。这种策略极大地促进了新活性化合物的发现,为进一步发掘微生物次级代谢产物多样性提供新思路。下面我们主要介绍在合成生物学指导下,重组基因或重构生物合成途径在异源表达方面的应用。
Zhang等[52]借助基于“脱氧单糖的重构”和“糖基模块替换” 对红霉素的基因簇进行了重构整合,在大环内酯苷元上引入不同的糖基化修饰,生产了一系列对红霉素耐药菌株具有抑制活性的红霉素类似物。Huffman等[53]从细菌核苷酸合成途径中的补救合成途径入手,设计了逆向合成路线,通过定向进化获得5种稳定的合成酶,在4种辅酶的辅助下实现了抗HIV药物伊斯拉曲韦(islatravir)生物全合成,相比于化学全合成工艺,步骤更少,产量更高。
随着启动子工程、基因簇克隆以及基因编辑技术的不断发展,未来在合成生物学理论指导下的异源表达技术在微生物次级代谢产物多样性发掘中具有广阔的应用前景。
3.3 人工智能指导合成生物学技术应用
生物元件的设计是合成生物学的基石,其固有的复杂性限制了对其工程化的改造,因此如何实现合成核心元件的标准化并提高其与底盘细胞的适配性是合成生物学领域的一个核心的研究方向。
生物信息学与人工智能(artificial intelligence,AI)不断发展,人们有望于在设计和优化生物元件、代谢工程和合成途径等方面取得更进一步的突破。Li等[54]通过人工智能技術对芽胞杆菌Bacillus sp.中催化碳-碳双键的不对称氢胺化中所使用的天冬氨酸酶YM55-1进行重新设计,获得了具有高度选择性的β-氨基酸合成酶,从而构建了高效合成β-氨基酸的工程菌,首次完成了由计算指导来发现新型合成酶的应用,实现了人工智能技术在微生物次级代谢产物多样性挖掘方面的成功运用。Yeh等[55]开发了一种基于深度学习的蛋白设计策略——family-wide hallucination,该策略可以产生大量不同结合口袋的理想蛋白结构,并设计出编码这些结构的序列,通过该策略,团队成功实现了可选择性催化化学发光的人造荧光素酶的从头设计,该策略的成功应用,在人工设计酶领域堪称里程碑意义的突破,这一突破意味着利用计算机可以实现尚未在自然界中发现的人工酶的从头设计,从而完成对化学反应定制酶的需求。
此外,人工智能还可以用于合成生物学的预测,极大地减少逆向设计的大量试错成本。然而,人工智能对合成生物学的影响依旧有限,仍局限于特定的数据集和研究问题。
目前,研究者们面临的挑战仍是如何使其适用于更广泛的应用程序和其他数据集。对此,人工智能技术已经结合了生物物理、机器学习和强化学习模型,以求更有效地进行预测,如:ReFeaFi[56]、生成对抗网络(generative adversarial network,GAN)[57]等,为调控元件的识别、活性强度预测、元件从头设计等方面提供了新思路[58]。
4 筛选方法的优化
微生物次级代谢产物的筛选方法是影响发掘效率的关键因素之一。一旦获得或即将获得这些产物,高效地筛选和分离目标化合物成为亟须解决的问题。针对这一问题,研究者们致力于改进筛选体系,力求简化和加速筛选、分离过程,以提高微生物次级代谢产物多样性的发掘效率。
4.1 高通量筛选策略
高通量筛选技术(high throughput screening,HTS)将多种技术(如组合化学、基因组学、生物信息学、自动化和机器人技术)相结合,实现快速、高效、自动化的筛选。这种方法已广泛应用于微生物次级代谢产物的筛选,随着应用范围的扩大,其重要性逐渐凸显,在微生物定向进化中也發挥着至关重要的作用。
定向进化(directed evolution)是利用实验室的条件来模拟自然进化的过程[59],通过对突变体次级代谢产物的筛选,可以用于发掘微生物次级代谢产物的多样性。这一过程需要对大量的突变体进行筛选,因而一个灵敏、可靠且快速的高通量筛选方法十分重要。荧光激活细胞分选技术(fluorescence-activated cell sorting,FACS)[60],即一种将酶活性转化为荧光信号以对单细胞进行高效快速分选的技术。Tan等[61]开发了一种荧光激活细胞的分选系统,运用超高通量筛选的方法(107个突变体/h)对突变体进行筛选,成功从幽门螺杆菌(Helicobacter pylori) strain NCTC 11639的突变体中筛选得到了α-1,3-岩藻糖基转移酶活性提高14倍的突变体M32。近年来,微流控技术(fluidics)也从最初的微电子工业应用逐渐发展到生命科学领域当中。微流控技术是一种将单个细胞包埋于液滴中,以实现对单细胞及其胞外代谢产物的分析技术。应用微流控技术,张道远等[62]综合考虑了基于液滴的数字PCR(Droplet Digital PCR,ddPCR)和基于芯片的ddPCR的优势及不足,设计并制备了高集成度的数字PCR微流控芯片,完成了PCR的高通量、高通用及高进样效率的要求,为PCR反应的高通量筛选提供了可能。高通量筛选技术为定向进化赋能将有助于高效解析生物合成的基因簇,并将其运用于微生物次级代谢产物多样性的发掘中。通过对微生物定向进化中突变体的高通量筛选,可以定向得到合成生物学的进化元件,其筛选效率直接决定了微生物次级代谢产物多样性发掘的效率。
随着计算机技术的不断进步,人工智能同样在辅助筛选领域得到越来越多的应用。如朱尤卓等[63]详述了深度学习模型——卷积神经网络、生成对抗网络和循环神经网络等在抗菌肽药物筛选和设计中的应用,但由于深度学习生成的抗菌肽缺乏指导理论,仍存在临床毒副作用过大等弊端;宋益东等[64]针对目前较新的蛋白质功能及结合位点的预测方法进行了总结及优劣分析,并提出了对目前预测方法的改进方向,为更好地预测蛋白质功能及结合位点提供了新的思路;胡如云等[65]详阐了机器学习在合成生物学领域的调控元件设计、酶催化元件的设计、功能多肽设计和代谢过程等方面的应用。可见计算机技术为高效筛选微生物次级代谢产物提供了新思路。
4.2 分子网络技术
近年来,随着生化信息学的快速发展,一大批强大的工具和网络平台不断涌出,掀起了一场“天然产物分离艺术”的革命。其中,基于质谱的分子网络分析技术是最具代表性的一类多功能便捷工具,其在天然产物分离工作中表现出的快速去重复化、低成本、高效性,极大地简化了传统的分离工作,为研究者发现新的生物活性分子提供了一条更为科学和便捷的方法。
基于串联质谱(MS/MS)的分子网络是一种生物信息学策略,用于可视化和解释非目标MS数据。MS/MS分子网络利用药物代谢物中分子间结构的相似性,通过比较每个MS/MS光谱之间的光谱相似程度,将MS/MS光谱作为分子网络映射到数据集中。分子网络在天然产物分离上的应用大致可以分为3类:①利用现有质谱数据库对已知化合物进行自动查询;②利用已知的光谱特征对待查询的光谱进行分析;③利用现有数据库对所有化合物进行全面注释并生成推定结构。关于分子网络技术的原理及应用进展近期已有多篇综述进行评述总结[66-68],这里就不再赘述。
在MS/MS分子网络的指导下,本实验室[69]从海洋沉积物中衍生的放线菌Nocardiopsis sp. HDN154086中获得了5个新颖的对三联苯并噻唑衍生物(p-terphenyl derivatives)nocarterphenyls D-H(1~5),其中化合物1对MRSA具有显著的抗菌。Han等[70]采用分子网络方法从海绵衍生的杂色曲霉(Aspergillus versicolor)中靶向分离出一系列新的柄曲霉素衍生物sterigmatocystins A~C。本单位Shao等[71]在MS/MS分子网络的指导下,从珊瑚衍生真菌花斑曲霉中分离出4个新的环七肽(cycloheptapeptides)与3个曲霉酰胺类似物asperversiamide A~C,并在此基础上半合成出一系列衍生物,进而系统评价了其潜在的抗结核活性。
5 总结与展望
一直以来,从微生物中寻找活性次级代谢产物就是先导化合物开发的重要途径,但是现有的技术和方法在产物多样性的开发及高通量筛选等方面仍存在局限性,尤其是常规培养中无法被激活的沉默生物合成途径,更是微生物次级代谢产物研究的最大阻碍。为此,研究者开发了多种策略,试图从不同层面解决这一问题。
在改变培养条件层面,OSMAC策略与共培养技术通过改变培养环境来激活菌株代谢潜能,表观遗传修饰则可通过简单地添加化学调控剂挖掘多种结构新颖的次级代谢产物。在基因转录和表达层面,启动子工程和核糖体工程可以在转录和翻译水平上实现对目标基因更为精细化的调控,以达到提高产量,获取更多新颖次级代谢产物的目的。此外,通过合成生物学技术对功能基因实施突变改造,并完成对生物合成流水线的重构甚至异源表达能够实现对天然产物的精准合成,高效获得多样化的活性天然产物。在筛选方法优化层面,基于测序技术的高通量筛选新策略与分子网络技术的指导为高效筛选微生物次级代谢产物提供了新思路、指明了发展方向。
然而方法与技术的应用与发展并不是彼此割裂、相互独立的,在实际科研中,研究人员常采用多方法联用、多技术交叉的策略,多角度挖掘和改良微生物的代谢潜力与代谢能力。同时,基因组学、代谢组学和生物信息学的蓬勃发展也为微生物代谢潜力探索挖掘提供了强有力的支持。随着更多更全面的次级代谢产物的基因信息被解析,其生物合成途径与机制逐渐被揭示,越来越多的沉默基因簇将会被发现并激活,迎来微生物次级代谢产物快速开发的新时代。
参 考 文 献
Keller N P, Hohn T M. Metabolic pathway gene clusters in filamentous fungi[J]. Fungal Genet Biol, 1997, 21(1): 17-29.
Scherlach K, Hertweck C. Mining and unearthing hidden biosynthetic potential[J]. Nat Commun, 2021, 12(1): 3864.
Reen F J, Romano S, Dobson A D, et al. The sound of silence: Activating silent biosynthetic gene clusters in marine microorganisms[J]. Mar Drugs, 2015, 13(8): 4754-4783.
Van Lanen S G, Shen B. Microbial genomics for the improvement of natural product discovery[J]. Curr Opin Microbiol, 2006, 9(3): 252-260.
Othoum G, Bougouffa S, Bokhari A, et al. Mining biosynthetic gene clusters in Virgibacillus genomes[J]. Bmc Genomics, 2019, 20(1): 696.
Schiewe H J, Zeeck A. Cineromycins, gamma-butyrolactones and ansamycins by analysis of the secondary metabolite pattern created by a single strain of Streptomyces[J]. J Antibiot (Tokyo), 1999, 52(7): 635-642.
Chen Y, Yang W, Zou G, et al. Cytotoxic bromine- and iodine-containing cytochalasins produced by the mangrove endophytic fungus Phomopsis sp. QYM-13 using the OSMAC approach[J]. J Nat Prod, 2022, 85(5): 1229-1238.
Lei H, Bi X, Lin X, et al. Heterocornols from the sponge-derived fungus Pestalotiopsis heterocornis with anti-inflammatory activity[J]. Mar Drugs, 2021, 19(11): 585.
徐德陽, 王莉莉, 杜春梅. 微生物共培养技术的研究进展[J]. 微生物学报, 2015, 55(9): 1089-1096.
Okada B K, Seyedsayamdost M R. Antibiotic dialogues: Induction of silent biosynthetic gene clusters by exogenous small molecules[J]. Fems Microbiol Rev, 2017, 41(1): 19-33.
陈雷, 申琳溪, 王光玉. 海洋动物来源微生物的共培养研究进展[J]. 微生物学通报, 2021, 48(1): 278-287.
Thissera B, Alhadrami H A, Hassan M, et al. Induction of cryptic antifungal pulicatin derivatives from Pantoea agglomerans by microbial co-culture[J]. Biomolecules, 2020, 10(2): 268.
Li X, Zhou Z, Li W, et al. Design of stable and self-regulated microbial consortia for chemical synthesis[J]. Nat Commun, 2022, 13(1): 1554.
杨志超, 颜挺威, 邱薇, 等. 共培养条件下群体感应系统对乳酸菌产细菌素的研究进展[J]. 中国农学通报, 2023, 39(15): 139-146.
李洪涛, 周皓, 丁中涛. 微生物共培养产生新颖活性次生代谢产物的研究进展[J]. 云南大学学报(自然科学版), 2023, 45(2): 493-512.
Shwab E K, Bok J W, Tribus M, et al. Histone deacetylase activity regulates chemical diversity in Aspergillus[J]. Eukaryot Cell, 2007, 6(9): 1656-1664.
黄禄. 三株真菌次级代谢产物的化学表观遗传调控研究[D]. 杭州: 浙江工业大学, 2019.
Mohammad H P, Barbash O, Creasy C L. Targeting epigenetic modifications in cancer therapy: erasing the roadmap to cancer[J]. Nat Med, 2019, 25(3): 403-418.
Zhao S, Allis C D, Wang G G. The language of chromatin modification in human cancers[J]. Nat Rev Cancer, 2021, 21(7): 413-430.
Ye Y, Li L, Dai Q, et al. Comprehensive analysis of histone methylation modification regulators for predicting prognosis and drug sensitivity in lung adenocarcinoma[J]. Front Cell Devel Biol, 2022, 10: 991980.
Millan-Zambrano G, Burton A, Bannister A J, et al. Histone post-translational modifications-cause and consequence of genome function[J]. Nat Rev Genet, 2022, 23(9): 563-580.
朱静轩, 王婷婷, 周珂欣, 等. 表观遗传试剂诱导海洋真菌Aspergillus versicolor DJ013产生次级代谢产物的研究[J]. 中国海洋药物, 2017, 36(1): 14-18.
Wu J S, Shi X H, Yao G S, et al. New Thiodiketopiperazine and 3,4-dihydroisocoumarin derivatives from the marine-derived fungus Aspergillus terreus[J]. Mar Drugs, 2020, 18(3): 132.
Wu J S, Shi X H, Zhang Y H, et al. Benzyl furanones and pyrones from the marine-derived fungus Aspergillus terreus induced by chemical epigenetic modification[J]. Molecules, 2020, 25(17): 3927.
吳金涛, 彭晓月, 缪莉, 等. 表观遗传修饰法增加真菌次级代谢产物化学多样性的应用[J]. 中国海洋药物, 2021, 40(4): 64-71.
Pacheco Tapia R, Vásquez-Ocmín P, Duthen S, et al. Chemical modulation of the metabolism of an endophytic fungal strain of cophinforma mamane using epigenetic modifiers and amino-acids[J]. Fungal Biol-Uk, 2022, 126(5): 385-394.
Bok J W, Keller N P. LaeA, a regulator of secondary metabolism in Aspergillus spp.[J]. Eukaryot Cell, 2004, 3(2): 527-535.
Yu J, Han H, Zhang X, et al. Discovery of two new sorbicillinoids by overexpression of the global regulator LaeA in a marine-derived fungus Penicillium dipodomyis YJ-11[J]. Mar Drugs, 2019, 17(8): 446.
Wang L, Zhang X, Zhang K, et al. Overexpression of global regulator pbrlaea leads to the discovery of new polyketide in fungus Penicillium brocae HDN-12-143[J]. Front Chem, 2020, 8: 270.
Wang L, Jiao J, Liu D, et al. Cytotoxic meroterpenoids from the fungus Alternaria sp. JJY-32[J]. Chem Biodivers, 2020, 17(7): e2000226.
王云胜. 过表达llm1基因对冠突散囊菌次级代谢及发育的影响[D]. 贵阳: 贵州师范大学, 2022.
Blazeck J, Alper H S. Promoter engineering: Recent advances in controlling transcription at the most fundamental level[J]. Biotechnol J, 2013, 8(1): 46-58.
Blazeck J, Alper H. Systems metabolic engineering: genome-scale models and beyond[J]. Biotechnol J, 2010, 5(7): 647-659.
Wu Y, Chen T, Liu Y, et al. Design of a programmable biosensor-CRISPRi genetic circuits for dynamic and autonomous dual-control of metabolic flux in Bacillus subtilis[J]. Nucleic Acids Res, 2020, 48(2): 996-1009.
Wang W, Li X, Wang J, et al. An engineered strong promoter for streptomycetes[J]. Appl Environ Microbiol, 2013, 79(14): 4484-4492.
Gilman J, Singleton C, Tennant R K, et al. Rapid, heuristic discovery and design of promoter collections in non-model microbes for industrial applications[J]. Acs Synth Biol, 2019, 8(5): 1175-1186.
Wei L, Xu N, Wang Y, et al. Promoter library-based module combination (PLMC) technology for optimization of threonine biosynthesis in Corynebacterium glutamicum[J]. Appl Microbiol Biotechnol, 2018, 102(9): 4117-4130.
Wei P L, Fan J, Yu J, et al. Quantitative characterization of filamentous fungal promoters on a single-cell resolution to discover cryptic natural products[J]. Sci China Lif Sci, 2023, 66(4): 848-860.
Lafleur T L, Hossain A, Salis H M. Automated model-predictive design of synthetic promoters to control transcriptional profiles in bacteria[J]. Nat Commun, 2022, 13(1): 5159.
Wang Y, Wang H, Wei L, et al. Synthetic promoter design in Escherichia coli based on a deep generative network[J]. Nucleic Acids Res, 2020, 48(12): 6403-6412.
Van Brempt M, Clauwaert J, Mey F, et al. Predictive design of sigma factor-specific promoters[J]. Nat Commun, 2020, 11(1): 5822.
Ochi K. From microbial differentiation to ribosome engineering[J]. Biosci Biotechnol Biochem, 2007, 71(6): 1373-1386.
Hosaka T, Ohnishi-Kameyama M, Muramatsu H, et al. Antibacterial discovery in actinomycetes strains with mutations in RNA polymerase or ribosomal protein S12[J]. Nat Biotechnol, 2009, 27(5): 462-464.
Wu C J, Yi L, Cui C B, et al. Activation of the silent secondary metabolite production by introducing neomycin-resistance in a marine-derived Penicillium purpurogenum G59[J]. Mar Drugs, 2015, 13(4): 2465-2487.
Fu J, Bian X, Hu S, et al. Full-length RecE enhances linear-linear homologous recombination and facilitates direct cloning for bioprospecting[J]. Nat Biotechnol, 2012, 30(5): 440-446.
Wang H, Li Z, Jia R, et al. RecET direct cloning and redalphabeta recombineering of biosynthetic gene clusters, large operons or single genes for heterologous expression[J]. Nat Protoc, 2016, 11(7): 1175-1190.
Wang H, Li Z, Jia R, et al. ExoCET: Exonuclease in vitro assembly combined with RecET recombination for highly efficient direct DNA cloning from complex genomes[J]. Nucleic Acids Res, 2018, 46(5): e28.
Song C, Luan J, Li R, et al. RedEx: a method for seamless DNA insertion and deletion in large multimodular polyketide synthase gene clusters[J]. Nucleic Acids Res, 2020, 48(22): e130.
Yamanaka K, Reynolds K A, Kersten R D, et al. Direct cloning and refactoring of a silent lipopeptide biosynthetic gene cluster yields the antibiotic taromycin A[J]. Proc Natl Acad Sci U S A, 2014, 111(5): 1957-1962.
Jiang W, Zhao X, Gabrieli T, et al. Cas9-assisted targeting of chromosome segments CATCH enables one-step targeted cloning of large gene clusters[J]. Nat Commun, 2015, 6: 8101.
Liang M, Liu L, Xu F, et al. Activating cryptic biosynthetic gene cluster through a CRISPR-Cas12a-mediated direct cloning approach[J]. Nucleic Acids Res, 2022, 50(6): 3581-3592.
Zhang G, Li Y, Fang L, et al. Tailoring pathway modularity in the biosynthesis of erythromycin analogs heterologously engineered in E. coli[J]. Sci Adv, 2015, 1(4): e1500077.
Huffman M A, Fryszkowska A, Alvizo O, et al. Design of an in vitro biocatalytic cascade for the manufacture of islatravir[J]. Science, 2019, 366(6470): 1255-1259.
Li R, Wijma H J, Song L, et al. Computational redesign of enzymes for regio- and enantioselective hydroamination[J]. Nat Chem Biol, 2018, 14(7): 664-670.
Yeh A H, Norn C, Kipnis Y, et al. De novo design of luciferases using deep learning[J]. Nature, 2023, 614(7949): 774-780.
Umarov R, Li Y, Arakawa T, et al. ReFeaFi: Genome-wide prediction of regulatory elements driving transcription initiation[J]. PLoS Comput Biol, 2021, 17(9): e1009376.
Repecka D, Jauniskis V, Karpus L, et al. Expanding functional protein sequence spaces using generative adversarial networks[J]. Nat Mach Intell, 2021, 3(4): 324-333.
Eslami M, Adler A, Caceres R S, et al. Artificial intelligence for synthetic biology[J]. Assoc Comp Mach, 2022, 5(65): 88-97.
Kilgore M B, Kutchan T M. The amaryllidaceae alkaloids: Biosynthesis and methods for enzyme discovery[J]. Phytochem Rev, 2016, 15(3): 317-337.
Yang G, Withers S G. Ultrahigh-throughput FACS-based screening for directed enzyme evolution[J]. ChemBiochem, 2009, 10(17): 2704-2715.
Tan Y, Zhang Y, Han Y, et al. Directed evolution of an alpha1,3-fucosyltransferase using a single-cell ultrahigh-throughput screening method[J]. Sci Adv, 2019, 5(10): w8451.
張道远, 林树靖, 王菲, 等. 用于高集成度数字PCR平台的微流控芯片[J]. 微纳电子技术, 2022, 59(3): 242-249.
朱尤卓, 刘红玉, 游宇豪, 等. 深度学习在抗菌肽药物研究中的应用进展[J]. 中国抗生素杂志, 2023, 48(4): 374-380.
宋益东, 袁乾沐, 杨跃东. 深度学习在蛋白质功能预测中的应用[J]. 合成生物学, 2023, 4(3): 488-506.
胡如云, 张嵩亚, 蒙海林, 等. 面向合成生物学的机器学习方法及应用[J]. 科学通报, 2021, 66(3): 284-299.
Panditrao G, Bhowmick R, Meena C, et al. Emerging landscape of molecular interaction networks: Opportunities, challenges and prospects[J]. J Biosci, 2022, 47(2): 1-26.
Fox R A, Evanno L, Poupon E, et al. Natural products targeting strategies involving molecular networking: Different manners, one goal[J]. Nat Prod Rep, 2019, 36(7): 960-980.
Zhang M, Otsuki K, Li W. Molecular networking as a natural products discovery strategy[J]. Acta Materia Medica, 2023.
Chang Y, Che Q, Xing L, et al. Antibacterial p-terphenyl with a rare 2,2-bithiazole substructure and related compounds isolated from the marine-derived actinomycete Nocardiopsis sp. HDN154086[J]. J Nat Prod, 2021, 84(4): 1226-1231.
Han X, Tang X, Luo X, et al. Isolation and identification of three new sterigmatocystin derivatives from the fungus Aspergillus versicolor guided by molecular networking approach[J]. Chem Biodivers, 2020, 17(6): e2000208.
Chao R, Hou X M, Xu W F, et al. Targeted isolation of asperheptatides from a coral-derived fungus using LC-MS/MS-based molecular networking and antitubercular activities of modified cinnamate derivatives[J]. J Nat Prod, 2021, 84(1): 11-19.
基金項目:国家自然科学基金面上项目(No. 41976105);山东省泰山学者青年专家项目(No. tsqn202103153)
作者简介:方岫琴,女,生于2002年,主要研究方向为微生物天然产物,E-mail: fangxiuqin@stu.ouc.edu.cn
*通信作者,E-mail: zhangguojian@ouc.edu.cn
