酿酒酵母细胞cdc50基因敲除及其转录组测序分析
2024-05-14李鑫玉唐秀琴李子航
李鑫玉,唐秀琴,李子航,刘 佳,王 琦,邹 伟*
(1.昆明医科大学 公共卫生学院,云南 昆明 650500;2.昆明医科大学 基础医学院,云南 昆明 650500)
酿酒酵母(Saccharomyces cerevisiae)是一种单细胞真核微生物,常用于制作面包、啤酒、白酒、葡萄酒等产品。酿酒酵母已完成全基因组测序、转录组分析和蛋白相互作用网络图的绘制,为基因组遗传和进化等各个方面提供了丰富的信息[1]。酿酒酵母细胞生命周期短,繁殖较快,易于培养,且大部分基因表达调控和信号转导机制与高等动植物具有高度同源性,也存在很多与哺乳动物细胞相似的保守生化机制[2]。
Cdc50p属于TMEM家族,是一种保守的完整跨膜蛋白,主要定位于晚期内体或前空泡腔室[3]。TMEM家族蛋白属于整合膜蛋白中的一种,是一类至少含有一段完全通过生物膜或部分通过生物膜的跨膜段蛋白质[4]。Cdc50p作为P4-腺嘌呤核苷三磷酸(adenosine-triphosphate,ATP)酶的β亚基,确保了P4-ATP酶从内质网输出并正确定位[5-6]。此外,Cdc50p还参与磷脂易位、内吞作用和其他细胞过程[7-8]。Cdc50的同源基因也存在于其他物种中,在人类中发现了tmem30a和tmem30b两个同源基因,tmem30a是与p4-ATP酶相互作用的主要β亚基,tmem30a的缺失会导致骨骼肌再生障碍[9],内皮细胞增殖减少和视网膜血管发育受损[10]。目前已知cdc50基因对于P4-ATP酶发挥正常生理功能十分重要,其人类同源基因tmem30a也参与多种细胞的生长发育调控。Cdc50基因同属于TMEM家族,该家族部分基因与癌症发生发展有关[11-12],但cdc50是否参与了某些癌细胞的发生发展尚不可知。
转录组学是从整体水平上研究细胞中基因转录的情况及其转录调控规律[13],基于高通量测序技术的转录组测序是通过对组织中的核糖核酸(ribonucleic acid,RNA)[包括信使核糖核酸(messenger ribonucleic acid,mRNA)和非编码RNA]进行测序,全面快速地检测特定环境和时间条件下的微生物基因表达信息,目前已普遍应用于转录组学相关研究工作[14-15]。本研究以PYM14质粒为模板,采用一步聚合酶链式反应(polymerase chain reaction,PCR)法扩增cdc50同源重组双交换片段,醋酸锂(lithium acetate,LiAc)转化法敲除cdc50基因,测定cdc50基因敲除对细胞生长的影响,基于Illumina NovaSeq 6000高通量测序平台对野生型酵母BY4742菌株和Δcdc50突变菌株进行转录组分析,筛选二者差异表达基因(differentially expressed gens,DEGs),并对其进行基因本体论(gene ontology,GO)和京都基因与基因组百科全书(Kyotoencyclopediaofgenes and genomes,KEGG)通路富集分析,以期揭示Δcdc50突变菌株基因表达谱和信号通路变化,为进一步揭示cdc50基因的分子调控机制提供参考依据。
1 材料与方法
1.1 材料与试剂
1.1.1 菌种与引物
野生型酿酒酵母(Saccharomyces cerevisiae)BY4742、质粒PYM14[带有遗传霉素418(geneticin 418,G418)的KanMX筛选标记,在大肠杆菌中具有氨苄青霉素(ampicillin,Amp)抗性]:云南生物资源保护与利用国家重点实验室提供。
根据酵母基因组数据库(Saccharomyces genomedatabase,SGD)的cdc50基因(S000000690)和PYM14的质粒序列设计基因敲除引物、敲除验证引物和实时荧光定量PCR(real-time fluorescent quantitative PCR,RT-fqPCR)引物,引物由擎科生物有限公司合成,见表1。
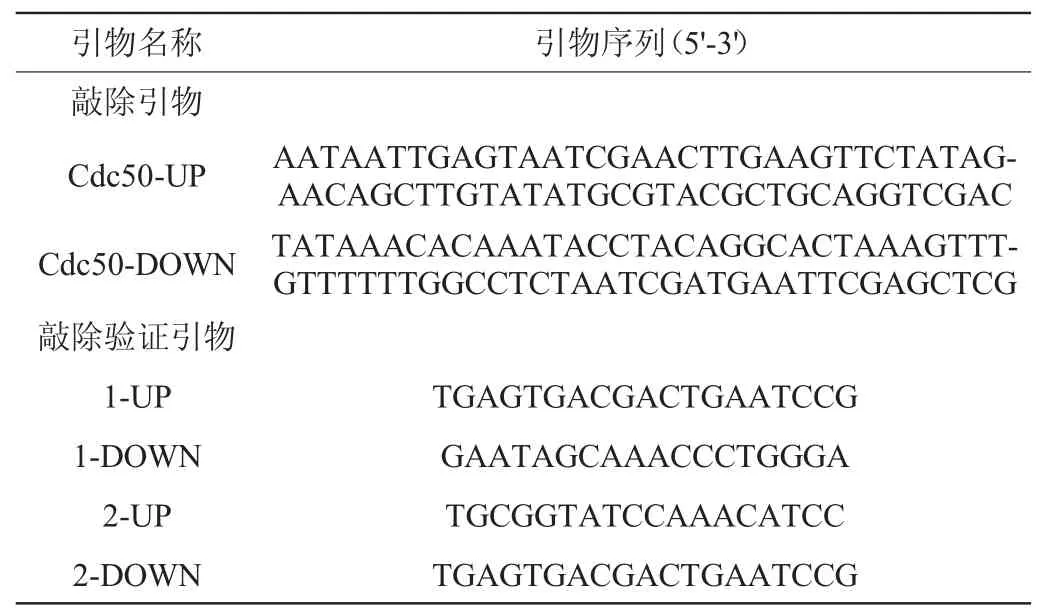
表1 本研究所用引物序列Table 1 Primer sequence used in this study
1.1.2 化学试剂
胶回收试剂盒、PCR混合酶:北京聚合美生物科技有限公司;琼脂糖:赛国生物科技有限公司;G418:北京兰杰柯科技有限公司;核糖核酸(RNA)提取试剂盒、酵母基因组提取试剂盒、互补脱氧核糖核酸(complementary deoxyribonucleic acid,cDNA)合成试剂盒:北京天根生物科技公司;鲑鱼精脱氧核糖核酸(salmon sperm DNA,SS-DNA)、聚乙二醇3350(polyethylene glycol 3350,PEG3350)、醋酸锂:北京索莱宝科技有限公司。本研究所用试剂均为分析纯或生化试剂。
1.1.3 培养基
酵母浸出粉胨葡萄糖(yeast extract peptone dextrose,YPD)培养基[16]:2%葡萄糖、2%蛋白胨、1%酵母提取物溶解于去离子水中。YPD固体培养基:在液体YPD基础上加入1.5%琼脂糖,121 ℃高压灭菌15 min。
含G418的YPD培养基:每150 mL YPD培养基加入质量浓度为100 mg/mL的G418母液450 μL,终质量浓度为300 μg/mL,用于酵母转化子的筛选。
1.2 仪器与设备
Allegra X-30低温高速离心机:美国贝克曼库尔特公司;BiOmetra PCR扩增仪器:德国耶拿公司;SPX-70B恒温培养箱:天津市泰斯特仪器有限公司;TS-100B恒温摇床:上海天呈实验仪器制造有限公司;UV-15紫外灯切胶仪:北京兰杰柯科技有限公司;DZKW水浴锅:上海东星建材试验设备有限公司;MQX200酶标仪:美国伯腾仪器有限公司;Light Cycler96实时荧光定量PCR仪:美国Life Technologies公司;Ts2R-FL显微镜:日本NIKON公司。
1.3 方法
1.3.1 酿酒酵母cdc50基因敲除
以质粒PYM14为模板,Cdc50-UP、Cdc50-DOWN为引物进行PCR扩增,获得cdc50基因敲除组件[17]。PCR扩增体系和条件按照PCR混合酶说明书进行,扩增完成后取10 μL PCR产物进行1.5%琼脂糖凝胶电泳检测,检测成功后进行胶回收。采用醋酸锂转化法[18]将敲除组件转化到酿酒酵母BY4742中,取转化后的菌液200 μL涂布于含300 μg/mL G418的YPD抗性平板上,30 ℃培养3~4 d观察是否有转化子。
1.3.2 提取转化子基因组验证
挑取在抗性平板上生长出来的转化子接种于含300 μg/mL的G418的YPD液体培养基中180 r/min、30 ℃培养24 h,按照酵母基因组提取试剂盒提取转化子基因组后采用验证引物1-UP、1-DOWN和2-UP、2-DOWN按照PCR混合酶说明书体系进行扩增,采用琼脂糖凝胶进行检测。
1.3.3 实时荧光定量PCR验证
将野生型酿酒酵母BY4742和阳性转化子分别在YPD液体培养基和含300 μg/mL的G418的YPD液体培养基中180 r/min、30 ℃培养24 h,根据RNA提取试剂盒提取酵母RNA,使用紫外可见微量分光光度计于波长260 nm处对所提取的RNA吸光度值进行检测,之后根据反转录试剂盒进行反转录得到cDNA,参照qPCR试剂盒说明书要求进行扩增,每组样本设3个复孔,以β-肌动蛋白(β-actin)为内参对照,采用2-ΔΔCt法[19]计算各基因转录水平差异。
1.3.4 细胞形态和生长情况
挑取适量对数期突变菌株Δcdc50和野生型酿酒酵母BY4742细胞,接种到对应的YPD液体培养基中,保证初始样本OD600nm值相同,置于摇床中180 r/min、30 ℃培养,每隔2 h取适量培养液测其OD600nm值,绘制生长曲线,24 h后取少量菌株BY4742和Δcdc50细胞菌液于载玻片上在显微镜下观察其形态。以菌株BY4742培养24 h后测定的OD600nm值为对照[20],按以下公式计算基因敲除组酵母细胞相对生长率:
1.3.5 转录组测序及分析
取酿酒酵母BY4742和Δcdc50菌株于5 mL YPD液体培养基中180 r/min、30 ℃培养24 h,12 000 r/min离心1 min收集沉淀,用双蒸水洗涤2次,提取酵母细胞的总RNA后将样本送至北京诺禾致源科技股份有限公司,采用Illumina NovaSeq 6000测序平台进行文库构建和转录组测序工作。测序片段被高通量测序仪测得的图像数据经CASAVA碱基识别转化为序列数据,为保证数据分析的质量及可靠性,需要对原始数据(raw data)进行过滤,同时对纯净数据(Clean Data)进行Q20、Q30和GC含量计算,后续所有分析均是基于纯净数据进行的高质量分析[21]。使用DESeq2软件进行基因差异表达分析,同时结合进行多重假设检验校正后的P<0.05和|log2Fold Change|≥1筛选差异表达基因。通过Cluster Profiler软件实现差异表达基因GO富集分析和KEGG通路富集分析,从而获得差异表达基因显著相关的生物学功能和代谢通路[22]。
1.3.6 数据分析
组间采用t检验分析差异性,应用Graphpad prism9进行绘图。
2 结果与分析
2.1 cdc50基因敲除及验证
Cdc50基因敲除组件的PCR扩增产物的电泳图见图1A。由图1A可知,得到1.7 kb的双交换同源重组片段,该片段包含cdc50基因上游48 bp同源序列、质粒片段和cdc50基因下游48 bp同源序列,胶回收该片段可见片段浓度高,可用于后续的cdc50基因敲除实验。双交换片段具有KanMX筛选标记编码序列,可在G418平板上进行阳性转化子筛选。

图1 cdc50基因敲除双交换片段扩增(A)、转化子基因组验证(B、C)、转化子测序(D)和实时荧光定量聚合酶链式反应验证(E)Fig. 1 Gene cdc50 knockout double exchange fragment amplification (A), transformation genome validation (B,C), transformation sequencing (D) and real-time fluorescence quantitative polymerase chain reaction validation (E)
醋酸锂转化法转化酿酒酵母3 d后,G418平板长出3个转化子,提取转化子基因组。采用验证引物1进行PCR扩增的电泳图见图1B。由图1B可知,1、2号转化子基因组得到大小约为2.4 kbp的正确条带,3号转化子基因组和野生型酿酒酵母BY4742菌株基因组得到约1.9kbp的条带。筛选扩增条带正确且明亮的2号转化子基因组采用验证引物2-UP/Z-DOWN扩增,扩增结果见图1C。由图1C可知,得到大小约为1.7 kbp的正确条带。由图1D测序结果表明,2号克隆菌株在cdc50基因中插入了G418编码序列。同时提取野生型酿酒酵母BY4742和2号转化子RNA进行实时荧光定量PCR检测,由图1E可知,cdc50基因在该转化子中不表达,采用drs2基因作为对照,drs2基因在该转化子中相对表达水平为0.77。结果表明2号转化子酵母细胞的cdc50基因已被成功敲除。
2.2 细胞生长和形态测定
以培养24 h的野生型BY4742细胞的OD600nm值为对照,分别测量突变菌株Δcdc50在不同时间点的OD600nm值,计算相对生长率,结果见图2A。
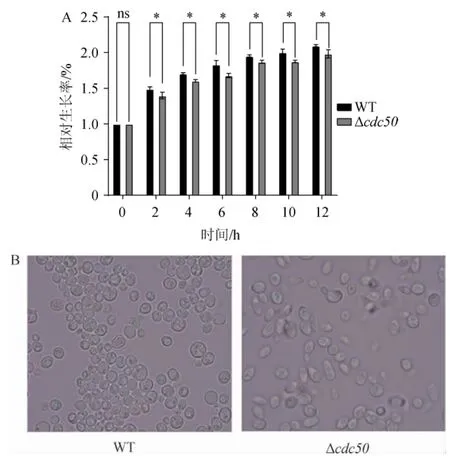
图2 野生型酿酒酵母菌株BY4742和突变菌株Δcdc50的相对生长率(A)和细胞形态(B)Fig. 2 Relative growth rate (A) and cell morphology (B) of wild type Saccharomyces cereviniae strain BY4742 and mutant strain Δcdc50
由图2A可知,培养2 h、4 h、6 h、8 h、10 h、12 h的突变菌株Δcdc50的相对生长率较野生型酵母BY4742菌株降低,均具有显著差异(P<0.05),其中培养12 h后,野生型BY4742菌株培养的相对生长率为2.10%,突变菌株Δcdc50为1.98%。菌株BY4742和Δcdc50细胞形态见图2B。由图2B可知,显微镜下也显示突变菌株Δcdc50细胞形态与野生型BY4742对比发生了明显的改变。野生型BY4742酿酒酵母细胞为卵圆形,细胞壁完整,边缘清晰;突变菌株Δcdc50细胞多为长杆状,其边缘较为模糊。
2.3 转录组测序数据质控分析
野生型酿酒酵母BY4742菌株和突变菌株Δcdc50进行转录组测序分析后结果见表2。由表2可知,6个样本共获得264 628 560条纯净序列,总长度约39.63 Gb碱基,其Q20均>97%,Q30均>91%,表明原始数据的质量较高,可用于后续的生物信息学分析[23]。
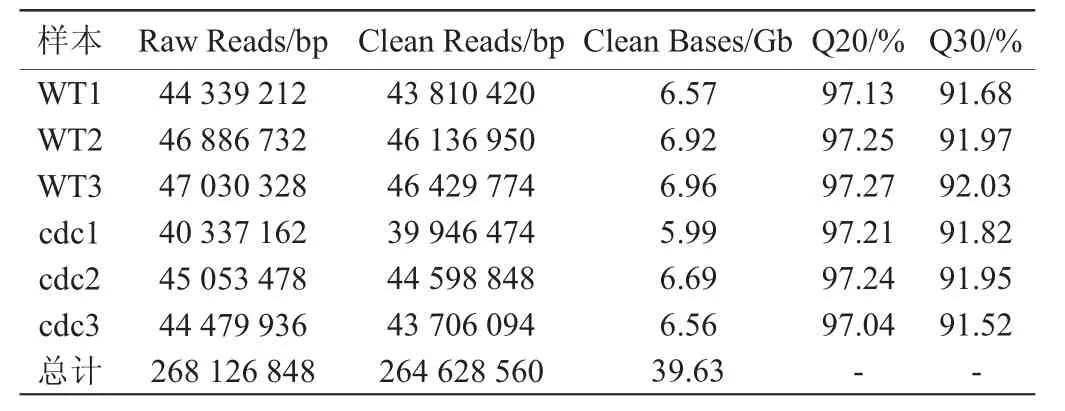
表2 测序数据统计结果Table 2 Statistics results of sequencing data
2.4 差异表达基因分析
野生型酿酒酵母BY4742和突变菌株Δcdc50组基因共表达结果见图3A。由图3A可知,均表达6 336个基因,其中两组共表达基因6 251个。具有显著统计学差异的基因火山图见图3B。由图3B可知,两组共筛选出581个差异表达基因(DEGs),其中cdc50基因敲除后发生上调的基因有271个,下调的基因有310个。
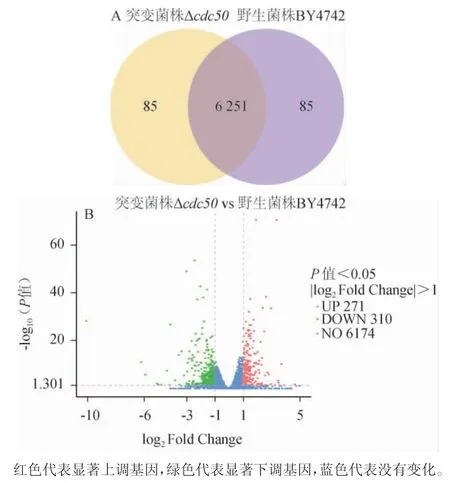
图3 基因共表达韦恩图(A)和差异基因火山图(B)Fig. 3 Venn diagram of gene co-expression (A) and volcano map of differential gene (B)
2.5 GO富集分析
差异表达基因GO富集分析结果见图4。由图4可知,Δcdc50菌株的差异表达基因(DEGs)主要富集在生物学过程(biological process,BP)的氧化还原过程、碳水化合物代谢、细胞氮化合物分解代谢、杂环分解代谢和有机环状化合物分解代谢等;细胞组成(cellular component,CC)结果显示DEGs主要分布于薄膜、细胞壁和外部封装结构等部位;分子功能(molecular function,MF)富集结果显示,DEGs主要影响氧化还原酶活性、辅助因子结合、跨膜转运体的活性以及水解酶活性等来发挥作用。其中差异表达基因最主要影响的是细胞氧化还原过程,在BP中富集到氧化还原过程的DEGs有44个,在MF中也有33个DEGs富集到氧化还原酶活性。参与表达的氧化还原酶有6-磷酸葡萄糖酸脱氢酶(Gnd1p)、铁螯合还原酶(Fre7p)、C-24(28)甾醇还原酶(Erg4p)和血红素加氧酶(Hmx1p)等。除此之外,在MF中也有29个DEGs富集到跨膜转运体活性,参与被动跨膜转运蛋白活性的有Hsp30p、Aqy3p、Yro2p和Mrh1p。转运蛋白介导生物膜内外化学物质的跨膜转运及信号交换,对细胞摄取营养物质、外排代谢产物及信号转导等方面起着重要作用[24-25]。通过GO富集分析推测酿酒酵母cdc50基因可能与细胞的氧化还原过程和跨膜转运密切相关。

图4 差异表达基因基因本体论富集分析结果Fig. 4 Results of Gene Ontology enrichment analysis of differentially expressed genes
2.6 KEGG分析
从KEGG富集结果中选取最显著的20个KEGG通路绘制柱状图进行展示,以P<0.05作为显著性富集的阈值,结果见图5。由图5可知,差异表达基因(DEGs)主要参与了糖酵解/糖异生、脂肪酸降解、碳代谢、次生代谢物的生物合成等过程,也与过氧化物酶体的作用密切相关。其中,富集通路中DEGs富集水平最显著的糖酵解/糖异生代谢通路,该通路涉及到的基因为cdc19、pcd1、adh2、tdh3和pox1等,其中Cdc19p是酵母糖酵解的丙酮酸激酶[16]。除此之外,次生代谢物的生物合成富集DEGs数目最多,碳代谢通路次之。通过KEGG富集分析推测cdc50基因对酿酒酵母细胞糖代谢过程具有重要调控作用。

图5 差异表达基因京都基因与基因百科全书(KEGG)富集分析散点图Fig. 5 Scatter plot of Kyoto Encyclopedia of Genes and Genomes enrichment analysis of differentially expressed genes
3 结论
酿酒酵母细胞cdc50基因缺失会抑制细胞生长,改变细胞形态。利用RNA-seq转录组测序技术对野生型酵母菌株BY4742和突变菌株Δcdc50进行比较分析后表明,两组差异表达基因共有581个,其中cdc50基因敲除后上调基因有271个,下调基因有310个。GO功能分析表明,差异表达基因主要富集在生物学过程中的氧化还原过程等,分子功能中的氧化还原酶活性、辅助因子结合和跨膜转运体活性等。KEGG富集分析表明,差异表达基因主要富集在糖酵解/糖异生、脂肪酸降解和碳代谢通路等。本研究为揭示cdc50基因的生物学作用和相关分子机制的提供了理论基础。
