奶牛乳房炎相关IFNE基因的克隆、表达及功能研究
2024-04-24王正兴罗仍卓么王晋鹏包斌武王兴平
王正兴,罗仍卓么,王晋鹏,包斌武,王兴平
(1.宁夏大学动物科技学院,银川 750021;2.宁夏回族自治区反刍动物分子细胞育种重点实验室,银川 750021)
【研究意义】奶牛乳房炎通常是由病原体感染乳腺组织引起的炎症反应,是奶牛最常见的疾病之一[1],可导致奶牛的产奶量下降和乳品质降低[2-3],使得牛奶的经济效益下降,并且会增加疾病处理成本,以及奶牛过早淘汰而增加总体生产成本,给牧场造成巨大的经济损失。【前人研究进展】研究发现,造成奶牛乳房炎的病原体大致分为传染性病原体和环境性病原体,其中大肠杆菌(Escherichiacoli)、金黄色葡萄球菌(Staphylococcusaureus)、无乳链球菌(S.agalactiae)和发育不良链球菌(S.pyogenes)被认为是传染性病原体,可在乳腺中定植,感染乳腺组织,产生炎症[4-8]。在众多病原体中,大肠杆菌被认为是导致乳房炎的主要病原体之一[9]。脂多糖(LPS)是革兰氏阴性菌细胞壁的主要结构成分,也是大肠杆菌的内毒素,乳腺组织接触LPS会引发炎症的发作[10]。炎症基因表达的一个重要转录调节因子是转录的Janus激酶/信号转导器和转录激活剂(JAK-STAT)[11],在特异性皮炎免疫应答中,胸腺基质淋巴细胞生成素(Thymic stromal lymphopoietin,TSLP)是一种具有致炎作用的细胞因子,其介导的反应通过JAK-STAT信号通路[12]。据报道,JAK-STAT在LPS、干扰素-γ(Interferon γ,IFNγ)和其他细胞因子激活的炎症信号通路中起作用[13]。JAK-STAT调节编码促炎细胞因子和趋化因子靶基因的转录[14]。大量哺乳动物细胞因子,包括白细胞介素和干扰素,通过JAK-STAT信号通路来调节细胞内信号传导[15]。干扰素(Interferon,IFN)是参与细胞生长调节和免疫激活的多功能分泌蛋白[16]。根据感染病原体的不同,IFN信号传导可能对细菌感染产生保护作用或有害作用[17]。干扰素ε(IFNE)是一种I型IFN[18],Choo等[19]在对哺乳动物如穿山甲的研究中发现,IFNE仅在上皮细胞中表达,主要在粘膜免疫、皮肤感染以及炎症方面也起着一定的作用[20-21]。【本研究切入点】目前,奶牛IFNE的序列信息以及分子功能尚不清楚。【拟解决的关键问题】本试验克隆奶牛IFNE基因的编码区(CDS),并通过qPCR及RNAi技术对IFNE基因在奶牛乳腺上皮细胞(bMECs)炎症反应中的表达模式及功能进行分析,以期为奶牛乳房炎的分子治疗提供参考。
1 材料与方法
1.1 试验材料
1.1.1 主要仪器 采用高速冷冻离心机(Eppendorf)、普通PCR仪(Bio-Rad公司)、多功能酶标仪(Bio-Rad公司)、电泳凝胶成像仪(Bio-Rad公司)、核酸凝胶电泳仪(Bio-Rad公司)、CFX 96 Touch 荧光定量PCR仪(Bio-Rad公司)和倒置荧光显微镜(Olympus)。
1.1.2 主要试剂 采用DMEM/F12培养基(Hyclone)、PBS(Hyclone)、0.25%胰酶消化液(Hyclone)、胎牛血清(BI)、Trizol(Takara)、PCR 2×TaqMaster Mix(诺唯赞生物科技有限公司)、胶回收试剂盒购(Omega公司)、2×M5 HiPer SYBR Premix EsTaq(北京聚合美生物科技有限公司)、X-treme GENE HP DNA转染试剂(Roche)、OPTI-MEM(Gibco)、2×TaqMaster Mix(诺唯赞生物科技有限公司)和RNase-free ddH2O(北京天根生化科技有限公司)。
1.1.3 奶牛乳腺上皮细胞 选用宁夏回族自治区反刍动物分子细胞育种重点实验室冻存的经鉴定的bMECs[22]。
1.2 研究方法
1.2.1 乳腺上皮细胞培养及炎症诱导 在液氮中取出bMECs后接种于含10% FBS的DMEM/F12培养基,在37 ℃含5% CO2和100%饱和湿度条件下培养,当单层细胞融合达70%时,采用LPS(工作浓度为50 ng/μL)诱导bMECs,使其产生炎症反应。在LPS诱导的第0、3、6、12和24小时收集细胞,用于RNA提取。
1.2.2 引物设计、RNA提取与cDNA合成 利用Primer Permier 5.0软件设计IFNE基因的PCR扩增引物(表1)。采用Trizol法提取LPS诱导的bMECs总RNA。用琼脂糖凝胶电泳仪和多功能酶标仪检测RNA的完整性和浓度。选择OD260/280值在1.8~2.0且较完整的RNA进行反转录得到cDNA。反转录体系及条件:①去除基因组DNA的反应:向无RNA酶离心管加入5×g DNA Eraser Buffer 2.0 μL,gDNA Eraser 1.0 μL,总RNA为1000 ng,最后加RNase free ddH2O 补至10.0 μL,混匀,瞬时离心,反应条件为42 ℃ 15 min,85 ℃ 5 s,4 ℃保存。②反转录反应:向步骤①中10 μL的反应液中加入PrimerScript RT Enzyme mix I 1.0 μL,RT Primer Mix 1.0 μL,5×primerscript Buffer 2(for Real Time)4.0 μL,RNase free ddH2O 补至20.0 μL,混匀,瞬时离心,反应条件为37 ℃ 15 min,85 ℃ 5 s,4 ℃保存。

表1 引物序列Table 1 Primer sequences
1.2.3IFNE基因的PCR扩增与回收、测序 以单链cDNA为模板,利用基因特异性引物扩增IFNE基因片段。反应体系:2×TaqPCR Master 10.0 μL,上、下游引物各0.8 μL,cDNA 1.0 μL,RNAse free ddH2O补至20.0 μL。PCR反应条件:95 ℃预变性3 min; 95 ℃变性15 s,54 ℃退火15 s,72 ℃延伸1.5 min,39个循环; 72 ℃终延伸5 min,4 ℃保存。PCR产物经1.5%琼脂糖凝胶电泳进行检测后,用DNA凝胶回收试剂盒进行扩增目标片段的回收,与pMDTM19-T载体进行连接。反应体系:Solution buffer 5.0 μL,纯化产物4.0 μL,pMDTM19-T 1.0 μL,4 ℃连接过夜,将连接产物进行细菌转化,并通过菌液PCR,选取扩增出条带单一且大小符合预期的菌液,送往杨凌天润奥科生物科技有限公司进行菌液测序。
1.2.4IFNE基因的生物信息学分析 根据DNAMAN软件将IFNE基因菌液测序所得结果与GenBank数据库进行比对,并利用ORF Finder(https://www.ncbi.nlm.nih.gov/orffinder/)在线软件分析基因的开放阅读框,并推导其所编码的氨基酸的序列;利用在线工具ExPASy-ProtParam(https://web.expasy.org/protparam/)分析IFNE基因所编码蛋白质的理化性质;利用在线工具Conserved Domains(https://www.ncbi.nlm.nih.gov/Structure/cdd/wrpsb.cgi)对IFNE蛋白进行保守结构域分析。
1.2.5IFNE基因干扰片段的合成与转染 为探究IFNE基因对bMECs炎症反应中的影响,委托生工生物工程(上海)股份有限公司合成3个奶牛IFNE基因的特异性干扰片段(si-152、si-653和si-730)以及对照组si-NC片段(表2)。细胞转染方法:将生长状态良好而且无污染的bMECs接种至6孔细胞培养板中,当单层细胞融合达70%时,配制甲液(si-干扰组:5 μL 干扰片段+100 μL OPTI;NC组:5 μL si-NC+100 μL OPTI)和乙液(3 μL X-tremeGENE HP转染试剂+100 μL OPTI);甲乙液均静止5 min,将甲和乙液等体积混合,静止20 min,每组进行3个重复;转染24 h后,采用荧光倒置显微镜观察转染情况;转染48 h后,在6孔板中加入1 ug/mL的LPS刺激bMECs炎症的发生,6 h后收集细胞进行RNA提取。

表2 奶牛IFNE的干扰片段序列Table 2 Interference fragment sequences of IFNE in dairy cows
1.2.6 qPCR实验 采用Primer Permier 5.0软件设计qPCR引物(表1),利用qPCR技术检测IL-6、IL-8、IL-1β、IFNE、IFNB、IFNK、CASP3、CASP9、BAD和IL-10RB基因mRNA的表达水平。qPCR反应体系:2×M5 HiPer SYBR Premix EsTaq10.0 μL,上、下游引物各0.8 μL,cDNA 1.5 μL,RNase free ddH2O补至20 μL;反应条件:95 ℃ 30 s,95 ℃ 5 s,55 ℃ 30 s,40个循环。
1.2.7 数据统计分析 以GADPH为内参,所有试验均进行3个重复,采用2-△△Ct法计算各检测基因的表达量,通过GraphPad Prism 8.3.0软件的独立样本T检验进行两组间差异显著性分析。P<0.05表示具有统计学意义,*表示差异显著(P<0.05);**、***和****表示差异极显著(P<0.01,P<0.001和P<0.0001)。
2 结果与分析
2.1 奶牛IFNE基因编码区的克隆
利用IFNE基因PCR引物进行扩增,扩增产物经琼脂糖凝胶电泳检测,结果显示,有清晰且单一的目的条带,其扩增片段大小为838 bp(图1),该序列已递交GenBank(ID:OQ909434)。测序结果显示,测序序列各碱基均为单峰,且582 bp的CDS区序列与牛基因组序列比对结果的同源性为100%,表明本试验成功克隆出奶牛IFNE基因的CDS区。利用ORF Finder对IFNE基因的测序序列进行分析发现,IFNE基因的CDS全长为582 bp(546~1127 bp),可编码193个氨基酸(图2)。

图1 奶牛IFNE基因的扩增产物电泳Fig.1 Electrophoresis of amplification products of IFNE gene in dairy cows

下划线表示扩增引物结合位置。The underline indicates the binding sites for primer sequences.图2 奶牛IFNE基因所编码的氨基酸序列Fig.2 Amino acid sequence encoded by IFNE in dairy cows
2.2 奶牛IFNE蛋白的生物信息学分析
利用ExPASy-ProtParam软件对IFNE基因所编码蛋白质的特性分析结果表明,IFNE蛋白的分子式为C1017H1631N279O288S10,原子总数为3225个,理论等电点为8.59。在IFNE蛋白的氨基酸组成中,亮氨酸(Leu)和谷氨酰胺(Gln)所占比例较高,分别为16.6%和11.4%;带负电荷的残基总数天冬氨酸(Asp)+谷氨酸(Glu)为19个,带正电荷的残基总数精氨酸(Arg)+赖氨酸(Lys)为22个;不稳定性指数(II)为48.56。跨膜结构域分析结果表明,奶牛IFNE蛋白存在于细胞内,无跨膜结构域。根据对其保守结构域的分析,IFNE蛋白质包含一个IFab超家族结构域,属于I型干扰素成员之一,初步推测IFNE在炎症反应的发生和发展过程中可能起到作用。使用DNAMAN软件对本研究获得的奶牛IFNE蛋白序列与野牛(XP_005887920.1)、水牛(XP_006054631.1)、绵羊(XP_011982517.1)、马鹿(XP_043746624.1)、山羊(XP_013821322.2)、非洲狒狒(XP_047623512.1)、蓝鲸(XP_036711910.1)、黑猩猩(XP_054513956.1)和马(XP_014686768.1)9个物种进行同源性比对,结果显示各物种之间IFNE蛋白的同源性较高(图3),表明各物种的IFNE蛋白可能具有相似的生物学功能。

图3 不同物种 IFNE 蛋白质的同源聚类树Fig.3 Homologous cluster tree of IFNE proteins in different species
2.3 奶牛乳腺上皮细胞炎症细胞模型的构建及IFNE基因表达量的检测
通过体外培养bMECs(图4-A),采用LPS诱导bMECs进行炎症细胞模型构建。为验证炎症细胞模型是否构建成功,采用qPCR检测炎症因子IL-6、IL-8和IL-1β基因mRNA水平的表达量。结果表明,与0 h相比,IL-6和IL-1β在LPS诱导3、6、12和24 h的bMECs中表达量显著上调(P<0.05)(图4-B、4-D),IL-8在LPS诱导的3、6、12和24 h的表达量极显著上调(P<0.01)(图4-C),表明成功构建了细胞炎症模型。同时,IFNE基因在LPS诱导bMECs 6 h的表达量与对照组0 h的表达量相比显著上调(P<0.0001)(图5),用于后续试验。
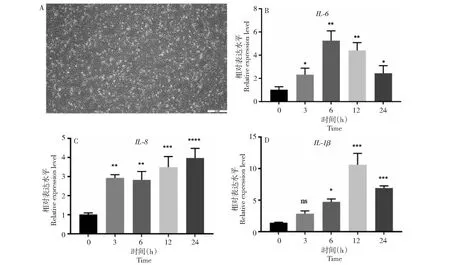
A.培养的bMECs;B. IL-6在LPS诱导bMECs不同时间的表达量;C. IL-8在LPS诱导bMECs不同时间的表达量;D. IL-1β在LPS诱导bMECs不同时间的表达量。*表示差异显著(P<0.05);**、***和****表示差异极显著(P<0.01、P<0.001、P<0.0001)。下同。A. Culture of bMECs; B. Relative expression of IL-6 in LPS induced bMECs at different time; C. Relative expression of IL-8 in LPS induced bMECs at different time; D. Relative expression of IL-1β in LPS induced bMECs at different time. * indicates significant difference(P<0.05);**, ***and****indicate extremely significant difference(P<0.01, P< 0.001,P<0.0001).The same as below.图4 bMECs炎症模型构建的验证Fig.4 Validation of the construction of inflammatory model for bMECs

图5 IFNE在bMECs炎症诱导不同时间的表达Fig.5 Relative expression of IFNE of inflammation induction in bMECs at different times
2.4 IFNE干扰片段的筛选与检测
为研究干扰IFNE后在LPS诱导的bMECs炎症反应中基因的表达量,将IFNE的3条特异性的IFNE干扰片段转染至bMECs后设置转染Cy3标记的对照组(NC)。转染24 h后,倒置荧光显微镜的成像结果显示Cy3所标记的对照组(NC)的细胞中均有清晰的细胞轮廓(图6-A、6-B),结果表明转染成功。此外,qPCR结果显示,与对照组(NC)相比,si-152、si-653和si-730均能够极显著抑制IFNE的表达(P<0.01),其中si-730片段的干扰效果最好(图6-C)。因此,选择si-730片段进行后续的IFNE基因干扰试验研究。

A.Cy3标记的对照组(NC)转染bMECs的明场图像;B.Cy3标记的对照组(NC)转染bMECs的荧光图像;C.干扰片段转染组细胞的IFNE表达量检测结果。A.Cy3-labeled control(NC)transfected into bMECs; B.Fluorescent image of Cy3-labeled control(NC)transfected into bMECs; C.qPCR results of three interfering fragments. 图6 IFNE干扰效率的qPCR检测Fig.6 Detection of IFNE interference efficiency by qPCR
2.5 IFNE基因干扰对炎性bMECs中炎症因子基因表达的影响
为研究干扰IFNE对bMECs炎症反应的影响,本试验转染si-730至LPS诱导6 h的bMECs中,在此基础上,采用qPCR法检测bMECs内IFNE基因、炎症因子IL-1β、IL-6和IL-8在IFNE基因干扰后的表达水平。结果表明,与对照组(NC)相比,干扰组的IFNE基因和IL-8的表达量显著下调(P<0.05)(图7-A、7-C),IL-6和IL-1β的表达量极显著下调(P< 0.01)(图7-B、7-D)。上述结果表明,干扰IFNE基因可通过下调促炎症因子(IL-1β、IL-6和IL-8)的表达,从而缓解bMECs炎症反应。

A. 干扰IFNE后IFNE基因在炎性乳腺上皮细胞中的表达量; B. IL-6在干扰IFNE后炎性乳腺上皮细胞的表达量;C. IL-8在干扰IFNE后炎性乳腺上皮细胞的表达量; D. IL-1β在干扰IFNE后炎性乳腺上皮细胞的表达量。A. Relative expression of IFNE in inflammatory bMECs after interference with IFNE; B. Relative expression of IL-6 in inflammatory bMECs after interference with IFNE; C. Relative expression of IL-8 in inflammatory bMECs after interference with IFNE; D. Relative expression of IL-1β in inflammatory bMECs after interference with IFNE. 图7 干扰IFNE 对LPS诱导炎性bMECs主要炎症因子基因表达的影响Fig.7 Relative expression of inflammatory factors in inflammatory bMECs after interference with IFNE
2.6 IFNE干扰对IFNE相关基因及细胞凋亡相关基因表达的影响
为研究干扰IFNE对IFNE相关基因及细胞凋亡相关基因表达的影响,利用qPCR技术检测干扰IFNE后bMECs内JAK-STAT信号通路中的IFNE相关基因以及细胞凋亡相关基因的表达量(图8),结果表明,与对照组(LPS+NC)相比,IFNB、IFNK和IL-10BR基因的表达量均显著下调(P<0.05)。此外,CASP3、CASP9和BAD基因的表达量均极显著下调(P<0.01)。上述结果表明,IFNE相关基因参与促进调节bMECs的凋亡,IFNE可能促进细胞炎症反应。

图8 干扰IFNE对炎性乳腺上皮细胞内IFNE相关基因表达的影响Fig.8 Relative expression of IFNE related genes in inflammatory bMECs after interference with IFNE
3 讨 论
3.1 IFNE基因克隆及蛋白生物信息学分析
奶牛乳房炎严重影响乳品质,对牧场的经济收益造成损失,其中病原微生物E.coli是诱发临床型乳房炎的主要病原体之一[24]。JAK-STAT参与调节细胞因子的产生,以及调控炎症反应和免疫反应,其与乳房炎的发生、发展和调控密切相关[15],而IFNE基因在JAK-STAT信号通路中起着至关重要的作用[25]。本研究通过克隆测序获得了奶牛IFNE基因CDS区序列,发现其可以编码一条由193个氨基酸组成的不稳定亲水性蛋白。在IFNE蛋白的保守结构域分析中,IFNE属于IFab超家族中,IFN-α和IFN-β与IFNE属于同一超家族基因,免疫系统长期暴露于IFN-α和IFN-β经常导致自身抗体和自身免疫性疾病的发生,如系统性红斑狼疮的发生[26]。基于IFNE超家族基因的分析,结果显示IFNE与IFN-α和IFN-β在免疫反应中起到相似的作用,推测IFNE在炎症反应的发生和发展过程中可能起到作用[26]。在山羊乳腺炎的研究中发现,干扰素(IFN)参与山羊乳房炎的调节[27],本研究奶牛与山羊的IFNE蛋白高度同源,推测奶牛IFNE蛋白可能参与调节bMECs炎症反应,与奶牛乳房炎的发病率相关。
3.2 乳腺上皮细胞炎症模型构建及IFNE的表达模式分析
bMECs不仅参与乳汁的合成与分泌,还与乳腺的应答有关[28]。外源性病原微生物感染奶牛乳腺时会被bMECs表面的特异性受体识别并会激活细胞免疫应答机制,产生炎症细胞因子等介质,进而导致炎症反应的发生[28]。有报道显示,LPS可诱导bMECs发生炎症反应[29],并且JAK-STAT信号通路可参与促炎细胞因子对LPS诱导的细胞炎症的调节[20]。在奶牛乳腺炎的研究中,通常以LPS诱导的bMECs为研究材料,构建炎症细胞模型[30]。此外,炎症因子IL-6、IL-1β和IL-8都会引发对免疫稳态、炎症、发育和代谢的生理调控[31]。本研究用LPS诱导bMECs产生炎症反应,发现炎症因子IL-6、IL-1β和IL-8的表达量都显著上调,这与杨箭等[32]成功建立炎症模型的结果相似,表明本研究建立的bMECs炎症模型可靠。
3.3 IFNE基因在bMECs炎症中的功能
IFNE基因是哺乳动物免疫反应中的重要调节因子[33]。迄今为止,有关于IFNE调控乳房炎的分子机制研究在奶牛和小鼠中还未报道。JAK-STAT信号通路内,IFNB、IFNK和IFNE同属于干扰素家族基因[34],在人体免疫中,IFNB和IFNK在抗病毒与抗炎症免疫和系统性自身免疫性疾病反应中发挥着重要作用[35-36]。IL-10RB基因是一种多效性因子,在早发性肠炎[37]和系统性红斑狼疮(SLE)[38]的发病机制中起重要作用。本研究发现,奶牛IFNE基因所编码的蛋白质与山羊的同源性较高,推测奶牛IFNE蛋白在炎症反应中也起着相似的功能。此外,本研究在炎性bMECs中进行IFNE基因干扰,可使IFNB、IFNK和IL-10RB基因表达量显著下调,表明干扰奶牛IFNE基因可缓解bMECs的炎症反应。CASP3与CASP9是细胞凋亡早期和晚期公认的标志基因,与乳腺炎风险有关[39-40];BAD是BCL2家族的促凋亡成员,其蛋白质是细胞凋亡的关键调节因子[41]。本研究发现在炎性bMECs中进行IFNE基因干扰可显著下调CASP3、CASP9和BAD基因的表达量,表明干扰IFNE基因的表达,可抑制炎性bMECs的凋亡。
综上所述,IFNE不仅参与调节bMEC炎症反应,还参与调节bMECs增殖与凋亡,本研究结果可为奶牛乳房炎抗性育种提供基础资料。
4 结 论
本研究成功克隆了奶牛IFNE基因的CDS序列,可编码由193个氨基酸组成的不稳定性亲水性蛋白质。IFNE基因在LPS诱导的bMECs炎症反应中表达量显著上调;干扰IFNE可下调炎症因子IL-6、IL-1β和IL-8基因以及IFNB、IFNK、CASP3和CASP9基因的表达水平,进而缓解bMECs的炎症反应。该结果可为奶牛乳房炎的分子调控机制研究提供参考。
