克氏原螯虾虾青蛋白A2 基因克隆、组织分布及原核表达
2024-03-15吉宏武刘书成宋文奎郝记明
陈 浩, 吉宏武*, 张 迪, 刘书成,宋文奎, 郝记明
(1. 广东海洋大学食品科技学院, 广东 湛江 524008;2. 广东省水产品加工与安全重点实验室, 广东 湛江 524008;3. 广东省海洋生物制品工程实验室,广东 湛江 524008;4. 广东省海洋食品工程技术研究中心,广东 湛江 524008;5. 水产品深加工广东普通高等学校重点实验室,广东 湛江 524008)
虾青蛋白(crustacyanin,CR)是甲壳动物中特有的一种载脂蛋白,属于脂钙蛋白(lipocalin)家族,常与虾青素特异性结合, 从而为甲壳等组织提供颜色。 Wade 等人研究发现,当提高饲料中类胡萝卜素的水平并养殖于黑色底质, 斑节对虾(Penaeus monodon)表皮中的虾青蛋白含量显著增加,体色随之变得更蓝更黑[1]。 而Budd 等人采用RNA 干扰技术沉默虾青蛋白基因,斑节对虾体色由蓝色转变为红色[2];显微结果显示,实验虾表皮上只有红色的虾青素颗粒,却未见有蓝色的蛋白质颗粒。Gao 等人采用类似方法干扰脊尾白虾虾青蛋白的表达,其体色也发生了较大的改变[3]。 由此可见,虾青蛋白在提供甲壳水产品体表颜色以及参与色泽调控等方面发挥着重要作用。
虾青蛋白存在多种类型的亚基,依据相对分子质量、氨基酸组成和序列等性质的不同,主要分为CR-A 亚型(包含A2 和A3) 和CR-C 亚型(包含A1、C1 和C2)。目前,从不同甲壳动物体表结构中纯化的天然虾青蛋白,均是由CR-A 或CR-C 亚型的一种或多种组合而成的多聚体,呈现蓝色、黄色等多种颜色,如来自欧洲龙虾(Homarus gammarus)甲壳的β-虾青蛋白呈现紫色,由A1 和A3 亚基组成[4]。近年来,天然虾青蛋白多聚体的呈色机制越来越受到学者的关注,研究者运用晶体学、量子力学等方法对天然虾青蛋白多聚体结构进行了剖析,陆续提出了极化作用、共平面效应、激子耦合、烯醇化等理论[5-8]。 但是,虾青素与虾青蛋白亚基之间相互作用引起复合物光谱红移的机制仍无定论。 为此,Chayen 等将虾青蛋白多聚体脱去虾青素,纯化后得到单一的亚基并研究该亚基与虾青素的相互作用,取得了较好的效果[9]。 但是该方法纯化难度较大,耗时费力。 因此,快速简便制备虾青蛋白亚基能够为虾青蛋白呈色机制研究奠定良好基础。 作为一种成熟的表达系统,大肠杆菌早已广泛应用于藻蓝蛋白等其他色素结合蛋白质的研究中[10-11]。 大肠杆菌表达系统制备重组蛋白质,具有操作简便、经济快捷、纯度较高等优点。 然而,虾青蛋白亚基异源重组表达的报道很少,关于克氏原螯虾虾青蛋白亚基的表达至今未有人研究。 因此,以大肠杆菌为表达宿主,研究克氏原螯虾虾青蛋白亚基异源重组表达具有重要意义。
克氏原螯虾(Procambarus clarkii),属甲壳纲、十足目、鳌虾科,原产于北美洲,现已广泛养殖于长江中下游水域。 近年来,因其色泽红亮、味道鲜美而广受消费者的追捧。2020 年总产量已达240 万t,约占我国甲壳类水产品总养殖产量的56.2%[12]。 目前,克氏原螯虾相关研究多集中在养殖模式、食品安全监测、病毒与免疫等方面,而色泽与虾青蛋白相关研究较少[13-15]。 作者所在课题组前期从克氏原螯虾甲壳中纯化得到一个蓝色的虾青素蛋白多聚体,亚基相对分子质量为21000(命名为PcCRA2),经质谱鉴定以及氨基酸比对分析推测该亚基属于虾青蛋白家族。 迄今,关于PcCRA2 编码基因、组织分布以及异源表达还未有研究报道。 作者通过基因克隆获得PcCRA2 基因(命名为cra2)的DNA 和cDNA序列,采用实时荧光定量PCR(RT-qPCR)分析cra2基因在不同组织的表达特征,构建原核表达载体并在大肠杆菌中进行重组表达,为进一步研究虾青蛋白呈色机制及虾类色泽调控奠定基础。
1 材料与方法
1.1 供试样品
克氏原螯虾: 购自潜江市楚淼水产品有限公司,平均体长(11±1) cm,平均体质量(32±4) g,实验前暂养1 d。 随机挑取6 只健康的克氏原螯虾进行解剖,分别取表皮、肌肉、心脏、血淋巴、胃、肝胰腺、肠道、神经、眼柄等9 种组织用液氮速冻后,置于-80 ℃保存待用。
1.2 菌种和质粒
大肠杆菌表达菌株BL21(DE3)、大肠杆菌感受态Top10、质粒pET28a(+)、pMD19-T 载体:安徽通用生物系统有限公司。
1.3 主要试剂
Ezup 柱式DNA 抽提试剂盒、 柱式Trizol 总RNA 抽提试剂盒、柱式DNA 胶回收试剂盒、T-载体PCR 产物克隆试剂盒: 上海生工生物工程有限公司;HiScript 1st Strand cDNA Synthesis Kit、AceQ qPCR SYBR Green Master Mix: 南京诺唯赞生物科技股份有限公司;LB 肉汤、LB 琼脂: 北京陆桥技术股份有限公司;镍-次氮基三乙酸(Ni-NTA)琼脂糖凝胶:德国Qiagen 公司;限制性内切酶Xho I 和Nco I、T4 DNA 连接酶:宝日医生物技术(北京)有限公司;SDS-PAGE 凝胶快速配制试剂盒、 蛋白质相对分子质量标准: 上海碧云天生物技术有限公司;虾青素(ATX):上海阿拉丁生化科技股份有限公司;卡那霉素 (Kan)、 异丙基-β-D-硫代吡喃半乳糖苷(IPTG)、曲拉通(Triton X-100)、尿素(Urea)、咪唑:北京索莱宝科技有限公司。
1.4 主要仪器
GeneAmp 9700 基因扩增仪: 美国Applied Biosystems 公司;CFX96 实时荧光定量PCR 仪、GelDoc XR+凝胶成像系统: 美国Bio-Rad 公司;Cary 60 紫外可见分光光度计: 美国Agilent 公司;JY92-2D 超声波细胞粉碎机:宁波新芝生物科技股份有限公司;SW-CJ-2FD 超净工作台:苏州净化设备有限公司;HZQ-X100 全温振荡培养箱:苏州市培英实验设备有限公司。
1.5 克氏原螯虾cra2 基因克隆
使用Ezup 柱式DNA 抽提试剂盒提取克氏原螯虾肌肉的基因组总DNA。根据美国国家生物信息中心数据库 (NCBI) 公布的红螯螯虾虾青蛋白A(GenBank:ALC79588.1)和凡纳滨对虾虾青蛋白A2(GenBank:XP_027238673.1) 高度保守氨基酸序列MFTT(L/V)(V/I)AA 和TAECVYRA,设计一对简并引物A2-Fw 和A2-Rev 用于PCR 扩增,见表1。 以克氏原螯虾基因组总DNA 为模板进行PCR 扩增,然后将PCR 扩增产物纯化回收后与克隆载体pMD19-T 进行连接反应,得到的反应产物转化至大肠杆菌Top10。 通过菌液PCR 验证获得阳性转化子, 提取阳性转化子质粒进行酶切和测序验证,最终获得cra2 基因的DNA 序列。

表1 实验中所用引物序列Table 1 Primers used in experiments
根据已获得的cra2 基因DNA 序列设计一对引物CA2-Fw 和CA2-Rev 用于cra2 基因cDNA 克隆。使用柱式Trizol 总RNA 抽提试剂盒提取克氏原螯虾肌肉总RNA, 通过HiScript 1st Strand cDNA Synthesis Kit 试剂盒进行反转录, 以反转录的产物为模板, 以引物CA2-Fw 和CA2-Rev 进行PCR 扩增。 扩增产物的回收、连接以及筛选验证均和cra2基因DNA 序列实验过程一致, 最终获得cra2 基因cDNA 序列。
1.6 基因序列分析
用软件DNAMAN 6.0 查找开放阅读框(ORF),并推导出氨基酸序列。 通过NCBI BLASTp 进行同源比对, 并下载其他虾种虾青蛋白的氨基酸序列,用DNAMAN 6.0 进行氨基酸序列多重对比, 并用MEGA 6.0 构建系统进化树。 用ProtParam(http://web.expasy.org/protparam/)预测蛋白质理化性质;用SignalP 4.0 Server (http://www.cbs.dtu.dk/services/SignalP-4.0/)分析信号肽;用Tmpred program(https://www.expasy.org/resources/tmpred) 分析跨膜区;用ScanProsite(http://prosite.expasy.org/)搜索蛋白质功能结构域;用CDD Search(https://www.ncbi.nlm.nih.gov/cdd/)进行保守结构域预测。
1.7 克氏原螯虾不同组织cra2 基因表达分析
按照柱式Trizol 总RNA 抽提试剂盒分别提取克氏原螯虾9 种组织的总RNA,采用1.0 g/dL 琼脂糖凝胶电泳检测总RNA 质量和完整性。 分别以9种组织总RNA 为模板, 使用HiScript 1st Strand cDNA Synthesis Kit 试剂盒反转录合成cDNA 第一链,-20 ℃保存。根据已获得的cra2 基因cDNA 序列设计引物CRA-F 和CRA-R,同时以克氏原螯虾βactin 基因序列(GenBank:D14612.1)为内参设计引物ACT-F 和ACT-R。 以获得的cDNA 为模板,通过RT-qPCR 测定cra2 基因在9 种组织中的表达情况。 RT-qPCR 反应体系10 μL:cDNA 2 μL,上、下游引物(10 μmol/L)各0.5 μL,2×AceQ qPCR SYBR Green Mix 5 μL,ddH2O 2 μL。 扩增程序:95 ℃5 min;95 ℃10 s,60 ℃35 s, 进行40 个循环;95 ℃15 s,60 ℃60 s,95 ℃30 s,95 ℃15 s。 每个样品重复3 次。 根据荧光定量数据,采用2-ΔΔCt法计算各组织中cra2 基因的相对表达量, 结果表示为平均值±标准差。 采用SPSS 20.0 进行单因素方差分析和Duncan 多重比较分析,P<0.05 表示差异显著。
1.8 PcCRA2 原核重组表达
1.8.1 表达载体的构建与转化 用在线软件JAVA Condon Adaption Tool(https://www.jcat.de/)将克氏原螯虾cra2 基因cDNA 序列按照大肠杆菌密码子偏好性进行优化。 优化后的基因序列由安徽通用生物系统有限公司合成,合成过程中额外添加了限制性内切酶Xho I 和Nco I 的酶切位点和6×His 标签。合成好的基因和pET28a 载体分别用Xho I 和Nco I进行双酶切,并将酶切产物分别纯化回收后进行连接反应。将连接产物转化至大肠杆菌Top10,经菌液PCR 鉴定后,选取阳性克隆单菌落质粒进行双酶切和测序验证。 最后将验证正确的质粒转化至表达菌株E. coli BL21(DE3)中。
1.8.2 重组PcCRA2 诱导表达条件优化 将构建成功的重组菌E. coli BL21 (DE3) 划线于含Kan(100 mg/L)的LB 固体平板上,挑取单菌落,接种于4 mL 含Kan(100 mg/L)的LB 液体培养基中,37 ℃、200 r/min 振荡培养12 h(OD600约为1.0),然后按体积比1∶100 接种于4 mL 含Kan(100 mg/L)的LB 液体培养基中。
1)最佳IPTG 诱导终浓度的确定 接种后培养3 h, 加入IPTG 并使其终浓度分别为0.1、0.5、1.0、1.5、2.0 mmol/L,37 ℃、200 r/min 振荡诱导4 h。
2)最佳诱导起始点的确定 分别向接种后培养2、3、4、5、6 h 的菌液中加入终浓度为0.5 mmol/L的IPTG,37 ℃、200 r/min 振荡诱导4 h。
3)最佳诱导温度的确定。 向接种后培养4 h 的菌液加入终浓度为0.5 mmol/L 的IPTG,分别在16、23、30、37、44 ℃的条件下200 r/min 振荡诱导4 h。
4)最佳诱导时长的确定 向接种后培养4 h 的菌液加入终浓度为0.5 mmol/L 的IPTG,30 ℃、200 r/min 下分别诱导2、4、6、8、10 h。 分别取出1 mL菌液,经4 ℃、5000 g 离心3 min,收集菌体,超声(300 W 超声10 s,间隔15 s,共30 次)处理后,进行SDS-PAGE 电泳分析,并使用设备自带软件分析电泳条带灰度来确定目标蛋白质表达量。
1.8.3 重组蛋白的分离与纯化 按最优条件扩大至1 L 培养。 所得菌体按1 g∶50 mL 用包涵体洗涤液(2 mol/L Urea,体积分数1%的Triton X-100,50 mmol/L 的pH 7.4 PBS)洗涤2 次,4 ℃、5000 g 离心3 min。 所得沉淀按1 g∶10 mL 加入变性液(8 mol/L Urea,50 mmol/L 的pH 7.4 的PBS) 于4 ℃搅拌溶解。按说明书使用Ni-NTA 柱进行亲和层析纯化,8 mol/L 尿素—不同浓度咪唑(10、20、50、100、200 mmol/L)溶液由低至高进行梯度洗脱,分别收集洗脱液,进行SDS-PAGE 电泳检测。 收集符合要求的洗脱液,使用尿素梯度(6、4、2、0 mol/L)透析复性,超滤浓缩,得到重组PcCRA2。
1.8.4 重组PcCRA2 与虾青素特异性结合 参照PAN 等[16]的方法,稍作调整。 将40 mmol/L Tris-HCl(pH 7.0)、0.2 mol/L 硫酸铵溶液和12 mmol/L 虾青素丙酮溶液等体积混合,加入重组PcCRA2 并使其终浓度为5 mmol/L,混合均匀,装入透析袋置于20 mmol/L PBS 溶液中,4 ℃过夜。 超滤除去游离虾青素后,测定其在220~800 nm 的紫外可见光谱。
2 结果与分析
2.1 cra2 基因的克隆及序列分析
DNA 和cDNA 扩增结果见图1。 电泳检测分别在750~1000 bp 和500~750 bp 处有单一条带。 通过测序分别获得cra2 基因DNA 序列和cDNA 序列。 将cra2 基因DNA 序列和cDNA 序列分别提交至NCBI 数据库, 序列登录号分别为MW727506 和MW727507。

图1 克氏原螯虾cra2 基因的扩增图谱Fig. 1 Amplification of the cra2 gene from P. clarkii
cra2 基因cDNA 序列全长为573 bp, 共编码190 个氨基酸,见图2。 由ProtParam 预测分析可知,PcCRA2 蛋白分子式为C960H1433N237O286S9, 相对分子质量为21158.9,理论等电点为5.59,属酸性蛋白质。不稳定系数39.29,推测该蛋白质为稳定蛋白质。 脂肪系数为66.74,平均亲水性系数-0.162,说明该蛋白质为亲水蛋白质。 SignalP4.0 和SMART 分析显示,PcCRA2 蛋白为分泌蛋白, 信号肽序列为1~16 氨基酸,ARM 保守结构域 (lipocalin) 为43~184。 通过Tmpred蛋白质跨膜区分析发现,PcCRA2 蛋白存在2个由内向外的跨膜区(第1~19、102~122 氨基酸位)和1 个由外向内的跨膜区 (第1~19 氨基酸位)。ScanProsite 分析结果显示,PcCRA2 蛋白存在1 个肌钙蛋白标记(lipocalinsignature),位于36~49 氨基酸处。
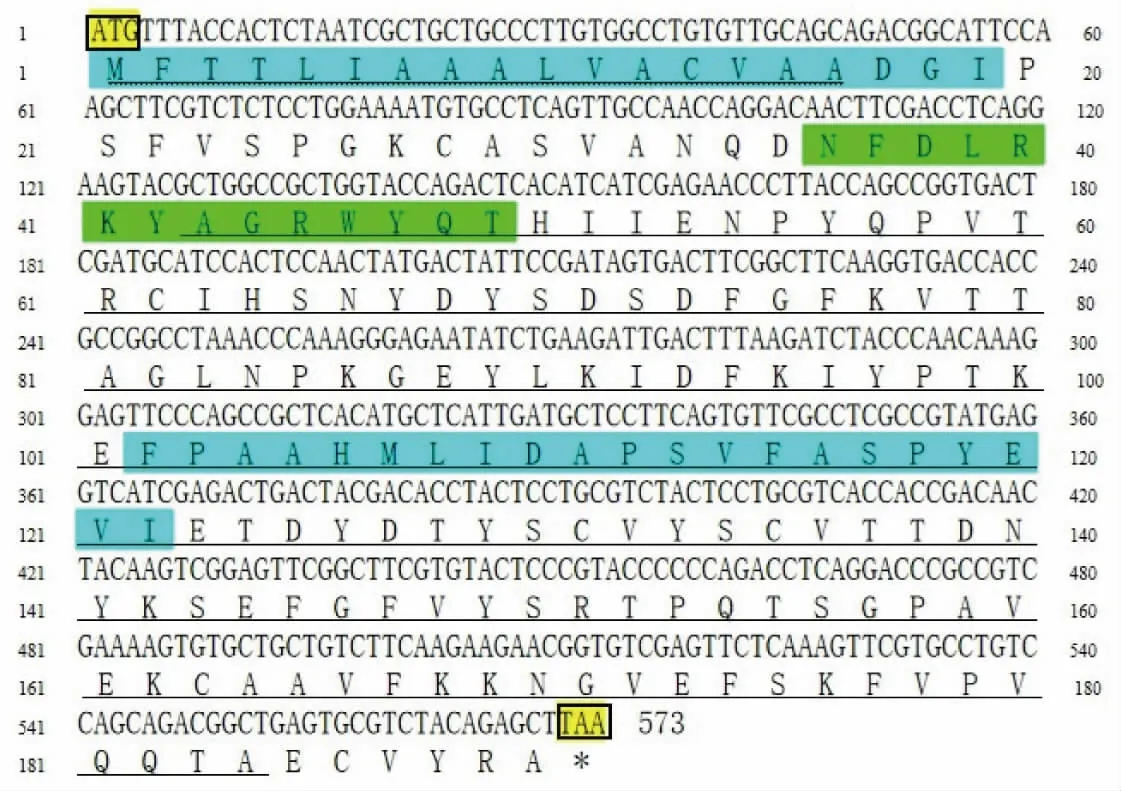
图2 克氏原螯虾cra2 基因cDNA 序列及推导的氨基酸序列Fig. 2 Nucleotide and deduced amino acids sequences of cra2 gene from P. clarkii
2.2 氨基酸同源性比较及系统发育分析
BLAST 比对结果表明,克氏原螯虾PcCRA2 与NCBI 公布的其他100 种虾类虾青蛋白氨基酸序列的相似性在57.89%~91.58%。 其中,与红螯螯虾虾青蛋白A(Cherax quadricarinatus,ALC79588.1)的相似度最高,达91.58%;与欧洲龙虾虾青蛋白A2(H.gammarus,P80007.1)、 美洲龙虾虾青蛋白A2(Homarus americanus,XP_042227234.1)和天鹅龙虾(Panulirus cygnus,ACL37112.1) 相似度分别达到87.93%、87.37%和73.68%。 多重比较氨基酸序列发现,克氏原螯虾与其他9 种虾的虾青蛋白均具有脂钙蛋白(lipocalin)家族典型区域SCR-1(G-X-W,X代表不同的氨基酸)、SCR-2 (T-D-Y) 和SCR-3(R), 而且这些区域在不同物种间具有高度保守性,见图3。
利用MEGA5.0 软件进行系统进化分析, 结果显示,克氏原螯虾与红螯螯虾先聚在一起,再与欧洲龙虾、美洲龙虾、天鹅龙虾聚为一大支,而同属对虾科的日本对虾、凡纳滨对虾、宽沟对虾、刀额新对虾、斑节对虾聚为另一大支,见图4。此外,遗传距离分析结果表明,克氏原螯虾与红螯螯虾具有最近的亲缘关系。

图4 虾青蛋白系统进化树Fig. 4 Phylogenetic tree of crustacyanin
2.3 cra2 基因组织表达分布
运用RT-qPCR 检测cra2 在克氏原螯虾9 种组织中的表达水平见图5。 cra2 mRNA 在各组织中均有表达,其中在表皮中的相对表达量显著高于其他组织(P<0.05),其次是肠道、肝胰腺,而肌肉中的表达量最低。
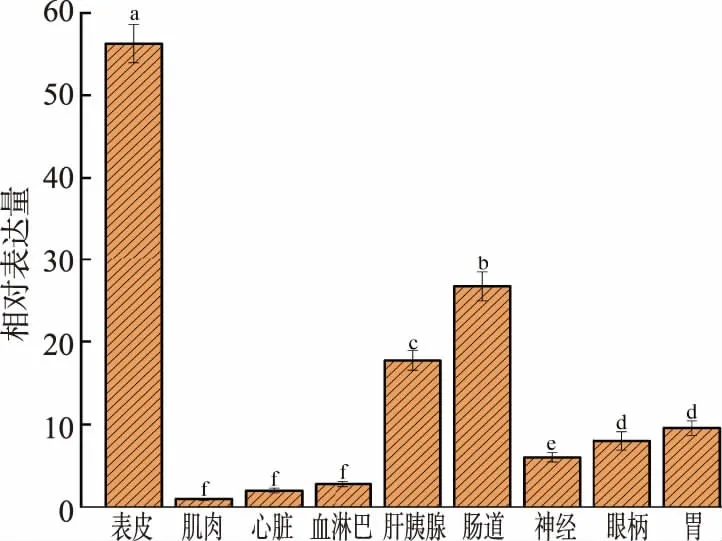
图5 克氏原螯虾cra2 基因mRNA 在不同组织中表达水平Fig. 5 Expression level of cra2mRNA in different tissues of P. clarkii
2.4 PcCRA2 原核重组表达
2.4.1 原核表达载体的构建与转化 根据大肠杆菌密码子偏好性, 优化了克氏原螯虾cra2 基因cDNA 序列。 GC 比例由优化前的54%减少至46%,密码子适应指数(codon adaptation index)由0.69 提高至0.94,与理想值1.0 更为接近,有利于目标基因高效表达[17]。 经双酶切和测序验证,表达载体pET28a-cra2 未发现移码、碱基突变等情况,与预期目标基因一致,表明cra2 基因cDNA 序列成功插入表达载体pET28a 中。
2.4.2 重组蛋白诱导表达条件的优化 如图6(a)所示,除了对照组(未诱导),不同终浓度的IPTG 诱导下目标蛋白质均有表达(22000),但表达量不同。当IPTG 终浓度为0.1 mmol/L 时, 目标蛋白质表达量为18.9%;IPTG 继续增加浓度至0.5、1.0 mmol/L,目标表达量均明显增加,但是相差不大;当IPTG 浓度增至1.5、2.0 mmol/L 时, 目标蛋白质表达反而减少。 综合考虑目标蛋白质表达量和成本, 选择0.5 mmol/L 为最佳IPTG 诱导终浓度。
如图6(b)所示,接种后4 h 时开始诱导,目标蛋白质表达量最高,约占菌体总蛋白质的31.3%。接种后生长2 h 和6 h,目标蛋白质占菌体总蛋白质比例均不高。 因此,选择接种生长4 h 为最佳诱导起始点。
如图6(c)所示,随着温度的升高,目标蛋白质表达量先升高后降低,30 ℃时达到最大。 当16 ℃和23 ℃诱导时,目标蛋白质电泳条带在22000 处,颜色较浅,但在菌体总蛋白质中的比例分别为23.5%,25.6%。 当诱导温度升高至44 ℃,目标蛋白质表达量降低。 因此,选择30 ℃为最佳诱导温度。
如图6(d)所示,诱导时长在2~6 h 时,目的蛋白质表达量随诱导时间而逐渐增加;超过6 h 时,目标蛋白质表达逐渐降低。 因此,选择6 h 为最佳诱导时长。
诱导最佳条件为: 接种4 h 后加入0.5 mmol/L的IPTG,于30 ℃诱导6 h。
2.4.3 重组PcCRA2 与虾青素特异性结合 重组PcCRA2 的Tris-HCl 溶液在270 nm 处出现单峰,见图7。 虾青素的丙酮溶液在474 nm 处有最大吸收峰。 超滤除去游离虾青素后,重组PcCRA2 与虾青素反应溶液在265 nm 和505 nm 处均出现最大吸收峰, 说明重组PcCRA2 与虾青素发生特异性结合。 相比游离虾青素,可见光区最大吸收峰发生了红移,从474 nm 移至520 nm 处。

图7 重组PcCRA2 与虾青素特异性结合的UV-vis 图Fig. 7 UV-vis spectra of the specific binding of recombinant PcCRA2 to astaxanthin
2.5 讨论
通过同源克隆获得了克氏原螯虾cra2 基因的DNA 和cDNA 序列, 并通过cDNA 序列获得PcCRA2 氨基酸序列。 PcCRA2 氨基酸序列中含16个氨基酸(N 端) 的信号肽和174 个氨基酸的成熟肽,其中43~184 氨基酸位为lipocalin 保守结构域,而且具有脂钙蛋白家族3 个典型区域SCR-1 (GX-W)、SCR-2(T-D-Y)和SCR-3(R),表明PcCRA2属于脂钙蛋白家族。 这些典型区域在不同物种虾青蛋白中高度保守,说明虾青蛋白该区域在进化上相对保守,这可能与虾青蛋白均具有β-桶形空间结构有关[2]。 另外,通过基因和氨基酸序列比对,可以确定PcCRA2 属于虾青蛋白A2 亚基。 迄今为止,在甲壳动物中只发现了2 个编码虾青蛋白的基因:cra和crc 基因, 它们同时或单独存在于同一物种中表达CR-A 亚型或CR-C 亚型虾青蛋白[18]。而且,同一亚型虾青蛋白可能在翻译后发生酰胺化或糖基化等修饰,从而导致它们的结构、性质等存在差异[19]。由此,克氏原螯虾cra2 基因属于cra 基因,但是克氏原螯虾中是否还存在crc 基因有待进一步研究。系统进化树显示,PcCRA2 与红螯螯虾、 欧洲龙虾、美洲龙虾等聚为一支,说明其与鳌虾科虾类有血缘关系,而与对虾科虾类有明显区分。
克氏原螯虾cra2 基因的表达具有组织特异性,在表皮中表达量最高。 Tlysty 等人认为虾青素主要在表皮中与虾青蛋白特异性结合形成结合蛋白质,而后转移至甲壳中继续修饰成不同颜色的多聚体[20]。在罗氏沼虾中,crc 基因在表皮中的表达量高于其他组织,且在蜕壳期间比其他生长阶段表达高[21]。干扰该基因后,其在表皮中的表达量明显减少,此时虾的体色转变成红色。 另外,李清清等人发现cra1基因在中华绒螯蟹表皮中表达相对较高,而crc1 基因和crc2 基因却在肝胰腺和卵巢中表达更高[22]。 他认为前者可能主要参与蟹的色泽调控,后者更多地参与卵巢色泽形成和发育。 由此,克氏原螯虾cra2基因可能主要参与其体色的形成以及色泽的调控,但是具体调控机制有待后续研究。
原核系统具有基因修饰简便、 生产周期较短、成本较低等优点,因而在各种蛋白质表达研究中被广泛使用。为获得大量重组PcCRA2,作者采用大肠杆菌原核表达系统, 并使用目前调控T7 启动子效果最优的乳糖类似物IPTG 作为诱导剂。 考虑到表达元件和外界环境均能影响原核表达效果,对诱导剂终浓度、诱导起始点、诱导温度和诱导时间等条件进行了优化。结果表明,IPTG 终浓度为0.5 mmol/L和1.0 mmol/L 时目标蛋白质表达效果较好。 尼罗罗非鱼Galectin-4 基因的原核表达条件优化结果显示,IPTG 浓度在0.4 mmol/L 表达量最大, 而在0.4~1.0 mmol/L 内没有明显增加, 与本研究结果相似[23]。 当IPTG 浓度超过1.0 mmol/L 时,目标蛋白质的表达量下降,这可能是IPTG 具有潜在毒性,过量会抑制菌体生长和蛋白质表达[24]。以接种后生长4 h作为起始诱导点,重组PcCRA2 表达量最大,此时重组菌处于对数期。 诱导处于生长迟缓期和稳定期的菌体均不利于目标蛋白质的表达和积累[25]。 在30 ℃诱导时,目标蛋白质表达效果最好。温度过低,重组菌生长缓慢,菌体密度不高;温度过高,重组菌加速增殖,不利于蛋白质的表达,而且容易使目标蛋白质形成包涵体,增加了后续纯化的难度[26]。诱导6 h 时,重组PcCRA2 表达量达到最大,这与哈维氏弧菌PspF 基因的异源表达条件优化结果一致[27]。
由于大肠杆菌系统表达的蛋白质主要以包涵体形式存在,容易在生成过程中出现未折叠或错误折叠等情况而使重组蛋白质丧失活性,纯化时需要增加复性操作[28]。 本研究获得的目标蛋白质包涵体占78.2%, 故采用尿素浓度梯度透析法对其进行了复性。 复性后的纯化重组PcCRA2 能与虾青蛋白特异性结合, 在265 nm 和505 nm 处出现最大吸收峰。 而Ferrari 等人制备的重组美洲龙虾虾青蛋白H2 与虾青素结合后,不仅在580 nm 处出现最大吸收峰,还在520 nm 处有肩峰[29]。 此外,重组PcCRA2与虾青素结合率(A505/A265)为0.38,略高于Ferrari 等的实验结果。 虽然结合效率不高,但是为后续研究虾青蛋白亚基与虾青素的相互作用提供可能。
3 结语
采用同源克隆获得了克氏原螯虾cra2 基因的DNA 和cDNA 序列,RT-qPCR 发现该基因在克氏原螯虾9 种组织中均有表达, 其中表皮表达量最高。 构建了表达载体pET28a-cra2, 并获得重组PcCRA2,证实了该蛋白质能与虾青素特异性结合。本研究结果可为获取重组虾青蛋白亚基、探究其生物功能提供参考。
