芋淀粉合成酶基因家族的鉴定、生物信息学及表达分析
2024-03-13何芳练刘莉莉邱祖杨董伟清
何芳练,刘莉莉,邱祖杨,董伟清
(1.广西壮族自治区农业科学院蔬菜研究所,南宁 530007;2.荔浦市农业农村局,广西 荔浦 546600)
【研究意义】淀粉是大多数植物的主要储存碳水化合物,是人类饮食中热量的重要来源,也是一种廉价、可再生的原材料,可用于许多非食品用途,包括纸张、纺织品、药品、醇基燃料、染料、黏合剂和建筑材料制造等[1-2]。淀粉由直链淀粉和支链淀粉组成,其中直链淀粉对淀粉的特性有重要影响,由α-1,4糖苷键连接形成的葡萄糖链占天然淀粉的5%~35%[2-3]。芋[Colocasiaesculenta(L). Schott ]是一种富含淀粉的块茎类作物,在热带和亚热带地区广泛栽培。我国有2000多年的芋栽培历史,是世界第二大芋生产国和最大出口国[4-5],但我国芋主产区均存在品种退化,产量、品质下降等问题[6]。球茎是芋的主要食用器官,其淀粉占球茎干物质含量的70%~80%[5, 7]。目前,芋淀粉生物合成已有相关报道,一些参与淀粉生物合成相关的酶基因已被鉴定[8-9],包括ADP-葡萄糖焦磷酸化酶(ADP-glucose pyrophosphorylase,AGPase)[10-12]、淀粉合成酶(Starch synthase,SS)[13-14]、淀粉分支酶(Starch branching enzyme,SBE)[15-16]等,其中对AGPase和SBE基因家族的结构特征和表达模式已经进行了较深入的研究,但针对芋SS(CeSS)基因家族信息的了解非常有限,因此,有必要在全基因组范围内鉴定CeSS基因家族的候选基因并明确其表达模式,以促进芋的产量提高和品质改良。【前人研究进展】淀粉在植物中的生物合成已有许多相关报道,淀粉由不同系列的酶协同作用合成,包括AGPase、SS、SBE、淀粉脱分支酶(Starch debranching enzyme,DBE)和淀粉磷酸化酶(Starch phosphorylase,Pho)等[17-21],其中,SS在直链淀粉和支链淀粉的生物合成过程中发挥重要作用[21-22]。SS家族属于碳水化合物活性酶第5家族的旁系家族,由至少6个保守糖基转移酶(Glycosyl transferase,GT)组成,C-末端具有GT5和GT1组成的保守GT催化结构域,N-末端具有可变结构域,包括盘绕结构域(Coiled-coil domain)和碳水化合物结合模块(Carbohydrate-binding module,CBM)等[23]。SS家族分为5种类型,分别为SSI、SSII、SSIII、SSIV、GBSS[24]。SSI在支链淀粉生物合成中将聚合度((Degree of polymerization,DP)为6~7的短葡聚糖链延长成DP 为8~12的葡聚糖链[25-28];SSII进一步延长支链淀粉中DP小于10的短葡聚糖链[19,29-30];SSIII产生长葡聚糖链并连接多个支链淀粉簇[31-33];SSIV影响淀粉颗粒的大小和形状,也具有延长葡聚糖链的功能[34-35];GBSS主要参与直链淀粉生物合成,具有2个成员(GBSSI和GBSSII),GBSSI负责胚乳的直链淀粉生物合成,而GBSSII负责叶片的直链淀粉生物合成[3,22,36]。SS在植物淀粉生物合成和品质改良方面发挥着重要作用,如抑制小麦SSI或SSIIIa基因的表达会导致淀粉颗粒形态、大小和精细结构发生显著变化[28,33];SSII基因缺失会使淀粉理化性质发生改变,增加直链淀粉的含量[30,37-39];GBSSI基因缺失不会显著改变淀粉含量,但会使支链淀粉含量减少[24];通过CRISPR/Cas9技术对水稻、木薯和马铃薯的GBSSI基因进行编辑,成功创制低直链淀粉或无直链淀粉的新种质[40-42];通过对大麦和小麦的SSIIa进行基因编辑,成功创制了高直链淀粉新种质[43-44]。【本研究切入点】虽然芋SSI和SSII基因的cDNA序列已被测定和分析,但CeSS基因家族结构特征和表达模式仍不清楚,亟待研究。【拟解决的关键问题】使用HMMER 3.12b软件鉴定芋基因组中CeSS基因家族信息,并对其分子结构特征、系统发育关系、顺式作用元件及表达模式进行分析,以加强对CeSS基因家族成员的了解,并为芋产量、品质和营养性状的遗传改良提供基础。
1 材料与方法
1.1 试验材料
供试芋材料为芋新品种荔浦芋1号,该品种为魁芋类型,具有产量高、品质优和耐储藏等特点。芋在播种90 d(3月龄)后,球茎淀粉含量迅速上升,为整个生育期淀粉含量上升最快的阶段,此时球茎也进入快速膨大发育期[8,15]。因此,选择播种90 d的球茎、叶片、叶柄和根用于CeSS基因的空间表达分析;选择播种30、60、90 d的球茎用于CeSS基因的时序性表达分析。选择播种90 d的芋植株进行干旱处理,对照组保持每天浇水,处理组自然干旱7 d(2张新叶保持正常发育,其余叶片从边缘开始干枯),采集对照组和处理组根、叶片和球茎用于CeSS基因在干旱胁迫下的表达分析。所有样品均设置3次生物学重复,液氮速冻后置于-80 ℃冰箱保存备用。
1.2 试验方法
1.2.1 CeSS蛋白鉴定和系统发育分析 从国家基因库生命大数据平台(https://db.cngb.org/)下载芋参考基因组数据,使用HMMER 3.12b以SS基因家族典型保守结构域Glyco_transf_5(Pfam登录号:PF08323)为模型预测CeSS蛋白,E值阈值设为小于2.7e-15。使用NCBI保守结构域数据库(CDD v3.20,E值设置为0.01)(https://www.ncbi.nlm.nih.gov/cdd)和SMART数据库(http://smart.embl.de)进一步验证候选CeSS蛋白序列的保守结构域。分别从TAIR数据库(http://www.arabidopsis.org)和RGAP数据库(http://rice.plantbiology.msu.edu)下载拟南芥和水稻的SS家族蛋白序列,使用MEGA 7.0对芋、水稻和拟南芥的SS蛋白序列进行序列比对,使用邻接法构建系统发育树(Bootstrap值为1000次)。
1.2.2 CeSS蛋白特性和基因结构分析 ExPASy软件(http://web.expasy.org/protparam)可预测蛋白序列的各种物理和化学参数,包括分子量、理论等电点、氨基酸组成、原子组成、消光系数、估计半衰期、不稳定指数、脂肪族指数和平均亲水性(GRAVY)等 ,使用该软件预测CeSS蛋白的分子量和理论等电点;MEME 5.5.1(https://meme-suite.org/meme/tools/meme)是可预测蛋白质motif的在线工具,使用该软件预测CeSS蛋白的保守motif(E阈值设置为小于2.60e-04);InterPro数据库(http://www.ebi.ac.uk/interpro)通过将蛋白分类为家族并预测结构域和重要部位来提供蛋白质的功能分析,使用该数据库对预测的motif进行功能注释;使用NCBI数据库(https://www.ncbi.nlm.nih.gov)和InterPro数据库预测CeSS蛋白的保守结构域;GSDS 2.0(http://gsds.gao-lab.org)可用于基因注释特征的可视化,使用该软件预测CeSS基因结构特征;PlantCARE数据库(http://bioinformatics.psb.ugent.be/webtools/plantcare/html)是一个预测植物顺式作用元件的在线工具,使用该软件对CeSS基因上游2 kb序列预测启动子区域的顺式作用元件。
1.2.3CeSS基因转录组分析 对CeSS基因在正常生长条件下芋不同组织、球茎不同发育阶段及干旱胁迫下的转录表达情况进行分析。对播种90 d的球茎、叶片、叶柄和根开展转录组测序,从转录组数据中提取CeSS基因的FPKM(Fragments per kilobase per million mapped reads)值,分析CeSS基因在不同组织的表达差异;对发育30、60、90、120、150 和240 d的球茎组织开展转录组测序,从转录组数据中提取CeSS基因的FPKM值,分析CeSS基因在球茎不同发育阶段的表达差异。对干旱胁迫试验中对照组和处理组的球茎、叶片和根进行转录组测序,从转录组数据中提取CeSS基因的FPKM值,分析CeSS基因在干旱胁迫下的表达差异。利用CeSS基因的FPKM值,在百迈客云平台(https://international.biocloud.net/zh/software/tools/detail/small/305)绘制CeSS基因在正常生长条件下不同组织、球茎不同发育阶段及干旱胁迫下的聚类热图。
1.2.4CeSS基因实时荧光定量PCR分析 使用植物多糖多酚总RNA提取试剂盒[天根生化科技(北京)有限公司,DP441]提取所有样品总RNA,使用TIANScript Ⅱ cDNA 第一链合成试剂盒[天根生化科技(北京)有限公司,KR107]将提取的总RNA 反转录成cDNA 第一链。以正常生长条件下不同组织、球茎不同发育阶段以及干旱胁迫下对照组和处理组的cDNA为模板,使用Analytik Jena qTOWERE2.2 荧光定量PCR仪进行实时荧光定量PCR试验。根据CeSS基因序列信息,利用Primer 5.0设计实时荧光定量PCR引物(表1),以芋Actin基因为内参基因[11]。使用ChamQ Universal SYBR qPCR Master Mix试剂盒(南京诺唯赞生物科技有限公司,Q711-02)进行实时荧光定量PCR检测。反应体系(20.0 μL):2 × ChamQ Universal SYBR qPCR Master Mix 10.0 μL,上下游引物(10 μmol/L)各0.4 μL,模板cDNA 1.0 μL,双蒸水补足20.0 μL。反应程序:95 ℃预变性30 s;95 ℃ 10 s,60 ℃ 20 s,进行40个循环。每个样品3次重复,采用2-ΔΔCT法计算基因的相对表达量[45]。

表1 CeSS基因的实时荧光定量PCR引物序列
2 结果与分析
2.1 CeSS蛋白鉴定及系统发育分析
使用SS基因家族典型保守结构域Glyco_transf_5(Pfam登录号PF08323)为模型鉴定芋SS基因家族。通过HMMER 3.12b软件在芋基因组中共鉴定出6个CeSS基因,并根据拟南芥中的同源基因分别命名为CeSS1、CeSS2、CeSS3-1、CeSS3-2、CeSS4和CeGBSS1。6个预测的CeSS基因长度为4980~61 347 bp,对应的CDS长度为1275~2895 bp,编码蛋白的氨基酸残基数量为424~964个,分子量为48 067.43~109 391.75 Da,理论等电点为5.18~6.11(表2)。CeSS基因在基因长度、编码蛋白大小、分子质量和理论等电点的差异可能反映其在各种生物过程中的功能差异。
系统发育关系对于了解基因家族的结构及多个植物物种的进化历史至关重要。为了揭示芋SS蛋白的进化关系,本研究构建了芋、水稻和拟南芥SS蛋白的系统发育进化树。23个SS蛋白被分成5个亚家族,CeSS蛋白在5个亚家族均有分布,其中CeSS3-1蛋白和CeSS3-2蛋白分布在亚家族3。在水稻和拟南芥中,5个亚家族也均有SS蛋白分布,其中拟南芥在亚家族4包含2个SS蛋白(AtSS4-1和AtSS4-2);而在水稻中,除了亚家族1只包含1个SS蛋白(OsSS1)外,其余4个亚家族包含至少2个SS蛋白(图1)。
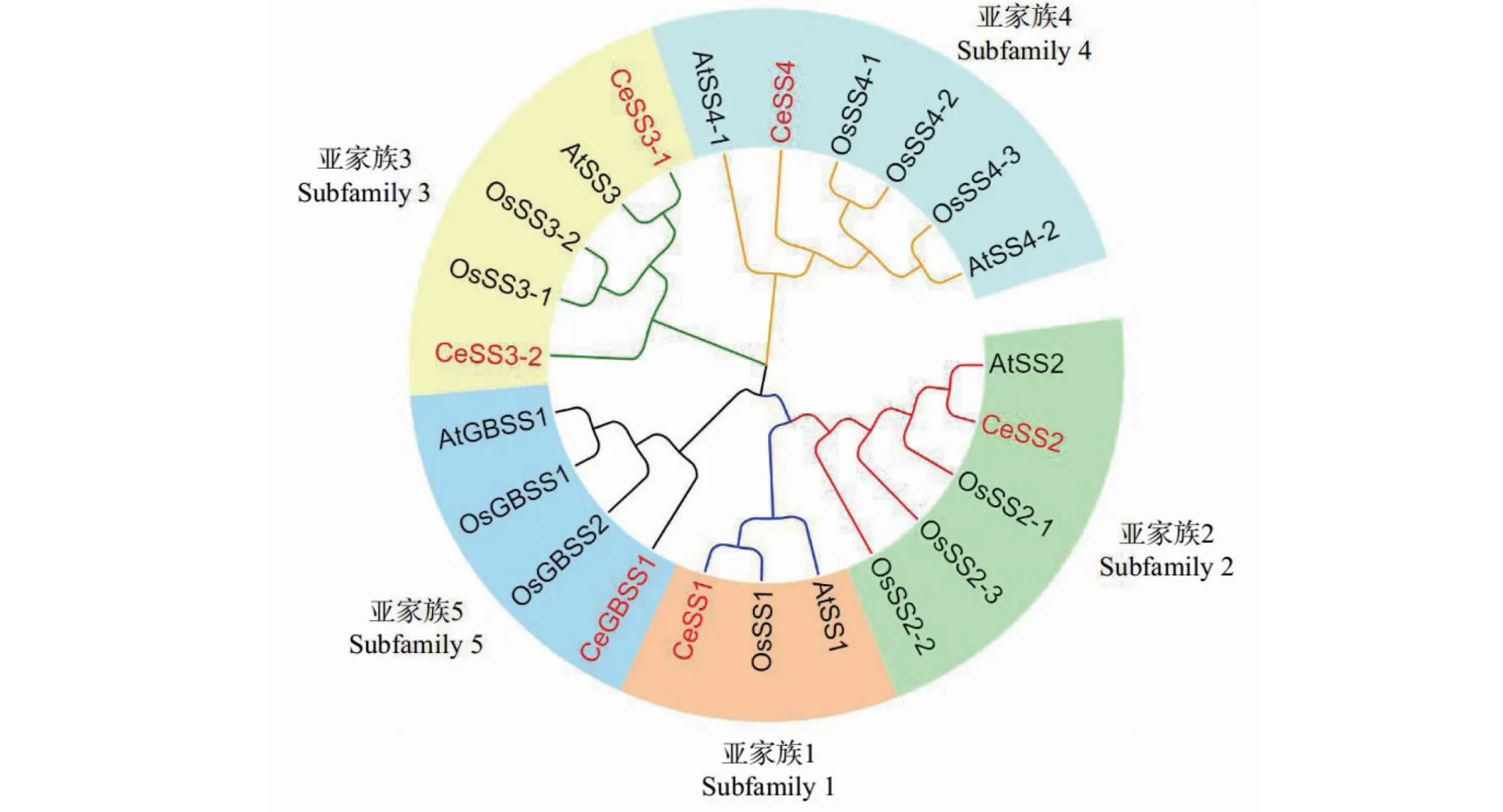
CeSS1、CeSS2、CeSS3-1、CeSS3-2、CeSS4和CeGBSS1为芋SS蛋白,OsSS1、OsSS2-1、OsSS2-3、OsSS3-1、OsSS3-2、OsSS4-1、OsSS4-2、OsSS4-3、OsGBSS1和OsGBSS2为水稻(Oryza sativa)SS蛋白,AtSS1、AtSS2、AtSS3、AtSS4-1、AtSS4-2和AtGBSS1为拟南芥(Arabidopsis thaliana)SS蛋白。CeSS1, CeSS2, CeSS3-1, CeSS3-2, CeSS4 and CeGBSS1 are taro SS proteins, OsSS1, OsSS2-1, OsSS2-3, OsSS3-1, OsSS3-2, OsSS4-1, OsSS4-2, OsSS4-3, OsGBSS1 and OsGBSS2 are rice (Oryza sativa) SS proteins, AtSS1, AtSS2, AtSS3, AtSS4-1, AtSS4-2 and AtGBSS1 are Arabidopsis thaliana SS proteins.
2.2 CeSS基因结构和保守motif分析
使用GSDS 2.0软件对6个CeSS基因结构进行分析,结果如图2-A所示,6个CeSS基因外显子数量为7~15个,说明这些基因在进化和功能上产生了差异,进一步支持了CeSS基因的进化分析结果。为进一步探究CeSS蛋白结构多样性并预测其功能,使用MEME 5.5.1软件在6个CeSS蛋白中鉴定出10个保守motif,并在InterPro数据库中进行注释。结果有7个motif获得了注释,其中motif 2和motif 5注释为淀粉合成酶催化结构域(Starch synthase catalytic domain,PF08323),motif 1和motif 3注释为糖基转移酶群组1(Glycosyl transferases group 1,PF00534/PF13692),mitof 4、motif 8和motif 10注释为糖原磷酸化酶B(Glycogen phosphorylase B,G3DSA:3.40.50.2000);大部分CeSS蛋白同时含有motif 2和motif 5(淀粉合成酶催化结构域),而CeSS3-1蛋白和CeSS3-2蛋白仅包含其中1个;除CeSS3-2蛋白外,其余CeSS蛋白均含有motif 1和motif 3(糖基转移酶群组1)、mitof 4和mitof 8(糖原磷酸化酶B)(图2-B,表3)。使用NCBI数据库和SMART数据库对CeSS蛋白的保守结构域进行预测,结果如表4所示,所有CeSS蛋白均含有淀粉合成酶催化结构域;除CeSS3-2蛋白外,所有CeSS蛋白都含有糖基转移酶群组1。6个CeSS基因在外显子数量、保守motif和保守结构域的差异可能反映了不同CeSS基因在进化历史和功能方面的差异。

A: 基因结构分析;B: 保守motif分析。A: Gene structure analysis; B: Conserved motif analysis.

表3 CeSS蛋白保守motif及功能注释情况

表4 CeSS蛋白的保守结构域分析
2.3 芋CeSS基因启动子区域顺式作用元件分析
为了研究CeSS基因家族的调控作用,提取6个CeSS基因起始密码子上游2 kb的序列,使用PlantCARE数据库预测该区域的顺式作用元件。共预测到73个顺式作用元件,其中36个具有功能注释。6个CeSS基因启动子除了具有核心元件 TATA-box和CAAT-box外,还存在大量与光响应、激素响应、胁迫响应及生长发育等相关元件(表5)。所有CeSS基因启动子均含有5~8个光响应元件,说明这些基因可能受光的调控。

表5 芋CeSS基因启动子区域顺式作用元件分析
6个CeSS基因启动子共有生长素、水杨酸、赤霉素、茉莉酸甲酯和脱落酸5种激素响应元件,每个基因至少含有3个以上激素响应元件。其中只有CeSS3-1基因含有生长素响应元件TGA-element;除CeSS3-2基因外,其余基因启动子均含有水杨酸响应元件TCA-motif;除CeSS4基因外,其余基因启动子均含有茉莉酸甲酯响应元件CGTCA-motif和TGACG-motif;除CeSS3-1基因外,其余基因启动子均含有脱落酸响应元件ABRE;CeGBSS1基因含有赤霉素响应元件GARE-motif和P-box,而CeSS2和CeSS4基因仅含有P-box。CeSS基因家族启动子含有多种激素响应元件,且不同基因在激素响应元件方面存在差异,说明这些基因可能受到多种激素调控,且不同基因可能存在不同调控方式。
在生长发育相关元件中,CeSS1、CeSS2、CeSS3-2和CeSS4基因启动子含有与分生组织表达相关的调节元件CAT-box;CeSS1、CeSS3-2和CeSS4基因启动子含有与蛋白代谢相关的调节元件O2-site;CeSS3-2基因启动子含有种子特异性调控元件RY-element;CeGBSS1基因启动子含有昼夜节律调节元件circadian。CeSS基因家族启动子含有与生长发育相关的元件,说明这些基因可能与芋生长发育密切相关。
在胁迫响应相关元件中,所有CeSS基因启动子至少含有1个厌氧诱导相关调节元件(ARE和GC-motif);CeSS1基因启动子含有干旱诱导相关元件MBS;CeGBSS1基因启动子含有低温响应元件LTR;CeSS1和CeSS3-1基因启动子含有防御和压力响应元件TC-rich repeats。CeSS基因家族启动子含有胁迫响应相关的元件,说明这些基因可能响应厌氧胁迫、干旱胁迫、低温胁迫及防御和压力胁迫等逆境胁迫。
2.4 芋CeSS基因在正常生长发育条件下的转录表达分析
为了研究CeSS基因在芋正常生长发育中的功能,对植株正常发育90 d的不同组织(球茎、叶片、叶柄和根)和球茎不同发育阶段(30、60、90、120、150 和240 d)的样品进行转录组测序,从转录组数据中提取CeSS基因家族的FPKM值,对其在不同组织和球茎不同发育阶段的表达模式进行分析。在不同组织中,CeGBSS1基因在球茎、叶片和叶柄中高表达。其中,以叶片的表达量最高,FPKM值为1036.25;其次为球茎和叶柄,FPKM值分别为529.28和255.90;根的表达量最低,FPKM值为51.06。CeSS2、CeSS3-1和CeSS3-2基因在叶片(FPKM值为64.34~98.84)和球茎(FPKM值为27.30~34.24)的整体表达量高于叶柄(FPKM值为18.12~27.09)和根(FPKM值为10.33~13.02)。CeSS1基因在所有组织中均有表达,但表达量较低(FPKM值为1.02~7.25)(图3)。在芋球茎不同发育阶段中,CeGBSS1基因在所有发育阶段均有较高的表达量(FPKM值为65.81~332.50),表达量呈先升高后降低趋势,其中120 d时表达量最高;与30 d相比,120和150 d的表达量差异达显著水平(P<0.05,下同)。CeSS1、CeSS2、CeSS3-1、CeSS3-2和CeSS4基因在芋球茎所有发育阶段均有表达,但表达量较低,FPKM值低于20.00(图4)。

聚类热图根据CeSS基因的FPKM值绘制,基因表达变化的差异以绿色-红色标度显示,图例数字是对FPKM值进行归一化的数值,下同。The clustering heat map was drawn according to the FPKM value of the CeSS gene. The difference in gene expression changes was shown on a green-red scale. The legend number was the normalised value of the FPKM value. The same as below.
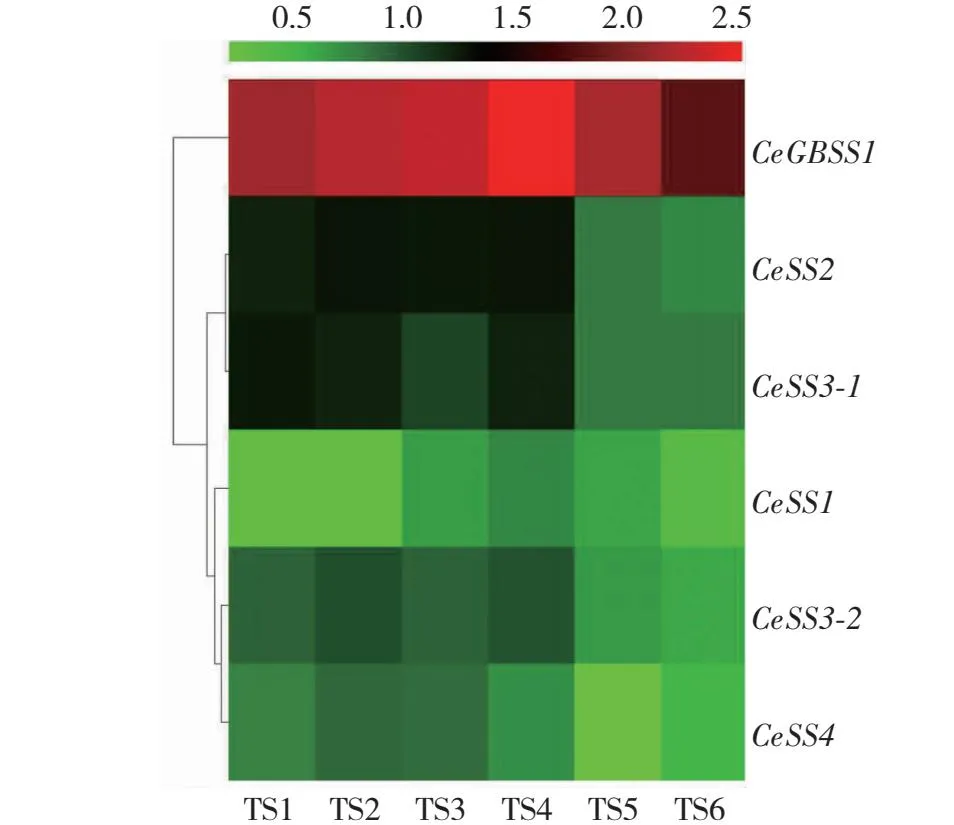
TS1、TS2、TS3、TS4、TS5和TS6分别代表发育30、60、90、120、150和240 d的球茎。TS1, TS2, TS3, TS4, TS5 and TS6 represented taro corms developing at 30, 60, 90, 120, 150 and 240 days, respectively.
2.5 CeSS基因在干旱胁迫下的转录表达分析
为了研究CeSS基因响应干旱胁迫的转录表达,对播种90 d的植株进行干旱胁迫,对照组保持每天浇水,处理组自然干旱7 d,分别对对照组和处理组的球茎、叶片和根进行转录组测序,从转录组数据中提取CeSS基因家族的FPKM值,对其在干旱胁迫下的表达模式进行分析。如图5所示,在球茎中,CeSS基因家族在干旱胁迫下的转录表达均下降,但下降幅度低于叶片,其中CeGBSS1基因的FPKM值从1360.31下降到933.51;CeSS2、CeSS3-1和CeSS3-2基因的FPKM值分别从148.44、65.35和64.51下降到49.66、45.91和47.70。在叶片中,CeSS基因家族在干旱胁迫下的转录表达也均下降,其中CeGBSS1基因的FPKM值从1451.21下降到493.08;CeSS2、CeSS3-1和CeSS3-2基因的FPKM值分别从153.45、99.06和105.03下降到72.80、63.64和59.78。在根中,CeSS基因家族在干旱胁迫下的转录表达略有降低,其中CeGBSS1基因的FPKM值从184.53下降到170.32。

DCC1、DCL1和DCR1分别代表对照组的球茎、叶片和根,DCC2、DCL2和DCR2分别代表干旱胁迫处理组的球茎、叶片和根。DCC1, DCL1 and DCR1 represented corm, leaf and root in controls, respectively, and DCC2, DCL2 and DCR2 represented corm, leaf and root in drought treatments, respectively.
2.6 CeSS基因的实时荧光定量PCR检测
使用实时荧光定量PCR进一步验证转录组数据的准确性。在正常生长发育条件下,CeSS基因在叶片和球茎的整体表达量均高于叶柄和根,其中CeSS2、CeSS3-1、CeSS3-2和CeGBSS1基因在叶片和球茎的相对表达量极显著高于根和叶柄(P<0.01,下同);CeSS4基因在球茎的相对表达量极显著高于其他组织;CeSS1基因在叶片的相对表达量显著高于其他组织(图6)。在球茎不同发育阶段,除了CeSS4基因在球茎发育90 d的表达量略低于60 d外,其余CeSS基因表达量在球茎发育30~90 d的发育过程中均呈上升趋势,其中CeGBSS1基因在球茎发育60和90 d的相对表达量均极显著高于30 d;CeSS2和CeSS3-2基因在球茎发育90 d的相对表达量极显著高于其他发育阶段;CeSS3-1和CeSS4基因分别在球茎发育90和60 d的相对表达量显著高于其他发育阶段(图7)。在干旱胁迫下,所有CeSS基因在叶片的相对表达量较对照组显著或极显著降低;CeSS基因在球茎的表达量有不同的表达模式,CeSS2和CeSS4基因表达量下降,而CeSS1、CeSS3-1、CeSS3-2和CeGBSS1基因表达量略有上升,但均未达显著水平;所有CeSS基因在根的表达量略有降低,其中CeSS4基因达显著水平,其余基因未达显著水平(图8)。综上所述,除了干旱胁迫下CeSS基因在球茎的表达略有差异外,实时荧光定量PCR检测结果与转录组表达分析结果一致,验证了转录组表达分析结果的准确性。

图7 CeSS基因在芋球茎不同发育阶段的相对表达量Fig.7 Relative expression of CeSS genes in taro corms at different developmental stages

图8 CeSS基因在干旱胁迫下的相对表达量Fig.8 Relative expression of CeSS genes in response to drought stress
3 讨 论
虽然芋也是富含淀粉的块茎类作物,但与其他淀粉类块茎作物(如马铃薯、木薯和甘薯等)相比,芋的研究相对较少,研究深度还有差距,尤其在淀粉(直链淀粉和支链淀粉)生物合成关键基因的挖掘与利用及淀粉品质改良方面[41,46-47]。SS参与直链淀粉和支链淀粉的生物合成,影响淀粉的结构和理化性质[3,19,48]。本研究使用HMM模型(PF08323)从芋基因组中鉴定了6个SS基因,与学者在拟南芥的研究结果一致,但也与一些植物的SS基因数量有差异,如水稻、高粱、小麦和大麦的SS基因分别为11、11、9和27个[22,49]。根据系统发育分析,6个芋SS基因分为5个亚群,与前人研究结果一致[24,50-51],其中有2个SS基因分布在亚群3,可能与基因组中片段重复事件有关,在水稻基因组中,SSII、SSIII和SSIV 3个亚家族发生了片段重复事件[22]。系统发育分析得到了基因结构和保守结构域分析的进一步支持。外显子-内含子结构特征分析显示6个SS基因的外显子数量不同,为7~15个,与在水稻[22]和高粱[49]的报道一致。在保守结构域上,CeSS1、CeSS2、CeSS4和CeGBSS1蛋白均具有淀粉合成酶催化结构域(motif 2和motif 5)和糖基转移酶群组1(motif 1和motif 3),而分布在亚家族3上的CeSS3-1基因编码蛋白具有motif 1、motif 3和motif 5,CeSS3-2基因的编码蛋白只含有motif 2,与水稻[22]的研究结果一致。
顺式作用元件参与基因的表达调控。对芋淀粉合成酶基因家族启动子区域顺式作用元件进行分析,发现该区域均存在光响应元件、激素响应元件等,高粱淀粉合成酶基因家族启动子区域也存在光响应元件和激素响应元件[49],说明淀粉合成酶基因可能受到光和激素的调控。同时,在淀粉生物合成途径中,其他淀粉生物合成相关基因如AGPase、SBE等基因家族的顺式作用元件也存在大量光响应元件和激素响应元件[15,52],说明淀粉合成代谢也可能受到光和激素的调控。
基因表达分析能在转录水平了解基因的功能。芋在播种60~120 d为淀粉快速积累期,淀粉含量从4.51 g/100 g增加到21.5 g/100 g,尤其60~90 d是其中的关键阶段,90 d淀粉含量比60 d淀粉含量增加221.5%[8,15];同时,播种90 d后,球茎开始进入快速膨大阶段[7]。因此,播种90 d阶段对芋产量和品质有重要影响,本研究选取该阶段进行不同组织的转录表达分析和干旱胁迫研究是合适的。在本研究中,CeGBSS1基因在叶片、球茎和叶柄等组织及球茎不同发育阶段均具有较高的表达量,说明其可能在芋淀粉生物合成中发挥重要作用,与之前在水稻[22]和高粱[49]等作物的报道一致。CeSS1和CeSS2基因在叶片和球茎具有较高的表达量,与之前在芋的报道一致[13-14]。干旱对淀粉生物合成的影响极大,是非生物胁迫中限制作物生产的主要环境因素[53]。在本研究中,干旱胁迫下所有芋SS基因在叶片和根的表达量均下降,其中在叶片的下降与对照组相比达显著水平,而在球茎中有不同的表达模式,但差异不显著。在对水稻SS基因家族的研究中,不同SS基因在干旱胁迫下表达模式有差异,淀粉合成酶活性及淀粉含量在不同组织中的积累也不同[54]。芋在干旱胁迫下SS基因的表达与淀粉合成酶活性、淀粉含量及与其他淀粉生物合成相关基因的协同关系还有待进一步研究。鉴于SS基因在直链淀粉和支链淀粉生物合成中的作用,已经在许多作物如水稻、木薯、马铃薯、大麦和小麦中对SS基因进行编辑,创制出不同淀粉类型的新种质[40-44],为芋淀粉品质改良和新种质的创制提供了参考和研究思路。
4 结 论
在芋基因组中鉴定出6个CeSS基因,明确其分子结构特征和表达模式。CeGBSS1基因在播种90 d的不同芋组织及球茎不同发育阶段呈现较高表达量,推测是芋淀粉生物合成的关键基因。
