辣椒小G蛋白CaROP的生物信息学分析
2024-02-23马思洁朱天生杨叔青
马思洁,朱天生,2,何 璐,杨叔青,2
(1.塔里木大学农学院,新疆阿拉尔 843300;2. 南疆农业有害生物综合治理兵团重点实验室,新疆阿拉尔 843300)
0 引 言
【研究意义】辣椒(CapsicumannuumL.)为茄科辣椒属植物[1],其果实含有丰富的营养成分[2]。我国辣椒总产量高达6 400×104t[3]。辣椒抗逆品种的选育是保证辣椒产业健康可持续发展经济有效途径,其中挖掘辣椒的抗逆元件是抗逆育种的基础。【前人研究进展】小G蛋白可以参与调控真核生物中细胞信号的转导[4],其中植物含有一类特有的亚家族Rho GTPases ROP(Rho-related GTPases of plants)[5]。ROP蛋白作为信号传导中一类重要的调节因子,它既可以参与植物的生长发育,也可调控植物适应外部环境的刺激[6-7]。ROP蛋白还可参与响应植物抵抗干旱、高盐、低温、病虫等多种逆境胁迫[8],AtROP1负向调控植物防卫反应的建立过程,在马铃薯中超表达失活型的AtROP1蛋白可以抑制马铃薯致病疫霉的生长[9]; AtROP2通过与MAP18互作进而调控植物根毛的伸长[10],AtROP2还可通过抑制下游效应因子RIC1参与调控植物抵抗盐胁迫[11];AtROP11通过抑制植物激素ABA信号的转导进而促进植物抵抗干旱胁迫[12]。ROP蛋白在参与植物生长发育和响应植物抵抗逆境胁迫的过程中具有重要作用。【本研究切入点】前期研究以拟南芥中的11个ROP蛋白的氨基酸序列为模板,对茄科基因组数据库中6种茄科植物的ROP蛋白进行了全基因组筛查等工作,共筛查到66个ROP蛋白,其中有9个ROP蛋白来自辣椒(CaROP)[13],需对辣椒中小G蛋白CaROP进行生物信息学分析。【拟解决的关键问题】研究辣椒内小G蛋白CaROP的生物学功能、蛋白理化性质、蛋白结构及系统发育关系,为分析辣椒小G蛋白CaROP参与调控辣椒生长发育及响应、不同逆境胁迫的功能及分子机理奠定理论基础。
1 材料与方法
1.1 材 料
9个CaROP蛋白的基因代码分别为CA01g27430、CA03g28070、CA02g04310、CA00g84620、CA02g05500、CA02g21300、CA04g05500、CA08g19280和CA00g82910,其氨基酸及核苷酸序列自数据库solgenomics(https://solgenomics.net/)中所获得。
1.2 方 法
1.2.1 CaROP蛋白的Rho功能域、开放阅读框及氨基酸序列相似度预测
运用SMARTS在线软件(http://smart.embl-heidelberg.de/)对9个CaROP蛋白的Rho功能域进行预测分析,其次使用在线软件ORF finder(ORFfinder Home - NCBI(nih.gov))预测9个CaROP核苷酸序列的开放阅读框长度,使用DNAMAN软件对9个CaROP蛋白氨基酸序列进行相似度比较分析[9]。
1.2.2 CaROP蛋白基本理化性质
使用Expasy数据库内ProtParam软件(http://www.exPasy.org/tools/)对9个CaROP蛋白的理化性质进行预测分析,包括氨基酸数量、分子式、分子量、原子总数、带正(负)电荷氨基酸残基及理论等电点等[12]。
1.2.3 CaROP蛋白亲水性、疏水性
使用Expasy数据库内ProtScale软件(https://web.expasy.org/protscale/)对9个CaROP蛋白的亲水性、疏水性进行预测分析[11]。
1.2.4 CaROP蛋白信号肽预测和跨膜结构域
利用SignalP5.0软件(http://www.cbs.dtu.dk/)对9个CaROP蛋白的信号肽进行预测分析;利用TMHMM在线软件(http://www.cbs.dtu.dk/services/TMHMM/)对9个CaROP蛋白的跨膜结构域进行预测分析[13]。
1.2.5 CaROP蛋白磷酸化位点和糖基化位点
利用NetPhos3.1在线软件(http://www.cbs.dtu.dk/services/NetPhos/)对9个CaROP蛋白的磷酸化位点进行预测分析[14];利用NetCGlyc1.0在线软件(http://www.cbs.dtu.dk/services/NetCGlyc/)对9个CaROP蛋白的糖基化位点进行预测分析[15]。
1.2.6 CaROP蛋白二级、三级结构预测
利用SOPMA在线软件(https://www.expasy.ch/)对9个CaROP蛋白的二级结构进行预测分析[16];利用SWISS-MODEL软件(https://swissmodel.expasy.org/)对9个CaROP蛋白的三级结构进行预测分析[17]。
1.2.7 CaROP蛋白系统发育关系
利用MEGA11软件并选用最大似然法(Maximum Likelihood)的计算方法对辣椒9个CaROP和拟南芥11个AtROP的氨基酸序列构建系统进化树,其中重复次数(Bootstrap-Replications)设置为1 000次。图1
图 1 系统进化树参数设置
2 结果与分析
2.1 9个CaROP的Rho功能域、开放阅读框及氨基酸序列相似度预测
研究表明,9个CaROP蛋白均存在1个Rho功能域,均属于Rho家族。CA01g27430开放阅读框长度为633 bp,CA03g28070开放阅读框长度为525 bp,CA02g04310开放阅读框长度为594 bp, CA00g84620开放阅读框长度为594 bp,CA02g05500开放阅读框长度为594 bp,CA02g21300开放阅读框长度为594 bp,CA04g05500开放阅读框长度为633 bp,CA08g19280开放阅读框长度为597 bp,CA00g82910开放阅读框长度为675 bp。9个CaROP蛋白氨基酸序列相似度高达73.02%。图2

图 2 9个CaROP蛋白氨基酸序列 相似度的比较
2.2 9个CaROP蛋白理化性质
研究表明,CA01g27430蛋白由210个氨基酸组成,分子式为C1037H1648N282O309S6,分子量为23 202.56,原子总数为3282,带正电荷的氨基酸残基(Asp + Glu)为19个,带负电荷的氨基酸残基(Arg + Lys)为28个,理论等电点(pI)为9.33;CA03g28070蛋白由174个氨基酸组成,分子式为C861H1333N225O256S6,分子量为19 124.77,原子总数为2 681,带正电荷的氨基酸残基(Asp + Glu)为17个,带负电荷的氨基酸残基(Arg + Lys)为16个,理论等电点(pI)为6.28;CA02g04310蛋白由197个氨基酸组成,分子式为C986H1575N263O286S7,分子量为21 914.36,原子总数为3 117,带正电荷的氨基酸残基(Asp + Glu)为18个,带负电荷的氨基酸残基(Arg + Lys)为26个,理论等电点(pI)为9.32;CA00g84620蛋白由197个氨基酸组成,分子式为C971H1543N255O283S5,分子量为21 477.77,原子总数为3 057,带正电荷的氨基酸残基(Asp + Glu)为17个,带负电荷的氨基酸残基(Arg + Lys)为25个,理论等电点(pI)为9.3;CA02g05500蛋白由197个氨基酸组成,分子式为C969H1538N256O284S5,分子量为21 478.72,原子总数为3 052,带正电荷的氨基酸残基(Asp + Glu)为18个,带负电荷的氨基酸残基(Arg + Lys)为25个,理论等电点(pI)为9.21;CA02g21300蛋白由197个氨基酸组成,分子式为C973H1546N256O284S5,分子量为21 534.82,原子总数为3 064,带正电荷的氨基酸残基(Asp + Glu)为17个,带负电荷的氨基酸残基(Arg + Lys)为25个,理论等电点(pI)为9.3;CA04g05500蛋白由210个氨基酸组成,分子式为C1050H1649N271O301S5,分子量为23 045.58,原子总数为3 276,带正电荷的氨基酸残基(Asp + Glu)为18个,带负电荷的氨基酸残基(Arg + Lys)为25个,理论等电点(pI)为9.17;CA08g19280蛋白由198个氨基酸组成,分子式为C982H1566N262O283S5,分子量为21 731.12,原子总数为3 098,带正电荷的氨基酸残基(Asp + Glu)为18个,带负电荷的氨基酸残基(Arg + Lys)为27个,理论等电点(pI)为9.39;CA00g82910蛋白由224个氨基酸组成,分子式为C1130H1775N305O324S13,分子量为25 234.15,原子总数为3 547,带正电荷的氨基酸残基(Asp + Glu)为21个,带负电荷的氨基酸残基(Arg + Lys)为28个,理论等电点(pI)为9.04。表1
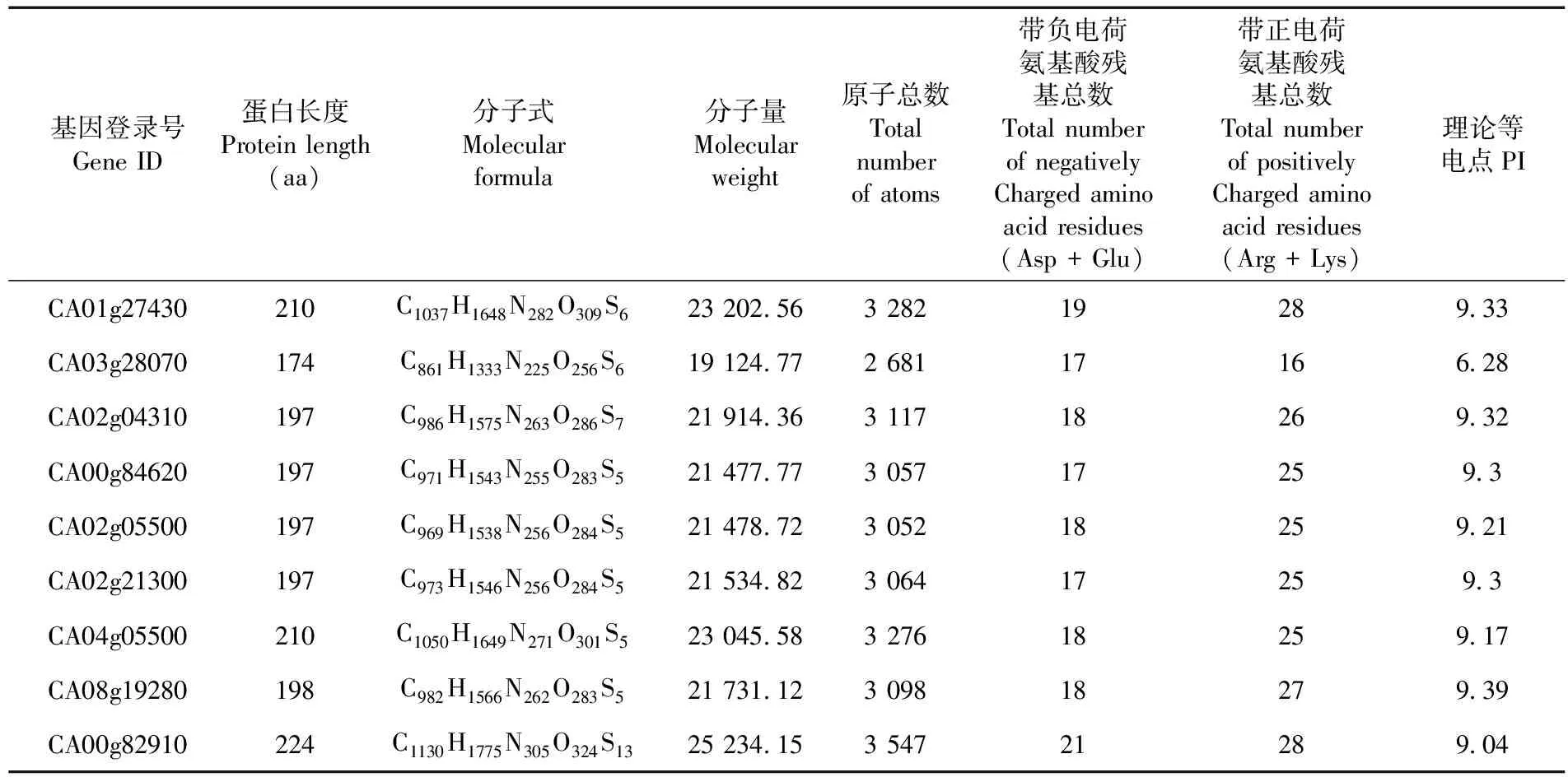
表1 9个CaROP蛋白的理化特性相关信息
2.3 9个CaROP蛋白亲水性及疏水性
研究表明,CA01g27430蛋白的不稳定指数为47.19,脂肪系数为82.14,亲水性平均系数为-0.277,属不稳定的亲水蛋白;CA03g28070蛋白的不稳定指数为43.18,脂肪系数为85.69,亲水性平均系数为-0.04,属不稳定的亲水蛋白;CA02g04310蛋白的不稳定指数为38.96,脂肪系数为90.56,亲水性平均系数为-0.138,属稳定的亲水蛋白;CA00g84620蛋白的不稳定指数为33.45,脂肪系数为88.53,亲水性平均系数为-0.096,属稳定的亲水蛋白;CA02g05500蛋白的不稳定指数为36.01,脂肪系数为87.56,亲水性平均系数为-0.121,属稳定的亲水蛋白;CA02g21300蛋白的不稳定指数为37.31,脂肪系数为88.53,亲水性平均系数为-0.122,属稳定的亲水蛋白;CA04g05500蛋白的不稳定指数为29.96,脂肪系数为91.43,亲水性平均系数为-0.029,属稳定的亲水蛋白;CA08g19280蛋白的不稳定指数为42.05,脂肪系数为90.61,亲水性平均系数为-0.12,属不稳定的亲水蛋白;CA00g82910蛋白的不稳定指数为33.89,脂肪系数为33.89,亲水性平均系数为-0.054,属稳定的亲水蛋白。表2,图3
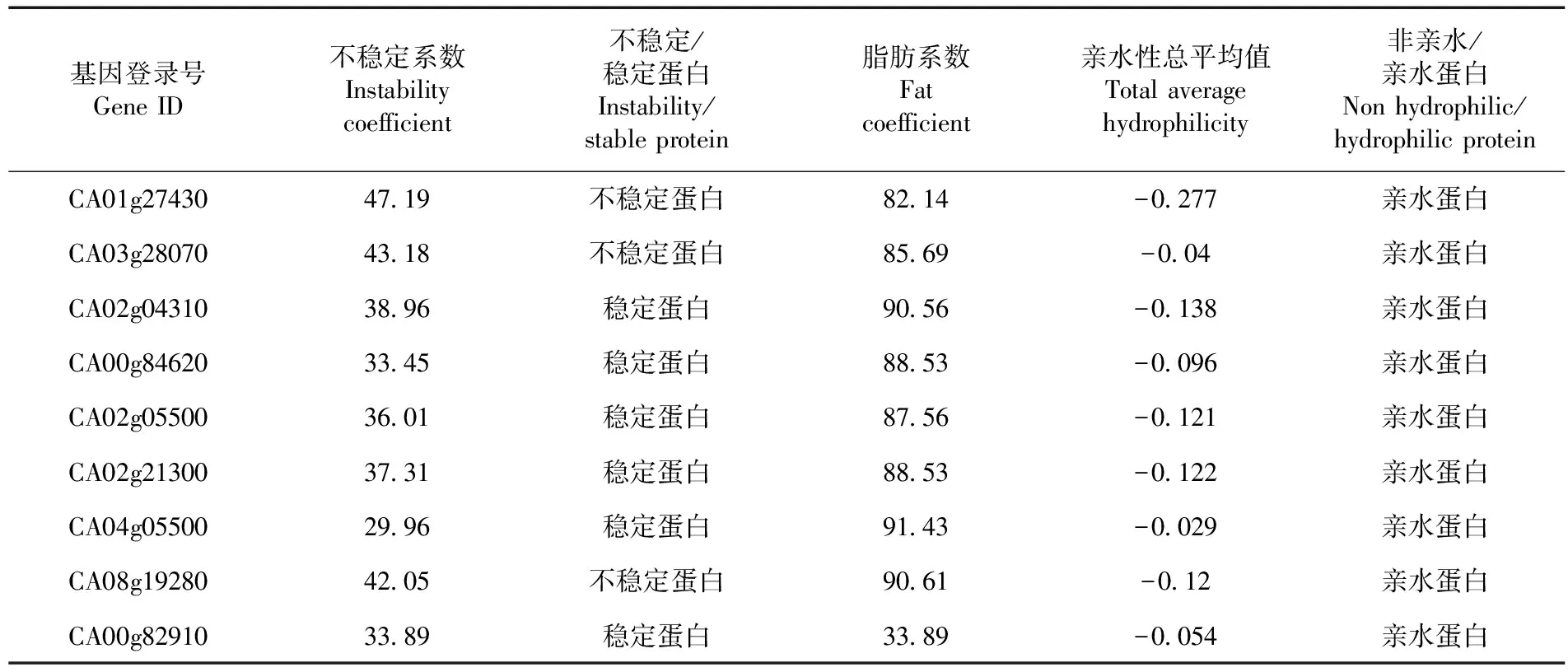
表2 9个CaROP蛋白的亲疏水性参数信息
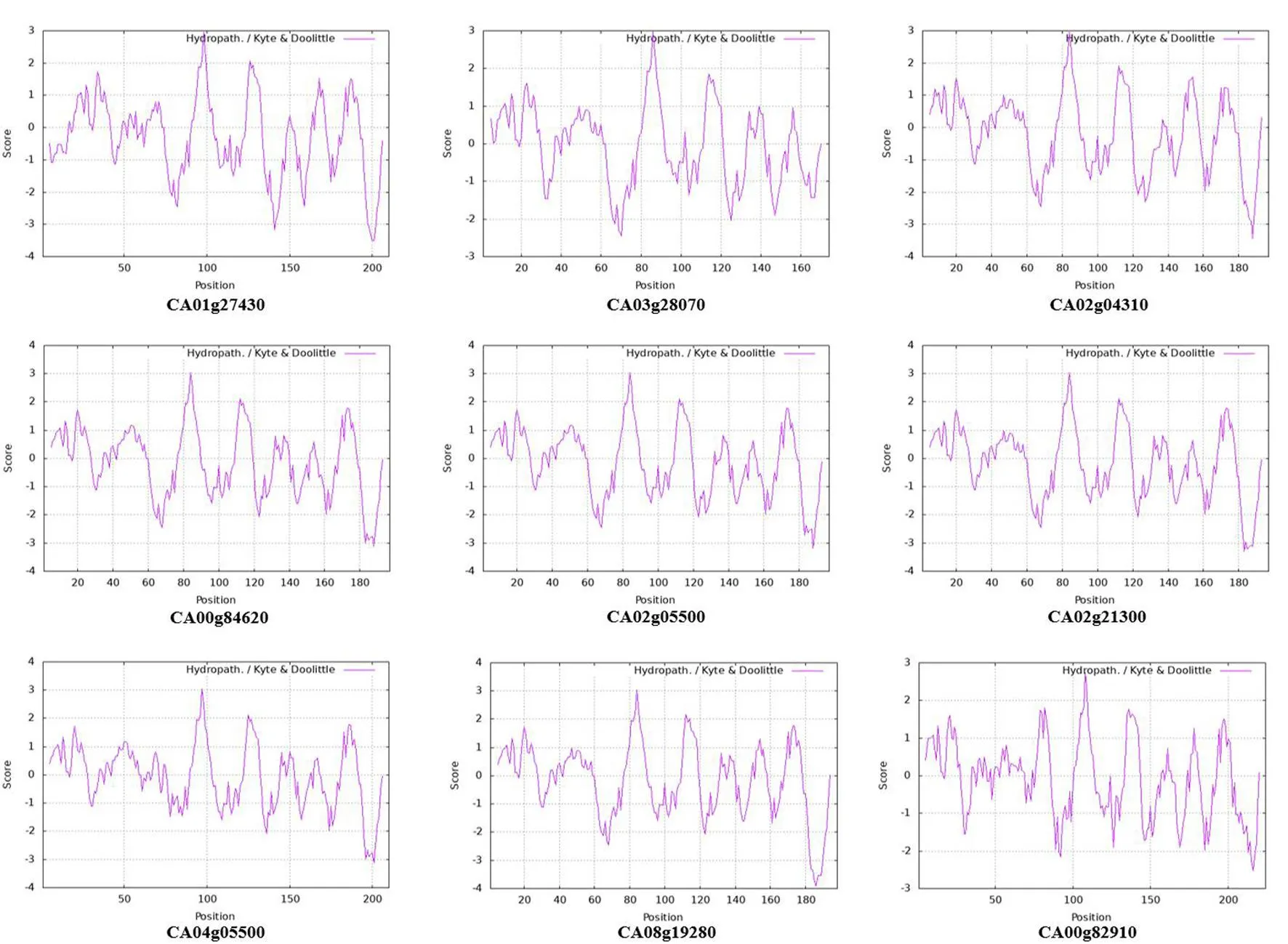
图 3 9个CaROP蛋白的亲水性和疏水性
2.4 9个CaROP蛋白的信号肽及跨膜结构域预测
研究表明,9个CaROP蛋白均无信号肽,均非分泌蛋白。图4
9个CaROP蛋白均跨膜结构域,均非跨膜蛋白。图5
2.5 9个CaROP蛋白磷酸化位点和糖基化位点的预测
研究表明,CA01g27430蛋白含有11个丝氨酸,5个苏氨酸,2个络氨酸;CA03g28070蛋白含有5个丝氨酸,4个苏氨酸,4个络氨酸;CA02g04310蛋白含有8个丝氨酸,6个苏氨酸,6个络氨酸;CA00g84620蛋白含有9个丝氨酸,4个苏氨酸,4个络氨酸;CA02g05500蛋白含有11个丝氨酸,5个苏氨酸,4个络氨酸;CA02g21300蛋白含有10个丝氨酸,3个苏氨酸,4个络氨酸;CA04g05500蛋白含有10个丝氨酸,4个苏氨酸,3个络氨酸;CA08g19280蛋白含有10个丝氨酸,2个苏氨酸,4个络氨酸;CA00g82910蛋白含有9个丝氨酸,5个苏氨酸,5个络氨酸。9个CaROP蛋白均不存在糖基化位点。表3,图6
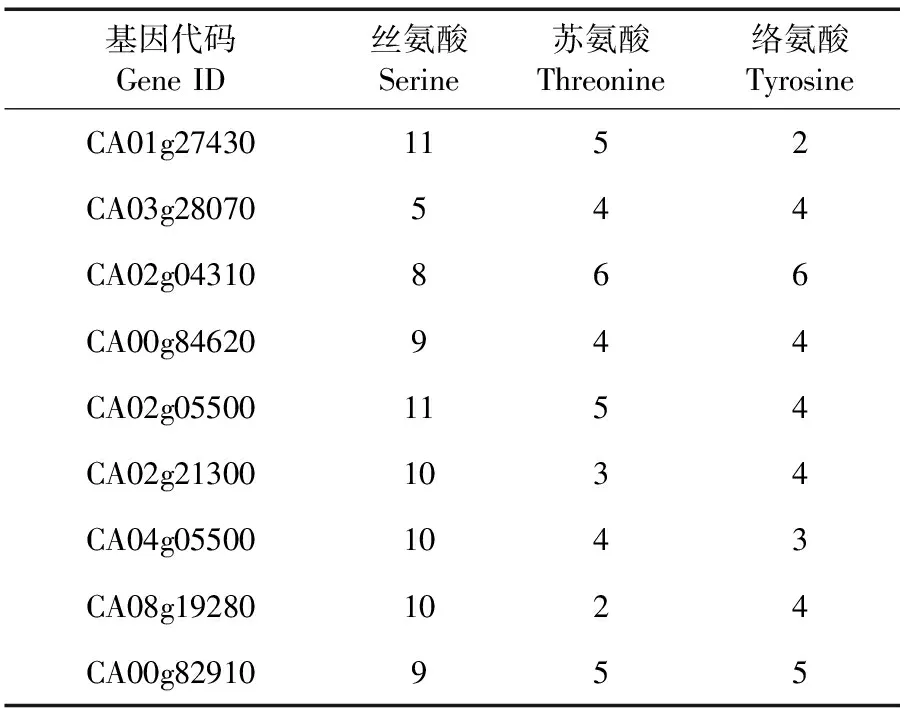
表3 9个CaROP蛋白磷酸化位点相关信息
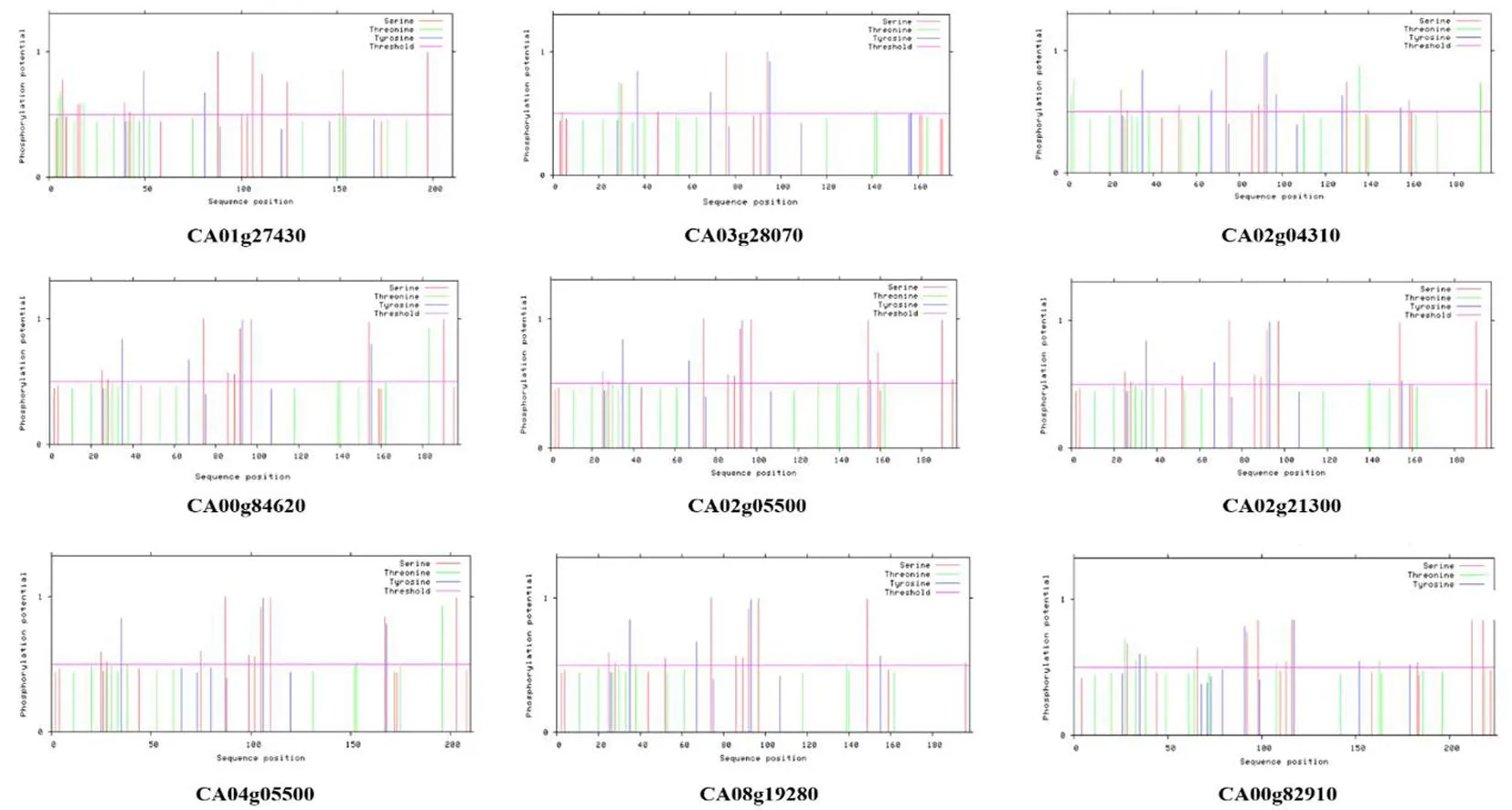
图 6 9个CaROP蛋白磷酸化位点的预测
2.6 9个CaROP蛋白二级、三级结构的预测
研究表明,CA01g27430蛋白含有77个α-螺旋(Hh),占比36.67%;含40个β-折叠(Ee),占比19.05%;10个β-转角(Tt),占比4.76%;含83个无规则卷曲,占比39.52%。CA03g28070蛋白含有68个α-螺旋(Hh),占比39.08%;含35个β-折叠(Ee),占比20.11%;12个β-转角(Tt),占比6.90%;含59个无规则卷曲,占比33.91%。CA02g04310蛋白含有66个α-螺旋(Hh),占比33.50%;含41个β-折叠(Ee),占比20.81%;12个β-转角(Tt),占比6.09%;含78个无规则卷曲,占比39.59%。CA00g84620蛋白含有69个α-螺旋(Hh),占比35.03%;含42个β-折叠(Ee),占比21.32%;13个β-转角(Tt),占比6.60%;含73个无规则卷曲,占比37.06%。CA02g05500蛋白含有72个α-螺旋(Hh),占比36.55%;含40个β-折叠(Ee),占比20.30%;11个β-转角(Tt),占比5.58%;含74个无规则卷曲,占比37.56%。CA02g21300蛋白含有72个α-螺旋(Hh),占比36.55%;含40个β-折叠(Ee),占比20.30%;11个β-转角(Tt),占比5.58%;含74个无规则卷曲,占比37.56%。CA04g05500蛋白含有75个α-螺旋(Hh),占比35.71%;含43个β-折叠(Ee),占比20.48%;11个β-转角(Tt),占比5.24%;含81个无规则卷曲,占比38.57%。CA08g19280蛋白含有67个α-螺旋(Hh),占比33.84%;含40个β-折叠(Ee),占比20.20%;12个β-转角(Tt),占比6.06%;含79个无规则卷曲,占比39.90%。CA00g82910蛋白含有82个α-螺旋(Hh),占比36.61%;含56个β-折叠(Ee),占比25.00%;11个β-转角(Tt),占比4.91%;含75个无规则卷曲,占比33.48%。表4,图7
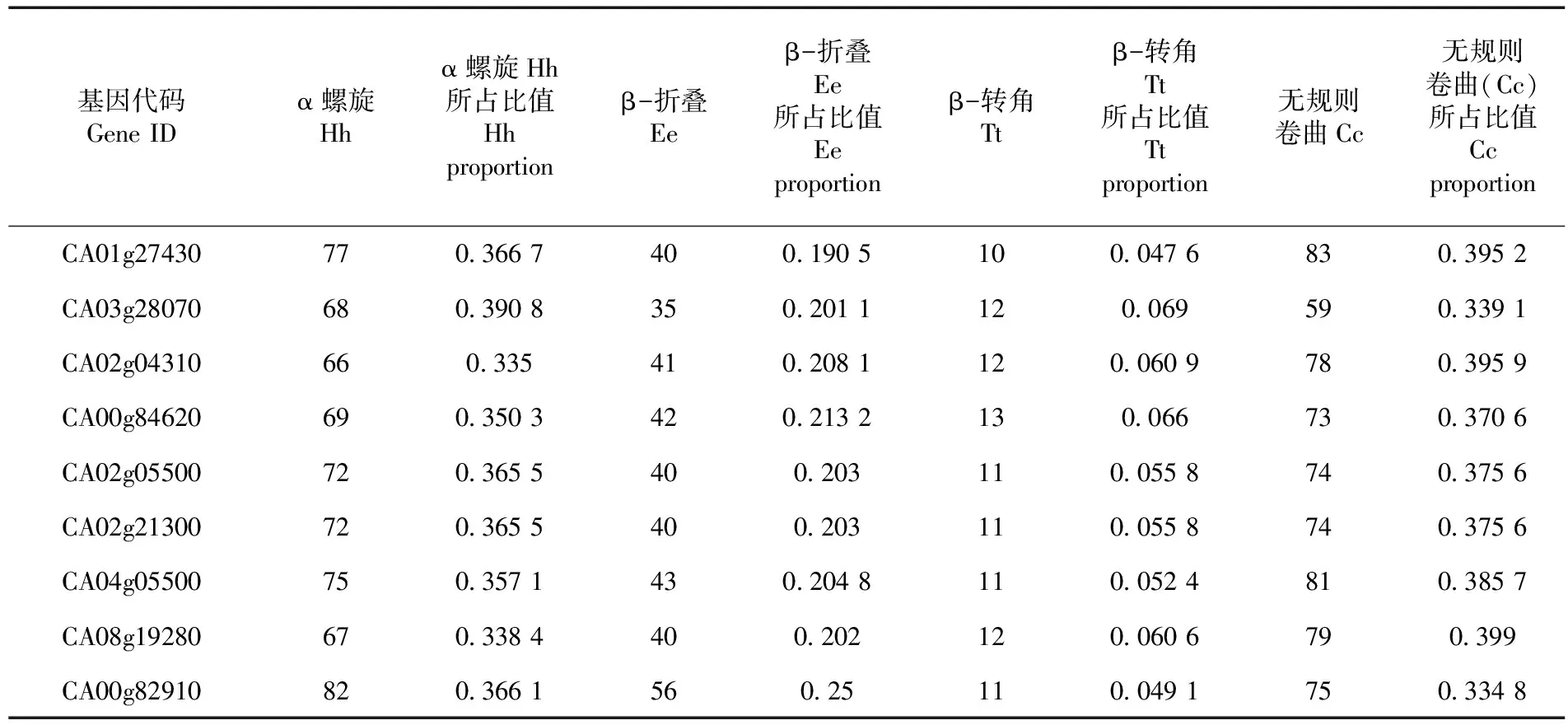
表4 9个CaROP蛋白二级结构的参数信息

图 7 9个CaROP蛋白的二级结构
CA01g27430蛋白的全局模型质量评估(GMQE)为0.77,QMEAN评分为0.82;CA03g28070蛋白的GMQE评分为0.85,QMEAN评分为0.82;CA02g04310蛋白的GMQE评分为0.81,QMEAN评分为0.82;CA00g84620蛋白的GMQE评分为0.81,QMEAN评分为0.83;CA02g05500蛋白的GMQE评分为0.81,QMEAN评分为0.84;CA02g21300蛋白的GMQE评分为0.81,QMEAN评分为0.84;CA04g05500蛋白的GMQE评分为0.76,QMEAN评分为0.79;CA08g19280蛋白GMQE评分为0.81,QMEAN评分为0.83;CA00g82910蛋白的GMQE评分为0.72,QMEAN评分为0.72。GMQE和QMEAN的整体模型质量测量值范围在0~1,数字越高表示预期质量越高,9个CaROP蛋白的全局模型质量评估(GMQE)和QMEAN评分均大于0.7,结果表明9个CaROP蛋白的三级建模预期均比较可靠,但CA00g82910蛋白与其他8个CaROP蛋白三级结构相比差异较大,推测可能与其脂肪系数有关。图8
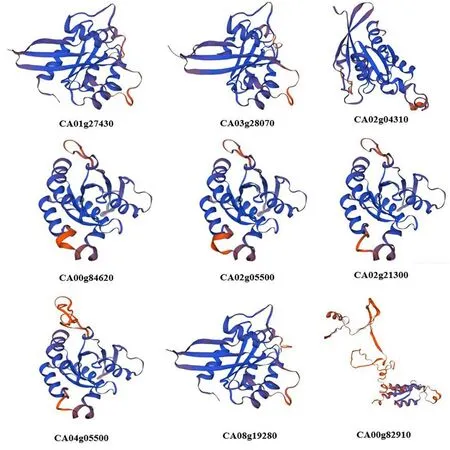
图 8 9个CaROP蛋白的三级结构
2.7 9个CaROP蛋白系统发育关系
研究表明,20个ROP蛋白聚为4个分支,不同颜色代表不同分支,红色代表分支Ⅰ,橙色代表分支Ⅱ,绿色代表分支Ⅲ,蓝色代表分支Ⅳ,其中CA01g27430和CA02g04310分别聚于分支Ⅰ和分支Ⅲ中,CA00g82910和CA03g28070聚于分支Ⅱ中,剩余5个CaROP(CA00g84620、CA04g05500、CA02g05500、CA02g21300及CA08g19280)均聚于分支Ⅳ中。图9
3 讨 论
3.1蛋白的亲水性可以保持结构及水分的稳定也可预测跨膜区,蛋白的脂肪系数越高蛋白相对越稳定,蛋白的稳定性可使蛋白在不良条件下仍然具备优良的理化性能[18-22]。运用生物信息学手段预测分析蛋白质的亲水性、稳定系数等理化特性及蛋白结构,可为后续深入研究蛋白的功能奠定理论基础,并且可减少试验中的盲目及失败[23]。研究蛋白预测分析结果显示,9个CaROP蛋白氨基酸序列相似度高达73.02%,其均具有磷酸化位点,均为非分泌性蛋白。9个CaROP蛋白亲水值均小于0,其均为亲水蛋白;9个CaROP蛋白其均无跨膜结构域和信号肽,表明均为非跨膜蛋白。9个CaROP蛋白的主要二级结构原件为无规则卷曲和α-螺旋,其次是β-折叠,最后是β-转角,其三级结构建模的预测评分均高于0.7,结果都较为可靠,但CA00g82910蛋白相比其他8个CaROP蛋白的三级结构模型差异较大。
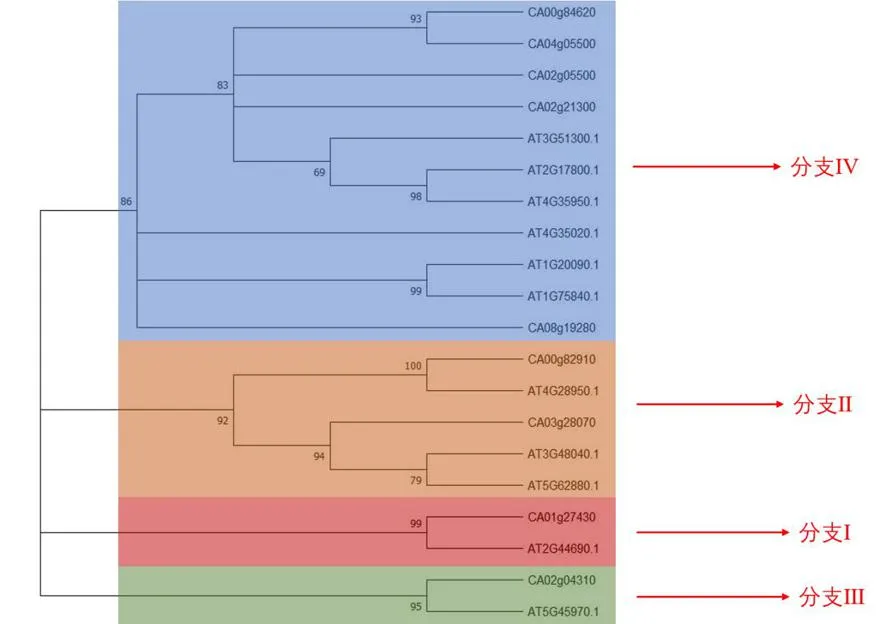
图 9 辣椒和拟南芥ROP蛋白的 系统发育关系
3.2系统发育关系分析结果表明,9个CaROP蛋白共聚为4个分支,其中CA01g27430和CA02g04310分别聚于分支Ⅰ和分支Ⅲ中,CA00g82910和CA03g28070聚于分支Ⅱ中,剩余5个CaROP(CA00g84620、CA04g05500、CA02g05500、CA02g21300及CA08g19280)均聚于分支Ⅳ中。Yang Z等人将拟南芥中的11个AtROP蛋白划分为4个进化分支[24],研究将辣椒中的9个CaROP和拟南芥中的11个AtROP也划分为4个进化分支,分支结果与Yang Z等[25]的研究结果一致。Yang S等将6种茄科物种中的66个ROP蛋白和11个拟南芥AtROP蛋白划分为5个进化分支,Yang Z等[24]的研究中,第IV分支具有2个明显的亚分支,但是随着Yang S等[25]将植物物种数增加后,第IV分支彻底分为2个分支,即新的分支II和分支V。研究中的第IV分支也具有明显的2个亚分支,其中CA08g19280单独聚为一个亚分支,其他3个CaROP蛋白的聚于另一个亚分支,预示其生物学功能可能也不尽相同。
4 结 论
9个CaROP蛋白具有完整的Rho功能域,均为非分泌性亲水蛋白,其中6个ROP蛋白CA02g04310、CA00g84620、CA02g05500、CA02g 21300、CA04g05500和CA00g82910为稳定的亲水蛋白。9个CaROP蛋白在系统进化树中共聚为了4个分支,其三级结构建模的预测结果也均较为理想,9个CaROP蛋白结构较稳定。
