三阴性乳腺癌m6A相关lncRNA的免疫预后模型构建*
2024-01-30鲍淑梅管浩钦苏莹刘沛吕小毅
鲍淑梅,管浩钦,苏莹,刘沛,吕小毅†
(1.新疆大学软件学院,新疆 乌鲁木齐 830091;2.新疆大学信息科学与工程学院,新疆 乌鲁木齐 830017)
0 引言
根据最新全球癌症统计报告显示乳腺癌(BRCA)已超过肺癌,成为世界上确诊人数最多的癌症,占全球报告病例的11.7%.2020年,全球仅女性乳腺癌就有230万例,同时有超过68万人死于乳腺癌[1].三阴性乳腺癌(TNBC)是一种特殊的乳腺癌分子亚型,约占乳腺癌的15%,其特征是雌激素受体(ER)、孕激素受体(PR)和人表皮生长因子受体2(HER2)的缺失[2].TNBC的复发和死亡主要发生在确诊后的前5年,尤其是确诊后的1~3年内.确诊后的前5年,TNBC患者内脏器官转移的发生率远高于非TNBC患者且预后较差,5年生存率不到30%[3-4].由于TNBC的异质性和缺乏分子靶点,TNBC患者对内分泌治疗或HER2靶向治疗不敏感,导致化疗仍然是TNBC患者的标准治疗方法[5-6].因为肿瘤对化疗的耐药性,导致TNBC患者在接受化疗后肿瘤迅速复发或转移,所以TNBC仍然是一种预后较差和治疗方法有限的疾病[7].N6-甲基腺苷(m6A)是真核细胞最丰富的核糖核酸(RNA)修饰,在各种生物过程和信使核糖核酸(mRNA)代谢中至关重要,例如RNA加工、转运和稳定性[8-9].m6A修饰包括甲基化转移酶、信号转导和去甲基化酶,它们分别被称为写入者、读取者和擦除者.此外,m6A修饰也是一个可逆的RNA表观遗传过程[10].RNA结构的变化可以影响各种细胞过程.因此,m6A调控的长链非编码RNA(lncRNAs)的作用可能对癌细胞的增殖和迁移至关重要[11].
有研究表明,m6A修饰可调节肿瘤发生和肿瘤发展.例如,甲基转移酶3(METTL3)的表达减少通过m6A甲基化介导的III型胶原蛋白α1链(COL3A1)上调促进三阴性乳腺癌的转移[12].此外,YTH N6-甲基腺苷RNA结合蛋白F3(YTHDF3)通过以m6A依赖性方式稳定锌指E-box绑定同源盒1(ZEB1)促进三阴性乳腺癌进展和转移[13].最近,一些生物信息学研究表明,m6A调节器的失调与TNBC有关[14-15].m6A调节因子在lncRNA中的具体作用尚不明确;因此,了解m6A相关lncRNA在TNBC发展中的机制可能对预后目标有用.
本文首先从癌症基因组图谱(TCGA)数据集中提取了10 357个lncRNA和23个m6A基因的表达谱.接下来使用皮尔逊相关性分析确定了m6A相关的lncRNA.构建的模型是基于m6A的新型预后模型,该模型旨在预测TNBC患者的总生存期(OS).使用公开的药物敏感性数据库,发现了针对这种m6A相关lncRNA特征的候选药物,并探讨了与免疫治疗反应的关系.最后,建立了列线图来预测TNBC患者的OS.
1 材料与方法
1.1 TNBC患者信息获取
TCGA数据库(https://cancergenomenihgov/)是目前最大的公开资源数据库,也是国际上最常用的数据库.该数据库提供数以千计的与肿瘤样本相关的基因表达、体细胞突变、基因甲基化等数据集.世界各地的癌症研究人员可以在TCGA数据库中免费获取相关数据,识别可能在癌症发展中发挥作用的重要基因.本文从TCGA数据库中获取了TNBC患者的RNA序列转录组数据、相关临床信息和突变数据.筛选三阴性乳腺癌患者样本的标准为:(1)组织病理学诊断为乳腺癌;(2)按TNBC诊断标准进行筛查免疫组织化学中ER、PR、HER2表达缺失患者.
1.2 m6A基因和m6A相关lncRNA的选择
本文从TCGA数据库中获得了lncRNA和m6A基因的表达情况.根据前人研究,从TCGA中检索到23个m6A基因的表达矩阵,包括甲基化转移酶(METTL3、METTL14、METTL16、VIRMA、RBM15、RBM15B、ZC3H13和WTAP)、结合蛋白(IGFBP1、IGFBP2、IGFBP3、LRPPRC、FMR1、YTHDC1、YTHDC2、YTHDF1、YTHDF 2、YTHDF3、HNRNPA2B1、HNRNPC和RBMX)和去甲基化酶(ALKBH5和FTO)[16].通过皮尔逊相关性分析筛选了m6A相关的lncRNA,确定1 149个m6A相关的lncRNA.该过程使用了|Pearson R|的标准大于0.4且p<0.001.
1.3 风险签名的建立和验证
整个TCGA数据集被随机分为训练集和测试集.利用训练集构建m6A相关的lncRNA模型,并应用整个集和测试集来验证所建立的模型.结合TCGA中的TNBC生存信息,本文采用单变量Cox回归从TCGA数据集中的275个m6A相关lncRNAs中筛选出m6A预后相关的lncRNAs(p<0.05).使用R包glmnet进行LASSO回归(使用通过10倍交叉验证估计的惩罚参数)筛选候选基因,同时使用R包randomForestSRC构建RSF算法模型筛选候选基因.对LASSO与RSF算法筛选出的候选基因取交集,获得5个m6A相关的lncRNAs与来自TCGA数据集的TNBC患者的OS明显相关.应用多因素Cox回归分析5个m6A相关的lncRNAs,最终建立了基于3个m6A相关的lncRNA风险模型.风险评分计算公式如下:Risk score=coef (lncRNA1)×expr (lncRNA1)+coef (lncRNA2)×expr (lncRNA2)+···+coef (lncRNAn)×expr (lncRNAn).其中:coef表示系数,coef (lncRNAn)是与生存相关的lncRNA的系数,expr (lncRNAn)是lncRNA的表达量.根据中位风险评分,建立了包括低风险组和高风险组在内的亚组[17].
1.4 差异基因富集分析
进行了基因本体论(GO)富集分析以确定高低风险组之间差异表达的基因所富集的相关生物学通路.此过程使用R包clusterProfiler.分析阈值由p值确定,p<0.05表示相关生物功能显著丰富[18].
1.5 免疫治疗模式探索
使用R包maftools评估和汇总突变数据.根据肿瘤特异性突变基因测量肿瘤突变负荷(TMB)[19].使用肿瘤免疫功能障碍和排斥(TIDE)算法计算TIDE评分,通过TIDE评分预测免疫治疗反应的效果[20].
1.6 PCA和KM生存分析
使用PCA根据3个m6A基因的表达模式,对全基因表达谱、23个m6A基因、275个m6A相关的lncRNA和风险模型相关的lncRNAs的高维数据进行有效降维、模型识别和分组可视化[21].使用KM生存分析评估高风险组和低风险组之间OS的多样性.使用R包survMiner和survival完成实验.
1.7 临床治疗中靶向m6A相关lncRNA模型的潜在化合物探索
为了在临床上获得用于TNBC治疗的潜在化合物,本文在TNBC数据集中计算了从GDSC网站获得的化合物的IC50.R包pRRhetic用于预测从GDSC网站获得的化合物在TNBC患者中的IC50.
1.8 m6A相关lncRNA模型的独立性
考虑到TNBC患者的其它临床特征,如年龄、原发肿瘤的范围和大小(T)分期、淋巴结播散情况(N)分期等,进行多变量和单变量Cox回归分析以检验预后模型是否为独立变量.
1.9 建立和证明预测列线图
建立列线图使用预测因子(年龄、风险评分、T分期、N分期)预测2年、3年和5年TNBC患者的OS.应用基于Hosmer-Lemeshow检验的校正曲线来说明实际结果和模型预测结果之间的一致性.
2 实验结果
2.1 TNBC患者m6A相关lncRNA的鉴定
风险模型构建和分析的详细工作流程如图1所示.从TCGA数据库中提取了23个m6A基因和10 357个lncRNA的矩阵表达.本文将m6A相关lncRNA定义为与大于或等于23个m6A基因之一显著相关的lncRNA(|Pearson R|>0.4和p<0.001).最后,使用图2(a)中的桑基图可视化m6A-lncRNA共表达网络,将275个m6A相关lncRNA识别为m6A相关的lncRNA.TCGA所有m6A基因与m6A相关lncRNA的相关性如图2(b)所示.

图1 工作流程
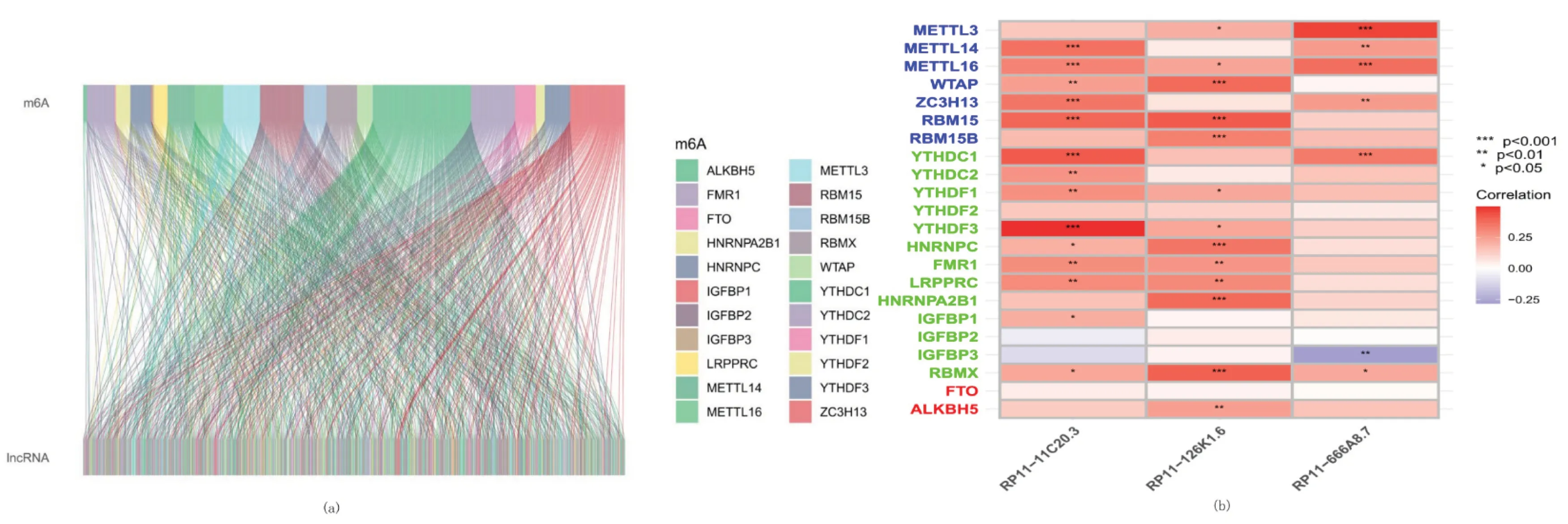
图2 TNBC患者中m6A相关lncRNA的鉴定
2.2 根据TNBC患者的m6A相关lncRNA构建和验证风险模型
本文使用单变量Cox回归分析从TCGA训练集中的275个m6A相关lncRNA中筛选出m6A相关预后lncRNA.TCGA数据集中的11个m6A相关lncRNA与OS显著相关(图3(a)).
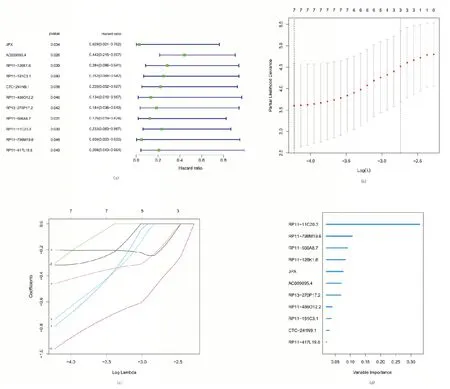
图3 基于m6A相关lncRNA的TNBC患者风险模型
LASSO分析是多元回归分析的常用方法.该方法的应用不仅提高了统计模型的预测精度和可解释性,而且可以同时进行变量选择和正则化.该方法广泛应用于相关性较差、预测值突出的高维数据中特征的最优选择,避免过拟合.因此,LASSO可以有效地辨别最可用的预测标记并产生预后指标来预测临床结果.RSF是一种基于决策树集合的生存分析机器学习方法,已经证明其对嘈杂的变量数据具有较好的鲁棒性.此外生存树既能检测非线性关系的变化(变量和预测目标值之间),又能检测变量之间相互作用.所以适用于从包含生存状态的基因表达数据中筛选出关键基因[22].之后,对LASSO和RSF选出的候选基因取交集,获得5个m6A相关的lncRNA用于后续的多变量Cox分析(图3(b~d)).接下来使用多变量Cox回归分析来筛选基因构建模型.最终3个m6A相关lncRNA用于构建风险模型以评估TNBC患者的预后风险.
根据预后风险等级的中值,将TNBC样本分为低风险组和高风险组.二者之间的风险等级分布如图4(a)所示,两个不同风险组患者的生存状态和生存时间如图4(b)所示.每个患者的3个m6A相关lnc-RNA的相对表达标准如图4(c)所示.生存分析表明,低风险组的OS长于高风险组(p<0.01)(图4(d)).
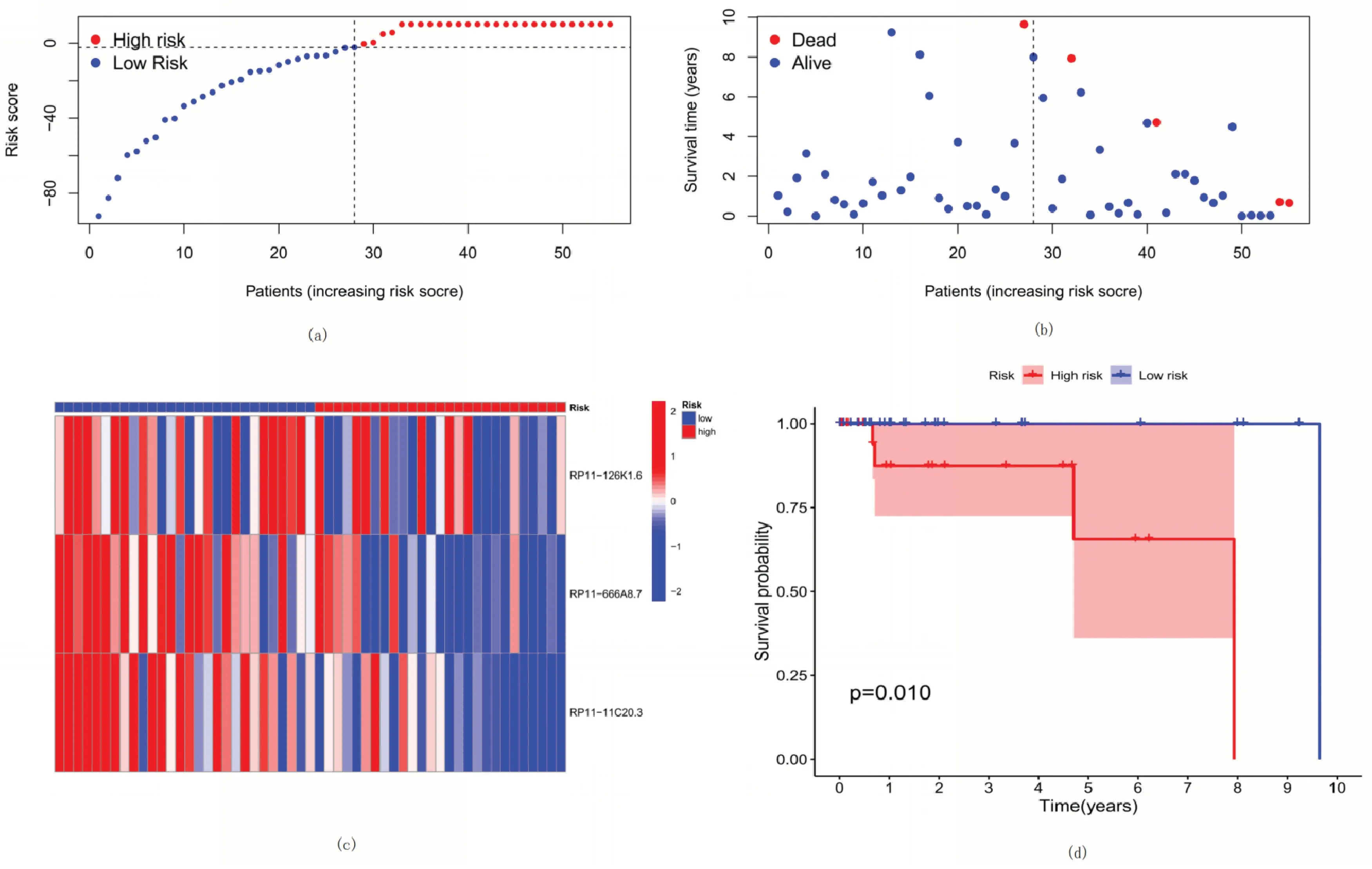
图4 TCGA训练集中3个m6A相关lncRNA风险模型的预后价值
为了测试这个已建立模型的预后能力,本文使用统一公式计算了测试组和整个组中每个患者的风险评分.图5描述了风险等级的分布、生存状态和生存时间的模式,以及测试集(图5(a~c))和整个集(图5(e~g))中m6A相关lncRNA的表达.对测试集和整个集进行的KM生存分析显示与TCGA训练集的结果没有差异:具有较高风险评分的TNBC患者的OS比具有较低风险评分的患者更差(p<0.05)(图5(d)和图5(h)),表明m6A相关lncRNA模型水平影响TNBC患者的预后.

图5 TCGA测试和整个组中3个m6A相关lncRNA风险模型的预后价值
2.3 PCA进一步验证了m6A相关lncRNA模型的分组能力
基于全基因表达谱、23个m6A基因、23个m6A相关lncRNA以及按3个m6A相关lncRNA表达谱分类的风险模型,进行PCA检验低危组和高危组的差异(图6).图6(a~c)显示高风险和低风险人群的分布相对分散.然而,根据本文模型获得的结果表明,低风险和高风险群体具有不同的分布(图6(d)).这些结果表明预后特征可以区分低风险和高风险人群.

图6 高风险组和低风险组之间的主成分分析
2.4 使用m6A相关lncRNA模型估计肿瘤免疫微环境和癌症免疫治疗反应
基于来自108个TNBC样本的m6A相关lncRNA模型,进一步分析了TNBC中高低风险组之间几种免疫细胞、通路或功能的活性.低危组和高危组在免疫指标的表达上表现出显著差异(图7(a~b)).为探索基于m6A模型的潜在分子机制,本文进行了GO富集分析,揭示了许多免疫相关生物过程的参与(图7(c)).接下来研究了m6A相关lncRNA模型与免疫治疗生物标志物之间的相关性.不出所料,发现高风险组比低风险组更有可能对免疫疗法产生反应,表明这种基于m6A的分类器指数可以作为预测TIDE的指标(图7(d)).使用R包maftools分析和总结突变数据.根据变异效应预测因子对突变进行分层.图7(e)和图7(f)显示了高风险和低风险亚组之间突变频率最高的前20个驱动基因.

图7 在TCGA整个数据集中使用m6A相关lncRNA模型估计肿瘤免疫微环境和癌症免疫治疗反应
2.5 鉴定靶向m6A相关lncRNA模型的新型候选化合物
为了确定针对本文用于治疗TNBC患者的lnc-RNA模型的潜在药物,使用pRRhetic算法根据每个样本的GDSC数据库中可用的半数最大抑制浓度(IC50)来估计治疗反应.筛选出了3种化合物在高低风险组之间估计的IC50存在显著差异,高风险组对这些化合物都更敏感.图8显示了可用于TNBC患者进一步分析的3种化合物.
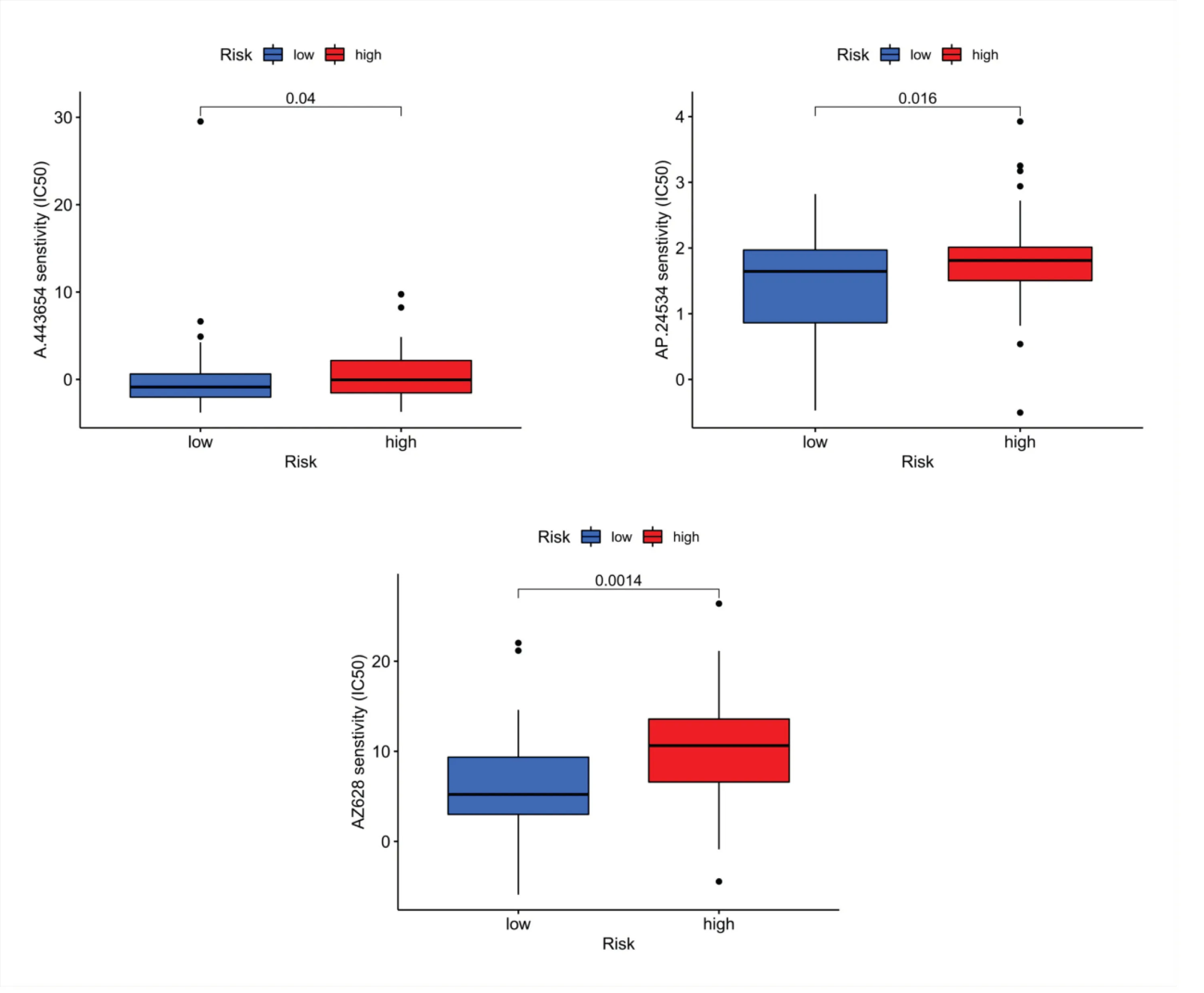
图8 三种新型候选化合物在高低风险组之间的IC50
2.6 m6A相关lncRNA预后风险模型评估
本文进行了单变量和多变量Cox回归分析,以此评估3个m6A相关lncRNA的风险模型是否具有TNBC的独立预后特征.在单变量Cox回归分析中,风险评分的风险比(HR)和95%置信区间(CI)分别为1.050和1.011~1.090(p<0.011).在多变量Cox回归分析中,HR为1.092,95%CI为1.002~1.189(p<0.044)(图9),表明3种m6A相关lncRNA的风险模型与临床病理参数无关,如种族、年龄和肿瘤/淋巴结/转移(TNM)分期等.
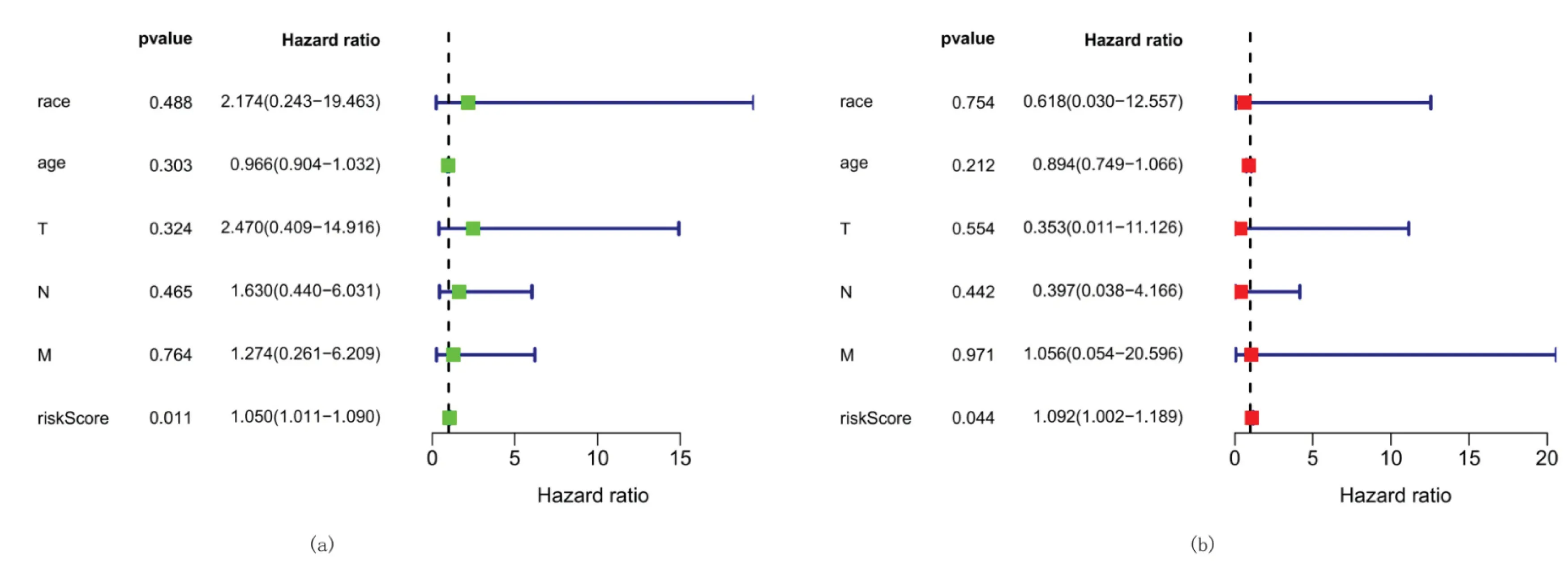
图9 TCGA整个组TNBC中m6A相关lncRNA和临床特征的预后风险模型评估
2.7 预后列线图的构建和评估
构建了包含风险等级和临床风险特征的列线图来预测2年、3年和5年的OS发生率(图10(a)).相关实验结果显示,2年、3年和5年OS率与预测率表现出理想的一致性(图10(b~d)).对于2年、3年和5年OS率,受试者工作特征曲线(ROC)的AUC值分别为0.984、0.805和0.853(图10(e)).
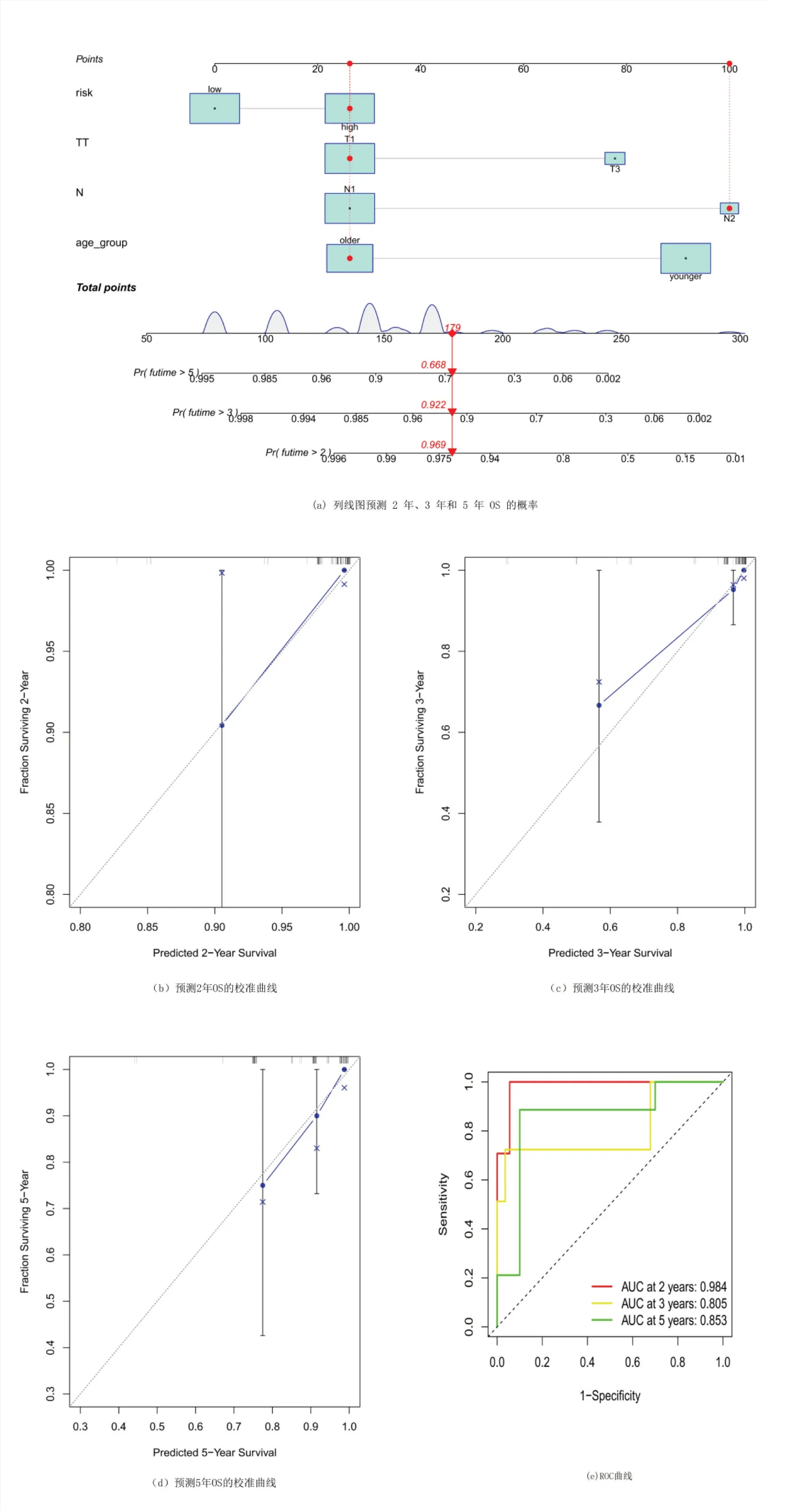
图10 预后列线图的构建和评估
3 分析与讨论
与其它乳腺癌相比,TNBC是一种更具侵袭性的亚型,与其它亚型相比存活率较低.TNBC患者中只有30%~45%达到了与其它乳腺癌亚型相似的完全病理反应和生存率.在预后方面,由于缺乏有效的治疗靶点,TNBC患者的预后效果一般不佳.因此,越来越多的研究专注于识别非编码RNA的特征,以预测TNBC患者的生存和免疫治疗反应.
本文从TCGA数据集中鉴定了275个m6A相关的lncRNA,以探索m6A相关的lncRNA的预后功能.TCGA数据集证实了11个m6A相关lncRNA的预后价值,其中3个用于构建m6A相关lncRNA模型以预测TNBC患者的OS.其中RP11-126K1.6与卵巢癌中基于铂的化学耐药性相关[23].此外,其它lncRNA首次被揭示与癌症相关.随后,根据中间风险评分将TNBC患者分为高风险组和低风险组,高风险组的临床结果明显较差.多变量Cox回归分析表明,m6A相关lncRNA模型是OS的独立风险因素.本文还建立了列线图,显示2年、3年和5年OS的观察率和预测率之间的完美一致性.ROC分析表明,该模型在TNBC的2年、3年和5年OS预测率显示出较好的敏感性和特异性.最后,观察到的2年、3年和5年OS预测率显示出极好的一致性.基于与OS独立相关的3个m6A相关lncRNA的风险模型相当准确,该预测模型可以为后续研究识别新的生物标志物.
TMB是体细胞编码突变的总数,与触发抗肿瘤免疫的新抗原的出现有关[24].最近的研究表明,TMB是预测程序性细胞死亡配体1(PD-L1)治疗反应的有效生物标志物[25].本文发现TMB在高低风险组之间具有差异.
此外,越来越多的研究使用了TIDE预测评分,这是一种为免疫治疗预测开发的计算框架,其预测功能已成功验证[26].本文中,TIDE算法的预测表明,高风险亚型患者对免疫疗法有更好的反应.根据以上结果,推断该预测模型可能为肿瘤治疗提供可靠的免疫生物标志物.此外,本文为TNBC中m6A相关lncRNA的分子生物学机制提供了新的见解.
在临床上,病理分期是影响TNBC预后的决定性因素[27].然而,同一分期的TNBC患者往往有不同的临床结局,提示目前的分期系统在提供可靠的预测和反映TNBC的异质性方面是不准确的[28].因此,应探索潜在的预测和治疗生物标志物.本文建立的m6A相关lncRNA模型为TNBC患者的预后预测提供了一种新方法.该结果也为未来研究lncRNAs m6A修饰的过程和机制提供了思路.
4 结论
本文为TNBC患者的预后预测提供了线索,并可能有助于阐明m6A调节的lncRNA的过程和机制.此外,该预后模型在2年、3年和5年的OS预测中表现出良好的性能.这些发现将为今后TNBC的预后和免疫治疗提供一定的借鉴.
