猪miR-23a-27a-24簇启动子预测及活性分析
2024-01-03李清春孙晓梅孙敬礼黄涛
李清春 孙晓梅 孙敬礼 黄涛
摘要:目的 本研究旨在對猪miR-23a-27a-24簇(miR-23a-27a-24 cluster)的潜在启动子区域进行预测和分析,为进一步探索miR-23a-27a-24基因对猪转录调控机制的研究奠定基础。方法 利于生物信息学分析猪miR-23a-27a-24簇的启动子区域、CpG岛及其潜在的转录因子;利用截短突变和PCR技术克隆得到5个逐级缺失的miR-23a-27a-24基因启动子片段,构建双荧光素酶报告载体, 通过检测各载体的双荧光素酶活性获得miR-23a-27a-24基因的核心启动子区域。结果 pGL3-cMiR启动子在猪miR-23a-27a-24簇上游341bp至994bp区荧光素酶活性最强,为pGL3-basic空载体活性的5.02倍(P<0.05),视为miR-23a-27a-24簇核心启动区。生物信息学分析表明:猪miR-23a-27a-24簇启动子区不含CpG岛,其核心启动子区可能存在SP1、VDR、MAZ、YY1、E2F1和ELF1六种潜在的转录因子结合位点。
结论 本研究构建并确定了猪miR-23a-27a-24簇核心启动子区域,可为今后开展猪miR-23a-27a-24簇的转录调控机制奠定基础。
关键词:猪;miR-23a-27a-24簇;启动子;转录调控
中图分类号:中图分类号S828文献标志码:A文献标识码
Prediction and activity analysis of the promoter of the porcine miR-23a-27a-24 cluster
LI Qingchun,SUN Xiaomei,SUN Jingli,HUANG Tao*
(School of Animal Science and Technology, Shihezi University,Shihezi,Xinjiang 832000, China)
Abstract: Objective The aim of this study was to predict and analyse the potential promoter regions of the porcine miR-23a-27a-24 cluster and to lay the foundation for further exploration of the function of the miR-23a-27a-24 gene on the transcriptional regulatery mechanism in pigs. Methods The promoter region of the porcine miR-23a-27a-24 cluster, the CpG island and its potential transcription factors were analyzed by bioinformatics; five promoter fragments of the miR-23a-27a-24 gene with stepwise deletion were cloned by using truncated mutation and PCR techniques, and a dual luciferase reporter vector was constructed, and the dual luciferase activity of each vector was assayed to obtain the miR-23a- The core promoter region of the miR-23a-27a-24 gene was obtained by assaying the dual luciferase activity of each vector. Results showed that the pGL3-cMiR promoter had the strongest luciferase activity in the 341bp to 994bp region upstream of the porcine miR-23a-27a-24 cluster, which was 5.02 times higher than that of the pGL3-basic empty vector (P<0.05), and was regarded as the core promoter region of the miR-23a-27a-24 cluster. Bioinformatics analysis showed that the promoter region of porcine miR-23a-27a-24 cluster did not contain CpG islands, and six potential transcription factor binding sites, SP1, VDR, MAZ, YY1, E2F1 and ELF1, might exist in its core promoter region. Conclusions This study constructed and identified the core promoter region of the porcine miR-23a-27a-24 cluster, which can lay the foundation for future work on the transcriptional regulatory mechanism of the porcine miR-23a-27a-24 cluster.
Key words: pig;miR-23a-27a-24 cluster;promoter;transcriptional regulation
MicroRNAs(miRNA)是一种内源性保守的长度为19-22个核苷酸的非编码RNA,通过翻译抑制和/或促进mRNA降解参与转录后调控进而参与一系列重要的生物学过程,如细胞增殖、分化、凋亡和细胞癌变等[1]。目前对于miRNAs的研究多集中在单一的miRNA上,而miRNA中的一部分是以miRNA基因簇的形式存在,与非成簇miRNA不同的是,这些miRNA簇往往由一个共同的启动子调控,转录后形成多个成熟的miRNAs[2],这些miRNA往往在功能上相同,它们或具有相同的靶基因或作用相同的信号通路[3]。因此,研究miRNA簇对调控细胞通路或决定细胞命运的协同作用是十分必要的,这将给我们一个细胞内miRNA调控的整体图景。
随着miRNA研究热度的不断攀升以及研究的深入,miRNA簇的作用开始逐渐被熟知。miR-23a-27a-24簇位于人19号染色体19p13.12,是由同一启动子转录后形成的3个成熟的miRNAs: miR-23a, miR-27a和miR-24[4]。miR-23a-27a-24簇被证明在多种癌症细胞中表达上调,包括肝细胞癌、乳腺癌、胃癌、胆管癌、胶质母细胞瘤[1]。但近年来的研究发现miR-23a-27a-24簇中的的成员miR-23a和miR-27a涉及卵巢卵泡發育,激素合成与分泌以及卵巢衰等生理过程[5]。例如,在小鼠的研究中发现, MⅡ期卵母细胞周围的颗粒细胞中miR-27a表达水平较MⅠ期卵母细胞明显降低, 上调mi R-27a可以提高卵母细胞的成熟率[6]。在人卵巢中,miR23a和miR27a可通过FasL-Fas凋亡途径调控颗粒细胞的凋亡过程,miR-24能够抑制雌激素的分泌参与调节颗粒细胞的凋亡[7]。而在家畜上,王思琪等[8]发现miR-23a-27a -24簇可以调控猪颗粒细胞的凋亡过程。提示miR-23a-27a-24可能作为猪繁殖育种的分子标记。但目前尚未见猪miR-23a-27a-24簇转录调控相关的报道。为此,本研究开展猪miR-23a-27a -24簇基因簇启动子位置的预测和活性分析等研究,旨在为分析猪miR-23a-27a-24簇的作用机制及其转录调控机制奠定基础。
1 材料与方法
1.1 试验材料
大肠杆菌DH5α感受态细胞(北京全式金生物有限公司),PK-15细胞(本实验室保存),萤光素酶报告基因载体(pGL3-basic)、海肾荧光素酶报告基因载体(pRL-TK)(美国Promega公司),Dual-Luciferase Reporter Assay System(美国Promega公司)。
1.2 试验试剂和仪器
无内毒素质粒提取试剂盒(北京全式金生物有限公司),限制性内切酶(Xho Ⅰ、Hind Ⅲ)、T4-DNA连接酶(TaKaRa大连宝生物有限公司),琼脂糖凝胶DNA回收试剂盒(北京天根生化有限公司),LipofectamineTM 2000转染试剂(美国Invitrogen公司),DMEM/Opti-MEN培养基(美国Invitrogen公司),CyclerTM Thermal Cycler PCR仪(美国Bio-Rad公司),DYY-6B型稳压稳流电泳仪(北京六一仪器厂,中国),微生物培养箱(Thermo Scientific,美国)。
1.3 方法
1.3.1 猪miR-23a-27a-24簇核心启动区域预测
利用UCSC(http://genome.ucsc.edu/)、RNAcentral(http://rnacentral.org/)和NCBI(https://www.ncbi.nlm.nih.gov/)分析猪miR-23a-27a-24簇前体序列;获取miR-23a-27a-24簇前体序列上游3500bp并利用Promoter 2.0 Prediction(http://www.cbs.dtu.dk/services/Promoter/)、phiSITE(http://www.phisite.org/)和softberry(http://www.softberry.com/)预测其核心启动子区域。
1.3.2 猪miR-23a-27a-24簇启动区域CpG岛预测
利用在线软件Methprimer(http://www.urogene.org/methprimer2/)和EMBOSS(http://emboss.bioinformatics.nl)对猪miR-23a-27a-24簇启动区CpG岛进行预测。
1.3.3 猪miR-23a-27a-24簇核心启动区域转录因子预测
利用AnimalTFDB 3.0 (http://bioinfo.life.hust.edu.cn/AnimalTFDB/#!/)、PROPO (http://alggen.lsi.upc.es/cgi-bin/promo_v3/promo/promoinit.cgi?dirDB=TF_8.3)和JASPAR (https://jaspar.genereg.net/)对猪miR-23a-27a-24簇核心启动子区转录因子结合位点进行预测,参数设定为Relative profile score threshold >90%。
1.3.4 引物设计
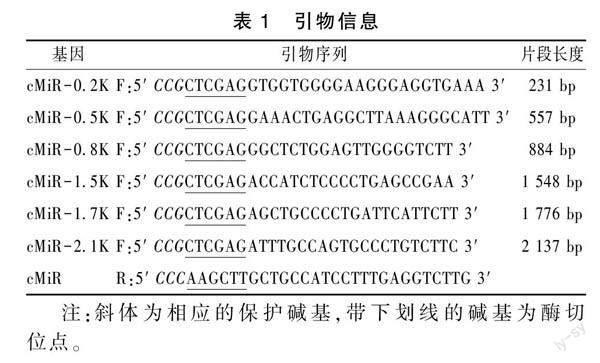
从NCBI和RNAcentral数据库上获取猪miR-23a-27a-24基因序列并利用Primer 5.0软件对miR-23a-27a-24基因序列上游5′端分别设计6条同向续减片段引物,即miR-23a-27a-24 基因簇前体序列上游-341bp/-110bp、-667bp/-110bp、-994bp/-110bp、-1658bp/-110bp、-1876bp/-110bp和-2247bp/-110bp,做为miR-23a-27a-24基因启动子区特异性引物,并在上下游引物序列5′端分别加入Xho Ⅰ、Hind Ⅲ限制性内切酶位点(表1)。
1.3.5 猪miR-23a-27a-24簇启动子报告载体的构建
以猪基因组DNA为摸板扩增miR-23a-27a-24基因上游6条续减片段。扩增产物经凝胶电泳鉴定和测序无误后,分别使用Xho Ⅰ和Hind Ⅲ对回
收的miR-23a-27a-24基因上游各续减片段及pGL3-Basic载体进行双酶切,胶回收miR-23a-27a-24基因上游各续减片段及pGL3-Basic线性载体并依次进行连接和转化,PCR筛选阳性克隆菌株送测序。测序结果比对无误后,根据北京天根生化有限公司无内毒素质粒大量提取试剂盒提取报告基因载体质粒,用于后续细胞转染。
1.3.6 PK15细胞的培养与转染
PK15细胞培养使用含10% FBS的DMEM完全培养基,培养条件为37℃、5%CO2。待PK15细胞汇合度达95%时,利用0.25%胰酶消化细胞,接种至12孔板(2.5×105个/孔)中,待细胞汇合度达70%时,分别将报告基因载体质粒pGL3-cMiR-0.2K、pGL3-cMiR-0.5K、pGL3-cMiR-0.8K、pGL3-cMiR-1.5K、pGL3-cMiR-1.7K及pGL3-Basic空报告基因载体各1000ng与50 ng的pRL-TK质粒混合,按Lipofec-tamineTM2000说明书共转染PK-15细胞,每组设置3个重复。
1.3.7 报告基因双荧光素酶活性检测
待PK15细胞转染48h后,吸弃培养液并用1×PBS润洗3次,在分别向每孔中加入200 μL 1× PLB细胞裂解液,充分裂解15 min后,转移至干净的离心管中,12 000g离心5min,取上清备用。分别向96孔检测板中加入20 μL上清液,随后立即加入100 μL LARII,检测萤火虫萤光素酶活性值,然后再加入100 μL Stop & Glo Substrate,检测海肾荧光素酶活性值。相对荧光素酶表达量为萤火虫荧光素酶活性/海肾荧光素酶活性的比值。
1.3.8 数据分析
使用SPSS 17. 0软件的单因素方差分析实验数据,所有实验设置3次重复,结果表示为±SD,当P<0.05表示为为差异显著,用*表示。
2 结果与分析
2.1 猪miR-23a-27a-24簇分析
利用UCSC、RNAcentral和NCBI查询猪miR-23a-27a-24簇前体序列,miR-23a-27a-24簇定位于猪2号染色体上(chr2:65,308,116-65,308,525),DNA序列全长408 bp;该基因簇具有3个串联排列的miRNA 前体,可编码3个成熟的miRNA,分别为ssc-miR-23a、ssc-miR-27a、ssc-miR-24-1-5p。
2.2 猪miR-23a-27a-24簇启动子核心区域预测
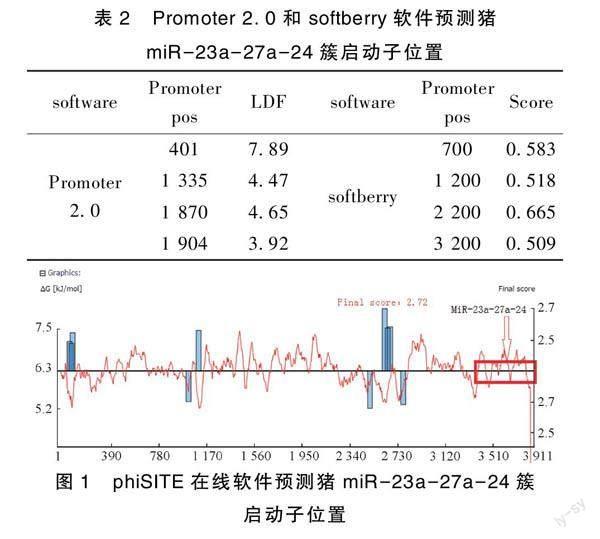
对猪miR-23a-27a-24簇前体序列上游3 500 bp进行启动子预测。结果显示,Promoter 2.0预测到4处启动子结合位点,分别位于miR-23a-27a-24簇上游700 bp、1 200 bp、2 200 bp和3 200 bp处,其Score分别为0.583、0.518、0.665和0.509(表2);softberry预测到4处启动子结合位点,即miR-23a-27a-24簇上游401 bp、1 335 bp、1 870 bp和2 204 bp,LDF分别为7.89、4.47、4.15和3.92(表2)。
phiSITE预测结果如图1所示:在miR-23a-27a-24簇启动子集中分布在上游500~900 bp,其中700 bp附近启动子预测得分最高,为2.72;尽管3个软件对miR-23a-27a-24簇的转录起始位点的预测结果不尽相同,但是3个软件的预测结果均集中在猪miR-23a-27a-24簇上游400 bp~900 bp、1 200 bp~1 400 bp和2 100 bp~2 300 bp区域内,并且在400 bp~900 bp区域的综合得分最高。因此,本研究推测miR-23a-27a-24簇前体序列上游400~900 bp区域可能是其潜在的核心启动子区域。
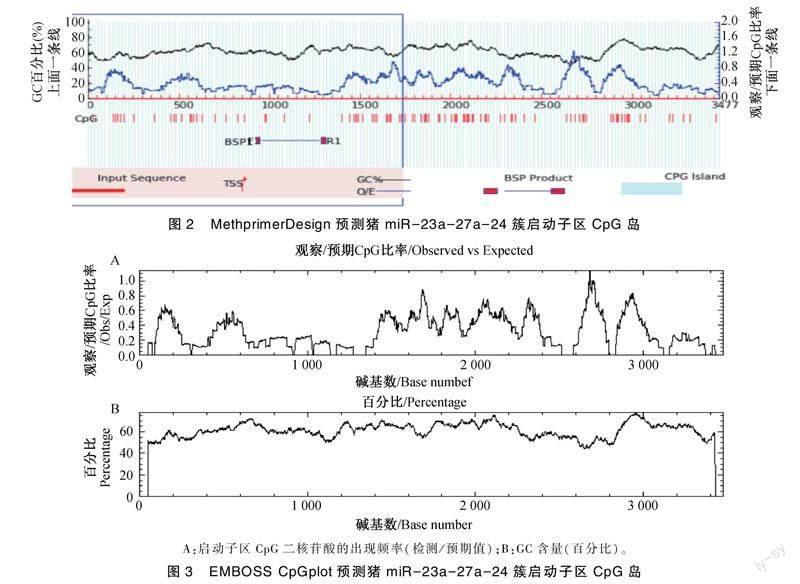
2.3 猪miR-23a-27a-24簇启动子区CpG岛预测
除顺式作用元件外,CpG岛也是真核生物pol Ⅱ型启动子的重要特征之一。本研究利用Methprimer和EMBOSSS在线软件对猪miR-23a-27a-24簇启动子区CpG岛进行预测,在猪miR-23a-27a-24簇前体序列上游3 500 bp序列中均没有发现CpG岛的存在,仅在2 800~2 900 bp区域(即猪miR-23a-27a-24簇上游600~700 bp区域)有一个CpG集中分布区域(图2,图3)。
2.4 猪miR-23a-27a-24簇启动子区克隆及荧光素酶报告载体构建
以猪基因组DNA为模板,成功扩增出miR-23a-27a-24簇5′上游6条续减片中前5条,大小分别为 231 bp、557 bp、884 bp、1 548 bp和1 776 bp,其中最長片段2 137 bp扩增失败。将克隆的miR-23a-27a-24基因上游5条续减片段和pGL3-Basic质粒同时用Xho Ⅰ、Hind Ⅲ进行双酶切,连接、转化DH5α感受态细胞,构建重组荧光素酶报告基因质粒。
经过测序结果验证,所有克隆的片段序列与目的序列完全一致。同时将构建好的重组荧光素酶报告基因质粒采用Xho Ⅰ和Hind Ⅲ进行双酶切,miR-23a-27a-24簇5′上游5条续减片段和pGL3-Basic质粒片断与原始序列位置相吻合,表明质粒构建成功(图4)。
2.5 猪miR-23a-27a-24簇核心启动区域确定
双荧光素酶报告结果显示:pGL3-cMiR启动子在PK-15细胞中均表现出荧光素酶活性,表明构建的miR-23a-27a-24启动子具有明显的转录活性, 且各段续减片段的荧光素酶活性并不相同(图5)。
其中最长片段pGL3-cMiR-1.7K(-1 886 bp/-110 bp)及稍短片段pGL3-cMiR-1.5K(-1 658 bp/-110 bp)沒有表现出最高的活性,其报告基因的活性分别为空载体(pGL3-basic)活性的3.91和3.56倍,相对于报告基因活性最高的片段pGL3-cMiR-0.8K(-994 bp/-110 bp)的活性逐渐减弱,表明猪miR-23a-27a-24簇上游(-997/-1 876 bp)可能存在负向调控因子。pGL3-cMiR-0.8K(-994 bp/ -110 bp)的活性最高,为空载体(pGL3-basic)活性的5.02倍(P<0.05);而pGL3-cMiR-0.2K(-341 bp/-110 bp)的活性只有pGL3-cMiR-0.8K(-994 bp/-110 bp)的45%(P>0.05)。说明miR-23a-27a-24簇上游(-341 bp-994 bp)区域存在强启动子活性,这与生物信息学预测的位置相一致。
2.6 猪miR-23a-27a-24簇核心启动区转录因子结合位点的预测
利用在线软件对猪miR-23a-27a-24簇核心启动子区的转录调控因子进行预测,结果显示:AnimalDB 3.0软件在目标序列中共预测到404个潜在的转录因子,JASPAR软件共预测到139个潜在的转录因子,PROPO软件共预测到59个潜在的转录因子(图6A)。3个软件共同预测到6个相同的转录因子,分别为SP1、VDR、MAZ、YY1、E2F1和ELF1。其中,SP1在目标序列中发现有6处潜在的结合位点、YY1发现3处潜在的结合位点、ELF1发现有2处潜在结合位点,SP1和MAZ的结合位点发生部分重叠(图6A,图6B)。
3 讨论
MiR-23a-27a-24簇是由同一启动子转录生成的3个相互邻近的一簇小分子miRNA[9]。该miRNA簇在脊椎动物中高度保守[4],常被报道与人类疾病相关[10]。但该基因簇在猪中的研究相对匮乏,本研究通过生物信息学和双荧光素酶试验预测并验证猪miR-23a-27a-24簇的核心启动子位置,为猪miR-23a-27a-24簇的转录调控机制的研究奠定基础。
荧光素酶报告基因系统因其敏感性高、操作简单等特点而广泛应用于监测细胞基因表达和调控的研究,核心启动子是调节基因转录活性的关键和核心要素,通过荧光素酶检测报告基因的表达量可直接反映所克隆片段的启动子活性[11]。本研究成功构建了猪miR-23a-27a-24簇上游的报告基因载体,双荧光报告基因系统显示猪miR-23a-27a-24簇上游341 bp至994 bp区域的荧光素酶活性最强。表明猪miR-23a-27a-24簇上游341 bp至994 bp可能是其核心启动子区域,这与生物信息学预测的位置相一致。然而,我们并不排除在该启动子上游含有另外的启动子的可能性。在相似研究中,Lee等[9]发现在人miR-23a-27a-24簇上游403~603 bp区域的荧光素酶活性最高,比本研究在猪miR-23a-27a-24簇上发现的启动子范围要小。这可能是由于物种间碱基序列之间差异造成的。
除了顺式作用元件外,CpG岛也是启动子序列的重要特征[12]。CpG岛作为表观调控的重要组成部分,在基因调控方面起着重要作用[13]。研究表明,CpG岛主要位于基因的启动子区和第1外显子区,CpG岛的GC含量>50%,长度>200 bp[13]。对真核生物而言,CpG岛主要位于管家基因的启动子调控区[14]。本研究在miR-23a-27a-24簇启动子区未检测到CpG岛,表明miR-23a-27a-24簇可能属于组织特异性基因。
基因表达的调节是一种协调有序的过程,基因表达调控的重要环节是转录水平的调控[15]。据报道,基因上游的启动子调控元件表达水平及结合区域可影响重组载体的转录活性[16],转录因子作为一种协助真核生物基因转录的调控元件,通过与目的基因启动子直接结合来调控基因的表达[17]。本研究中对miR-23a-27a-24簇的核心启动子区域进行转录因子预测,发现6个潜在的转录因子结合位点(SP1、VDR、MAZ、YY1、E2F1和ELF1)。但值得关注的是,转录因子SP1和YY1在猪miR-23a-27a-24簇核心启动子区域上存在多处结合位点。推测SP1和YY1可能是猪miR-23a-27a-24簇核心启动区的关键转录调控元件,但是本研究所发现的其他转录因子,如:VDR、MAZ、E2F1和ELF1对猪miR-23a-27a-24簇的调控作用也是不容忽视的。
阴阳因子1(Yin-Yang-1,YY1)是一种普遍表达的转录因子,通过占据靶基因活性增强子或启动子区域激活或抑制特定基因的表达进而调控其正常生理功能[18]。YY1可参与雌性动物卵母细胞成熟以及颗粒细胞的扩张过程[19]。在猪颗粒细胞中,YY1可激活TAC3基因转录活性[20]。同时,Dong等[21]发现YY1参与了克隆猪胚胎XIST和XCI的表达调控,抑制YY1的表达可以提高克隆猪胚胎的发育率。特异性蛋白(specificity protein 1,SP1)是一种进化上高度保守的转录因子,参与细胞的增殖、凋亡和分化等过程[22]。原始卵泡(PF)池对于动物生殖至关重要,SP1通过调节哺乳动物卵巢中的颗粒前细胞发育来控制卵巢储备的建立[23]。同时,研究发现SP1可通过调节卵巢中的原始到初级卵泡过渡[24].在湖羊中的研究表明,湖羊SP1基因可抑制人卵巢颗粒细胞 (KGN) 增殖、诱导其凋亡过程[25]。对此,本研究推测SP1和YY1基因作为真核生物基因启动子上的调控因子,可能与哺乳动物卵泡发育过程相关。但是本研究中,转录因子YY1和SP1是否与猪miR-23a-27a-24簇核心启动子区结合并调控其表达及影响猪卵泡的发育过程,还有待于今后通过基因突变、ChIP以及EMSA等技术,重点对猪miR-23a-27a-24簇的转录调控机制及其在猪特定生物学过程中调控作用进行研究。
4 结论
本研究构建并确定了猪miR-23a-27a-24簇核心启动子区域,并预测了SP1和YY1可能参与猪miR-23a-27a-24簇的转录调控过程。本研究结果可为猪miR-23a-27a-24簇的转录调控机制及其对猪特定生物学过程的研究奠定基础。
参考文献(References)
[1]崔梦莹. MicroRNA-23a-27a-24-2簇在肝细胞癌中的功能及调控机制研究[D]. 吉林:吉林大学, 2020.
[2]TANZER A, STADLER P F. Molecular evolution of a microRNA cluster[J]. J Mol Biol, 2004,339(2):327- 335.
[3]KIM Y K, YU J, HAN T S, et al. Functional links between clustered microRNAs: suppression of cell-cycle inhibitors by microRNA clusters in gastric cancer[J]. Nucleic Acids Res, 2009,37(5):1672- 1681.
[4]CHHABRA R, DUBEY R, SAINI N. Cooperative and individualistic functions of the microRNAs in the miR-23a~27a~24-2 cluster and its implication in human diseases[J]. Mol Cancer, 2010,9:232.
[5]聂明月,杨晓葵. miR-23a和miR-27a在卵巢中的生理与病理作用[J]. 国际生殖健康/计划生育杂志, 2014, 33(5): 367-369.
NIE M Y,YANG X K. Physiological and pathological roles of miR-23a and miR-27a in the ovary[J]. International Journal of Reproductive Health/Family Planning, 2014, 33(5): 367-369.
[6]KIM Y J, KU S Y, KIM Y Y, et al. MicroRNAs transfected into granulosa cells may regulate oocyte meiotic competence during in vitro maturation of mouse follicles[J]. Hum Reprod, 2013,28(11):3050- 3061.
[7]NIE M, YU S, PENG S, et al. miR-23a and miR-27a promote human granulosa cell apoptosis by targeting SMAD5. Biol Reprod, 2015,93(4):98.
[8]王思琪,张卓凡,杨柳,等. miR-23a-27a-24簇在猪卵巢颗粒细胞凋亡中的作用[J]. 畜牧与兽医, 2022, 54(4): 1-9.
WANG S Q,ZHANG Z F,YANG L,et al.The role of the miR-23a-27a-24cluster in the apoptosis of porcine ovarian granulosa cells[J].Animal Husbandry & Veterinary Medicine,2022, 54(4): 1-9.
[9]LEE Y, KIM M, HAN J, et al. MicroRNA genes are transcribed by RNA polymerase II[J]. EMBO J, 2004,23(20):4051-4060.
[10]LI X, LIU X, XU W, et al. c-MYC-regulated miR-23a/24-2/27a cluster promotes mammary carcinoma cell invasion and hepatic metastasis by targeting Sprouty2[J]. J Biol Chem, 2013,288(25):18121-18133. .
[11]ALAM J, COOK J L. Reporter genes: application to the study of mammalian gene transcription[J]. Anal Biochem, 1990,188(2):245-254.
[12]ZHOU X, RUAN J, WANG G, et al. Characterization and identification of microRNA core promoters in four model species[J]. PLoS Comput Biol, 2007,3(3):e37.
[13]陸杏蓉,段安琴,马小娅,等. 水牛HSD17B1基因启动子荧光素酶报告基因载体构建与分析[J]. 中国畜牧兽医, 2021, 48(10): 3512-3521.
LU X R,DUAN A Q,MA X Y,et al.Construction and analysis of the luciferase reporter gene vector of HSD17B1Gene promoter in buffalo[J].China Animal Husbandry & Veterinary Medicine,2021, 48(10): 3512-3521.
[14]薛梅,李旭,陈葳. 人UCA1基因转录调控的生物信息学分析与鉴定[J]. 南方医科大学学报, 2013, 33(11): 1596-1599.
XUE M,LI X,CHEN W.Bioinformatics analysis and identification of transcriptional regulation of human UCA1gene[J].Journal of Southern Medical University,2013, 33(11): 1596-1599.
[15]马冬梅,韩林强,白俊杰. 大口黑鲈GHRH基因启动子区域序列分析及其活性检测[J]. 海洋渔业, 2016, 38(4): 383-390.
MA D M,HAN L Q,BAI J J,et al. Sequence and activity analysis of GHRHpromoter region from Micropterus salmoides[J].Marine Fisheries, 2016, 38(4): 383-390.
[16]周琳,高春芳,王皓,等. 人B细胞活化因子基因启动子活性研究[C].中国免疫学会第五届全国代表大会暨学术会议论文摘要, 2006: 293.
[17]FITZ E, WANKA F, SEIBOTH B. The promoter toolbox for recombinant gene expression in trichoderma reesei[J]. Front Bioeng Biotechnol, 2018,6:135.
[18]ATCHISON M, BASU A, ZAPRAZNA K, et al. Mechanisms of Yin Yang 1 in oncogenesis: the importance of indirect effects[J]. Crit Rev Oncog, 2011,16(3-4):143-161.
[19]MARK B,JOHN S. Targeted inhibition of the transcription factor YY1 in an embryonal carcinoma cell line results in retarded cell growth, elevated levels of p53 but no increase in apoptotic cell death[J]. Eur J Cell Biol, 2005,84(5):543-553.
[20]李忠慧,袁晓龙,邢燕,等. 猪速激肽3基因启动子活性及其表达调控的初步研究[J]. 畜牧与兽医, 2018, 50(6): 12-18.
LI Z H,YUAN X L,XING Y,et al.Analysis of the promoter activity and its regulation of TAC3 gene expression in pig[J].Animal Husbandry & Veterinary Medicine,2018, 50(6): 12-18.
[21]DONG Y Z, WU X, PENG X T, et al. Knockdown of YY1 inhibits XIST expression and enhances cloned pig embryo development[J]. Mdpi Ag, 2022, 23(23): 14572.
[22]VELLINGIRI B, IYER M, DEVI S, et al. Understanding the role of the transcription factor sp1 in ovarian cancer: from theory to practice[J]. Int J Mol Sci, 2020,21(3):1153.
[23]HAN C,BINGYING L,HUARONG W, et al. SP1 governs primordial folliculogenesis by regulating pregranulosa cell development in mice[J]. Oxford University Press(oup), 2019, 12(3): 230-244.
[24]ZHOU J, LIN L, CAI H, et al. SP1 impacts the primordial to primary follicle transition by regulating cholesterol metabolism in granulosa cells[J]. FASEB J, 2023,37(2):e22767.
[25]姚一龍,李隐侠,安外尔·热合曼,等. 湖羊Sp1基因CDS区克隆及其对颗粒细胞增殖和凋亡的影响[J]. 畜牧兽医学报, 2017, 48(11): 2098-2106.
YAO Y L,LI Y X,REHEMAN·Anwaier,et al.Cloning of Sp1Gene CDSRegion of Hu sheep and its effect on proliferation and apoptosis of granulosa cells[J].Chinese Journal of Animal and Veterinary Sciences,2017, 48(11): 2098-2106.
(责任编辑:编辑唐慧)
收稿日期:中文收稿日期2022-11-16
基金项目:国家自然科学基金项目(31460586,31960645),新疆生产建设兵团科技创新人才计划项目(2020CB018),石河子大学动植物育种专项项目(XJ2019000401)
作者简介:李清春(1995—),男,硕士研究生,专业方向为动物遗传育种与繁殖。
*通信作者:黄涛(1978—),男,教授,从事猪分子育种研究,e-mail:taohuang100@sina.com。
