探究影响生物代谢物注释准确性的因素
2023-12-30卢雨欣
卢雨欣
渭南师范学院,陕西 渭南 714000
在生物学领域,代谢组学是研究生物体的重要学科,其中,代谢物注释扮演着关键的角色。代谢物通常在生物体内的生化反应中被消耗或生成,微小的环境变化都可能引起生物体内生化过程的变化,从而导致代谢物的浓度和种类发生实时变化[1]。因此,代谢物的变化直接反映了生物体的生化和功能状态。在代谢组学中,代谢物注释对研究生物体具有重要意义。由于代谢物的组成非常复杂,一份样品中可能包含大量不同种类的代谢物,这些代谢物中包含化学结构和浓度各异的多种化学物,因此代谢物注释的精确性显得至关重要。
代谢物的注释研究在研究生物体的各种调节机制[2]、微生物和植物分析[3]、基因功能的阐明[4]、疾病诊断[5]和药物毒性[6]等方面发挥了重要的作用。而影响代谢物注释的因素有很多,例如加合物列表的大小、p 值、皮尔逊系数,分子式列表的大小及保留时间等。因此,为了探究各项参数对代谢物注释准确性的影响进行本次试验,以求为生物代谢组学的相关领域提供参考。
1 试验方法
本次试验主要采用伯明翰大学自主研发的代谢物注释程序BEAMS 探究各项参数对代谢物注释准确性的影响。BEAMS 是依据代谢组学标准倡议报告标准的第三级进行的注释。
本次试验数据为某项目提供了三组不同的代谢物数据,分别通过加合物列表、P 值、皮尔逊系数、分子式列表及保留时间进行对比分析试验。BEAMS 的运行如图1 所示。
图1 BEAMS 的运行界面图
数据分析过程主要包括四个阶段,分别为峰值检测、峰值注释、分子式注释及统计分析。首先是对代谢物进行分组。根据保留时间(RT)对离子峰进行分组,提取这些峰的离子色谱图(EIC),再利用皮尔逊相关性对它们进行二次分组,随后根据不同的质荷比(m/z)使用不同的加合物列表、同位素列表或低聚物列表进行峰注释。如果组内每对峰的m/z 之差值较为合理,则认为这两个离子来自同一个代谢物[7]。化合物的准确分子质量可以通过分子式数据库的计算和搜索来获得并记录分子式,并根据质谱中的m/z 和加成离子形式计算出所有可能的分子式后进行过滤,得到最终代谢物注释结果[8]。本研究使用三个不同的代谢物数据集,分别改变加合物列表的大小、P 值、皮尔逊系数、分子式列表的大小及保留时间等,探究各项参数对于代谢物注释准确性的影响。试验步骤如图2 所示。
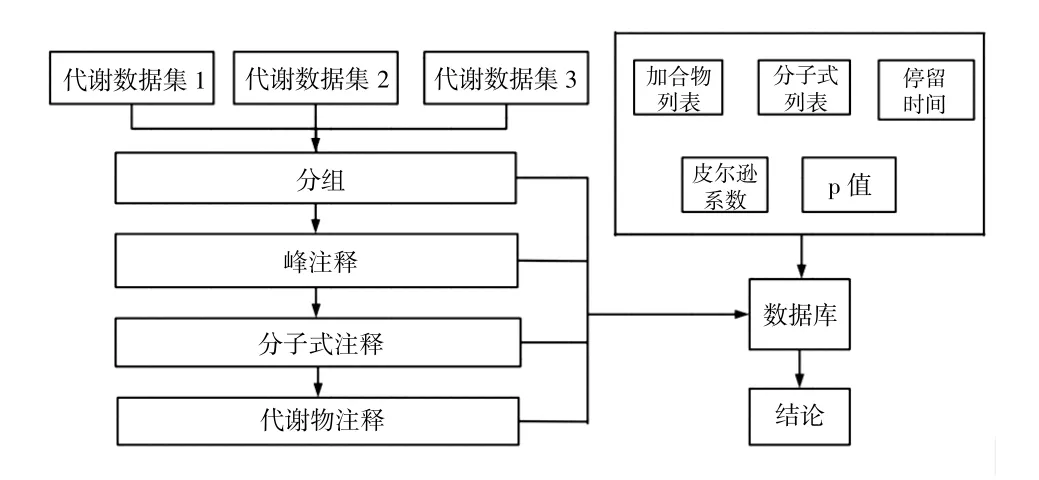
图2 试验流程图
2 试验结果
为了探究各项参数对代谢物注释准确性的影响,本实验采用了三个不同的代谢物数据集、两个不同的加合物列表、三个不同的分子式列表,并通过改变P 值、保留时间(RT)和皮尔逊系数进行了试验。其中,代谢物数据集1 是通过超高效液相色谱-质谱分析获得“人工”数据集,包含相对较少的数据;代谢物数据集2 和代谢物数据集3 则是“真实世界”中的生物样本代谢物数据集,包含较为丰富的数据。加合物列表1 包含较少的数据,加合物列表2 则包含较多的数据。分子式列表1、分子式列表2 和分子式列表3 分别包含较少、中等和较多的数据。图3展示了加合物列表、代谢物数据集文件和分子式列表文件所包含数据的数量。
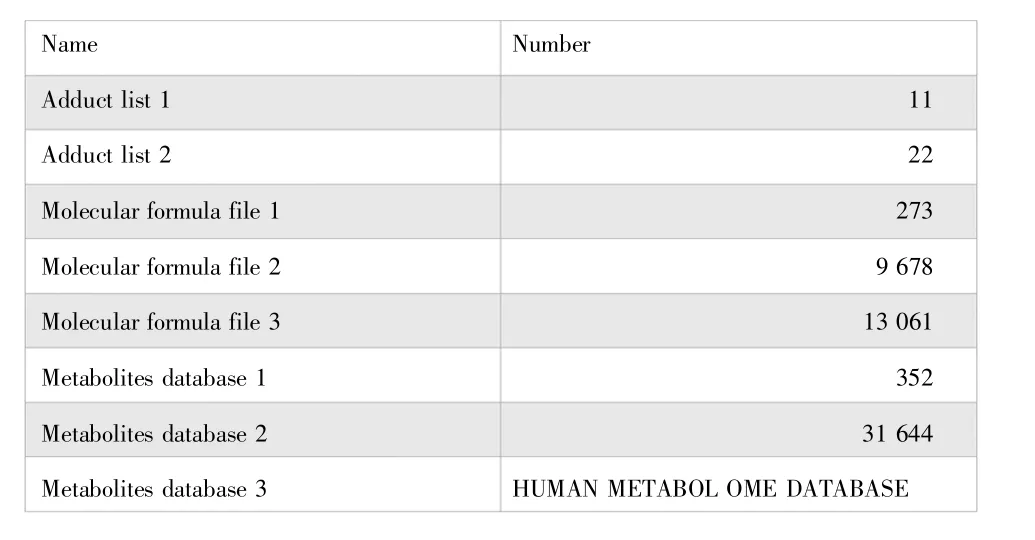
图3 加合物列表、分子式列表及代谢物数据集中包含的数据数量图
加合物列表1 和分子式列表1 只定义代谢物数据集1 中的已知代谢物,而加合物列表2、分子式列表2 与分子式列表3 则包含更多加合物与分子式。本研究将使用不同的数据库和参数进行试验,统计每个特征所包含的分子式占总数量的百分比,以确定代谢物注释的影响因素。
2.1 使用不同数据库对代谢物注释的影响
使用相同的P 值、皮尔逊系数和RT 更改加合物列表、分子式列表和代谢物数据集的结果如图4所示。当使用加合物列表1、分子式列表1 和代谢物数据集1 注释代谢物时,结果显示该组有733 个代谢物的特征包含一个分子式(理想情况),占代谢物特征的65%。使用加合物列表2、分子式列表1 和代谢物数据集1 时,有58.5%的代谢物特征包含一个分子式。除此之外,应用其他数据库时,只有18%-23%的代谢物特征报告中包含一个分子式。因此,使用分子式列表1 和代谢物数据集1 时,代谢物注释的准确性明显高于使用其他分子式列表和代谢物数据集,这证明大型分子式列表会造成多个假阳性,对代谢物注释的准确性有负面影响。
除此之外,在相同条件下,比较使用加合物列表1 和加合物列表2 的数据后发现,使用加合物列表1的结果普遍优于使用加合物列表2 的结果。在相同条件下,使用加合物列表2 时,只有占特征组总数9%~12%的特征组包含一个分子式,相较于使用加合物列表1 的结果低6%~14%。此外,使用加合物列表2 时,具有两个以上特征的组数明显增加,表明假阳性峰的数量明显增加,从而导致代谢物注释的准确性大大降低。然而,仅改变加合物列表时,其变化相对于改变其他条件较小。当加合物列表1 和分子式列表1 保持不变,代谢物数据集1 改为代谢物数据集2 时,含有一个分子式的特征组数量从733 个减少到260 个,占比从65%减少到22%。同样,当分子式列表1 和代谢物数据集1 不变,而加合物列表改变时,包含一个分子式的特征群数量便从733 个下降到662 个,占比从65%下降到59%。这表明分子式列表和代谢物数据集对代谢物注释准确性的影响相较于加合物列表更为显著。由于较大的分子式列表和代谢物数据集包含更多的分子式和更多种代谢物,它们能够注释代谢物的衍生物。因此,当使用加合物列表2、分子式列表3 和代谢物数据集3 时,结果最差,仅有113 个代谢物特征包含一个分子式,有1 010 个代谢物特征包含一个以上的分子式。
2.2 使用不同参数对代谢物注释的影响
在考察了分子式列表、加合物列表和代谢物数据集对代谢物注释的影响后,本研究转向使用代谢物数据集2,以研究不同参数对代谢物注释准确性的影响。与代谢物数据集1 不同,代谢物数据集2中的代谢物是未知的,因此选择了较大的分子式列表3。这是因为小型数据库中可能缺乏某些分子式或代谢物种类。在本次试验中,通过调整最大保留时间差、皮尔逊系数、加合物列表和P 值,分别研究了各种参数对代谢物注释的影响。
最大保留时间差、皮尔逊相关系数(Pearson correlation)、加合物列表和P 值都与特征组有关。理论上,相同代谢物的保留时间(RT)相同,但由于误差和其他原因,RT 会有微小的偏差,所以一起用最大保留时间差来调整时间差,并对峰进行分组。但仅根据保留时间进行分组并无法满足相关要求,还需要对峰形或峰丰度进行相关分析。如果两个峰之间的峰形或峰丰度的相关性高于预定的阈值,那么这两个特征就会被认定属于同一代谢物[9]。这种相似性通常用两个峰提取离子色谱(EIC)的皮尔逊系数衡量[10]。皮尔逊系数是用来计算两个变量之间的关系强度的[11]。其数值在负1 和1 之间,其中1 是正相关,负1 是负相关。一般来说,皮尔逊系数低于0.8被视为无效。P 值代表一个显著性水平,非常重要,如果P 值不显著,无论皮尔逊系数有多高,都可能是偶然结果。一般来说,P 值低于0.05 则表示成效显著。使用不同参数对代谢物注释的影响显示在以下图表中(图5,6)。
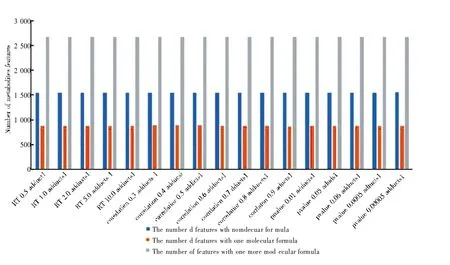
图5 数据集2 中分配给每个代谢物特征的分子式数量(使用加合物列表1)
如图5 和图6 所示,无论使用何种加合物列表、无论什么类型的参数发生变化,其结果都是相似的。例如,在图5 中,当最大RT 差值为2.0 和5.0 时,具有一个分子式的特征数量都是880 个;当P 值为0.01 和0.0005 时,具有一个分子式的特征数量也一样。该试验把皮尔逊系数分别调整为0.7、0.8 和0.9。这是因为0.7 和0.9 的值分别代表被认为无效和被认为有效。当我们使用这三个值时,结果仍然是一样的。虽然参数增加了五到十倍,但含有一个分子式的代谢物特征只是略有增加或保持稳定。无论最大RT 差值、P 值和皮尔逊系数如何增加,含有一个分子式的特征数量都是17%。不含分子式的特征组数量和含一个以上分子式的特征组数量也很稳定,比例保持在30%和52%。
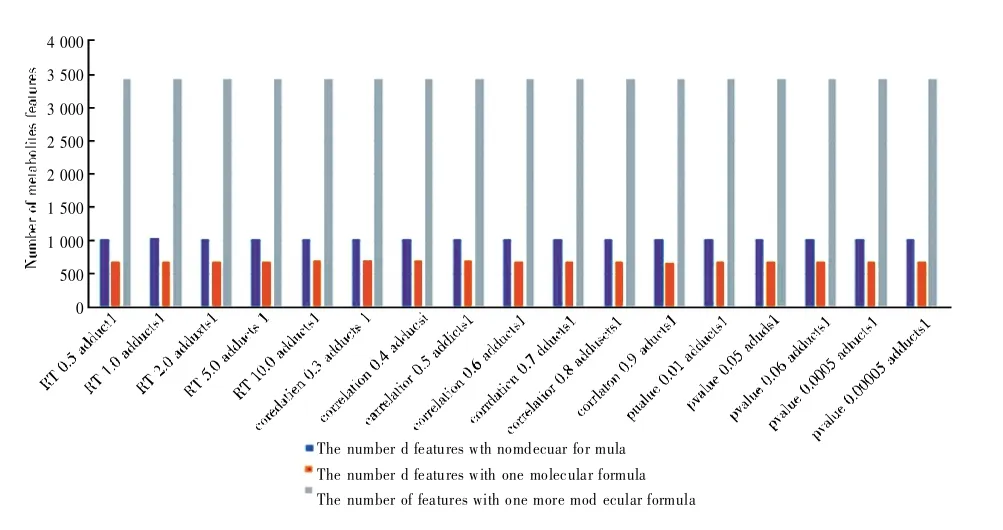
图6 数据集2 中分配给每个代谢物特征的分子式数量(使用加合物列表2)
3 结论与建议
试验数据表明,加合物列表、分子式列表和代谢物数据集文件对代谢物注释结果的影响极为显著。与此相反,最大RT 差异、皮尔逊系数和P 值对结果的影响几乎可以忽略不计。当使用含有大量数据的加合物列表、分子式列表和代谢物数据集时,对代谢物注释产生了负面影响,导致结果中含有多种代谢物的组数明显增加,这表明出现了大量假阳性峰。尽管每个代谢物都有大量离子,但只有其中一小部分能够通过数据库匹配得以识别,其他部分是同一组代谢物的衍生物。因此,使用大型的加合物列表、分子式列表和代谢组学数据集时,可能会注释出衍生物,从而在一个组中产生多种代谢物。
假阳性峰的产生在分子式注释和代谢物名称注释的过程中产生很多错误。合适的分子式列表和代谢物数据集包括适当的分子式和代谢物名称,因此当选取合适的分子式列表和代谢物数据集时,绝大部分假阳性峰无法被注释出来。而在较大的分子式列表和代谢物数据集中,假阳性峰可以被注释,导致含有两种以上代谢物的组增加,没有代谢物的组和只含有一种代谢物的组减少。形成假阳性峰的原因有三个:1. 两个具有相同电荷状态的前体离子在一个小的RT 窗口内的质量几乎相同;2.每个代谢物都包含大量离子,但只有其中一小部分可以通过数据库匹配来识别,另一部分是同一组代谢物的衍生物,如果不识别在电离过程中形成的碎片、同位素和被视为单同位素离子的加合物,可能会导致基于质量的方法在检测许多分子时出现错误,一些高强度的单同位素峰可能存在许多后续的同位素峰,其强度可能会超过规定的阈值;3.在收集中心点数据时,主峰周围可能会产生一些峰,如果强度阈值的水平低于新生峰,可能会造成假阳性的问题。
解决假阳性峰的问题是非靶向代谢组学的重点和难点,但目前还没有评估代谢物鉴定假发现率(FDR)的指标。目前的解决方案是:1. 控制RT。如果RT 很大,假阳性峰的数量会增加,但应该注意,太小的RT 会导致真正的候选峰产生损失。2. 根据实验结果选择最佳强度阈值有助于减少假阳性,重要的是,不会丢失真实的实验数据和结果。3.可以用一些软件来减少假阳性,如使用非靶向代谢组学的自动数据分析管道(ADAP)来建立EIC。ADAP 的峰值检测算法可以通过引入新的信噪比估计方法和其他一些过滤步骤来检测EIC 中的假峰,但它通常运用于GC-MS。
在这个实验中,分别采用了测试数据集和真实的生物数据集来测试各项参数对于代谢物注释准确性的影响。在实验过程中,使用了控制变量的方法对每个变量进行多次测试。例如,在测试1 中,使用加合物列表1、分子式列表1 和代谢数据集1 作为控制组,并与其他使用较大加合物列表、分子式列表和代谢物数据集的组进行比较,以使结果清晰易懂。在试验2 中,为了探究改变P 值的效果,对多个实验的P 值进行了调整和观察,从而避免误差,保证实验结果的准确性。但在测试参数对代谢物注释准确性的影响时,该实验只使用了一个数据集,可能会造成一些意外的误差。
在代谢组学研究中,代谢物的注释至关重要,错误率过高就需要花费大量的时间和金钱来识别错误。因此,在进一步研究中,建议使用更多数据集来测试各参数对代谢物注释准确性的影响。在之后的试验中可以使用更多的标准数据集进行测试,以避免因数据集问题产生的误差。本实验使用加合物列表1、分子式列表1 和代谢物数据集1 时,结果仍然存在一些特征组没有分子式或一个以上的分子式。这可能是因为在组特征阶段存在一些误差,如一些代谢物没有被分组,可以选择合适的参数和数据库进行注释,直到所有特征组都有一个分子式,然后在相同参数下测试使用不同加合物列表、分子式列表文件和代谢物数据集的效果并观察其结果,从而进行进一步的试验。
