棉花β-tubulin基因家族的鉴定及其在纤维发育中的表达
2023-12-28党媛玥马建江杨书贤宋吉坤贾冰冯盼陈全家于霁雯
党媛玥,马建江,杨书贤,宋吉坤,贾冰,冯盼,陈全家,于霁雯
棉花β-tubulin基因家族的鉴定及其在纤维发育中的表达
党媛玥1,2,马建江2,杨书贤2,宋吉坤1,2,贾冰1,2,冯盼2,陈全家1,于霁雯1,2
1新疆农业大学农学院/棉花教育部工程研究中心,乌鲁木齐 830052;2中国农业科学院棉花研究所/棉花生物育种及产业技术国家工程研究中心,河南安阳 455000
【目的】β-微管蛋白是棉纤维细胞形态建成的基本结构单位,在纤维发育过程中具有重要作用。通过鉴定棉花β-tubulin基因家族成员,并进行生物信息学和表达模式分析,为深入探究β-tubulin基因在棉花纤维发育中的作用提供理论依据。【方法】利用BLAST方法,在4个棉种基因组中鉴定β-tubulin基因家族成员,并结合ProtParam tool分析理化性质、MEGA7.0构建系统进化树、Mapchart2.2绘制染色体定位图、MEME分析保守基序、PlantCARE分析启动子顺式作用元件;根据39个材料发育纤维转录组数据分析陆地棉β-tubulin基因家族的表达水平,并利用Spearman相关性分析鉴定影响纤维品质性状形成的β-tubulin基因。【结果】在陆地棉(,AD1)、海岛棉(,AD2)、亚洲棉(,A2)和雷蒙德氏棉(,D5)基因组中分别鉴定到36、37、19和18个β-tubulin基因,且在四倍体棉种中的数目约是二倍体棉种数目的二倍,系统进化将其分为ClusterⅠ—ClusterⅤ共5个亚组。系统进化与共线性分析发现,与陆地棉β-tubulin基因家族相比,海岛棉与二倍体亚洲棉和雷蒙德氏棉亲缘关系更近。保守结构域均具有典型的Tubulin和Tubulin-C。理化性质分析表明,该家族基因编码的氨基酸数目为421—508,等电点为4.68—5.09。启动子顺式作用元件分析获得生长发育响应相关元件、激素响应相关元件和胁迫响应相关元件等,表明β-tubulin基因参与细胞的生长调节。利用TM-1转录组数据对36个陆地棉β-tubulin基因表达模式进行聚类分析,发现有42%基因在纤维中优势表达;此外,利用海陆群体动态纤维转录组数据筛选到1、6和11个β-tubulin基因分别与纤维马克隆值、比强度和纤维长度显著相关,其中4个基因同时影响纤维长度和比强度性状。【结论】4个棉种中共鉴定出110个β-tubulin基因家族成员,氨基酸理化性质和序列高度保守而启动子序列调控元件多样;筛选出陆地棉纤维优势表达的β-tubulin基因家族成员,并发掘潜在调控纤维发育的候选基因。
棉花;β-tubulin基因家族;生物信息学;表达分析;纤维发育
0 引言
【研究意义】棉花是全球最重要的天然纤维作物和主要的经济作物。随着人们生活水平的提高,对绿色健康的高品质纺织品的需求日益增加[1]。然而,近年来,中国棉花总产量随着植棉面积的减少呈现逐年下降趋势,其中,优质棉花品种的缺乏(纤维长度≥30 mm、纤维强度≥30 cN·tex-1)制约中国高品质棉纺产品的生产,为此,中国每年有高达200万t的优质棉依赖进口[2]。因此,改良棉纤维品质成为现阶段棉花育种的主要目标。【前人研究进展】微管是细胞骨架的主要成分,主要由微管蛋白构成[3]。在棉纤维细胞发育过程中微管随着纤维细胞的发育呈现不同的排列方式,因此,纤维细胞发育与微管密切相关[4]。微管作为细胞骨架的核心成分,在细胞迁移、有丝分裂、机械应激、细胞极性、细胞内运输、细胞分裂和细胞形态发生中起着关键作用[5-7]。组成微管的各类蛋白质统称为微管蛋白,现阶段研究证实,在真核生物中至少存在α、β、γ、δ、ε、ζ和η-微管蛋白[8],而在微管形成中起主要作用的是α微管蛋白(α-tubulin,TubA)和β-微管蛋白(β-tubulin,TubB)2种[9-10]。微管蛋白具有高度保守性,含有Tubulin与Tubulin-C 2个保守结构域[11],多数α-微管蛋白和β-微管蛋白的氨基酸序列同源性可达88%,这种高度保守的情况在植物微管蛋白中同样存在[12]。尽管微管蛋白家族在序列上高度保守,但不同微管蛋白成员在功能上可能存在较大差异。随着对拟南芥等模式植物微管蛋白研究的不断深入,发现植物基因组中常常有多个微管蛋白基因,而基因间的表达模式也因受不同生长信号调控存在一定差异,部分基因在特定组织和器官中特异表达或优势表达对特定组织的生长和发育具有重要作用[13-14]。当前,β-tubulin基因家族成员在哺乳动物、昆虫和微生物中研究较为广泛。在拟南芥(L)[15]、亚麻(L.)[16]、柳树(L.)[17]、慈竹(L.)[18]和龙眼(L.)[19]等植物部分基因家族成员中也得到了鉴定与分析。研究表明,β-tubulin基因家族成员在一些特定的植物组织中高表达以调控植物的生长发育。如,Li等[20]研究证明在棉铃发育早期优先在纤维中高表达,在胚珠中低表达,在棉花其他组织中表达量极低,说明可能在纤维发育时期特异性表达;Qin等[21]探索长纤维和短纤维棉材料之间的基因表达差异时发现,53个差异表达基因(DEGs)中有12个纤维发育的良好候选基因,包括2个β-tubulin基因,其中,在纤维细胞发育起始时期表达丰度高,而在纤维细胞伸长时期高度表达;棉花是纤维特异性较强的纤维发育后期基因,其在拟南芥中超表达抑制了根的伸长,在棉花中超量表达抑制了纤维的伸长,而反义抑制则促进纤维伸长[22];毛竹中的2个微管蛋白基因和经体外表达具有生物活性并能够促进细胞生长[23];在亚麻中,通过对β-tubulin基因的表达分析,发现一些β-tubulin基因可使细胞伸长,且细胞伸长早于细胞壁沉积和增厚[16]。【本研究切入点】棉纤维作为重要的经济产物,在人们日常生活中发挥着重要作用。β-微管蛋白是棉纤维细胞骨架形成的重要元件,而β-tubulin基因在4个棉种中的全基因组鉴定及在纤维中的表达分析还未见报道。【拟解决的关键问题】本研究通过对棉花β-tubulin基因家族系统分析及棉纤维发育中β-tubulin基因的表达水平与表型性状相关性分析,发掘参与棉纤维发育的重要β-tubulin基因,为进一步研究棉花β-tubulin基因在棉纤维发育中的重要作用奠定基础。
1 材料与方法
1.1 β-tubulin基因家族成员的鉴定及系统进化分析
从Cottongen(https://www.cottongen.org/)网站下载亚洲棉(CRI_V1.0)[24]、雷蒙德氏棉(JGI_v2.1)[25]、陆地棉TM-1(ZJU_v2.1)和海岛棉H7124(ZJU_v1.1)[26]的基因组数据库。从TAIR网站(http://www.arabidopsis. org)下载拟南芥基因组数据库。以拟南芥的β-tubulin的氨基酸序列为参考系列,4个棉种的氨基酸序列为库,利用本地blast(E-value为1e-5)比对得到4个棉种β-tubulin的待选基因。再利用Batch-CDD程序(https://www.ncbi.nlm.nih.gov/Structure/bwrpsb/bwrpsb.cgi)检索基因中是否存在β-tubulin蛋白保守结构域,利用在线SMART网站库[27](http://smart.embl- heidelberg.de/)对结构域进一步确定。用Clustal X2.0[28]对设定阈值以内的β-tubulin基因氨基酸序列进行多重序列比对,依据邻近法(NG),利用MEGA7.0[29]构建进化树,再利用网站EvolView(https://evolgenius. info//evolview-v2)对结果进行可视化。
1.2 β-tubulin基因家族成员的理化性质分析
利用在线软件ProtParam tool[30](https://web. expasy.org/)预测每个β-tubulin候选基因的蛋白长度、分子量和等电点。利用在线网站WoLF PSORT(https:// wolfpsort.hgc.jp/)预测亚细胞定位。
1.3 染色体定位及共线性分析
根据染色体注释文件GFF3调取基因的物理位置和染色体信息,利用Mapchart2.2[31]软件绘制染色体定位图。共线性分析首先利用TBtools[32]对β-tubulin基因家族的氨基酸序列进行比对;比对上的区域占较长氨基酸序列80%以上,对比区域的相似度大于80%。4种棉花的片段重复基因对用于Circos作图[33]。
1.4 基因结构与保守基序分析
根据下载的CDS序列与DNA全长序列,使用TBtools软件绘制外显子/内含子结构图。利用在线软件MEME[34](http://meme-suite.org/)分析每个蛋白序列的motif。
1.5 顺式作用元件分析
根据下载的基因组数据,利用TBtools提取基因上游的2 000 bp序列进行顺式作用元件分析。将提取的序列提交至在线网站PlantCARE[35](http:// bioinformatics.psb.ugent.be/webtools/plantcare/html/)进行启动子顺式作用元件分析。并利用TBtools将其可视化处理。
1.6 表达模式分析
从浙江大学棉花数据库(http://cotton.zju.edu. cn/index.htm)下载陆地棉不同发育时期胚珠、纤维和不同组织的转录组数据[26],对数据进行log2(FPKM+ 1)标准化处理,并使用TBtools对其进行可视化。
2 结果
2.1 棉花β-tubulin基因家族成员的鉴定及系统进化树的构建
以拟南芥β-tubulin基因家族的氨基酸序列为参考,运用本地blast,以下载的4个棉种基因组数据为库进行BlastP搜索。将得到的候选序列提交至NCBI Batch-CDD数据库,进一步分析确认候选基因的氨基酸保守结构域。最终在4个棉种中共鉴定得到110个β-tubulin基因,即,陆地棉36个、海岛棉37个、亚洲棉19个、雷蒙德氏棉18个。
通过对4个棉种(陆地棉、海岛棉、亚洲棉和雷蒙德氏棉)及拟南芥的β-tubulin基因家族氨基酸序列进行比对,构建系统发育树(图1)。结果表明,β-tubulin基因家族被分为5个亚组,其中ClusterⅠ成员最多,包含64个(53%)家族成员。
2.2 棉花β-tubulin基因家族成员的理化性质分析
将鉴定得到的β-tubulin基因进行理化性质分析(表1),蛋白序列长度最大值为508 aa(),且与其余成员长度相差较大,最小值为421 aa(),分子量在47.37()—57.42 kDa(),理论等电点为4.68— 5.09。亚细胞定位预测显示,19个β-tubulin基因定位于细胞质,其中,陆地棉7个(、、、、、和)、海岛棉6个(、、、、和)、亚洲棉3个(、和)、雷蒙德氏棉3个(、和);3个β-tubulin基因定位于叶绿体,其中,陆地棉1个()、海岛棉2个(和);2个β-tubulin基因定位于内质网,其中,陆地棉1个()、海岛棉1个()。其余β-tubulin基因均定位于细胞核。

续表1 Continued table 1
2.3 棉花β-tubulin基因家族染色体定位及共线性分析
将β-tubulin基因家族进行染色体定位分析。发现亚洲棉19个分布在Chr.01、Chr.03、Chr.05、Chr.06、Chr.07、Chr.08、Chr.09、Chr.11和Chr.13染色体上,其中,在Chr.01染色体上存在最多,5个(图2-A);雷蒙德氏棉18个家族成员分布在除Chr.08、Chr.11和Chr.12外的其余10条染色体上,其中,在Chr.03染色体上存在最多,4个(图2-B)。另外,陆地棉37个和海岛棉36个均定位到除A02、A04、A07、A10、A12、D04、D10和D12外的18个染色体上,在陆地棉和海岛棉的A03染色体均含有最多,5个(图2-C—D)。通过比较β-tubulin基因家族成员在陆地棉和海岛棉染色体上的分布,发现除海岛棉外,其余海岛棉和陆地棉β-tubulin基因家族成员在染色体的物理位置分布大体相近,表明β-tubulin基因家族在不同棉种之间的进化有相似性。
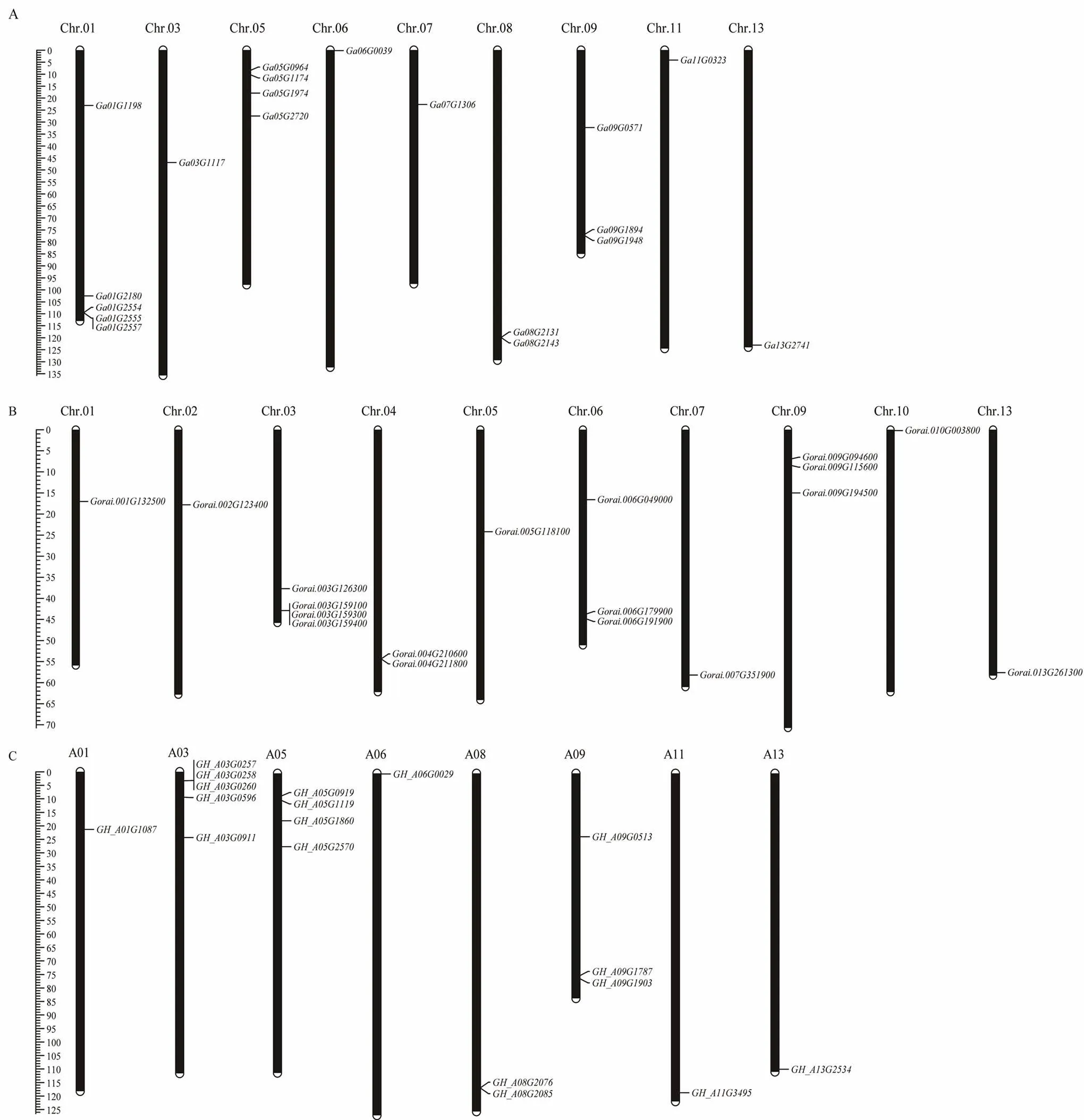
为了阐明4个棉种β-tubulin基因的扩增机制和进化情况,分别对亚洲棉和陆地棉(A亚组)、亚洲棉和海岛棉(A亚组),以及雷蒙德氏棉和陆地棉(D亚组)、雷蒙德氏棉和海岛棉(D亚组)进行共线性分析,发现26、23、32、37对共线性基因(图3)。陆地棉与亚洲棉和雷蒙德氏棉的共线性基因对比海岛棉少,而系统发育分析中,亚洲棉和雷蒙德氏棉也大都与海岛棉聚类,与陆地棉相比,海岛棉可能与亚洲棉和雷蒙德氏棉之间的亲缘关系较近。
2.4 β-tubulin基因家族的结构分析
为探究β-tubulin基因家族成员间的功能异同,对其基因结构和功能域进行分析,并将β-tubulin基因家族成员的motif、CDS及氨基酸序列进行了可视化分析(图4-A),有67个β-tubulin基因含有3个外显子,4个β-tubulin基因(、、和)含有2个外显子,2个基因(和)含有4个外显子。利用在线NCBIBatch CDD数据库和SMART数据库对β-tubulin蛋白进行蛋白结构域的预测和矫正(图4-B),所有β-tubulin蛋白都含有Tubulin与Tubulin-C 2个典型结构域。此外,还通过在线MEME软件预测β-tubulin基因家族成员的motif(图4-C)。结果表明,除中缺少motif1外,其余基序在β-tubulin蛋白中均保守。
2.5 顺式作用元件分析
利用PlantCARE对棉花的β-tubulin基因上游2 000 bp的序列进行顺式作用元件分析(图5),结果表明,β-tubulin基因家族成员启动子顺式作用元件主要分为3种:生长发育响应相关元件(如GGTTAA、GATGACATGG、TCAAACGGT等)、激素响应相关元件(如CGTCA、ACGTG、CCATCTTTTT等)和胁迫响应相关元件(如AAATTTCCT、GTTTTCTTAC、CAACTG等)。其中,光响应元件最多,达53%,此外,生长发育响应元件还包括分生组织表达、胚乳表达、玉米蛋白代谢、类黄酮生物合成、昼夜节律调控、栅栏叶肉细胞及细胞周期调控等元件;激素响应相关元件包括生长素、脱落酸、茉莉酸、赤霉素及水杨酸等;胁迫响应相关元件有干旱诱导、低温响应、应激响应、创伤反应及厌氧诱导等。在所有β-tubulin基因家族成员中,除的作用元件主要分布在起始密码子上游1 000 bp外,其他家族成员顺式作用元件多均匀分布在上游2 000 bp。
2.6 陆地棉β-tubulin基因家族成员表达模式分析
利用陆地棉TM-1根、茎、叶、花、胚珠和纤维的转录组数据对36个GhTUBs基因家族成员进行不同组织表达模式聚类分析(图6)。结果表明,将分为5个簇,簇Ⅰ中的3个基因(、和)在不同组织不同时期均有表达;簇Ⅱ中的15个基因在各组织表达量均较高且在纤维发育时期优势表达;簇Ⅲ中的4个基因(、、和)在叶、花及胚珠发育的各个时期低表达,而在根、茎及纤维组织中的表达量相对较高;簇Ⅳ中的7个基因主要在根、茎、叶、花等组织中高表达,其中和仅在根、茎、叶、花等组织中特异性高表达,胚珠和纤维发育的各个时期表达量都较低;簇Ⅴ中的7个基因在不同组织不同时期表达量均较低,其中,同源基因和在以上组织中几乎不表达。
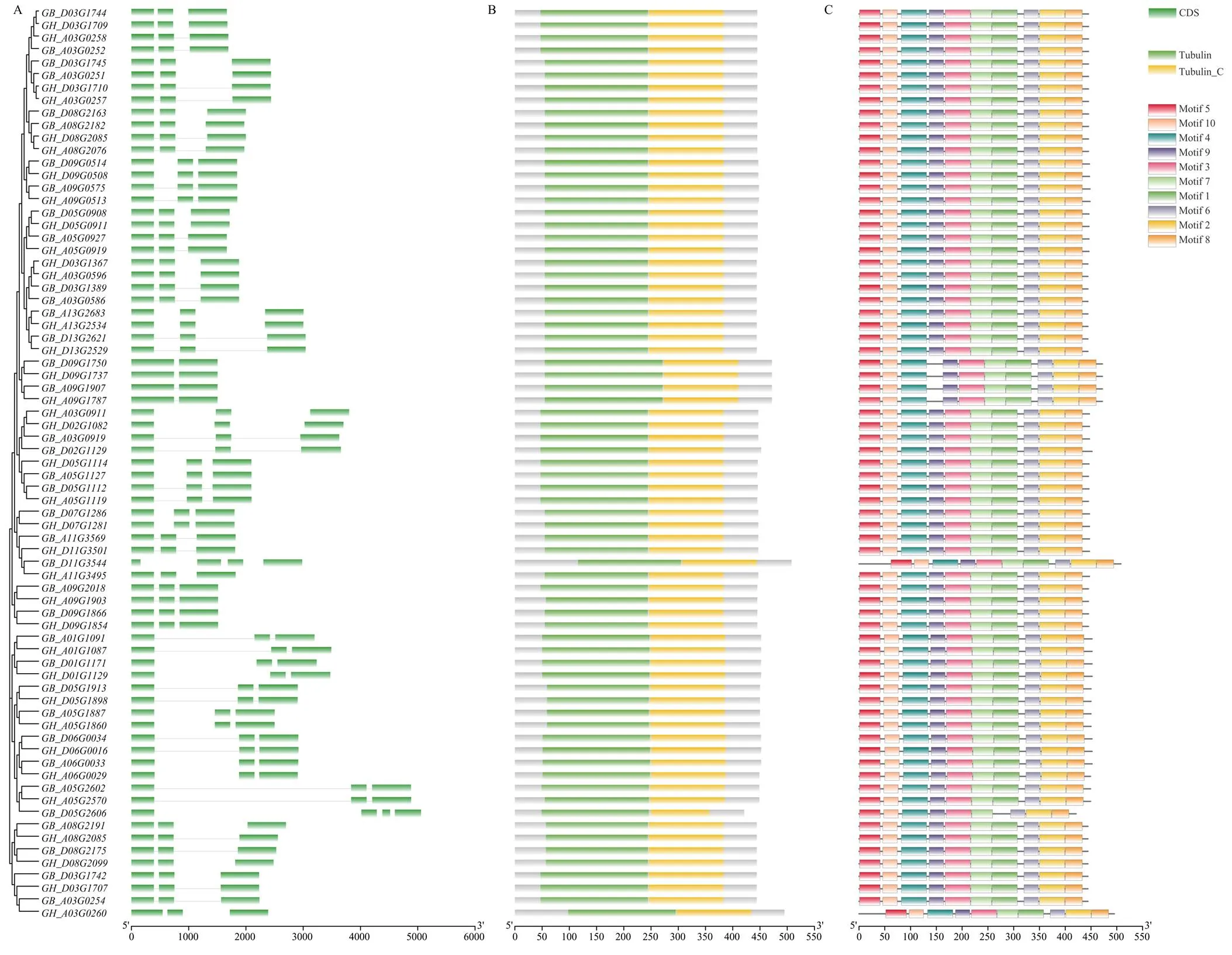
A:β-tubulin基因的内含子、外显子示意图;B:β-tubulin蛋白结构域预测;C:β-tubulin基因的motif预测分析
利用已有的39个海陆群体长、短纤维材料5、10和15 DPA的转录组数据及成熟纤维的马克隆值、强度、长度表型数据,分别对36个GhTUBs基因进行各个时期表达模式与纤维的马克隆值、强度、长度之间的相关性分析,鉴定到13个基因的表达与3个纤维性状的表型数据存在显著相关(图7)。其中,5 DPA纤维中的表达水平与马克隆值显著相关;5 DPA纤维中的表达水平与纤维强度显著相关,10 DPA纤维中和的表达水平与纤维强度显著相关,15 DPA纤维中、和的表达水平与纤维强度显著相关;10 DPA纤维中、、、和的表达水平与纤维长度显著相关,15 DPA纤维中和的表达水平与纤维长度显著相关。

图5 棉花β-tubulin基因启动子顺式作用元件

图6 36个GhTUBs在陆地棉不同组织中的表达模式
3 讨论
3.1 β-tubulin基因家族成员鉴定及分析
微管是植物细胞骨架的主要组成部分[3],参与植物细胞的形态建成。β-微管基因家族鉴定相关研究已在多种植物中完成,如拟南芥中有9个β-tubulin[13]、亚麻中有14个[16]、水稻中有8个[36]、柳树中有20个[37]。本研究利用生物信息学方法在二倍体和四倍体棉种中分别鉴定到18、19和36、37个β-tubulin基因家族成员。通过系统进化分析发现,4个棉种中的76(69.09%)个β-tubulin基因和拟南芥中的9个β-tubulin基因亲缘关系较近,聚类到Cluster Ⅰ、Cluster Ⅱ和Cluster Ⅲ,说明大多数棉花β-tubulin基因在不同物种中具有较高的功能保守性。此外,棉花中有34个基因聚在了Cluster Ⅳ和Cluster Ⅴ与拟南芥相比亲缘关系较远,推测这类家族基因与棉花发育的多样性相关。
3.2 候选基因表达模式与功能分析
棉花纤维品质性状改良是棉花育种工作的重点,本研究通过分析陆地棉的36个GhTUBs基因在根、茎、叶、花、胚珠和纤维的表达模式,可将其分为5个簇,其中簇Ⅱ的基因在纤维发育时期优势表达,占总数的42%,该结果暗示较多的β-tubulin基因家族成员潜在调控棉花纤维的生长发育。通过比对表达模式的5个簇与进化树分析的5个簇基因之间的异同发现,两者间存在较大程度重叠。进化树Cluster Ⅱ和ClusterⅤ中的所有陆地棉β-tubulin基因分别聚类到表达模式中的簇Ⅱ和簇Ⅳ中,同时表达模式簇Ⅰ中67%的基因位于进化树分析的ClusterⅠ中,表明亲缘关系较近的在不同组织或发育时期往往具有相似的表达模式。此外,结合海陆群体纤维表型数据与基因表达水平,筛选到1、6和11个β-tubulin基因分别与马克隆值、纤维比强度和纤维长度显著相关,其中4个基因(、、和)同时与纤维长度和比强度性状显著相关。近年来,已有研究表明β-tubulin基因潜在调控棉纤维的生长、发育[4]。如Li等[20]通过筛选棉花基因组DNA文库和组织化学分析,发现和在纤维发育早期高表达,在其他组织中表达量极低,说明该基因可能在纤维发育中起着重要作用。Qin等[21]通过分析长短纤维转录组数据和基因功能注释发现可能是纤维发育相关的β-tubulin基因,该基因在10 DPA时高表达,说明该基因在纤维伸长过程中起作用。曾志锋[22]发现和在纤维发育次生壁合成期特异性高表达,将该基因过表达转化拟南芥发现根长明显受到抑制,过表达转化棉花发现与野生型相比纤维长度明显降低,而反义抑制则促进了纤维的伸长;而CHEN等[38]通过分析陆地棉微管蛋白基因并结合转录组和qRT-PCR发现,该基因可能在调控纤维长度的同时还调控纤维强度。以上研究筛选出的β-tubulin基因为揭示其在纤维发育中的功能提供了一定的基因资源。然而,有关β-tubulin基因家族成员在较多材料中的表达模式及其与品质性状之间的关系研究尚未被系统阐述,其制约了对β-tubulin基因家族成员的较为全面的筛选。以上结果进一步表明,β-tubulin基因家族成员可能调控纤维发育,并存在一因多效。

图7 13个基因5、10、15 DPA纤维转录组数据与马克隆值、纤维强度、纤维长度相关性分析
4 结论
在4个棉种中共鉴定出110个β-tubulin基因家族成员,氨基酸理化性质和序列高度保守而启动子序列调控元件多样;筛选出15个陆地棉纤维优势表达的β-tubulin基因家族成员,并发掘出潜在调控马克隆值性状的基因,及潜在同时调控纤维长度和比强度性状的候选基因、、和。
[1] 肖水平, 宋国立, 余进祥. 棉花纤维品质相关基因挖掘及功能基因研究进展. 棉花科学, 2020, 42(2): 3-14.
XIAO S P, SONG G L, YU J X. Research progress of cotton fiber quality related gene mining and functional genes. Cotton Sciences, 2020, 42(2): 3-14. (in Chinese)
[2] 杨君, 马峙英, 王省芬. 棉花纤维品质改良相关基因研究进展. 中国农业科学, 2016, 49(22): 4310-4322.
YANG J, MA Z Y, WANG X F. Progress in studies on genes related to fiber quality improvement of cotton. Scientia Agricultura Sinica, 2016, 49(22): 4310-4322. (in Chinese)
[3] Dixon D C, Seagull R W, Triplett B A. Changes in the accumulation of [alpha]- and [beta]-tubulin isotypes during cotton fiber development. Plant physiology, 1994, 105(4): 1347-1353.
[4] Kost B, Chua N H. The plant cytoskeleton: vacuoles and cell walls make the difference. Cell, 2002, 108(1): 9-12.
[5] He X C, Qin Y M, Xu Y, Hu C Y, Zhu Y X. Molecular cloning, expression profiling, and yeast complementation of 19 β-tubulin cDNAs from developing cotton ovules. Journal of Experimental Botany, 2008, 59(10): 2687-2695.
[6] Nieuwenhuis J, Brummelkamp T R. The tubulin detyrosination cycle: Function and enzymes. Trends in Cell Biology, 2019, 29(1): 80-92.
[7] Meiring J C M, Shneyer B I, Akhmanova A. Generation and regulation of microtubule network asymmetry to drive cell polarity. Current Opinion in Cell Biology, 2020, 62: 86-95.
[8] McKean P G, Vaughan S, Gull K. The extended tubulin superfamily. Journal of Cell Science, 2001, 114(15): 2723-2733.
[9] Seagull R W. Cytoskeletal stability affects cotton fiber initiation. International Journal of Plant Sciences, 1998, 159(4): 590-598.
[10] Oakley R V, Wang Y S, Ramakrishna W, Harding S A, Tsai C J. Differential expansion and expression of α- and β-tubulin gene families in. Plant Physiology, 2007, 145(3): 961-973.
[11] Mubeen H, Shoaib M W, Raza S. In silico analysis and comparison of alpha tubulin gene with other tubulin families. Journal of Advance in Biology & Biotecnology, 2016, 7(4): 1-7.
[12] LI Y L, SUN J, LI C H, ZHU Y Q, XIA G X. Specific expression of agene () in developing cotton fibers. Science in China Series C: Life Sciences, 2003, 46(3): 235-242.
[13] Snustad D P, Haas N A, Kopczak S D, Silflow C D. The small genome ofcontains at least nine expressed beta-tubulin genes. The Plant Cell, 1992, 4(5): 549-556.
[14] Cheng Z G, Carter J V. Temporal and spatial expression patterns of, a β-tubulin gene of. Plant Molecular Biology, 2001, 47(3): 389-398.
[15] 杨建霞.对拟南芥生长发育的影响和基因新功能分析[D]. 兰州: 兰州大学, 2015.
YANG J X. The effects ofongrowth and development and the analysis of the new role of[D]. Lanzhou: Lanzhou University, 2015. (in Chinese)
[16] Gavazzi F, Pigna G, Braglia L, Gianì S, Breviario D, Morello L. Evolutionary characterization and transcript profiling of β-tubulin genes in flax (L.) during plant development. BMC Plant Biology, 2017, 17(1): 237.
[17] 睢金凯, 饶国栋, 张建国. 柳树β微管蛋白基因家族的克隆和序列分析. 西北植物学报, 2016, 36(5): 902-909.
SUI J K, RAO G D, ZHANG J G. Cloning and sequence analysis of β-tubulin gene families in willows. Acta Botanica Boreali- Occidentalia Sinica, 2016, 36(5): 902-909. (in Chinese)
[18] 黄胜雄, 胡尚连, 孙霞, 蒋瑶. 慈竹基因片段的克隆及序列分析. 福建林业科技, 2009, 36(1): 7-10, 42.
HUANG S X, HU S L, SUN X, JIANG Y. Cloning and sequence analysis ofgene fragments of. Fujian Forestry Science and Technology, 2009, 36(1): 7-10, 42. (in Chinese)
[19] 王亚婷, 林玉玲, 曾丽兰, 赖钟雄. 龙眼胚性愈伤组织微管蛋白基因-克隆及序列分析. 福建农林大学学报(自然科学版), 2014, 43(1): 49-54.
WANG Y T, LIN Y L, ZENG L L, LAI Z X. Cloning and sequence analysis of-gene from embryogenic callus inLour. Journal of Fujian Agriculture and Forestry University (Natural Science Edition), 2014, 43(1): 49-54. (in Chinese)
[20] Li X B, Cai L, Cheng N H, Liu J W. Molecular characterization of the cottongene that is preferentially expressed in fiber. Plant Physiology, 2002, 130(2): 666-674.
[21] Qin Y, Sun H R, Hao P B, Wang H T, Wang C C, Ma L, Wei H L, Yu S X. Transcriptome analysis reveals differences in the mechanisms of fiber initiation and elongation between long- and short-fiber cotton (L.) lines. BMC Genomics, 2019, 20(1): 633.
[22] 曾志锋.在棉纤维发育中的功能[D]. 重庆: 西南大学, 2016.
ZENG Z F. The function of
[23] 李彩丽. 毛竹细胞骨架微管蛋基因和研究[D]. 北京: 中国林业科学研究院, 2012.
LI C L. Study onandencoding micro tuhulin of cvtoskeleton in moso bamboo () [D]. Beijing: Chinese Academy of Forestry, 2012. (in Chinese)
[24] Du X M, Huang G, He S P, Yang Z E, Sun G F, Ma X F, Li N, Zhang X Y, Sun J L, Liu M, Jia Y H, Pan Z E, Gong W F, Liu Z H, Zhu H Q, Ma L, Liu F Y, Yang D G, Wang F, Fan W, Gong Q, Peng Z, Wang L R, Wang X Y, Xu S J, Shang H H, Lu C R, Zheng H K, Huang S W, Lin T, Zhu Y X, Li F G. Resequencing of 243 diploid cotton accessions based on an updated A genome identifies the genetic basis of key agronomic traits. Nature Genetics, 2018, 50(6): 796-802.
[25] Paterson A H, Wendel J F, Gundlach H, Guo H, Jenkins J, Jin D C, Llewellyn D, Showmaker K C, Shu S Q, Udall J, Yoo M J, Byers R, Chen W, Doron-Faigenboim A, Duke M V, Gong L, Grimwood J, Grover C, Grupp K, Hu G J, Lee T H, Li J P, Lin L F, Liu T, Marler B S, Page J T, Roberts A W, Romanel E, Sanders W S, Szadkowski E, Tan X, Tang H B, Xu C M, Wang J P, Wang Z N, Zhang D, Zhang L, Ashrafi H, Bedon F, Bowers J E, Brubaker C L, Chee P W, Das S, Gingle A R, Haigler C H, Harker D, Hoffmann L V, Hovav R, Jones D C, Lemke C, Mansoor S, ur Rahman M, Rainville L N, Rambani A, Reddy U K, Rong J K, Saranga Y, Scheffler B E, Scheffler J A, Stelly D M, Triplett B A, Van Deynze A, Vaslin M F S, Waghmare V N, Walford S A, Wright R J, Zaki E A, Zhang T Z, Dennis E S, Mayer K F X, Peterson D G, Rokhsar D S, Wang X Y, Schmutz J. Repeated polyploidization ofgenomes and the evolution of spinnable cotton fibres. Nature, 2012, 492(7429): 423-427.
[26] Hu Y, Chen J D, Fang L, Zhang Z Y, Ma W, Niu Y C, Ju L Z, Deng J Q, Zhao T, Lian J M, Baruch K B, Fang D, Liu X, Ruan Y L, Rahman M U, Han J L, Wang K, Wang Q, Wu H T, Mei G F, Zang Y H, Han Z G, Xu C Y, Shen W J, Yang D F, Si Z F, Dai F, Zou L F, Huang F, Bai Y L, Zhang Y G, Brodt A, Ben-Hamo H, Zhu X F, Zhou B L, Guan X Y, Zhu S J, Chen X Y, Zhang T Z.andgenomes provide insights into the origin and evolution of allotetraploid cotton. Nature Genetics, 2019, 51(4): 739-748.
[27] Letunic I, Doerks T, Bork P. SMART: recent updates, new developments and status in 2015. Nucleic Acids Research, 2015, 43(D1): D257-D260.
[28] Larkin M A, Blackshields G, Brown N P, Chenna R, McGettigan P A, McWilliam H, Valentin F, Wallace I M, Wilm A, Lopez R, Thompson J D, Gibson T J, Higgins D G. Clustal W and Clustal X version 2.0. Bioinformatics, 2007, 23(21): 2947-2948.
[29] Kumar S, Stecher G, Tamura K. MEGA7: Molecular evolutionary genetics analysis version 7.0 for bigger datasets. Molecular Biology and Evolution, 2016, 33(7): 1870-1874.
[30] Bjellqvist B, Basse B, Olsen E, Celis J E. Reference points for comparisons of two-dimensional maps of proteins from different human cell types defined in a pH scale where isoelectric points correlate with polypeptide compositions. Electrophoresis, 1994, 15(3/4): 529-539.
[31] Voorrips R E. MapChart: software for the graphical presentation of linkage maps and QTLs. Journal of Heredity, 2002, 93(1): 77-78.
[32] Chen C J, Chen H, Zhang Y, Thomas H R, Frank M H, He Y H, Xia R. TBtools: An integrative toolkit developed for interactive analyses of big biological data. Molecular Plant, 2020, 13(8): 1194-1202.
[33] Krzywinski M, Schein J, Birol I, Connors J, Gascoyne R, Horsman D, Jones S J, Marra M A. Circos: an information aesthetic for comparative genomics. Genome Research, 2009, 19(9): 1639-1645.
[34] Bailey T L, Boden M, Buske F A, Frith M, Grant C E, Clementi L, Ren J Y, Li W W, Noble W S. MEME Suite: tools for motif discovery and searching. Nucleic Acids Research, 2009, 37(supp1_2): W202- W208.
[35] Lescot M, Déhais P, Thijs G, Marchal K, Moreau Y, Van de Peer Y, Rouzé P, Rombauts S. PlantCARE, a database of plant cis-acting regulatory elements and a portal to tools for in silico analysis of promoter sequences. Nucleic Acids Research, 2002, 30(1): 325-327.
[36] Yoshikawa M, Yang G X, Kawaguchi K, Komatsu S. Expression analyses of β-tubulin isotype genes in rice. Plant and Cell Physiology, 2003, 44(11): 1202-1207.
[37] Rao G D, Zeng Y F, He C Y, Zhang J G. Characterization and putative post-translational regulation of- andgene families in. Scientific Reports, 2016, 6: 19258.
[38] CHEN B J, ZHAO J J, FU G Y, PEI X X, PAN Z E, LI H G, AHMED H, HE S P, DU X M. Identification and expression analysis of Tubulin gene family in upland cotton. Journal of Cotton Research, 2021, 4(3): 229-238.
Genome-Wide Identification and Expression Analysis of β-tubulin Family in Cotton fiber development
DANG YuanYue1,2, MA JianJiang2, YANG ShuXian2, SONG JiKun1,2, JIA Bing1,2, FENG Pan2, CHEN QuanJia1, YU JiWen1,2
1College of Agriculture, Xinjiang Agricultural University/Engineering Research Centre of Cotton, Ministry of Education, Urumqi 830052;2Institute of Cotton Research of Chinese Academy of Agricultural Sciences/National Engineering Research Center of Cotton Biology Breeding and Industrial Technology, Anyang 455000, Henan
【Objective】β-tubulin is the basic structural unit of cotton fiber, regulates fiber cell morphogenesis, and plays a vital role in fiber development. But there is less understood how β-tubulin gene influenced the distinct characteristic of fiber quality traits in cotton. In this study, members of the β-tubulin gene family were identified in cotton, their expression profiles were analyzed, and role of β-tubulin genes were explored for fiber quality. 【Method】BLAST method was used to identify members of the β-tubulin gene family in the genomes of four cotton species. ProtParam tool was utilized to analyze physicochemical properties, MEGA7.0 to construct phylogenetic tree, Mapchart2.2 to draw chromosomal localization map, MEME to analyze conserved motif, and PlantCARE to analyze promoter cis-acting elements. Expression levels of β-tubulin genes were characterized by using transcriptome data from 39 studies on fiber development. Spearman correlation analysis was used to identify candidate genes for fiber quality traits. 【Result】Importantly, 36, 37, 19 and 18 β-tubulin genes were identified in the genomes of(AD1),(AD2),(A2) and(D5), respectively. The number of β-tubulin genes in tetraploid cotton species is almost double than that of diploid cotton species. Phylogenetic analysis classified these genes into 5 main clusters. Phylogenetic and collinearity analysis revealed that β-tubulin genes inis closely related toandas compared to. Furthermore, all genes have typical conservative domains with Tubulin and Tubulin-C. The genes physicochemical properties showed amino acids range from 421 to 508 with isoelectric point of 4.68 to 5.09. The analysis of promoter cis-acting elements identified growth responsive, hormone responsive, and stress responsive elements which showed β-tubulin mediates various mechanisms of cell growth regulation. Interestingly, cluster analysis on 36 β-tubulin gene expression profiles showed 42% genes in cluster П had dominant expression in fiber. In particular, 1, 6, and 11 β-tubulin genes exhibited significant correlation with fiber micronaire value, fiber strength, and fiber length, respectively. Four genes were found to influence fiber length and fiber strength traits simultaneously. 【Conclusion】A total of 110 β-tubulin gene family members were identified in the four cotton species. Their physicochemical properties and sequences of amino acids were highly conserved and the promoter sequence had diverse regulatory elements. This study characterized the expression profiles as well as molecular function of β-tubulin gene family in cotton fiber. Further discovered the potential candidate genes that probably regulate fiber quality traits in cotton. Our results may have great potential for cotton fiber quality improvement by genetic engineering.
cotton; β-tubulin gene family; bioinformatics; expression analysis; fiber development

10.3864/j.issn.0578-1752.2023.23.003
2022-12-10;
2023-02-22
国家自然科学基金青年科学基金(32101716)、国家重点研发计划(2022YFD1200300)
党媛玥,E-mail:1015258312@qq.com。通信作者陈全家,E-mail:chqjia@126.com。通信作者于霁雯,E-mail:yujw666@hotmail.com
(责任编辑 李莉)
