基于COⅠ基因的5种新疆土著鱼类DNA条形码鉴定及聚类分析
2023-12-27强壮赵年桦张旦旦秦凯瑞聂竹兰李立伟钟馨胡仁云
强壮,赵年桦,张旦旦,,秦凯瑞,聂竹兰*,李立伟,钟馨,胡仁云
(1 塔里木大学生命科学与技术学院,新疆 阿拉尔 843300)(2 塔里木大学动物科学与技术学院,新疆 阿拉尔 843300)(3 武汉中科瑞华生态科技股份有限公司,湖北 武汉 430000)
裂腹鱼亚科属鲤形目(Cypriniformes)、鲤科(Cyprinidae),共11属,中国境内存在10属,主要分布于青藏高原及其附近水域,有明显的地域特征性[1]。扁吻鱼(Aspiorhynchuslaticeps)[2]、塔里木裂腹鱼(Schizothoraxbiddulphi)[3]、宽口裂腹鱼(Schizothoraxeurystomus)[4]、斑重唇鱼(Diptychusmaculatus)[5]等众多裂腹鱼亚科鱼类是新疆塔里木河流域重要的土著鱼[6],近年来,水库堤坝的大量修建,严重破坏土著鱼类产卵场、洄游通道以及索饵场,加之性成熟晚、繁殖力低、生存和繁殖环境要求高等众多因素制约,这些鱼类的野外种群数量急剧下降[7-8]。扁吻鱼于1988年在《中国濒危动物红皮书》中被列为国家Ⅰ类重点保护水生野生动物[9];斑重唇鱼于2021年列入《中国国家重点保护野生动物名录》二级[10];塔里木裂腹鱼被列为新疆维吾尔自治区Ⅱ类重点保护水生野生动物,又于2021年被列入国家二级重点保护水生野生动物[11]。
高原鳅属鱼类属鳅科(Cobitidae)、条鳅亚科(Nemachilinae)、高原鳅属(Triplophysa)[12],在青藏高原地区广泛分布,随着全球气候变暖,导致高原湖泊、水库、河流的水位逐年下降,水利工程建设及水域资源过量开发,许多高原鳅物种都失去了生存条件[13]。
DNA条形码技术通过利用生物体内能代表该物种的一段或几段共有DNA序列[14-15],再结合形态学鉴定分析,即可实现新物种、潜藏种以及物种有效性的鉴定[16]。正确的物种鉴定对资源管理和生态保护起着显著作用[17],尤其是在渔业资源匮乏地区,尽早采取系统科学的技术措施保护本地区的渔业资源变得尤为重要[18-19]。然而裂腹鱼亚科中很多种类间的形态差异极为微小[20],同属物种间的鉴定十分困难,过去简单的形态学分析因其具有低效率、低准确率、对鉴定者实践经验和技术手段过于依赖等局限性,已不再适应于当今时代科技发展水平的需求[17]。随着分子技术的快速发展、PCR技术的普及和测序成本的大幅降低,DNA条形码技术应时而生[16],在出现后的短短20余年内不断纠正物种鉴定中存在的同种异名或异种同名问题[21-22]。杨天燕等[2]基于COⅠ基因比较和分析了扁吻鱼和塔里木裂腹鱼遗传差异,表明两者间的亲缘关系较特化等级更高的新疆裸重唇鱼、斑重唇鱼更近。海萨·艾也力汗等[23]通过COⅠ基因对塔里木河流域4种裂腹鱼间的亲缘关系进行分析鉴定,表明塔里木裂腹鱼与厚唇裂腹鱼、重唇裂腹鱼、宽口裂腹鱼之间存在丰富的基因交流,并且表明这4种裂腹鱼可能尚未达到物种分化程度,仍属同一物种的不同亚种。
本研究通过对新疆5种土著鱼类的DNA条形码进行比较分析,验证COⅠ基因对当地土著鱼类物种鉴定的有效性,以此为DNA条形码数据库的建立奠定基础,并为该地区鱼类生物资源调查以及建立健全的种质资源保护体系提供理论参考依据。
1 材料与方法
1.1 样品采集
2021年4月于乌恰县克孜勒河采集样品鱼30尾、于拜城县克孜尔水库采集样品鱼3尾,采集工具为地笼网(网长10 m,33节20洞,框架2 cm×3 cm,网目4 mm),样品分类鉴定参照《新疆鱼类志》[9]。使用MS-222进行麻醉,取部分尾鳍浸泡在装有无水乙醇的离心管中,放入-20 ℃冰箱保存,以备后续的分子生物学鉴定。
1.2 新疆5种土著鱼类DNA提取、PCR扩增及测序
使用DNeasy Blood &Tissue Kit试剂盒提取基因组DNA,提取的DNA置于-20 ℃冰箱保存备用。PCR扩增引物如表1所示,反应体系为25 μL,包含2.5 μL 10×buffer,2 μL dNTPs (2.5 mmol/L),正反引物(10 μmol/L)各0.5 μL,0.5 μLTaq酶,DNA模板1 μL,其余用ddH2O补足至25 μL。扩增条件为95 ℃预变性5 min,循环数为1;95 ℃变性30 s,55 ℃退火30 s,72 ℃延伸7 min,35个循环;72 ℃延伸9 min,4 ℃保存。PCR仪为ABI-2720。PCR产物经1%的琼脂糖凝胶电泳(120 V,30 min),在紫外灯下观察,选取单一明亮的条带PCR产物送至青岛鹏翔生物科技有限公司进行测序。

表1 COⅠ基因扩增引物序列
1.3 新疆5种土著鱼类序列分析
测序结果使用Chromas软件编辑,人工辅助校对,然后用SeqMan软件拼接、人工核对。再使用Clustal W与鲤科同源序列多重比对,去除两端冗余区域,保留共有序列在NCBI数据库的Blast中检索一致性,确定物种确切的分类地位。若与NCBI数据库中确定物种一致性在98%以上,则视为同一种,一致性位于92%~98%为同属,85%~92%为同科。处理后的序列利用MEGA-X分析序列长度、核苷酸含量、变异位点数和保守位点数、密码子组成、简约信息位点等参数,并计算种内和种间的遗传距离,模型选择为Kimuar 2 parameter model双参数模型(k2p),使用邻接法(neighbor-joining,NJ)构建系统发育树,聚类树的可靠性利用Bootstrap软件进行检验,设置1 000次重复抽样评估。
2 结果与分析
2.1 新疆5种土著鱼类形态学分类鉴定
33尾样品初步鉴定为4属5种,具体信息如表2所示。
2.2 新疆5种土著鱼类序列比对和分析
33尾样品鱼的DNA条形码逐条与NCBI(https://www.ncbi.nlm.nih.gov)数据库中的Blast进行一致性比对。比对结果表明:33尾新疆土著鱼类经过与Genbank比对,属于4属5种(扁吻鱼、塔里木裂腹鱼、宽口裂腹鱼、斑重唇鱼、长身高原鳅)与形态学鉴定结果一致。
5种鱼类检测出13个单倍型,其中Hap_4和Hap_7为最优单倍型,均出现5次,其次为Hap_2(4次),第三为Hap_5和Hap_8(均出现3次)。33尾样品鱼的COⅠ基因测序一致,序列长度为674 bp(剔除引物区)。
分析COⅠ基因序列的平均碱基组成为G=18.4%、C=28.3%、A=25.4%、T=27.9%,(G+C)mol%值为46.7%,(A+T)mol%值为53.3%,出现反G偏倚现象。A、G、C、T在第1、第2、第3位点的平均含量差别较大(表3)。分析可知,密码子碱基的使用存在明显的偏向性,第1个密码子中,T碱基使用频率显著高于其他碱基,第2位密码子的A碱基使用频率最高,G碱基的使用频率在第3位密码子中最多。且第2密码子位点处(A+T)mol%值59.8%,明显高于第1位和第3位密码子的(A+T)mol%值,第3位密码子处的(C+G)mol%值55.9%,极高于第1和第2密码子处的(C+G)mol%值,说明第1、2、3位密码子中的碱基组成存在差异。

表3 新疆5种土著鱼类COⅠ基因序列碱基组成 %
MEGA-X分析表明,5种新疆土著鱼类COⅠ基因序列包含保守位点C(conserved sites)499个,变异位点V(variable sites)173个,简约性信息位点PI (parsimony informative sites)170个,3个单突变位点S(singleton variable sites)。分别占总碱基数的74.04%、25.67%、25.22%、0.45%。所分析的序列中(表4),不变位点(ii)包括599个,45个转换位点(si),21个颠换位点(sv),转换位点明显大于颠换位点,R值为2.10(转换/颠换),R值大于2,说明该段序列转换与颠换未达到饱和状态,且该序列可靠,可进行下一步的系统发育树构建。其中第1位密码子的不变位点最多,为222个,转换位点和颠换位点属第2位密码子居多。T和C是主要发生转换的碱基,颠换主要发生在A和C之间。

表4 新疆5种土著鱼类COⅠ基因碱基变异情况
如表5所示,从碱基组成分析,5种鱼类的(A+T)mol%值均大于(G+C)mol%值,表现出明显的反G偏倚;从遗传多样性分析(表5),斑重唇鱼的单倍型多样性指数最高,为0.833,说明斑重唇鱼的遗传多样性最高;从核苷酸相关参数分析,斑重唇鱼的核苷酸多样性指数和平均核苷酸差异数最高,分别为0.002 5和1.667 0,说明其分化速度最快,变异程度最高。

表5 新疆5种土著鱼COⅠ基因片段碱基组成与遗传多样性参数
2.3 新疆5种土著鱼类遗传距离
如表6所示,从NCBI的Genbank中选择相似度高于90%以上的同属物种,基于k2p模型分别计算新疆4属5种土著鱼类的遗传距离以及和其他种的遗传距离,结果表明,5种新疆土著鱼类种内遗传距离最小值为(0.07±0.06)%,最大为(0.25±0.14)%,分别存在于塔里木裂腹鱼和斑重唇鱼中。

表6 新疆5种土著鱼类的种内遗传距离 %
如表7所示,5种新疆土著鱼的种间遗传距离两两比对结果显示,最大种间遗传距离和最小种间遗传距离分别为(22.73±2.11)%和(0.24±0.16)%,前者存在于长身高原鳅和斑重唇鱼之间,后者为塔里木裂腹鱼和宽口裂腹鱼的种间遗传距离,均大于0.02的种间遗传分化临界值。种间平均遗传距离为(11.55±0.95)%,种内平均遗传距离为(0.14±0.10)%。前者远大于后者的10倍,根据Hebert提出鉴定物种的“10倍法则”,能符合该要求,说明COⅠ基因在不同的物种中形成了明显的“条形码间隙”。
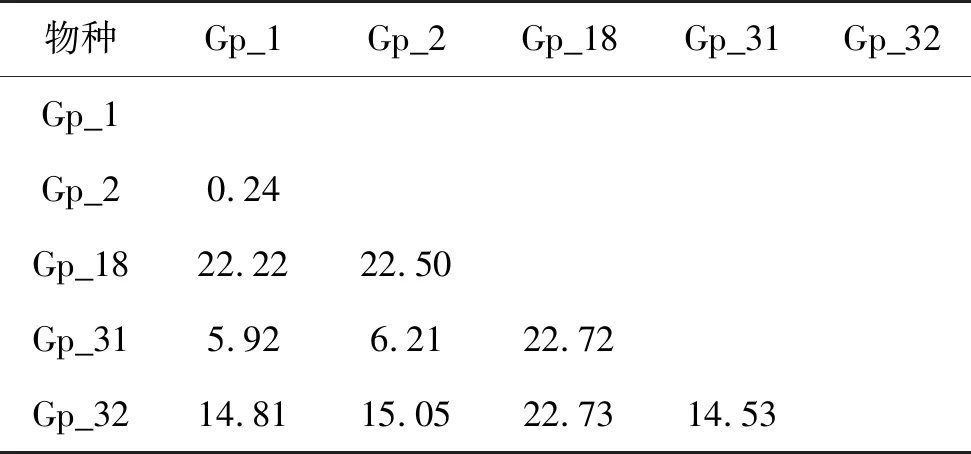
表7 新疆5种土著鱼类的种间遗传距离 %
如表8所示,基于COⅠ基因计算塔里木裂腹鱼和宽口裂腹鱼与其他同属裂腹鱼属的种间遗传距离,结果表明,塔里木裂腹鱼与齐口裂腹鱼(Schizothoraxprenanti)和中华裂腹鱼(Schizothoraxsinensis)的遗传距离最大,均为0.056 8,与宽口裂腹鱼的遗传距离最小;宽口裂腹鱼与弧唇裂腹鱼(Schizothoraxcurvila-biatus)、巨须裂腹鱼(Schizothoraxmacropogon)、澜沧裂腹鱼(Schizothoraxlantsangensis)、光唇裂腹鱼(Schizothoraxlissolabiata)的最大遗传距离为0.053 6。如表9所示,长身高原鳅与软口高原鳅(Triplophysachondrostoma)的遗传距离最大,为0.071 6,与梭形高原鳅(Triplophysaleptosoma)的遗传距离最小,为0.008 6。

表8 新疆土著鱼类裂腹鱼属与其他裂腹鱼属的种间遗传距离

表9 新疆土著鱼类高原鳅属与其他高原鳅属的种间遗传距离
2.4 新疆5种土著鱼类系统发育关系分析
本次构建的系统发育树包括4属32种,其中裂腹鱼属17种、高原鳅属13种,扁吻鱼属1种、重唇鱼属1种。由图1分析可知,进化树分为四大支,第一支为塔里木裂腹鱼、宽口裂腹鱼和其他同属裂腹鱼的3个种,且塔里木裂腹鱼和宽口裂腹鱼系统位置较近,符合进化分类地位;第二支为扁吻鱼和亲缘关系较近的裂腹鱼属;第三支为斑重唇鱼,形成单系,且支持度高达100%;第四支为高原鳅属和已发表的同属物种,符合进化地位。基于形态学特征,将裂腹鱼类划分为3个等级,即形态学原始等级、形态学特化等级和形态学高度特化等级。本研究中,塔里木腹鱼和宽口裂腹鱼属于原始等级裂腹鱼类,扁吻鱼属于特化等级裂腹鱼类,而斑重唇鱼属于高度特化等级裂腹鱼类。因此,本研究基于COⅠ基因构建的系统发育树拓扑结构合理,且进化分析显示,各物种都能有效区分,说明COⅠ基因条形码对于不同物种的鱼类具有较为准确的辨识力。
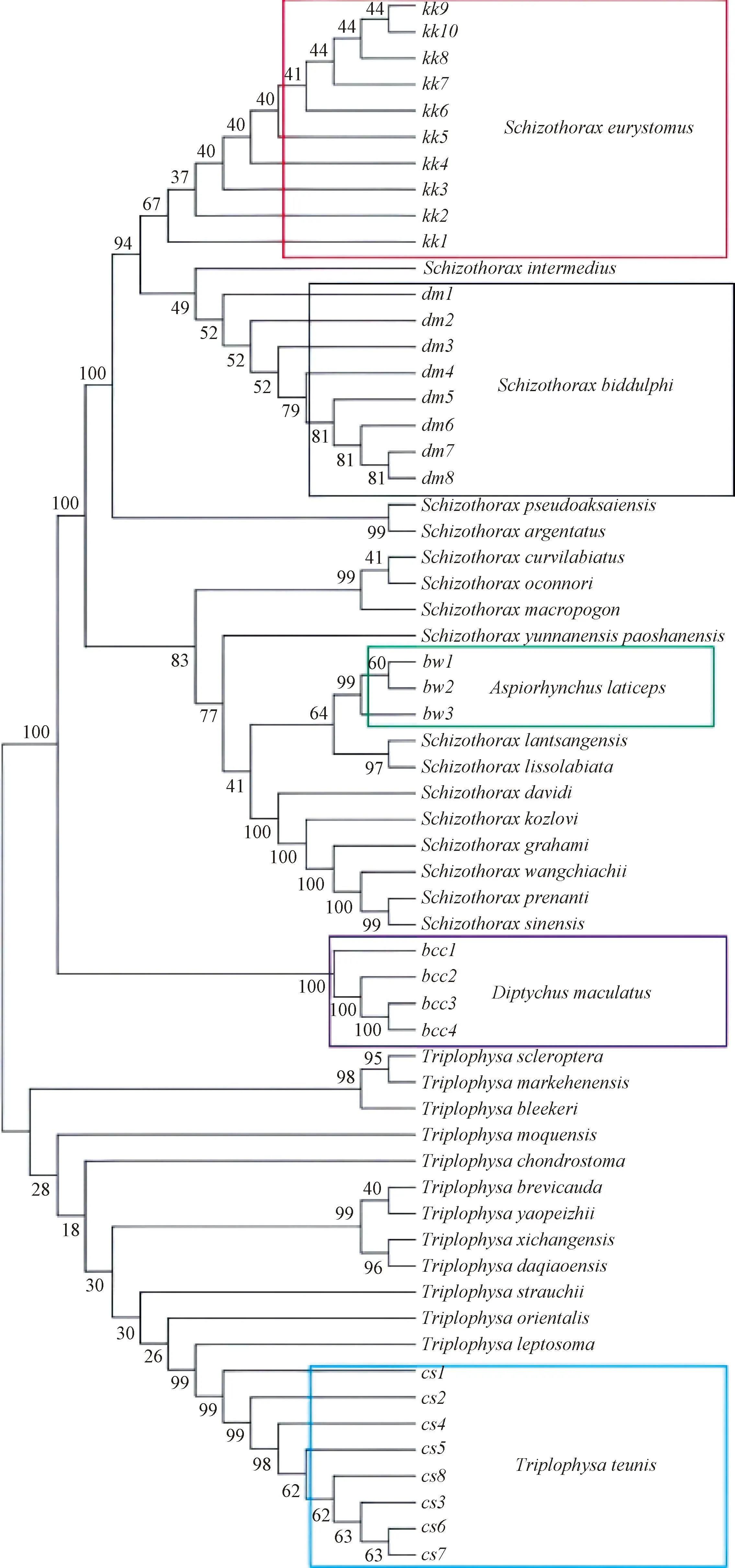
图1 4属5种新疆土著鱼类的系统发育树
3 讨论
3.1 新疆5种土著鱼类DNA条形码技术的适用性
本研究的5种裂腹鱼均具有特有的COⅠ基因序列,各物种间无共享的单倍型;系统发育树结果也表明,塔里木裂腹鱼和宽口裂腹鱼属于原始等级裂腹鱼类,扁吻鱼属于特化等级裂腹鱼类,而斑重唇鱼属于高度特化等级裂腹鱼类。各物种能够有效区分,这说明COⅠ基因条形码对于不同物种的鱼类具有较为准确的辨识力。因此,结果表明以COⅠ基因作为DNA条形码可鉴定5种新疆土著鱼类,并且具有较高的适用性和可行性。
3.2 新疆5种土著鱼类遗传变异分析
33尾新疆土著鱼类COⅠ基因一致序列长度674 bp,(G+C)mol%值表现出较为明显的反偏倚性(46.7%),符合硬骨鱼类线粒体COⅠ基因碱基组成的典型特征[24-25],与光唇鱼属(Acrossocheilus)[26]、沙带鱼(Lepturacanthussavala)[27]等鱼类的研究结果一致。其中克孜勒河塔里木裂腹鱼(G+C)mol%值的反偏倚性与阿克苏河水系、和田河的塔里木裂腹鱼研究结果一致[2,23],克孜尔水库的扁吻鱼与喀拉喀什河和野生扁吻鱼的碱基含量偏向性一致[9],斑重唇鱼与伊犁河厚唇裂腹鱼也存在相同的碱基偏向[28]。本研究通过对5种裂腹鱼线粒体COⅠ基因的序列进行分析,发现第三位密码子碱基变异最多,密码子变异程度最高,表明不同类群鱼类的DNA条形码碱基组成有所不同。
通过COⅠ基因序列的分析,斑重唇鱼的单倍型多样度和核苷酸多样性指数最高,分别为0.833和0.002 5,说明该物种分化程度较大分化较快,核苷酸变异程度较高,遗传资源也较为丰富,与杨天燕等[28]的结果相比(h=7,Hd=0.808,π=0.013 3),前者偏低,中间者近似且后者偏低,可能是本研究采样点分布较为单一或者采集样本数量较少导致。近年来,斑重唇鱼主要栖息地克孜勒河兴修水利设施,分为诸多河段,阻断了洄游路线,各个河段的斑重唇鱼长期无法进行基因交流,因而分化程度较大。宽口裂腹鱼只存在1个单倍型数,单倍型多样度和核苷酸多样性指数最低。扁吻鱼的单倍型数、单倍型多样度和核苷酸多样性指数与杨天燕等[2]的结果相比(h=2,Hd=0.381,Pi=0.000 61),单倍型数一致,其余指数均偏高,说明单倍型之间序列差异较大,同时也表明该物种丰富度较高,对环境的耐受能力较强。本研究中塔里木裂腹鱼的各指数与喀拉喀什河和玉龙喀什河的塔里木裂腹鱼群体相比(Hd=0.733,Pi=0.001 58)[2],Hd偏低,Pi将近一致,可能与COⅠ基因的保守性有关。长身高原鳅的核苷酸多样性指数为0.000 81,和宽口裂腹鱼相似。
3.3 遗传距离与系统发育树分析
4属5种新疆土著鱼类的种内平均遗传距离为(0.142±0.100)%,远低于HEBERT P N等[29]提出的2%阈值;种间平均遗传距离为(11.55±0.95)%,是种内平均遗传距离的43.8倍,远高于Hebert等提出的2%阈值和“10倍法则”。本研究采用邻接法(neighbor-joining,NJ)系统发育树对结果进行验证分析,可以得到裂腹鱼亚科和高原鳅属鱼类同种不同个体均聚在同一分支,形成单系。塔里木裂腹鱼、宽口裂腹鱼和厚唇裂腹鱼聚为一大支,扁吻鱼、斑重唇鱼、长身高原鳅也各自形成了独立的进化支。塔里木裂腹鱼和厚唇裂腹鱼又同聚为一小支,表明两者的亲缘关系更近。因此基于COⅠ基因的DNA条形码技术可应用于扁吻鱼、塔里木裂腹鱼和斑重唇鱼等5种新疆土著鱼类的物种鉴定。
4 结论
本研究证明了基于COⅠ基因的DNA条形码技术可以有效鉴定新疆5种土著鱼类,减少了形态学分析过程中人为错误和外界因素的影响,补充和丰富了裂腹鱼亚科和高原鳅属的DNA条形码库,有利于更加精确地鉴定各物种,对当地裂腹鱼的种质资源保护有着重要的意义。
