基于全基因组测序的弯曲菌食物中毒事件的病原学研究
2023-12-25何娇明马艳萍刘楚云俞慕华鞠长燕
何娇明,马艳萍,刘楚云,陈 辉,俞慕华,鞠长燕
空肠弯曲菌是一种人兽共患病原菌,人群普遍易感,可引起发热、腹痛和血性大便等临床症状。它是世界范围内大多数胃肠炎病例的病因,严重可导致吉兰-巴雷综合征[1]。弯曲菌属的传播途径,以食物、饮水、接触为多见,经口摄入是最主要的传播方式。市售家禽家畜的肉、奶、蛋类多被弯曲菌污染,如进食未加工或加工不适当,吃凉拌菜、生蛤、调味品、汉堡包等,均可引起传染[2]。食物中毒病原菌潜伏期短,对于弯曲菌引起的食物中毒,每年暴发数量呈指数级增长,随着时间的推移,弯曲菌的食物中毒分布变得更广[3]。据美国CDC统计,每年有超过30万人感染是由耐药性弯曲菌引起的[4]。
食源性致病菌溯源分析方法有很多种,常用的有脉冲场凝胶电泳(Pulsed Field Gel Electrophoresis,PFGE)、多位点序列分型(Multi Locus Sequence Typing,MLST)、全基因组测序(Whole Genome Sequencing,WGS)。2022年刘海波等[5]用PFGE方法进行弯曲菌疫情调查和病原菌快速鉴定,判断是由同一克隆株引起的食源性疾病。同年,刘绮明等[6]用相同的方法在中山市鉴定一起由弯曲菌引起的食源性疾病事件,证明是由不同克隆株的空肠弯曲菌感染引起的。深圳地区弯曲菌引起的食物中毒事件报道鲜见,2020年,王文祥等[7]曾用MLST方法对深圳市一起食物中毒事件进行溯源分析,证明是由ST8941空肠弯曲菌引起的急性胃肠炎。然而,基于WGS方法用于食物中毒应急事件的处置在我国深圳地区尚未有报道。WGS是一种基于基因水平的快速简便工具,可快速准确地进行物种鉴定、分析不同个体基因组间的差异、预测细菌耐药和毒力基因以及预测种群演化模型等[8]。
本研究采用全基因组测序的方法对分离的弯曲菌进行二代序列测定,利用The CLC Genomics Workbench(CLC) 12.0 version),工作平台中自带的workflow进行序列比对和菌株鉴定。利用K-mer Tree聚类方法及在线耐药基因数据库、毒力基因数据库等生物信息手段进行分析。分析腹泻病人和其他来源的弯曲菌之间的关联性,了解本地区引起食物中毒的弯曲菌的病原特征。
1 材料与方法
1.1 材 料
1.1.1 样本来源 2022年4月实验室收到南山区某小学食物中毒样本83份,其中病人肛拭子样本20份,厨工等工作人员肛拭子样本17份,剩余食品8份,餐具涂抹样10份,环境涂抹样28份。
1.1.2 主要仪器和试剂 CFX96 Real-Time PCR 仪(美国Bio-Rad公司)、基质辅助激光解吸电离飞行时间质谱仪(MALDI-TOF-MS)(德国BRUKER 公司)、ANOXOMAT MARK II 厌氧微需氧培养系统(荷兰MART 公司)、双箱体培养箱(Thermo公司)、天隆提取仪NP968-S(西安天隆公司)。
天隆DNA/RNA提取试剂盒(西安天隆公司)、双孔滤膜法弯曲菌检测试剂盒(青岛中创公司)、弯曲菌琼脂稀释法最低抑菌浓度MIC检测试剂盒(青岛中创公司)、哥伦比亚血平板(广东环凯公司)、诺如病毒核酸检测试剂盒(深圳澳东公司),所有试剂均在有效期内使用。
1.2 方 法
1.2.1 诺如病毒荧光PCR检测 对肛拭子样本进行诺如病毒荧光PCR检测。在肛拭子样本中各加入1 mL无菌生理盐水充分震荡混匀,吸取200 μL悬液进行核酸提取,采用天隆DNA/RNA提取试剂盒按照说明书进行操作,将提取的核酸用深圳澳东诺如病毒GⅠ/GⅡ型荧光PCR试剂盒进行检测。
1.2.2 其他病原菌检测 参照食品安全国家标准GB4789系列、食物中毒诊断标准及处理原则WS/T 9-1996和感染性腹泻诊断标准WS271-2007对食品、肛拭子、餐具等涂抹样进行副溶血性弧菌、沙门氏菌、金黄色葡萄球菌、志贺菌、蜡样芽胞杆菌、变形杆菌、致泻性大肠埃希氏菌检测。同时对增菌样本吸取1 mL菌悬液8 000 r/min离心1 min收集菌苔,利用煮沸法提取细菌DNA,对其进行荧光PCR检测。
1.2.3 弯曲菌增菌及分离鉴定 采用青岛中创汇科生物科技有限公司生产的弯曲菌检测试剂盒进行弯曲菌分离鉴定。吸取1 mL样本采集液或漂洗液加于4 mL促生长增菌液中,37 ℃微需氧(5% O2,10% CO2,85% N2)培养24 h,分别吸取300 μL滴加Karmali平板和哥伦比亚血平板的滤膜上,约30 min后揭去滤膜,翻转平板,置于微需氧环境37 ℃培养24 h,对可疑菌落进行质谱仪鉴定。
1.2.4 菌株药敏试验及结果判定 药敏试验采用琼脂稀释法为基础的商品化96孔板试剂盒,获得弯曲菌对临床常用的7大类11种抗生素的最小抑菌浓度(MIC)。成品试剂盒将含有不同浓度抗生素的MH琼脂包被到96孔板中。质控菌株为本实验室保存的空肠弯曲菌(ATCC33560)。采用美国国家耐药监测中心(National Antimicrobial Resisitance Monitoring System, NARMS)(http://ars.usda.gov/Main/docs.html docid=6 750)细菌耐药标准来判断弯曲菌的敏感、中介、耐药。耐药折点值:萘啶酸≥64 μg/mL、环丙沙星≥4 μg/mL、四环素≥16 μg/mL、氟苯尼考≥8 μg/mL、氯霉素≥32 μg/mL、克林霉素≥8 μg/mL、红霉素≥32 μg/mL、阿奇霉素≥8 μg/mL、泰利霉素≥16 μg/mL庆大霉素≥8 μg/mL、链霉素≥16 μg/mL。弯曲菌菌株对3类及3类以上抗生素耐药特征为多重耐药性。
1.2.5 菌株测序及溯源分析 鉴定后的弯曲菌,提取全基因组DNA,使用NovaSeq PE150二代测序平台(illumina, San Diego, CA, USA)进行测序。测序后,通过CLC基因组学工作平台v12(QIAGEN)对WGS原始数据进行修剪和组装。简单地说,将样品DNA随机中断以构建用于双端测序的DNA文库。具体参数如下: quality scores cutoff≤0.05,ambiguity number≤2,minimum frequency 2.0%,minimum forward/reverse balance 0.05,minimum average base quality 20.0,minimum contig length 500。3 consecutive bases ≤quality scores≤Q30,则去除低质量读取,然后通过CLC工作平台中自带的workflow对全基因组序列进行序列比对和菌株鉴定。利用K-mer Tree聚类方法,与不同来源标本分离的弯曲菌进行比较,分析传播风险。利用毒力因子数据库VFDB(http://www.mgc.ac.cn/cgi-bin/VFs/v5/main.cgi?func=VFanalyzer)在线分析平台,获得弯曲菌的相关毒力基因数据,通过耐药基因数据库Resfinder(https://cge.food.dtu.dk/services/ResFinder/)、CARD(https://card.mcmaster.ca/analyze)数据库比对获得耐药基因,同时将全基因组序列上传至PubMLST(http://pubmlst.org/)数据库,获取每个菌株7个管家基因的等位基因编号,根据等位基因编号得到ST分型。与数据库比对后没有ST分型结果,则需要将全基因组序列按照模板要求提交至全基因组版块等待系统管理者给出分型结果,即为新发现的ST型。弯曲菌的cgMLST分型是基于弯曲菌核心基因组的1 343个位点的等位基因序列进行分配的。通过cgMLST在线分型网站(https://cge.food.dtu.dk/services/cgMLSTFinder/)获得分型结果,结合Splits Tree4图像化展示菌株间差异,即为菌株的系统发育树。
2 结 果
2.1 病原菌分离鉴定结果 83份样本经过分离、纯培养、质谱仪鉴定,共检出17株弯曲菌,腹泻患者弯曲菌检出率80%(16/20)。其中空肠弯曲菌12株,结肠弯曲菌5株,均来源于病人肛拭子样本,1份病人样本中同时检出空肠弯曲菌和结肠弯曲菌。副溶血性弧菌、沙门氏菌、金黄色葡萄球菌、志贺菌、蜡样芽胞杆菌、变形杆菌、致泻性大肠埃希氏菌均未检出,同时病原菌的荧光PCR核酸结果均为阴性。37份肛拭子样本中,诺如病毒GⅠ型、GⅡ型核酸结果均为阴性。
2.2 菌株抗生素敏感性特征 通过琼脂稀释法测定17株菌株的耐药性,其中空肠弯曲菌最小抑菌浓度(MIC)和耐药菌株的百分比见表1。耐药率最高的是四环素(100%),其次是萘啶酸(91.67%)、环丙沙星(83.34%)和庆大霉素(58.33%)。58.33%(7/12)的空肠弯曲菌具有多重耐药(MDR,对至少3类抗生素药物耐药),空肠弯曲菌的多重耐药模式主要是喹诺酮类/四环素类/氨基糖苷类,而结肠弯曲菌中所有分离株对喹诺酮类、氨基糖苷类、四环素类、大环内酯类、环内酯类抗生素均为耐药。100%的分离株具有多重耐药,见表2。

表1 病人肛拭子样本中分离的空肠弯曲菌MIC和耐药情况Tab.1 MICs and drug resistance of C. jejuni isolated from patient anal swabs

表2 病人肛拭子样本中分离的结肠弯曲菌MIC和耐药情况Tab.2 MICs and drug resistance of C. coli isolated from patient anal swabs
12株空肠弯曲菌中,1株对氯霉素类耐药,其它菌株对红霉素、阿奇霉素、链霉素、氯霉素、氟苯尼考、泰利霉素和克林霉素均敏感。5株结肠弯曲菌中,1株对氟苯尼考耐药,其余4株敏感。4株对克林霉素均耐药,所有结肠弯曲菌对链霉素、氯霉素均敏感。
2.3 弯曲菌毒力基因携带情况 17株弯曲菌用全基因组测序,全部测序成功。17株弯曲菌的基因组大小在1 605 551 bp ~1 749 062 bp,GC含量在30.25%~31.41%,Contigs数量(>500 bp)在15~36。研究发现,100%分离株均携带与粘附相关的cadF、jlpA、porA、pebA基因,所有菌株均携带flaA、flaB、flaC等鞭毛基因,都携带与入侵有关的ciaB基因,然而,在所有分离株中未检测到ciaC基因。所有菌株均携带cdtA、cdtB、cdtC毒力基因,这些基因编码细胞致死性扩张毒素(CDT)。此外,41.18%(7/17)分离株携带跟粘附和侵袭相关的virB11基因,均来源于空肠弯曲菌分离株。
2.4 系统发育及溯源分析 根据系统发育分析,在空肠弯曲菌分离株中主要分为3个分支,58.3%菌株(7/12)集中在分支1,如图1所示。分支1中有6个分离株(S2.1、S3.1、S7.1、S10.1、S11.1、S13.1)均含有β-内酰胺基因、氨基糖苷类基因、四环素类基因和gyrA基因点突变。MLST结果显示空肠弯曲菌分为6个ST型, 8株分离株是新发现的ST型(6株ST-12294、1株ST-12295、1株ST-12296),具体见表3。分支1中的7个分离株携带跟粘附和侵袭相关的virB11基因。与参考菌株相比,分支1中的空肠弯曲菌与2021年克罗地亚地区的人源分离株、2018年深圳地区的鸡源分离株、1997年美国的鸡源分离株高度相关。此外,本次分离的菌株还有报道的ST-760(S14.1,分布于分支1)、ST-464(S9.1、分布于分支2)、ST-22(S4.1、S5.2,分布于分支3)。

图例粉色部分为分支1,绿色部分为分支2,蓝色部分为分支3。从内到外的圆圈分别代表国家、来源、年份、耐药基因及基因点突变。Cam代表深圳地区分离株。图1 12株病人来源空肠弯曲菌和19株参考株的K-mer tree系统发育分析Fig.1 K-mer tree phylogenetic analysis of 12 strains of C. jejuni from patients and 19 reference strains
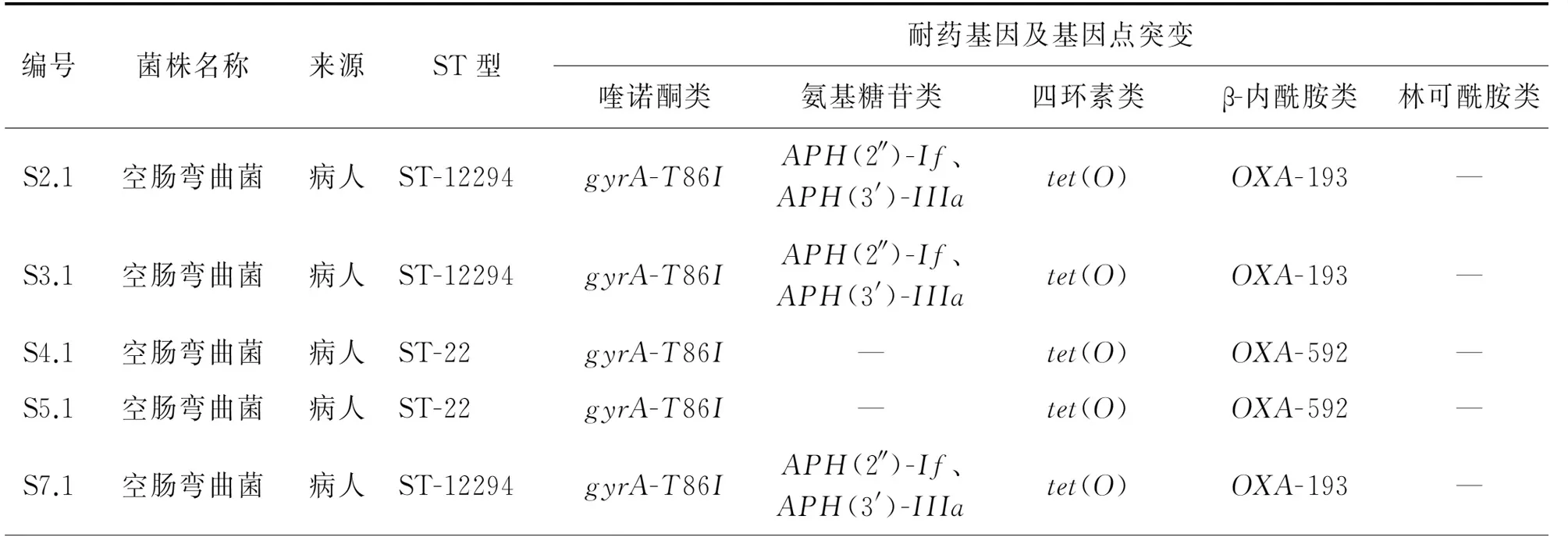
表3 17株弯曲菌分离株耐药基因和ST分型结果表Tab.3 Drug resistance genes and ST typing of 17 Campylobacter isolates
本研究分离的结肠弯曲菌均属于一个进化分支,如图2所示:5个分离株均携带β-内酰胺基因、氨基糖苷类基因、四环素类基因和gyrA基因点突变,在PubMLST数据库比对发现均归属于ST-828型。与参考菌株相比,本次食物中毒病人中分离的结肠弯曲菌与实验室中2018年蛇口市场鸡源分离株高度相关(coli3.1、coli132.2、coli142.1),而与浙江2020年牛粪便来源的分离株进化关系相对较远。

●代表本次分离的结肠弯曲菌,Coli代表深圳地区分离株图2 5株病人来源结肠弯曲菌和8株参考株的K-mer tree系统发育分析Fig.2 K-mer tree phylogenetic analysis of five strains of C. coli from patients and eight reference strains
弯曲菌的cgMLST分型中,5株结肠弯曲菌均属于cgST-40140型,相邻菌株间等位基因差异数在0~1,结合Splits Tree4图像化展示菌株间差异为一个点簇。12株空肠弯曲菌中有6株属于cgST-19646型(S2.1、S3.1、S7.1、S10.1、S11.1、S13.1),菌株间等位基因差异数在0~5;2株属于cgST-1625型(S4.1、S5.2),菌株间等位基因差异数0;1株属于cgST-22232型(S9.1),1株属于cgST-16510型(S14.1),2株属于cgST-44226型(S12.1、S16.1),菌株间等位基因差异数269,聚类分析如图3所示。

图3 基于cgMLST分析的12株病人来源空肠弯曲菌的系统发育树Fig.3 Phylogenetic tree of 12 C. jejuni strains from patients, on the basis of cgMLST analysis
2.5 耐药基因型及其与耐药表型的一致性 本研究发现了3种主要的耐药基因,100%四环素类耐药性、100%β-内酰胺类耐药性、70.59%氨基糖苷类耐药性。100%gyrA基因点突变,所有gyrA基因86位点发生T-I突变。17株弯曲菌均未携带大环内酯类耐药基因。
在空肠弯曲菌中,所有分离株携带gyrA基因点突变、四环素类耐药基因同时表现出100%表型耐药。7个分离株携带氨基糖苷耐药基因同时表现出相应(庆大霉素或链霉素耐药)的表型耐药,然而,有1个分离株携带氨基糖苷耐药基因并未表现表型耐药。研究发现,1个分离株携带5类耐药基因同时表现多重表型耐药。
在结肠弯曲菌中,5个分离株100%对氨基糖苷类、喹诺酮类、四环素类表型耐药,并检测到相应的耐药基因及gyrA基因点突变。然而,结肠弯曲菌分离株对大环内酯类、环内酯类、林克酰胺类表型耐药并未携带相应耐药基因。对17株弯曲菌进行23S rRNA突变位点分析,结果5株结肠弯曲菌中,1株筛查到A2075G位点突变,3株筛查到A2074G位点突变,1株未筛查到23S rRNA基因位点突变。12株空肠弯曲菌均未筛查到23S rRNA基因位点突变。
研究发现所有空肠弯曲菌均携带抗生素外排泵cmeR基因,而在结肠弯曲菌中并未找到。
3 讨 论
弯曲菌是食源性致病菌,大部分以空肠弯曲菌和结肠弯曲菌为主[4]。现代生活中,牲畜中抗生素的不受控制使用,可能会导致具有抗生素耐药的弯曲菌株产生[9]。用于治疗弯曲菌感染的抗菌药物氟喹诺酮类药物(如环丙沙星),也常用于其它腹泻病治疗。弯曲菌对抗菌药物的耐药性正在增加,已成为潜在的公共卫生问题[4]。
本文中,空肠弯曲菌耐药率最高的是四环素(100%),其次是萘啶酸(91.67%)、环丙沙星(83.34%)和庆大霉素(58.33%)。58.33%(7/12)的空肠弯曲菌具有多重耐药,这和Zhang P等[10]的研究一致。而王文祥等[7]在2020年深圳地区空肠弯曲菌食物中毒事件报道中,空肠弯曲菌对萘啶酸和环丙沙星耐药,对其他类药物均敏感。研究中, 80%(4/5)的结肠弯曲菌对除了氯霉素类、林克酰胺类外的其它抗生素均耐药,100%的分离株存在多重耐药现象。两种多重耐药结果和马艳萍等[11]在深圳地区研究腹泻病人和鸡源弯曲菌的耐药结果一致,不同的是,本研究中空肠弯曲菌对氟苯尼考药物均敏感,而马艳萍等研究显示空肠弯曲菌对氟苯尼考的耐药率高于75%。
耐药弯曲菌能够抵抗或降低抗菌药物治疗效果,这将成为严重的公共卫生威胁。弯曲菌在多种动物物种和栖息地中定植和生存的能力使其极难控制[12]。本文在病人中分离的弯曲菌,通过WGS结合耐药基因数据库获得菌株的耐药基因,同时结合琼脂稀释法检测耐药表型,分析耐药基因与耐药表型之间的一致性。研究发现分离株含有多种耐药基因和gyrA基因点突变,主要耐四环素类(100%)、喹诺酮类(100%)、氨基糖苷类(70.59%),同时这3类耐药基因与耐药表型基本一致。此外,所有分离菌均携带β-内酰胺耐药基因。Redondo N等[13]基于WGS对爱尔兰的弯曲菌临床分离株进行了特征分析,发现分离株对喹诺酮类药物(43%)、β-内酰胺类药物(71%)和四环素(25%)具有耐药基因,这与本研究有较大差别。结肠弯曲菌分离株对大环内酯类、环内酯类、林克酰胺类表型耐药并未携带相应耐药基因。据推测,一致性还受计算管道、基因组覆盖水平和抗生素耐药基因类型的影响[14]。研究发现所有空肠弯曲菌均携带抗生素外排泵cmeR基因,这与Rokney A[15]的研究一致。CmeR作为转录抑制因子调节空肠弯曲杆菌多药外排泵cmeABC的表达,cmeR功能缺失突变严重降低了空肠弯曲杆菌定植的能力[16]。然而在结肠弯曲菌分离株中并未找到。
研究发现,弯曲杆菌携带多种毒力因子,弯曲菌的毒力因子与其疾病的严重程度密切相关。实验显示100%的弯曲菌均携带与粘附相关的cadF、jlpA、porA、pebA基因,所有菌株均携带flaA、flaB、flaC等鞭毛基因,都携带与入侵有关的ciaB基因,所有菌株均携带cdtA、cdtB、cdtC毒力基因,这些基因编码细胞致死性扩张毒素(CDT)。此外,41.18%(7/17)分离株携带跟粘附和侵袭相关的virB11基因,均来源于空肠弯曲菌分离株。virB11是弯曲菌pVir质粒上与幽门螺杆菌四型分泌系统组成成分virB的同源性基因,virB11在弯曲菌的染色体上没有同源序列。pVir质粒上virB11的突变导致弯曲菌的黏附和侵袭能力显著降低[17]。Wysok B等[18]用HeLa细胞证实缺乏virB11的菌株表现出轻微的入侵减少。屠丽红等[19]对上海腹泻患者的弯曲菌检测中发现90%以上菌株均携带cadF、flaA、cdtA、cdtB、cdtC等毒力基因,只有7.0%的弯曲菌菌株virB11基因阳性,低于本研究中的携带率。
菌株的分子分型在病原体传播途径的流行病学研究中起着重要的作用。本文基于WGS技术用于食源性疾病暴发溯源,具有准确率高,省事省力等优点,WGS是研究食源性病原体的有力工具。如今在国外WGS数据常用于鉴定弯曲杆菌属,并比较耐药基因型和表型之间的一致性,同时可用于鉴定弯曲菌的毒力因子并进行溯源分析,已成为公共卫生监测和疫情检测的一种快速、经济的工具[20]。然而,基于全基因组测序用于食源性疾病暴发溯源在我国报道鲜见。2022年,章乐怡等[21]在温州地区用PFGE和WGS方法证实了一起食源性疾病事件源于空肠弯曲菌引起的,具有快速、准确的特点。本研究中,根据系统发育分析,在空肠弯曲菌分离株中主要分为3个分支,58.3%菌株集中在一个分支,MLST结果显示空肠弯曲菌分为6个ST型,大部分分离株是新的ST型。该分支中的7个分离株携带跟粘附和侵袭相关的virB11基因。与参考菌株相比,该分支中的空肠弯曲菌与2021年克罗地亚地区的人源分离株、2018年深圳地区的鸡源分离株、1997年美国的鸡源分离株高度相关。据Zhang P[10]等报道,北京地区2017-2018年临床感染病例空肠弯曲菌最常见的为ST-22和ST-760。Nielsen L N等[22]报告丹麦的一位吉兰-巴雷综合征患者感染空肠弯曲菌的型别归为ST-22克隆复合体。本文中5个结肠弯曲菌分离株归属于ST-828型。与参考菌株相比,本次食物中毒病人中分离的结肠弯曲菌与实验室中2018年蛇口市场鸡源分离株高度相关(coli3.1、coli132.2、coli142.1),而与浙江2020年牛粪便来源的分离株进化关系相对较远。菌株ST型多样化表明病原来源复杂,共同导致本次食源性疾病的暴发。本文对17株弯曲菌基于基因组水平的cgMLST分子分型,并与传统的MLST分型方法进行比较,两者分型结果一致性较高,区别在于空肠弯曲菌ST12295和ST12296划分为同一cgST型。弯曲菌cgMLST是以核心基因组的1 343个位点的等位基因序列进行分配的,具有准确性高等特点。该方法是针对弯曲菌暴发溯源分析、同时也是确定暴发的菌株之间关联分析的重要手段,各菌株之间等位基因差异数在cgMLST分型方法中得以显现。本研究是由多克隆型别、同一次事件中引起的食物中毒暴发,在我国报道极为罕见。
本次食物中毒事件未能在可疑餐次的食品中找到病原菌,未能进行食品溯源。但有研究报道,弯曲菌的食源性疾病多与禽畜肉的处理不当及案板、厨具等环境二次污染相关[21]。建议餐饮食品行业严格执行卫生管理制度,做好食品安全存储工作。弯曲菌食源性致病菌的检出为日常工作积累了宝贵的经验,同时此次事件也提醒弯曲菌引起的食源性疾病不容忽视。
利益冲突:无
