单细胞转录组测序分析实训平台架构研究
2023-11-08林长松邵娇芳武剑汪强虎
林长松,邵娇芳,武剑,汪强虎
(南京医科大学,江苏南京 211166)
近些年,随着单细胞转录组测序技术的日趋成熟,每天都会产生海量的单细胞测序数据,如何有效分析并应用这些数据是细胞测序的重点。将单细胞测序数据分析融入本科生课程是生物信息学教学中一个关键问题,理论和实验教学中涉及大量先进的软件和分析方法,教师在教学中发现,本科生在实验平台中进行多次训练可以掌握该知识点,因此,教学团队需要构建性能优越的实训化平台,以满足实验课教学需求。
1 单细胞实验教学中存在的问题
生物信息学专业具有较强的实践性,单细胞转录组测序技术的兴起对教学提出了更高的要求。但该课程的教学尚处于初步探索阶段,未形成一个完整的课程建设体系,仍面临着许多问题和挑战[1]。
1.1 配置生物信息分析环境难度大
单细胞转录组测序分析作为一门新兴学科,其一大特点是会产生海量的数据。不仅从下机数据到后续分析的处理过程复杂,而且配置分析环境也具有较大难度[2]:一是软件数量多,仅R 包就多达几百个;二是软件版本不同,不同版本的软件数据分析结果不尽相同。本科生需要花费大量时间用于配置环境,给教学带来巨大挑战。在实际教学中,为了解决实验分析环境统一化的问题,教学团队基于docker 技术构建了包含R 包和Python 软件的标准化实训平台环境,极大地提高了课堂教学效率。
1.2 课程自主学习难度大
单细胞转录组数据分析知识点复杂,要求学生具备系统的知识体系,而真正掌握该技术需要大量的练习。对于传统教学模式,学生在有限的学时内难以将知识点融会贯通,甚至会消磨学习兴趣,配置分析环境的复杂性更是加大了学习难度。实验课涉及的典型实例,学生在运行时无法理解深层含义,不能达到触类旁通的效果,在分析新的数据时依然感到困难。
1.3 代码纠错能力差
由于涉及大量的分析软件,而每个软件的参数又多而杂,学生难以在众多参数中抓住主要的参数,准确理解软件的各项功能,因此,容易出现无法解决软件系统报错的现象。这就需要引入视频类教学,学生可以通过多次回放视频来理解、记忆如何使用软件,更高效地解决实际运行软件报错的问题。
2 实训化平台的重要性
作为一种轻量级的虚拟化技术[3],docker 在生物信息学分析中被广泛使用,并且目前生物信息公司多使用docker 技术完成流程化分析过程[4]。由于其具有可移植、不依赖操作系统的优势,因此教学团队使用docker 建立了一个容器[5],在此基础上搭建了标准化的生物信息分析实训平台,立足高校实训场景和教学需求,集教学、实训、案例分析、讨论评价于一体。该实训平台在实践过程中不断完善,对于教学和科研具有重要意义。
2.1 实训化平台提供了稳定的生物信息分析环境
教学团队采用docker 技术建立实训平台,在学习过程中为学生提供了跨平台分析的便利,从而提高了教学效率,也极大方便了科研实践。在实验方面,平台不仅包含本科生学习用到的大部分软件,而且提供了相关的分析代码和配置文件等。此外,实训化平台稳定性较强,考虑了不同软件的兼容性和版本问题[6],使得学生在学习过程中专注于数据分析,不受分析环境问题的困扰。而对于学有余力的同学,可以进一步掌握docker 技术,从底层掌握容器构建过程,强化计算机底层知识,提高科研技能,这是传统教学模式所无法做到的[7]。
2.2 实训化平台提高了学生自主学习能力
实训化平台基于容器化技术及影音技术的基础构建而成[8],具有专业化和规范化的特点,整合了丰富的学习资源,学生可以依靠视频讲解进行多次训练,并且通过阅读纠错文档,实现自己修改错误,极大地提高了学生的自主学习兴趣。学生能够在不依赖教师的情况下进行自我探索、自我提高。
对于视频讲解:为了满足当下学生的学习需求,将视频讲解运用到数据分析的教学中,对关键代码的讲解和结果的解读有助于加深学生对知识的理解。图文并茂化的生物信息教学,提高了互动性和代码学习的趣味性,同时为学生提供了自主性的学习平台,使学生能够发挥主观能动性,促进教学优化,提升数据分析的实践质量[7]。
对于纠错文档:实训化平台倡导学生自主阅读纠错文档,减少依赖性,真正理解软件的运行原理,而不仅仅是模仿代码,从而提高学生自主解决问题的能力,会解决错误也是生物信息学人才必备的技能之一。
对于数据来源:实训化平台提供的数据集多来自公共数据集,案例分析具有真实的生物学意义,因此有助于激发学生自主学习和探索的兴趣,鼓励学生从科学问题出发,将生物信息学运用到实际当中[9]。
此外,实训平台提供个性化的课前预习实践,教学视频为学生的课前自主学习提供了帮助,减轻了课后压力。由于单细胞转录组数据分析需要大量的练习时间,所以课前学习有助于学生系统掌握知识体系,同时保证了教学效果。
生物信息学作为交叉学科,要求学生具有较强的自主学习能力,而实训化平台满足了专业未来的发展趋势,立足生物大数据的背景,目标是培养学生自主学习的能力和创新能力。
2.3 实训化平台促进了教学深入实践和探索
目前,国内高校开设单细胞转录组测序课程比较少[10],而且涉及的实验教学内容相对较少,因此可供参考的内容不多,亟需一个标准化教学平台。生物信息学教学团队将容器化技术融入课程教学,根据一线教师的建议及学生的反馈及时完善实训平台,取得了良好的教学效果:不仅实现了学生分析数据能力、自学能力、探索能力和团队合作精神的综合培养,而且进一步加强了实验课教学对理论课的补充与深化作用[11]。
实训化平台以促进教学、规范课程建设、学生实训为目标,提供了完整的单细胞转录组数据分析学习体系,并且充分考虑到了学生选择模块的灵活性和组学分析的个性化特点[12-14]。学生除了按照已有的示例数据和代码进行实验外,还可以基于已有的软件模块,自行设计实验方案,尝试自主写代码、复现文献的实验流程,在具体的案例分析中,使用生物信息学方法尝试解决科学问题。在这个过程中,教师引导学生在实验中发现问题并解决问题,采用文献汇报的方式进行讨论,教师对学生的实验提出建议,形成一个完整的教学模式。实训化平台能够有效帮助教师了解学生的技能掌握情况,提高教学质量,帮助学生掌握单细胞转录组分析的知识体系。
3 模块化结构是实训化平台的基础
生物信息学相关实训化平台的构建需要稳定的软件环境,而模块化结构则是实训化平台的基础。在教学中采用的策略是基于生物信息学分析流程构建实训化平台的模块化结构,从而引导学生系统掌握单细胞转录组数据分析流程,同时为其他生物信息学分析方法的教学如转录组数据分析方法提供借鉴。目前,教学上使用的单细胞转录组数据分析平台分为以下的模块化结构(见图1):
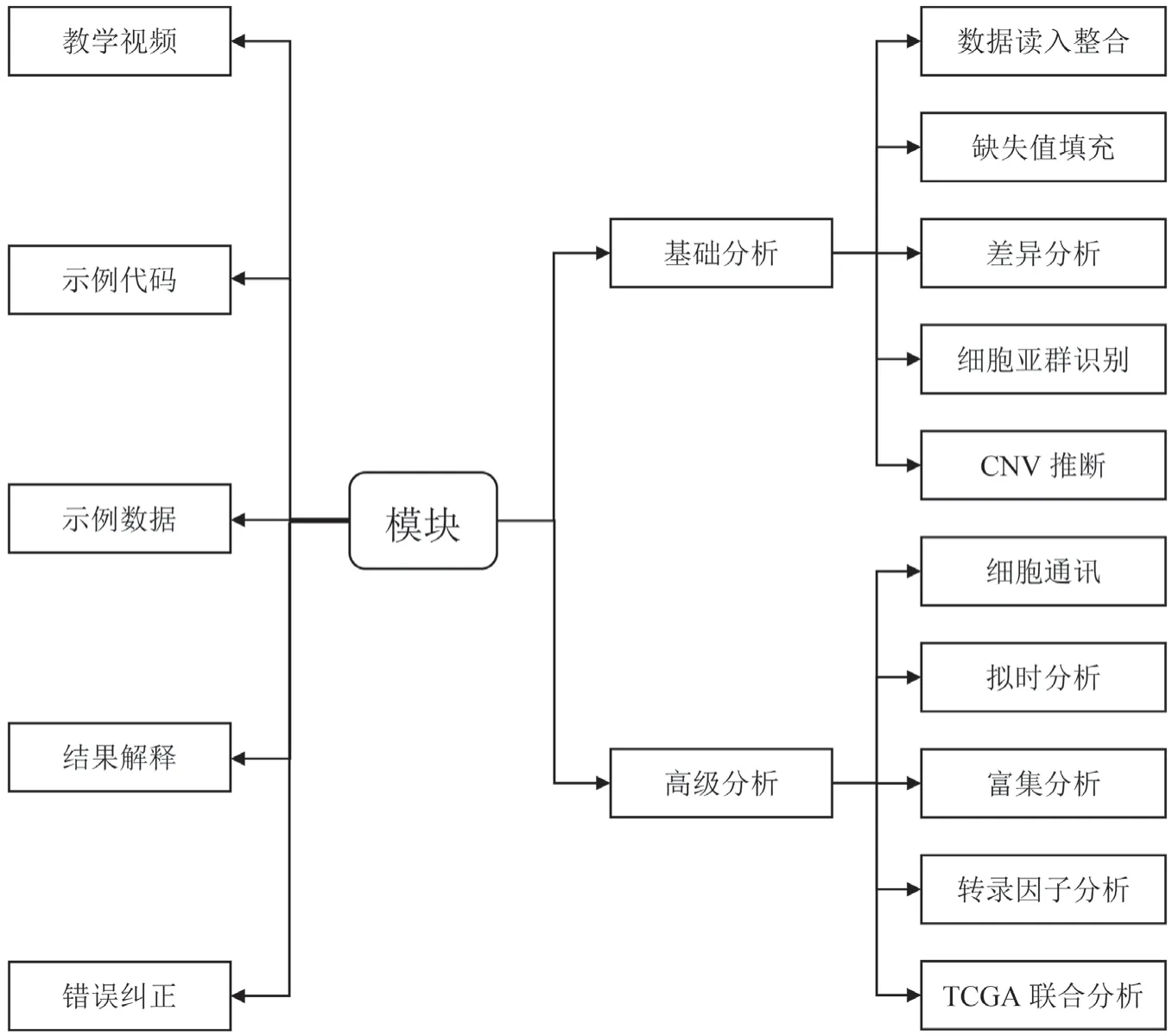
图1 单细胞转录组数据分析平台架构
3.1 单细胞转录组数据的基础分析
(1)数据读入整合。
在实训化平台的相关模块环境中利用Cellranger对单细胞测序数据进行比对,然后根据需要读取的数据格式,基于Seurat 包分别采取Read10X、Read10X_h5、read.table的方法读取比对后的数据。读入数据后,对数据采取以下3 种方法整合:Seurat 整合流程(CCA+ MNN)、Seurat(reciprocal PCA)、Harmony 软件整合操作。
(2)缺失值填充。
由于实际的单细胞测序数据不可避免地会遇到含有缺失值,即表达矩阵较为稀疏的情况,教学中常常会使用scImupute 或VIPER 方法来对数据进行降噪处理,实现缺失值填充的目的。
(3)差异分析。
分别使用FindNeighbors()和FindClusters()函数对整合后的数据做降维聚类分群。接着,对分群单细胞数据通常采用FindAllMarkers()函数做单细胞表达水平的差异分析,从而可以确定每个单细胞亚群特异性高表达的基因。
(4)细胞亚群识别。
基于细胞异质性、细胞共性、单细胞数据库,可以对单细胞亚群做识别鉴定分析。目前,使用较为广泛的单细胞亚群的鉴定方法主要分为以下四类:基于差异分析确定的单细胞亚群特异性高表达基因,利用Marker 基因数据库识别细胞亚群;利用SingleR 或scMCA 或scHCL 等相关R 包来比较样本数据的单细胞基因表达与已知细胞类型的bulk RNA,基于相似性指数的高低确定细胞类型;训练给定的数据集,基于训练的模型半监督地预测目标数据的细胞亚群,通常利用cellassign、axibet 等R 包;利用clusterprofiler、SCSA 等R 包,基于差异基因对marker 基因做富集分析,细胞类型的判断标准是富集程度。
(5)CNV 推断。
对于单细胞基因组拷贝数分析,教学中通常以inferCNV 和CopyKAT 为主要的教学方向。inferCNV主要是用于分析肿瘤样本单细胞转录组数据,分析体细胞水平上拷贝数大规模的变化,而CopyKAT 则是计算每个细胞的基因组拷贝数分析并确定其中的亚克隆结构。
3.2 单细胞转录组的高级分析
(1)细胞通讯。
对于单细胞转录组数据不同细胞亚群之间的相互作用关系,常使用CellPhoneDB、iTALK、CellChat、Garnett、Nichenet 等方法确定配体与受体细胞,从而实现预测配体调控的靶基因与相应受体的目的。
(2)拟时分析。
为获得细胞过渡状态的轨迹,使用monocle2、monocle3、velocyto 或scVelo 做拟时分析,即细胞轨迹分析,基于细胞的基因表达情况识别核心基因,实现对不同分化状态的细胞排序,从而确定细胞的发育轨迹。
(3)富集分析。
单细胞转录组的富集分析分别可以利用GSEA 对差异分析后的样本或GSVA 对表达矩阵计算特定基因集的变异分数。
(4)转录因子分析。
单细胞转录组数据可以使用SCENIC 软件研究转录因子并确定转录因子的调控单元及其调控的相关活性分数。
(5)TCGA 联合分析。
对于单细胞转录组数据分析得到的marker 基因或基因标签等数据,往往会利用公共数据库的bulk RNA 数据进行验证,如结合TCGA 或GEO 数据库对样本数据做生存分析验证。
4 模块的组成
单细胞转录组数据的实训化平台的每个模块都包括教学视频、示例代码、示例数据、结果解释、错误纠正。
(1)教学视频。
教学视频由授课教师按照单细胞转录组数据的分析流程分模块录制,教师在视频中展示如何进入容器的镜像环境、在环境中运行示例代码、运行结果与教学中的常见错误。这种教学方式便于学生课前预习与课后复习,极大地提高了教学效率。
(2)示例代码。
示例代码包含了常见的单细胞转录组数据的分析代码、针对特异性数据的特殊代码以及不同方式的结果呈现代码。
(3)示例数据。
教学中的示例数据覆盖范围较广,包括肺腺癌、宫颈癌、牙周炎等单细胞转录组数据,给予学生一定的自由度以个人科研兴趣为导向进行系统的单细胞转录组数据分析流程的学习,有较好的激励作用。
(4)结果解释。
实训化平台覆盖了对所有示例数据运行结果的解读,从单细胞转录数据本身的相关生物学意义的解释扩展到相关领域的前沿技术或文章的最新进展与研究思路的指导,拓宽了学生的研究思路,拓展了学生科研思维,为后续进一步的学习与深造奠定了良好的基础。
(5)错误纠正。
在错误纠正版块包含了以往教学中常见的错误示范文档与运行代码报错的指导意见,提高了学生的学习效率,有较高的用户友好性。
5 实训化平台在教学中取得的效果
基于docker 容器及多媒体技术构建的单细胞转录组测序数据分析的实训化平台以相关模块结构化为特点,使得学生在可移植的安全封装的稳定生物信息分析环境的基础上,实现示例数据的复现。此外,包括示例数据、结果解释、错误反馈等用户友好型模块,以数据覆盖范围广、联系科研前沿理论与技术、错误反馈及时为特色,拓展了学生的科研思路,提升了教学效率,促进了单细胞转录组数据相关教学的实践与探索。该实训化平台对其他生物信息学分析流程及相关学科的教学有良好的示范作用,有一定的借鉴意义。
