MRI增强序列列线图预测脑胶质瘤IDH1基因突变的研究
2023-11-01郑金晶董海波李明王超超卫雨果
郑金晶,董海波,李明,王超超,卫雨果
MRI增强序列列线图预测脑胶质瘤IDH1基因突变的研究
郑金晶1,董海波1,李明1,王超超1,卫雨果2
1.宁波大学附属李惠利医院 宁波市医疗中心李惠利医院放射科,浙江宁波 315046;2.通用电气药业有限公司,浙江杭州 310000
探讨T1WI MRI增强序列的临床–影像组学列线图预测脑胶质瘤异柠檬酸脱氢酶1(isocitrate dehydrogenase 1,IDH1)基因突变的价值。回顾性分析2016年2月至2022年2月宁波市医疗中心李惠利医院收治的98例经手术病理证实的脑胶质瘤(Ⅱ~Ⅳ级)T1WI MRI增强图像。其中,26例IDH1基因突变型(IDH1-M)、72例IDH1基因野生型(IDH1-W),以7∶3比例划分为训练集(=69)和测试集(=29),使用逻辑回归方法筛选特征并建立临床模型。勾画并测量脑胶质瘤的实质区及坏死区参数,建立Logistic回归影像组学模型,计算Radscore,生成列线图。采用校准曲线和受试者操作特征(receiver operating characteristic,ROC)曲线评价3个模型对脑胶质瘤IDH1基因突变状态的预测效能,进行决策曲线分析,评估列线图临床实用性。训练集经过特征筛选,最终选择6个影像组学特征和2个临床特征用于构建列线图。临床模型在训练集和测试集中的曲线下面积(area under the curve,AUC)分别为0.834和0.718,影像组学模型分别为0.902和0.831,临床–影像组学组合模型最高,分别为0.906和0.857。校准曲线表明,临床–影像组学列线图在训练集中IDH1基因型的预测值和观察值之间具有较好的一致性(=0.751)。决策分析曲线表明,组合模型的净收益在几乎整个Pt值范围内均高于临床模型及影像组学模型。基于MRI T1WI增强序列的临床-影像组学列线图能较精准地预测IDH1基因突变状态。
脑胶质瘤;磁共振成像;异柠檬酸脱氢酶;影像组学;基因型
脑胶质瘤是最常见的脑部原发性恶性肿瘤,约占中枢系统恶性肿瘤的80%,男性的发病率略高于女性,并以老年人多见。异柠檬酸脱氢酶1(isocitrate dehydrogenase 1,IDH1)是脑胶质瘤的一种重要基因分子生物学标志物,分为野生型(IDH1-W)和突变型(IDH1-M)两种表达状态。IDH1基因突变是胶质细胞瘤中最早可检测到的基因改变,突变的IDH蛋白被认为是通过竞争性抑制参与组蛋白和DNA去甲基化,从而阻断了细胞的分化,降低肿瘤细胞的增殖,因此具有相对良好的预后[1-3]。2016年世界卫生组织(World Health Organization,WHO)中枢神经系统肿瘤分类中新增了分子分型,它将IDH突变型弥漫性星形细胞瘤又根据组织学确定为3种不同的分类,即弥漫性星形细胞瘤、间变性星形细胞瘤和胶质母细胞瘤。而2021年第5版进一步推进了分子诊断的作用,它将所有IDH突变型弥漫性星形细胞瘤归为一种类型,再分为2~4级,与此同时,分子特征可以独立于组织学特征来确定是否归为4级。因此,治疗前精准预测脑胶质瘤IDH1是否突变,对指导个体化治疗和预后评估具有重要意义,成为近年来影像基因组学研究热点[4-5]。但是既往相关研究多以肿瘤整体和(或)水肿区作为感兴趣区提取影像组学特征,没有全面、完整地反映肿瘤异质性的特征,准确性有待于提高[6-7]。本研究分别勾画增强MRI肿瘤实质区域(region of contrast-enhanced tumor,rCET)、坏死区域(region of necrosis,rNec),并将临床特征、MRI影像学特征与影像组学标签相结合,构建预测模型、绘制列线图,评估基于T1WI MRI增强序列的临床–影像组学列线图预测脑胶质瘤IDH1基因突变的价值。
1 资料与方法
1.1 临床资料
选取2016年2月至2022年2月宁波市医疗中心李惠利医院收治的符合以下标准的患者:①经手术病理证实为脑胶质瘤;②获得患者的临床特征及IDH1免疫组织化学结果;③术前行常规T1增强MRI序列检查,图像无明显伪影;④术前无放化疗史。排除标准:①肿瘤内有出血或钙化者;②肿瘤无坏死区或强化程度较弱者。共98例脑胶质瘤患者入组,其中,男57例,女41例;IDH1突变型26例,IDH1野生型72例。按7∶3比例随机分为训练集(69例,其中IDH1-M型19例,IDH1-W型50例)和测试集(29例,其中IDH1-M型7例,IDH1-W型22例)。本研究经宁波市医疗中心李惠利医院伦理委员会批准(伦理审批号:KY2022PJ184),并获得免除知情同意书许可。
1.2 仪器与检查方法
采用GE Discovery 3.0T MRI磁共振扫描仪(美国GE Health Care公司)和8通道头线圈。①常规MRI平扫序列:T1WI序列:TE 24ms,TR 1850ms,TI 780ms;T2WI序列:TE 105ms,TR 6656ms;矩阵288×224,FOV 240mm×240mm,层间距1mm,层厚4mm,扫描层24层;②DWI序列:TR 4500ms,TE为最小值,b值设定1000s/mm2,矩阵160×160,FOV 240mm×240mm,层间距1.5mm,层厚5mm,扫描层数24层;③T1WI增强MRI序列:注入对比剂钆双胺注射液(德国Bayer Schering Pharma公司)0.2ml/kg后,横断面T1WI序列扫描,参数同平扫T1WI序列。
1.3 图像采集处理、影像组学特征的提取及模型建立
收集医院影像归档和通信系统(picture archiving and communication system,PACS)中的脑胶质瘤MRI影像后,应用美国GE公司AI-Kit(Artificial Intelligence Kit,Version:3.3)软件进行图像去噪、去骨、偏差矫正等预处理。由2位神经影像医师在开源软件ITK-SNAP(Version 3.4.0)上对T1WI增强图像逐层勾画,并由1名资深神经影像学专家复核,选择勾画感兴趣区(volume of interest,VOI)。实质区为T1增强图像呈强化的区域;坏死区为T1WI上低信号、T2WI上高信号且T1增强图像未强化的区域(图1)。
应用AI-Kit软件对MRI图像特征提取,提取了396个影像组学特征,包括直方图特征(histogram features,HF)、灰度共生矩阵(grey level co-occurrence matrix,GLCM)、灰度游程矩阵(grey level runlength matrix,GLRLM)等。每例患者包括实质区和坏死区2个VOI,共采集792个有效组学特征参数。
在R软件(v.4.1.0)中完成特征选择和模型构建。选择用于构建模型的非零系数特征参数用于预测IDH1基因表达类型,将具有非零系数的特征组合起来构建一个公式,该公式可用于计算每例脑胶质瘤的Radscore,使用Wilcoxon检验比较不同IDH1基因状态Radscore的差别。
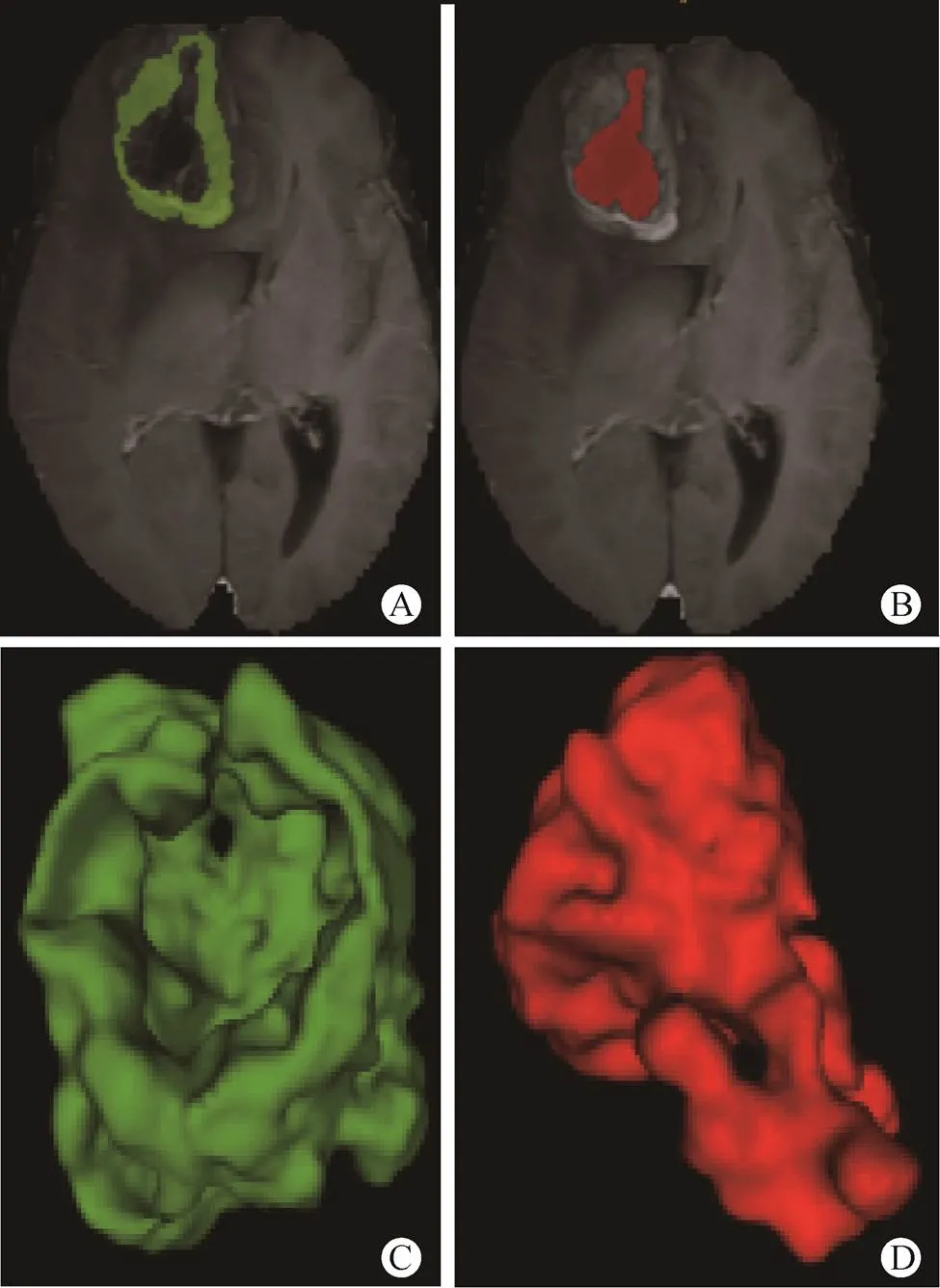
图1 VOI示意图
A.实质区的最大切面图;C.其对应的3D可视化图;B.坏死区最大切面图;D.代表其对应的3D可视化图
1.4 统计学方法
在R软件中将Radscore与筛选出的临床预测因子进行多元Logistic回归分析,构建预测IDH1基因表达类型的组合模型,利用受试者操作特征(receiver operating characteristic,ROC)曲线对以上3种预测模型诊断效能进行评估。校准曲线和Hosmer- Lemeshow检验用于评估IDH1基因型的列线图预测概率与实际结果之间的一致性,若校准曲线的拟合度较好,且>0.05,说明模型的预测性能较好。决策曲线用于评价临床决策的净获益情况,并与临床模型进行比较。
2 结果
2.1 构建临床模型及效能评估
训练集和测试集的IDH1基因状态、WHO分级情况及临床特征比较,差异均无统计学意义(表1)。经单因素Logistic回归分析,训练集中IDH1-M组和IDH1-W组的年龄(=0.023)、肿瘤位置2(=0.032)比较,差异有统计学意义。经多因素Logistic回归分析后,仍选择年龄(=0.007)和肿瘤位置2(=0.012)构建临床模型。临床模型在训练集和测试集中预测IDH1状态的曲线下面积(area under the curve,AUC)分别为0.834和0.718(图2);训练集的敏感度、特异性和准确率分别为68.4%、88.0%及82.6%,测试集分别为57.1%、81.8%及75.9%。
2.2 构建影像组学模型及效能评估
对792个有效组学特征参数进行最小冗余最大相关性(mRMR)算法,得到30个特征,再进行最小绝对收缩和选择算子(leastabsolute shrinkage and selection operator,LASSO)逻辑回归(图3),得到6个非零系数的组学特征来构建影像组学模型,分别计算训练集及测试集每个患者的Radscore,结果显示,IDH1-M组的Radsore低于IDH1-W,差异有统计学意义(图3D)。影像组学模型在训练集的敏感度、特异性和准确率分别为89.5%、82.0%、84.0%,测试集为71.4%、63.6%、65.5%。模型在训练集中的AUC值为0.902,测试集的AUC值为0.831。
2.3 构建临床-影像组学组合模型及列线图
在临床–影像组学组合模型的构建过程中,Logistic回归分析将Radscore、年龄和肿瘤位置2确定为独立预测因子,在此基础上构建可视化临床–影像组学列线图(图4)。与影像组学模型和临床模型相比,临床–影像组学组合模型达到最高AUC[训练集:0.906(95%:0.834~0.991);测试集:0.857(95%:0.707~0.995)],敏感度、特异性和准确率训练集分别为84.2%、90.0%、88.4%;测试集为60.0%、94.7%、82.8%。影像组学–临床列线图的校准曲线表明,训练集和测试集中IDH1基因突变的预测和实际测得值之间都具有较好一致性(分别为0.751、0.196,图5)。3个模型的决策分析曲线表明,组合模型的净收益在几乎整个Pt值范围内都高于其他2个模型(图6)。
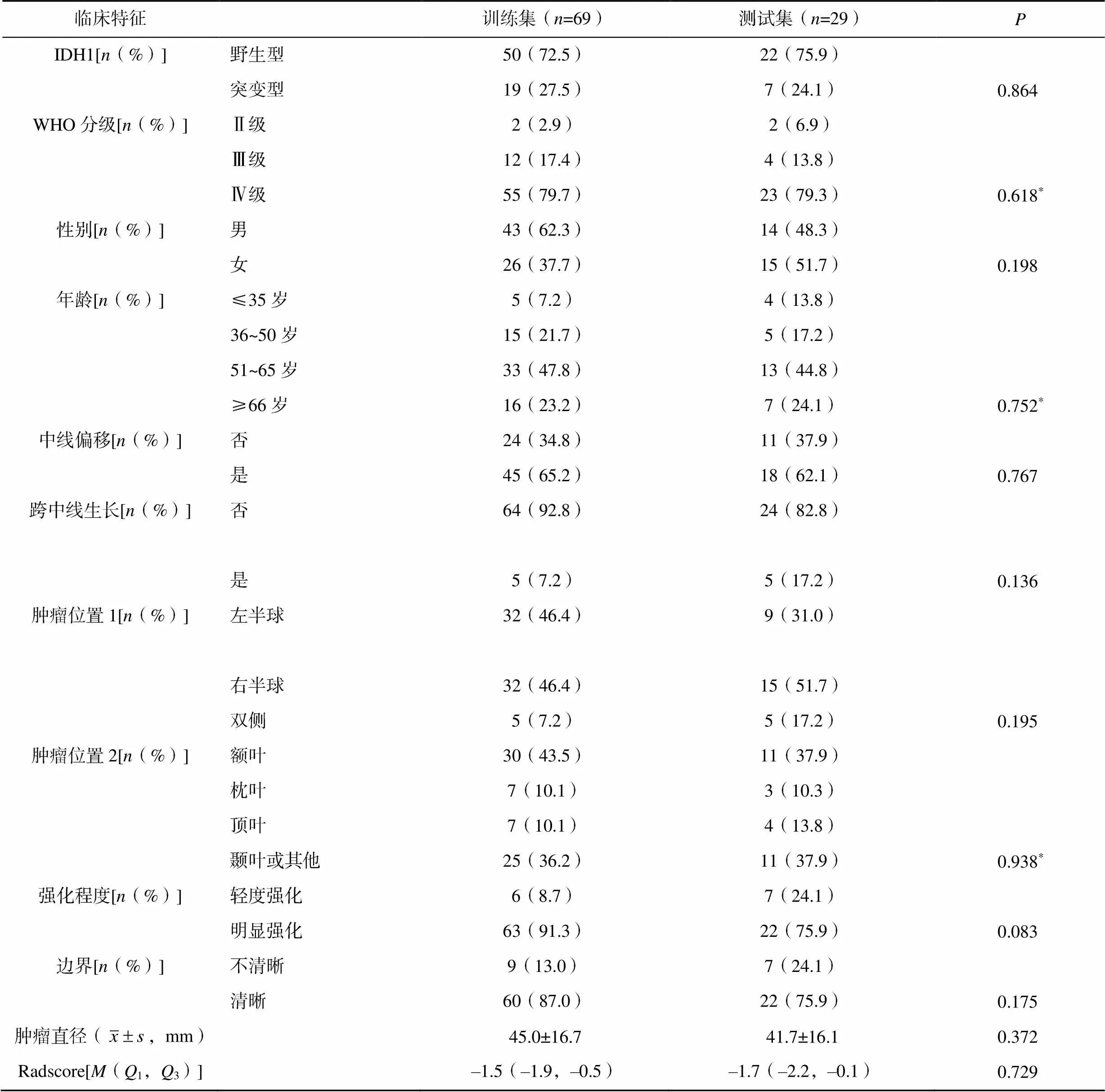
表1 训练集与测试集临床特征比较
注:*采用Fisher确切概率法
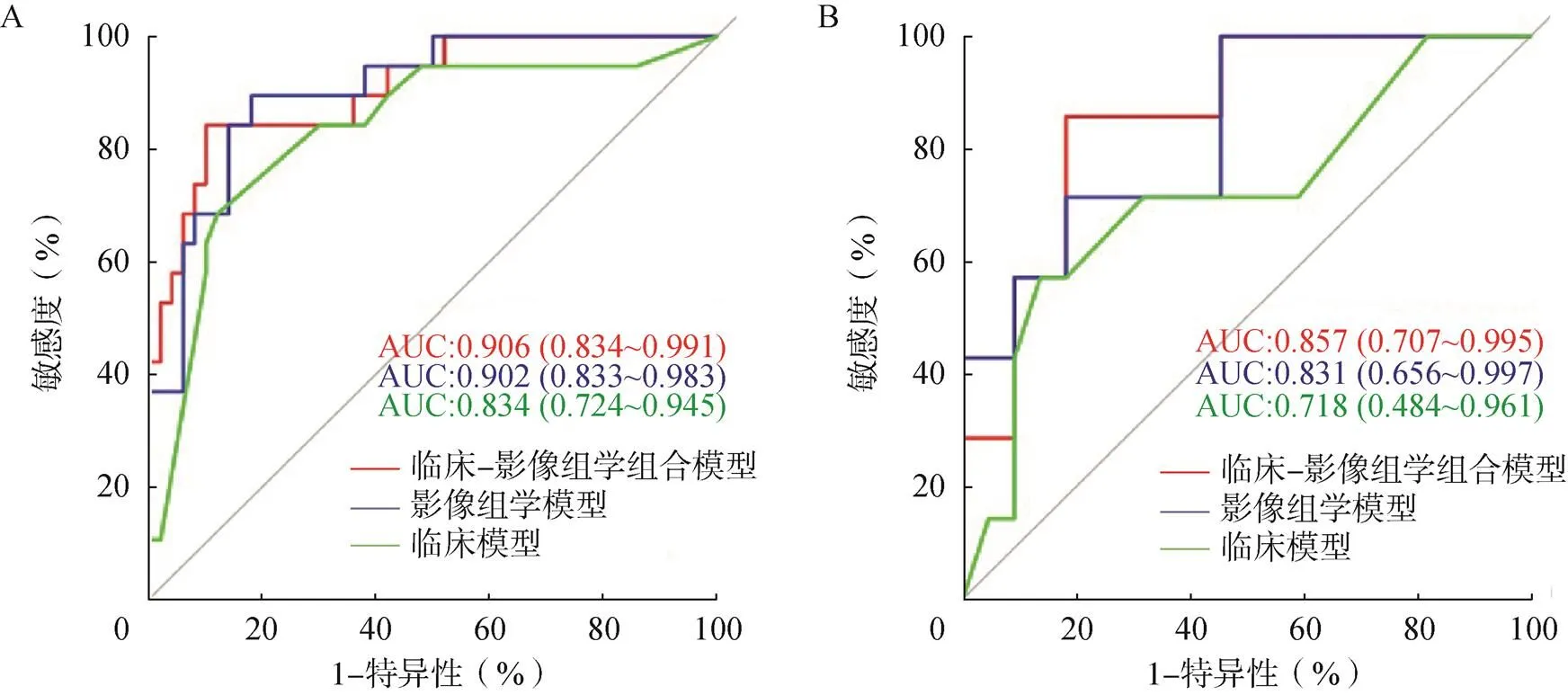
图2 训练集(A)和测试集(B)中3种模型的ROC曲线
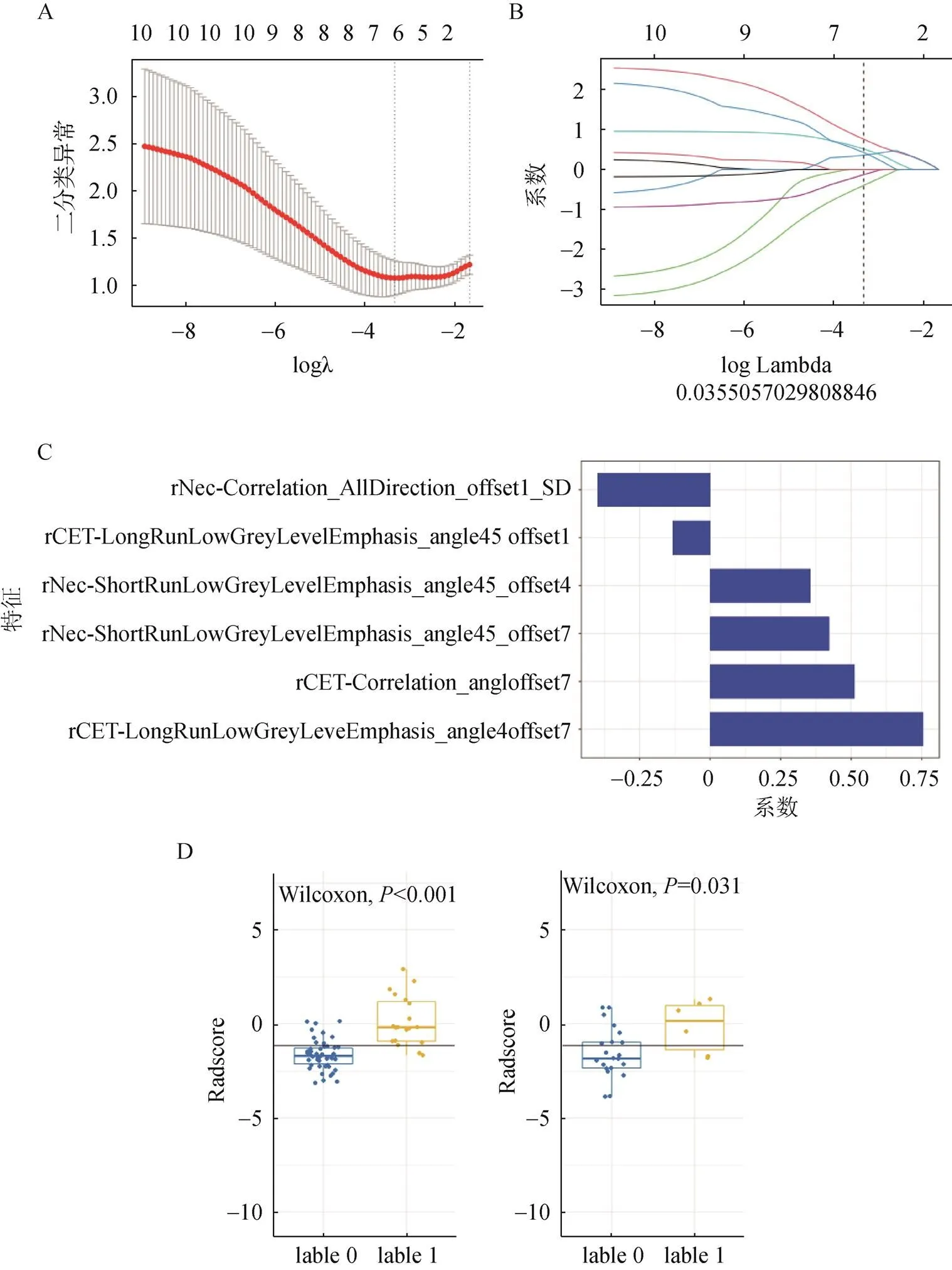
图3 影像组学特征LASSO降维图
A.利用十折交叉验证算法求得最佳惩罚系数λ,使其对应的特征集合具有最佳分类效能;B.系数收缩图,横轴是log λ,纵轴表示系数;C.最终筛选出的6个非零系数特征及其对应系数;D.Radscore在不同IDH1突变状态下的区别,lable0代表野生型,lable1代表突变型,左边为训练组,右边为测试组。Radscore的截止值为–1.15
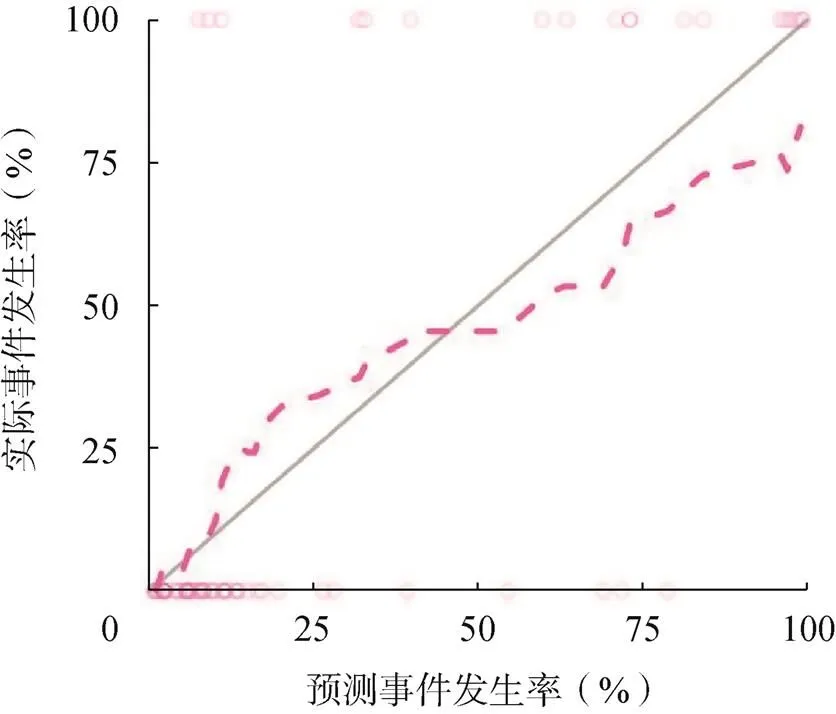
图5 临床-影像组学列线图的校准曲线
3 讨论
近年来,对脑胶质瘤分子结构的深入研究揭示了其特有的遗传特征和表观遗传学表现,并据此划分为不同的分子亚型,表观遗传与DNA的甲基化有关,IDH1基因能诱导去甲基化,因此,表观遗传调节IDH1基因成为肿瘤分类的关键生物学标志物,在脑胶质瘤进化和生物学表现起着关键的作用[8]。Yao等[9]关于IDH1-R132H突变与神经胶质瘤干细胞(glioma stem cells,GSC)的研究发现,IDH1-R132H的过表达会导致GSC增殖、迁移和侵袭减少,诱导细胞凋亡并改善GSC分化,而该位点的突变发生于80%以上的IDH1-M脑胶质瘤。因此,IDH1-M脑胶质瘤患者预后较好。Yan等[10]研究显示,IDH1-M脑胶质瘤患者的中位生存期明显高于IDH1-W。Su等[11]研究发现,IDH1-M脑胶质瘤患者平均年龄低于IDH1-W。本研究中,年龄特征也显示出了较强的预测强度,意味着结合年龄特征的列线图具有更好的预测能力[12]。Qi等[13]研究发现,IDH1-M脑胶质瘤主要位于额叶,本研究亦有类似发现。因此,本研究把年龄和位置加入到组合模型中来,期望提高其预测能力。
Yu等[14]关于Ⅱ级脑胶质瘤IDH1基因突变的影像组学研究,将脑胶质瘤分级作为重要特征纳入研究,构建的影像组学模型AUC值为0.860,但是脑胶质瘤的分级因素需要经过手术或活检后病理获取,本研究是基于无创的影像组学,因此未将胶质瘤分级作为临床特征纳入研究。
一部分相关研究是基于勾画脑胶质瘤的整体区域用于构建影像组学模型预测IDH1基因突变[14-15]。笔者认为,肿瘤rCET和rNEC包含细胞密度、微血管增殖和局部微环境的信息存在较大差异,分别提取rCET和rNEC影像组学特征可以更好地量化肿瘤的综合信息,表征脑胶质瘤的异质性。本研究中,提取的6个组学特征中各有3个来自于rCET和rNEC,来自rCET的特征系数总和为1.399,rNEC的系数总和为1.177,特征系数的绝对值可以看作是某个特征对预测IDH1基因突变风险的重要性,绝对值越大,对分类效果的影响越显著,笔者认为,rCET是肿瘤细胞密度较高、生长较活跃及异质性较明显的区域,因此该区域对于IDH1基因突变预测效能较rNEC贡献更大。在这6个特征中,包括了4个GLRLM特征和2个GLCM特征,是影像组学较常使用的特征类型,从图像的精细程度和均匀程度反映了肿瘤的影像信息,定量地描述肿瘤的内部特征。GLRLM特征表示纹理在预设方向上的粗糙程度,其特征系数总和达1.665,笔者推测其占比较高的原因可能是这一类型下多种特征的互补从不同灰度值及方向上反映rCET和rNEC的信号强度和肿瘤的不均质性,从而更好地实现模型的预测效能。GLCM特征系数总和0.911,均为相关性特征,该特征可以显示灰度值与GLCM中各自的体素之间的线性相关性,相关度愈大,矩阵元素值愈均匀,在本研究中,这意味着IDH1-M较IDH1-W的内部结构更加均匀,说明IDH1-M异质性低,而IDH1-W较高。
本研究构建的临床–影像组学组合模型在训练集和测试集中的AUC值分别为0.906和0.857,校准曲线的拟合度较好(=0.751),表明组合模型的预测能力好于单独的模型[11-12]。决策曲线分析显示,组合模型的临床净收益优于单一模型。因此,笔者认为基于分别勾画rCET和rNEC建立的临床–影像组学组合模型能够较全面、完整地反映胶质瘤的异质性,在预测IDH1基因状态方面可能具有较高的准确度。
本研究尚有一些局限性,首先,本研究为单中心研究,样本量还不够大,模型的效率和稳定性还有待于提高,下一步将加大样变量继续研究;其次,勾画VOI无法避免各种组织类型的重叠,例如,一些没有强化的肿瘤组织可能与水肿组织重叠,对结果可能造成影响。
综上所述,基于MRI T1WI增强序列的临床–影像组学列线图能较精准地预测IDH1基因突变状态。
[1] OHGAKI H, KLEIHUES P. The definition of primary and secondary glioblastoma[J]. Clin Cancer Res, 2013, 19(4): 764–772.
[2] TURCAN S, ROHLE D, GOENKA A, et al. IDH1 mutation is sufficient to establish the glioma hypermethylator phenotype[J]. Nature, 2012, 483(7390): 479–483.
[3] WAITKUS M S, DIPLAS B H, YAN H. Biological role and therapeutic potential of IDH mutations in cancer[J]. Cancer Cell, 2018, 34(2): 186–195.
[4] CHOI Y S, BAE S, CHANG J H, et al. Fully automated hybrid approach to predict the IDH mutation status of gliomas via deep learning and radiomics[J]. Neuro Oncol, 2021, 23(2): 304–313.
[5] WANG K, WANG Y, FAN X, et al. Radiological features combined with IDH1 status for predicting the survival outcome of glioblastoma patients[J]. Neuro Oncol, 2016, 18(4): 589–597.
[6] LAO J, CHEN Y, LI Z C, et al. A deep learning-based radiomics model for prediction of survival in glioblastoma multiforme[J]. Sci Rep, 2017, 7(1): 10353.
[7] NIU L, FENG W H, DUAN C F, et al. The value of enhanced MR radiomics in estimating the IDH1 genotype in high-grade gliomas[J]. Biomed Res Int, 2020, 2020: 4630218.
[8] GUSYATINER O, HEGI M E. Glioma epigenetics: from subclassification to novel treatment options[J]. Semin Cancer Biol, 2018, 51: 50–58.
[9] YAO Q, CAI G, YU Q, et al. IDH1 mutation diminishes aggressive phenotype in glioma stem cells[J]. Int J Oncol, 2018, 52(1): 270–278.
[10] YAN H, PARSONS D W, JIN G, et al. IDH1 and IDH2 mutations in gliomas[J]. N Engl J Med, 2009, 360(8): 765–773.
[11] SU X, SUN H, CHEN N, et al. A radiomics-clinical nomogram for preoperative prediction of IDH1 mutation in primary glioblastoma multiforme[J]. Clin Radiol, 2020, 75(12): 963.e7–963.e15.
[12] TAN Y, ZHANG S T, WEI J W, et al. A radiomics nomogram may improve the prediction of IDH genotype for astrocytoma before surgery[J]. Eur Radiol, 2019, 29(7): 3325–3337.
[13] QI S, YU L, LI H, et al. Isocitrate dehydrogenase mutation is associated with tumor location and magnetic resonance imaging characteristics in astrocytic neoplasms[J]. Oncol Lett, 2014, 7(6): 1895–1902.
[14] YU J, SHI Z, LIAN Y, et al. Noninvasive IDH1 mutationestimation based on a quantitative radiomics approach for grade Ⅱglioma[J]. Eur Radiol, 2017, 27(8): 3509–3522.
[15] HSIEH K L, CHEN C Y, LO C M. Radiomic model for predicting mutations in the isocitrate dehydrogenase gene in glioblastomas[J]. Oncotarget, 2017, 8(28): 45888–45897.
Prediction of IDH1 gene mutation in glioma using nomogram based on MRI enhanced sequence
ZHENG Jinjing, DONG Haibo, LI Ming, WANG Chaochao, WEI Yuguo
1.Department of Radiology, Ningbo Medical Center Lihuili Hospital, Ningbo University, Ningbo 315046, Zhejiang, China; 2.General Electric Pharmaceutical Co., Ltd, Hangzhou 310000, Zhejiang, China
To investigate the value of clinical-radiomics nomogram based on T1WI MRI enhanced sequence in predicting isocitrate dehydrogenase 1 (IDH1) gene mutation in glioma.T1WI MRI enhanced images of 98 cases with glioma (grade Ⅱ-Ⅳ) which were received and treated by Ningbo Medical Center Lihuili Hospital from February 2016 to February 2022, confirmed by operation and pathology were analyzed retrospectively, among them, 26 cases of IDH1 gene mutants type (IDH1-M) and 72 cases of IDH1 gene wild type (IDH1-W) were divided into training set (=69) and test set (=29) in a 7∶3 ratio, Logistic regression methods were used for screening the features and the clinical model was established. The parenchymal area and necrotic area of glioma were delineated and parameters were measured to establish a Logistic regression radiomics model, calculate Radscore, and generate the nomogram. Receiver operating characteristic (ROC) curve was used to evaluate the prediction efficiency of the three models for IDH1 gene mutation status, and the decision curve was analyzed to evaluate the clinical usefulness of the nomogram.After feature screening, six imaging features and two clinical features were selected to construct the nomogram. Area under the curve (AUC) of clinical model in training set and test set were 0.834 and 0.718 respectively. AUC of radiomics model were 0.902 and 0.831, respectively. AUC of clinical-radiomics combined model were 0.906 and 0.857, respectively. The calibration curve proved that there was a good agreement between the predicted and observed values of IDH1 genotypes in the training set (=0.751). The decision curve analysis curve showed that the net benefit of the combined model was higher than that of the clinical model and the radiomics model in almost the entire Pt range.Clinical-radiomics nomogram based on MRI T1WI enhanced sequence can accurately predict IDH1 gene mutation status.
Glioma of the brain; Magnetic resonance imaging; Isocitrate dehydrogenase; Radiomics; Genotype
R445.2
A
10.3969/j.issn.1673-9701.2023.29.001
浙江省医药卫生科技项目(2023KY1047,2017KY572);宁波市科技惠民项目(2016C51017)
董海波,电子信箱:donghb18@sina.com
(2023–04–07)
(2023–04–29)
