绿豆象热激蛋白超家族基因的鉴定及表达分析
2023-10-25张昕杨星宇张超然张冲郑海霞张仙红
张昕,杨星宇,张超然,张冲,郑海霞,张仙红
绿豆象热激蛋白超家族基因的鉴定及表达分析
张昕,杨星宇,张超然,张冲,郑海霞,张仙红
山西农业大学植物保护学院,山西太谷 030801
【目的】鉴定绿豆象()热激蛋白(heat shock protein,HSP)超家族基因成员,明确高、低温胁迫后HSP基因在绿豆象中的表达变化,为深入挖掘HSP基因功能提供理论依据。【方法】从Insect Base 2.0下载不同昆虫HSP基因的CDS和蛋白序列,并以此为参考在绿豆象全长转录组测序数据库中进行本地BLASTp和tBLASTn比对搜索,同时结合HMMER和关键词两种方法再次筛选目标序列,最终完成搜索结果的汇总。利用CDD、MEGA、ProtParam等在线分析工具对绿豆象HSP超家族基因进行生物信息学分析。根据绿豆象成虫高、低温转录组测序数据筛选出7个候选,采用实时荧光定量PCR(qRT-PCR)技术比较分析其在绿豆象不同虫态(幼虫、蛹、成虫)及不同温度胁迫下的表达特性。【结果】共鉴定出31个HSP基因,其中包括3个HSP90、8个HSP70、8个HSP60和12个sHSP(small HSP)。理化性质分析显示编码的蛋白质包含159—776个氨基酸残基(aa),分子量介于18.4—88.9 kDa,理论等电点为4.95—9.17。亚细胞定位结果显示多数定位于细胞质中,少数基因定位于线粒体基质、内质网和细胞核。系统发育分析表明绿豆象热激蛋白不同家族成员与其他昆虫的热激蛋白进化关系较近,显示了其在进化上的保守性。qRT-PCR分析发现,不同虫态在不同温度胁迫后7个候选差异表达。经高温胁迫后在雌、雄成虫体内的表达量分别上调1 000和500倍;-经高温胁迫后在雌、雄成虫体内的表达量分别上调500和450倍;幼虫经高、低温胁迫后,和-表达差异显著。【结论】通过绿豆象全长转录组测序数据共鉴定出31个完整的热激蛋白超家族基因成员,分为4个亚家族,家族间蛋白结构、保守结构域和基因表达特征存在差异。可能在成虫抵御高温胁迫中行使重要功能,而幼虫的高温耐受性可能与的表达差异有关。
绿豆象;全长转录组测序;热激蛋白;基因家族;生物信息学分析;表达分析
0 引言
【研究意义】绿豆象()属鞘翅目(Coleoptera)豆象科(Bruchidae)瘤背豆象属(),是世界范围内危害储藏豆类最严重的害虫之一。该虫对环境温度变化具有很强的适应能力,50和-20 ℃的极限温度并不会引起各虫态绿豆象的立即死亡,且蛹在50和-20 ℃的极限温度下仍可生存约3.71和0.85 h[1]。热激蛋白(HSP)是昆虫生存的重要调节因子,在昆虫抵抗环境胁迫以及生长、发育和繁殖过程中均发挥重要作用[2-5]。据报道,HSP基因的表达不同程度地被诱导上调是昆虫抵御温度胁迫的重要机制[6-8],因此对绿豆象热激蛋白超家族基因成员进行鉴定和表达模式分析,对明确绿豆象抵御温度胁迫的机制具有重要意义。【前人研究进展】当昆虫暴露于热、冷和其他环境胁迫刺激时,其体内HSP基因的表达被诱导和调节以响应环境的胁迫,如韭菜迟眼蕈蚊()在经历40 ℃高温处理时,其体内HSP70和sHSP等分子伴侣协同上调[9];美洲斑潜蝇()在热胁迫和冷胁迫下,编码热激蛋白和角质层蛋白的基因分别显著上调[10];亚洲柑橘木虱()中热激蛋白和解毒酶等基因在热胁迫后表达量上调,可能参与了对热胁迫的反应和保护[11]。此外,LI等[12]研究发现,沉默松墨天牛()其成虫的耐热性显著降低,可能在成虫的热抗性中发挥作用;Dong等[13]利用RNAi方法沉默二化螟()表达,发现与27 ℃饲养温度及阴性对照相比,39 ℃高温和-11 ℃低温均导致幼虫存活率降低,表明可提高二化螟的环境胁迫耐受性和生理调节活性。全基因组和转录组学测序分析是鉴定基因家族的主要方式,广泛应用于动、植物的基因鉴定及表达水平研究,如Xu等[14]基于全基因组分析对重要的蛀干害虫光肩星天牛()HSP超基因家族成员进行了鉴定;Wang等[15]确定了烟粉虱()HSP家族成员的数量,并明确热/冷胁迫诱导或者抑制了HSP的表达。【本研究切入点】绿豆象各虫态均具有很强的耐热性和耐寒性。45 ℃胁迫下完全消灭绿豆象需要32 h,而50 ℃下4 h即可完全防治绿豆象[1]。在极限低温下其幼虫、蛹和成虫分别可在-16 ℃ 45 min、-20 ℃ 30 min、-26 ℃ 20 min死亡率达100%[16]。但有关绿豆象HSP基因家族的鉴定及功能尚未见报道。【拟解决的关键问题】基于绿豆象三代全长转录组测序数据,鉴定其HSP超家族基因成员。在此基础上,利用实时荧光定量PCR(qRT-PCR)进行绿豆象不同虫态在不同温度胁迫下HSP基因的表达研究,为进一步探究绿豆象HSP超家族基因成员的功能及其耐热、耐寒机制提供理论依据。
1 材料与方法
试验于2021年4月至2022年6月在山西农业大学植物保护学院完成。
1.1 昆虫饲养与转录组测序
供试绿豆象为山西农业大学植物保护学院昆虫重点实验室连续饲养数代的稳定种群。饲养条件:光照培养箱(LRH-100CB,上海一恒科学仪器有限公司,中国),温度(28±1)℃,相对湿度75%—80%,光周期16L﹕8D。
挑选羽化24 h的绿豆象雌、雄成虫,配对后置于装有无卵绿豆的培养皿中,接虫24 h后取出绿豆象,将带卵绿豆置于上述恒温光照培养箱中进行饲养。根据前期研究结果,选取产卵后13 d(幼虫期)、23 d(蛹期)以及羽化1 d的成虫分别进行高温45 ℃(金属水浴锅HH-S2,常州国宇仪器制造有限公司,中国)和低温-3 ℃(低温培养箱LRH-150CA,上海一恒科学仪器有限公司,中国)胁迫处理3 h。处理后的绿豆象与27 ℃饲养的绿豆象各虫态样品均匀混合后液氮速冻进行三代全长转录组测序(百迈客生物科技有限公司,中国北京,测序数据未发表)。同样取上述高、低温胁迫后的绿豆象成虫与27 ℃饲养的成虫构建二代转录组测序,用于基因的定量分析(测序数据已上传至NCBI,生物项目编号:PRJNA810268,编录号:SRR18148874—SRR18148882)。共计10个样品用于转录组测序:1个混合样品(全长转录组测序);9个处理样品(3个样品为高温45 ℃处理组:B1、B2、B3;3个样品为低温-3 ℃处理组:C1、C2、C3;3个样品为27 ℃对照组:A1、A2、A3)。
1.2 绿豆象HSP超家族基因的鉴定和序列分析
1.2.1 基因家族的鉴定 为准确鉴定绿豆象HSP超家族基因成员,从昆虫基因组网站http://v2.insect- genome.com/Genomeo[17]基因家族模块中下载不同昆虫的HSP蛋白序列和CDS序列,以下载的HSP数据为目标序列进行本地BLASTp和tBLASTn比对。通过Pfam蛋白数据库(http://xfam.org/)下载HSP90(PF00183)、HSP70(PF00012)、HSP60(PF00118)和HSP20(PF00011)的隐马尔可夫模型(HMM)文件,并以-value<10-5作为筛选标准,使用HMMER3.0进行绿豆象蛋白序列中4个结构域的搜索[18]。同时,使用关键词“Heat shock protein”和“Hsp”于转录组测序数据中查找,利用NCBI的CDD数据库(https://www.ncbi.nlm.nih.gov/Structure/cdd/wrpsb.cg)和SMART数据库(http://smart.embl-heidelberg. de/)进一步对候选的绿豆象目标序列进行保守结构域的确定。
1.2.2 序列理化性质分析和系统发育树的构建 分别使用ORFfinder(https://www.ncbi.nlm.nih.gov/orffinder/)和ProtParam(https://web.expasy.org/protparam/)对鉴定出的绿豆象HSP超家族基因序列进行理化性质分析,并通过WoLF PSORT(https://www.genscript.com/ tools/psort)进行亚细胞定位预测。将候选的绿豆象HSP基因编码的蛋白序列通过NCBI在线BLAST比对,下载与其他昆虫比对后的fasta结果文件,然后使用MEGA 7.0软件中的ClustalW对HSP进行多序列比对生成树文件,采用邻接法构建系统发育树[19]。
1.2.3结构特征分析 基于绿豆象全长转录组测序的GFF3文件,利用GSDS(http://gsds.cbi.pku.- edu.cn/)对进行CDS和UTR结构分析。使用MEME在线程序(https://meme-suite.org/meme/index. html)进一步评估热激蛋白中的保守基序(Motif)。参数设置如下:Motif位点分布,每个序列零个或一个位点;最大Motif数目,10;Motif宽度,Hsp90、Hsp70和Hsp60设置为30—70个残基。由于Hsp20家族的序列都较短,因此设置的宽度为10—40个残基,预测结果通过TBtools软件可视化[20]。采用DNAMAN软件(Lynnon Bio-soft,Quebet,美国)进行多序列比对,并通过Swissmodel (https://swissmodel.expasy.org/)在线软件对目标基因进行PDB文件下载,再结合ESPript 3.0 (https://espript.ibcp.fr/)预测二级结构。
1.3 基因表达特性分析
1.3.1 候选的确定 绿豆象三代全长转录组测序与二代转录组测序的联合分析避免了差异表达基因(DEG)的假阳性。通过对照和高温胁迫下的绿豆象成虫转录组测序结果分析,筛选差异表达显著的HSP并作为候选基因。
1.3.2 RNA的提取与实时荧光定量PCR分析 收集高、低温胁迫处理和对照的绿豆象幼虫、蛹及雌、雄成虫,液氮迅速冷冻,分别使用UNIQ-10柱式总RNA抽提试剂盒(生工生物,中国上海)和HiScript® III第一链cDNA合成试剂盒(诺维赞,中国南京)进行样品的总RNA提取、cDNA模板的反转。超微量核酸蛋白测定仪和1%琼脂糖凝胶电泳用于检测RNA的纯度、浓度和完整性。使用Primer Premier 5.0(http://www. premierbiosoft.com/primerdesign/)设计7个候选HSP基因的qRT-PCR特异性引物,详细信息列于表1中。qRT-PCR使用Stratagene Mx3000P(安捷伦,美国)定量PCR仪进行扩增,20 µL的反应体系如下:2×ChamQ Universal SYBR qRT-PCR Master Mix(诺维赞,中国南京)10 µL,正反向引物各0.4 µL,cDNA模板0.8 µL,ddH2O 8.4 µL。扩增程序采用二步法:95 ℃预变性30 s;然后95 ℃变性5 s,60 ℃ 30 s,共40个循环;熔解曲线则为95 ℃ 15 s,60 ℃ 60 s,95 ℃ 15 s。作为内参基因,采用2-ΔΔct方法计算各基因的相对表达量[21],每个样品包括3次生物学重复和3次技术重复。
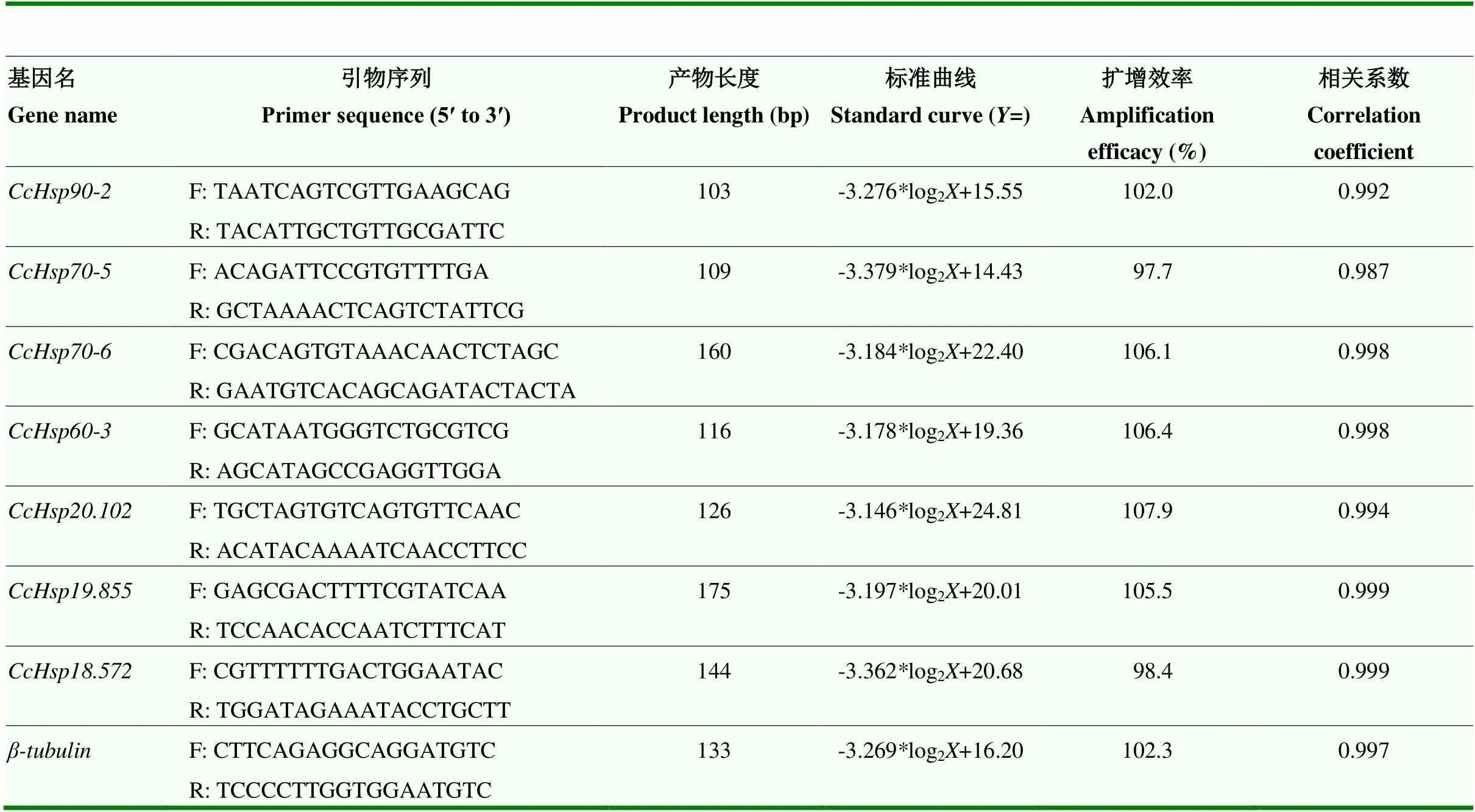
表1 qRT-PCR引物信息及结果分析
1.4 数据处理
使用SPSS 24.0(https://www.ibm.com/products/ spss-statistics)进行单因素(ANOVA)方差分析和最小显著性差异(LDS)检验,同时利用GraphPad Prism 9.0(https://www.graphpad.com/)进行双尾T检验(two-tailed test)并将结果可视化。以<0.05作为显著差异标准,数据均以平均值±标准误表示。
2 结果
2.1 绿豆象HSP超家族基因的鉴定
通过本地BLASTp、tBLASTn、HMM和关键词的搜索,分别获得178、209和165个HSP相关基因。去除重复序列84个及不完整序列109个,最终得到31个绿豆象HSP超家族基因序列,包括3个HSP90、8个HSP70、8个HSP60和12个sHSP(表2)。NCBI中的CDD以及SMART分析结果显示,鉴定出的序列均含有相应亚家族特定的保守区域。以“”命名绿豆象热激蛋白基因,根据分子量大小将小热激蛋白基因保留3位小数。编码的蛋白质,氨基酸长度介于159—776 aa,分子量大小为18.4—88.9 kDa,理论等电点为4.95—9.17。亚细胞定位预测显示多数被定位于细胞质中,但也有少数基因定位于线粒体基质、细胞核和内质网(表2)。
2.2 CcHsp的系统发育分析
为进一步研究的进化关系,将筛选出的31个HSP序列信息在NCBI数据库中进行BLAST同源比对分析,并使用MEGA7.0软件邻接法进行1 000次抽样分析构建系统进化树(图1)。结果显示,HSP基因家族分为HSP90、HSP70和HSP60 3支(图1-A)。3个各自单独成支,在进化距离上关系较远。-与赤拟谷盗()聚为一支,同源性关系较近;-与米象()和马铃薯甲虫()的氨基酸序列一致性高达90%以上;而-在进化关系上则与萤火虫()距离较近。8个分别聚类到两个大分支和一个单独的小分支。-、-、-和-聚为一支(A支);-、-和-聚为另一大支(B支);-则单独成支(C支)。在进化距离上,A支和B、C两支关系较远,而B与C支的进化距离关系较近。A支与光肩星天牛、松墨天牛和马铃薯甲虫聚为一支,同源性最高;B支与鞘翅目其他昆虫的HSP70基因也能较好地聚为一支;而C支则与黑腹果蝇()在进化距离上关系较近。8个共聚为4支,-、-和-分别单独聚为一小分支,并在进化距离上关系较远。剩余的5个聚为一支,同源性高,进化距离近。

表2 绿豆象HSP超家族基因编码蛋白质的理化性质分析
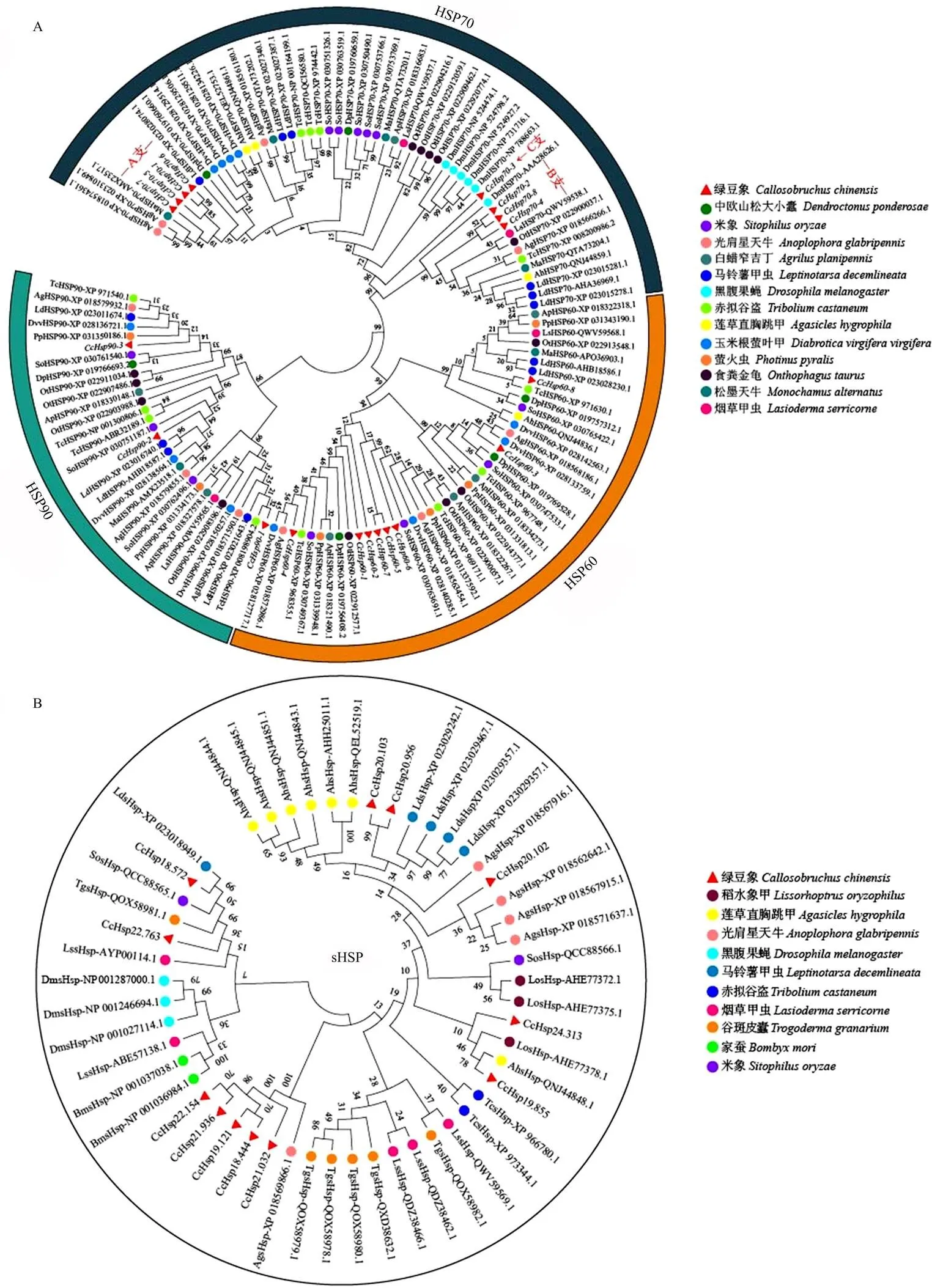
A: HSP; B: sHSP
由于sHSP家族成员的序列较短,无法与上述的HSP家族其他成员进行准确比对,因此单独构建了绿豆象sHSP与相关昆虫独立的系统发育进化树(图1-B)。结果显示,12个绿豆象sHSP基因间部分聚为一支,部分单独成支,聚类结果具有一定的分散性。由于sHSP家族基因在进化上的广泛性,导致部分绿豆象HSP基因与不同种昆虫的亲缘关系比同种昆虫更为密切。
2.3 基因结构和保守基序分析
利用MEGA7.0软件构建了编码蛋白间的进化关系(图2-A)。结果显示,分为4组,每一组代表一个亚家族,且同一亚家族中的基因很好地汇集。利用MEME工具预测了10个保守基序,以探索超家族基因的结构多样性,同时使用TBtools软件将基序进行可视化分析(图2-B)。结果表明,绿豆象HSP70家族中,除-缺少Motif 7外,其他蛋白序列均含有10个相同基序;HSP60家族中,-仅含有4个基序,-、-和-则缺少了一个相同的基序Motif 10;在HSP90家族中,3条蛋白序列都有完整的10个Motif,仅在位置上略有差异。在绿豆象sHSP家族中,蛋白序列均不完整,但个别序列之间的Motif组成高度相似,如、和均含有6个Motif基序,和均包含5个Motif基序等。绿豆象sHSP家族的多序列比对(图3-A)结果显示,所有的12个均具有保守的晶体结构域,并且在其保守区域内含有6条折叠结构域。使用相同的方法依次构建了绿豆象HSP60、HSP70和HSP90家族的多序列比对(图3-B、3-C和3-D),与绿豆象sHSP家族相同的是,各亚家族均具有该家族蛋白保守的特征结构域。
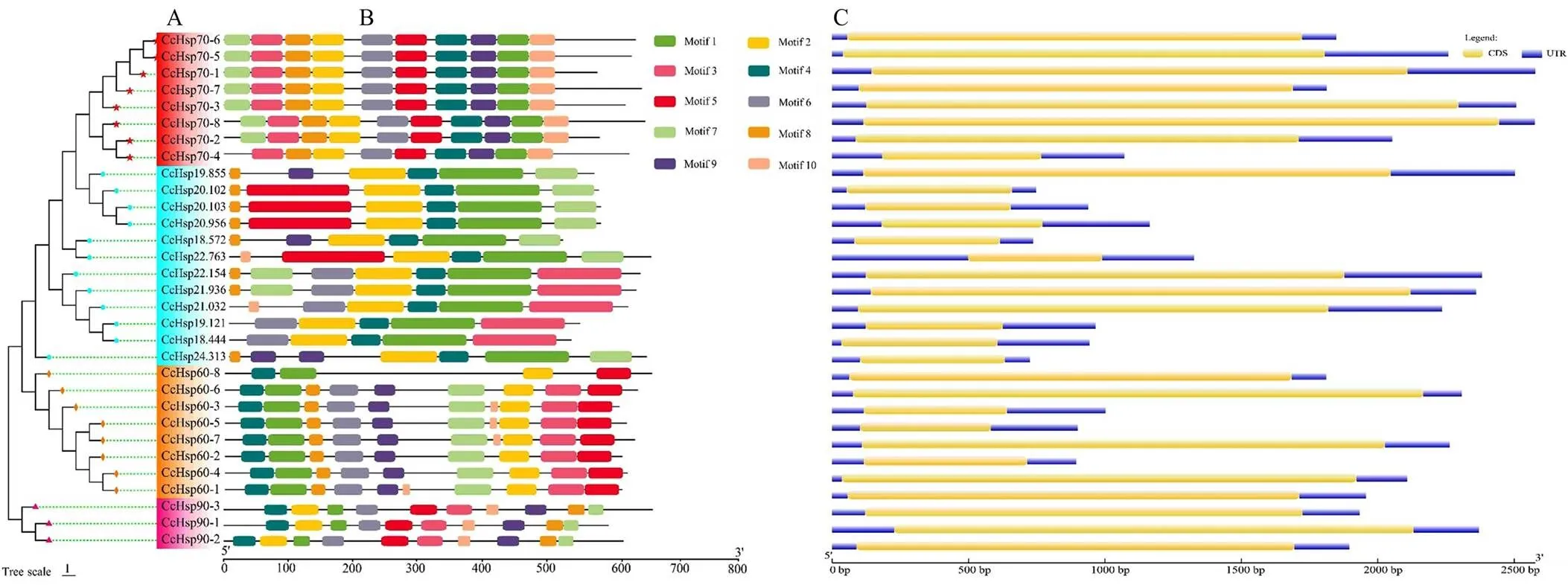
A:31个HSP基因编码的氨基酸序列构建的无根系统发育树,不同颜色区域代表不同CcHsp The unrooted phylogenetic tree was constructed from 31 amino acid sequences encoded by HSP genes, different CcHsps were marked with different colors;B:蛋白质保守基序分析,不同颜色方块代表不同基序Conserved motif of proteins, different color squares represented different motifs;C:绿豆象HSP超家族基因的CDS和UTR分析CDS and UTR analysis of the HSP proteins in C. chinensis
2.4 引物扩增效率检测
内参基因与目的基因扩增效率的一致性是qRT-PCR相对定量测定的前提,7个和内参基因的相关系数(2)均大于0.98,扩增效率和斜率分别介于90%—110%和-3.6—-3.1,且与内参基因扩增效率一致,满足试验要求,因此可进行后续试验(表1)。
2.5 高、低温胁迫后不同发育阶段CcHsp表达分析
利用绿豆象成虫高、低温胁迫的转录组测序数据,分析31个的表达谱。结果表明,所有的在转录组数据中均可被找到,且不同基因表达水平不同,如、、经高温胁迫后高水平表达,而、、、-、-和-经高温和低温胁迫后差异表达不显著(表3)。
比较对照组和高温处理组的转录组测序结果,共发现有51个具有HSP注释的DEG(表4)。根据基因的差异表达变化,在不同的亚家族分别选择、1、、-、-、-和-共7个作为候选基因进行后续的表达分析。
qRT-PCR分析了高温45 ℃和低温-3 ℃胁迫3 h后7个候选的表达变化,以27 ℃室温下7个候选基因在各虫态的表达量作为对照(图4)。结果显示,7个候选基因在绿豆象幼虫期、蛹期和成虫期均检测到表达。经-3 ℃低温诱导后,、-和-在绿豆象不同发育阶段的表达量无明显变化,而在蛹期差异上调表达。经历45 ℃高温胁迫后,7个候选基因表达量与对照组相比均上调表达,其中和-的表达量上调最为显著。经高温胁迫后在雌、雄成虫体内的表达量分别上调1 000和500倍;-经高温胁迫后在雌、雄成虫体内的表达量分别上调500和450倍。在同一发育阶段,不同温度胁迫后各的表达变化也不相同。大致可分为3类:Ⅰ无显著差异,包括幼虫期-和-,蛹期、-和-,雌性成虫期的-和雄性成虫期的-;Ⅱ 显著差异(*:<0.05;**:<0.01):包括幼虫期、、和-,蛹期、、-和-,雌性成虫期-和-,雄性成虫期、和-;Ⅲ 极显著差异(***:<0.001;****:<0.0001):包括幼虫期-,雌性成虫期、、和-,雄性成虫期。
3 讨论
3.1 绿豆象HSP基因超家族成员的鉴定与分析
本研究基于绿豆象全长转录组测序数据共鉴定出31个HSP基因,包括3个HSP90、8个HSP70、8个HSP60和12个sHSP。前人研究发现,在光肩星天牛、赤拟谷盗、褐飞虱()和烟粉虱中分别鉴定出47、20、18和26个HSP基因[14-15,17,20]。可见不同昆虫间热激蛋白基因的数量不同,这可能一方面与物种进化过程中基因的重复或缺失而导致基因家族成员的扩展和收缩有关[21],也可能与参考基因组的来源和数量不同有关[22]。由于目前绿豆象基因组测序还未完成,本文仅利用模式昆虫、近缘种昆虫及其他科和目共计10种昆虫基因组中的HSP基因作为目标序列,在绿豆象全长转录组测序数据中进行本地BLAST,最后完成HSP基因超家族成员的鉴定[23-24],但缺少本物种基因组作为参考可能会造成部分鉴定结果的不完整。
据Wang等[25]报道,HSP75是线粒体维护的关键调控因子,线粒体为真核生物提供了大部分的细胞能量,其功能的调整可能是环境适应的基础[26]。通过BLAST比对发现,绿豆象经高温胁迫后上调表达的-属于HSP75和G高温蛋白,亚细胞定位于线粒体,推测该基因的诱导表达可能在绿豆象应对高温胁迫中发挥重要作用。在筛选的8个中,仅-和-的氨基酸C末端与已知的HSP70蛋白家族C端特异性基序不同,这可能与亚细胞的定位有关[27]。HSP60家族包括Ⅰ型Hsp60和Ⅱ型TCP-1-a/CCT-a[14]。绿豆象8个中,仅-属于Ⅰ型HSP60,其余7个均属于Ⅱ型HSP60,而亚细胞定位于线粒体基质的-则是Ⅱ型HSP60的另一个亚家族。本研究中,除-—、-—、—以及-、—、外,其他亚家族成员均缺乏一些基序。因此,蛋白结构的改变可能导致了热激蛋白功能的差异[14]。
3.2 HSP的表达在绿豆象各发育阶段发挥不同功能
目前,许多与温度适应性和胁迫相关的sHSP基因已在不同昆虫中得到了研究,且大量的试验证实HSP家族基因的表达上调在昆虫抵御高温胁迫中发挥着重要作用[28-32]。此外,HSP基因上调倍数因胁迫程度及昆虫种类不同而存在差异。赤拟谷盗中的小热激蛋白HSP22.2在高温45 ℃胁迫1 h后表达量达到峰值,为对照组的15 000倍[2];40 ℃高温胁迫2 h引起三叶斑潜蝇()表达量的上调且为对照组的140倍[33]。本研究发现,高温胁迫后7个候选均被诱导表达,且雌、雄成虫经45 ℃处理后,雌成虫和-上调显著,分别为1 000和500倍,雄成虫两个基因上调分别近500倍,表明和-的上调表达可能在绿豆象成虫抵御高温胁迫中发挥作用。相似地,42.5 ℃高温胁迫3 h导致-2在松墨天牛雌、雄成虫体内的表达差异显著[12];在经历高、低温处理的幼虫中和-表达量差异显著,推测其可能有助于绿豆象幼虫抵御高温胁迫;绿豆象蛹期,、和-在低温处理后的上调水平显著高于高温胁迫,推测这3个基因可能在蛹应对低温胁迫中发挥作用。相比于高温胁迫,低温并未诱导7个基因的显著性响应,这与BAI等[34]研究低温处理烟粉虱后3个sHSP表达上调不显著的结果相似。
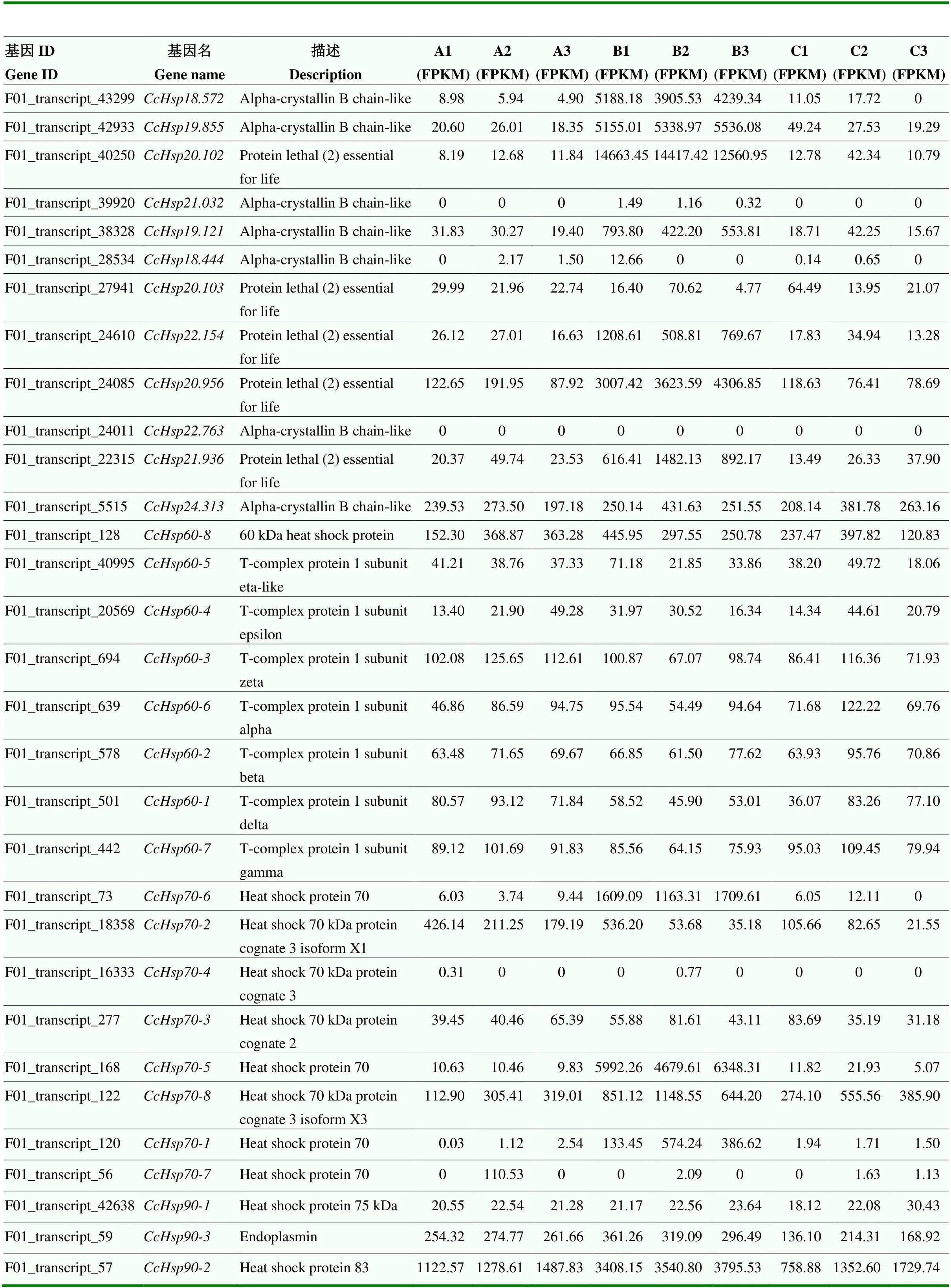
表3 31个CcHsp在绿豆象成虫高、低温转录组中的表达谱
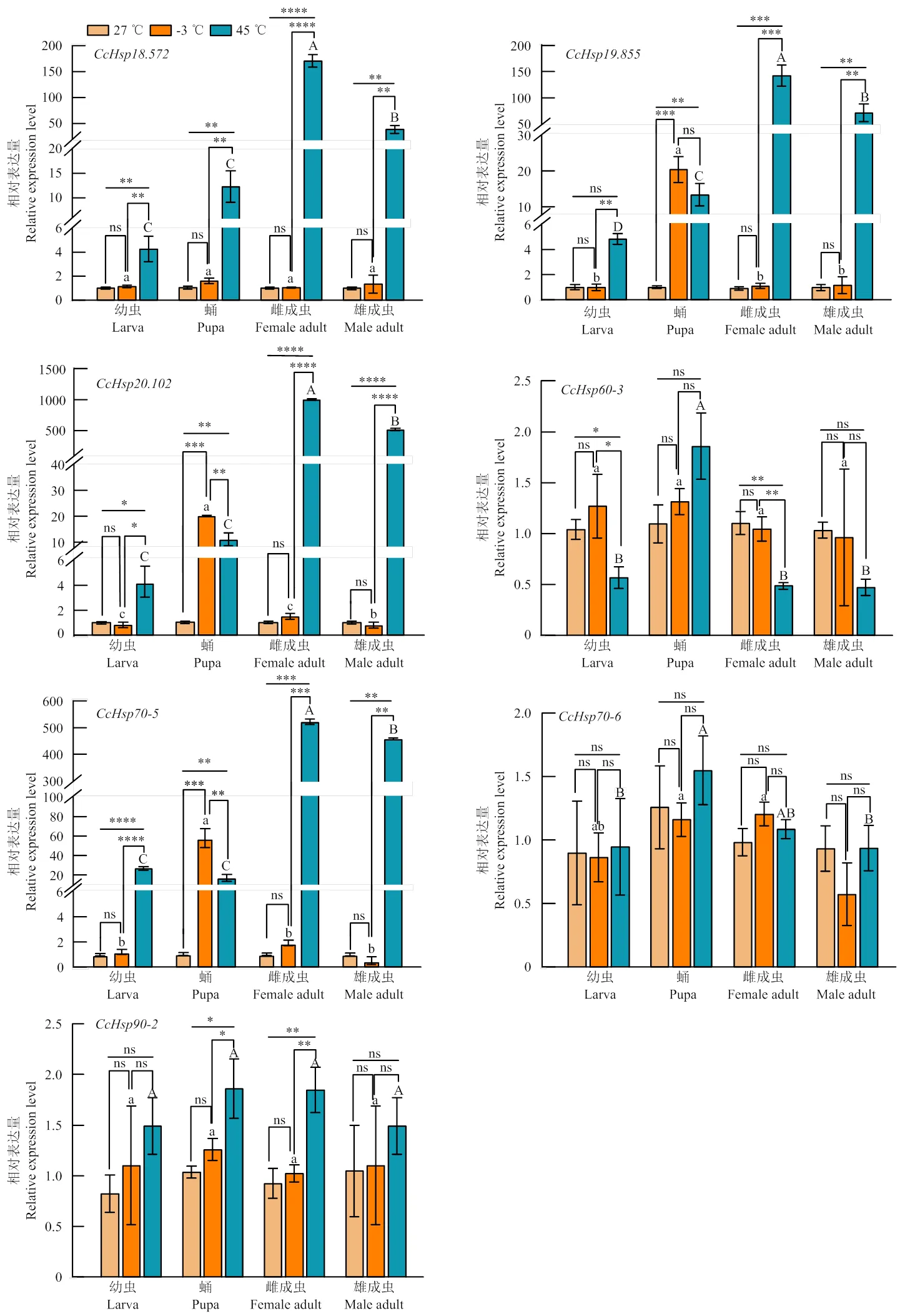
柱上不同大、小写字母分别代表高温(45 ℃)和低温(-3 ℃)条件下发育阶段间表达量差异显著(P<0.05,单因素方差分析和最小显著性差异检验);星号和ns分别代表同一发育阶段不同温度处理下表达量差异显著和不显著(P<0.05,双尾T检验)
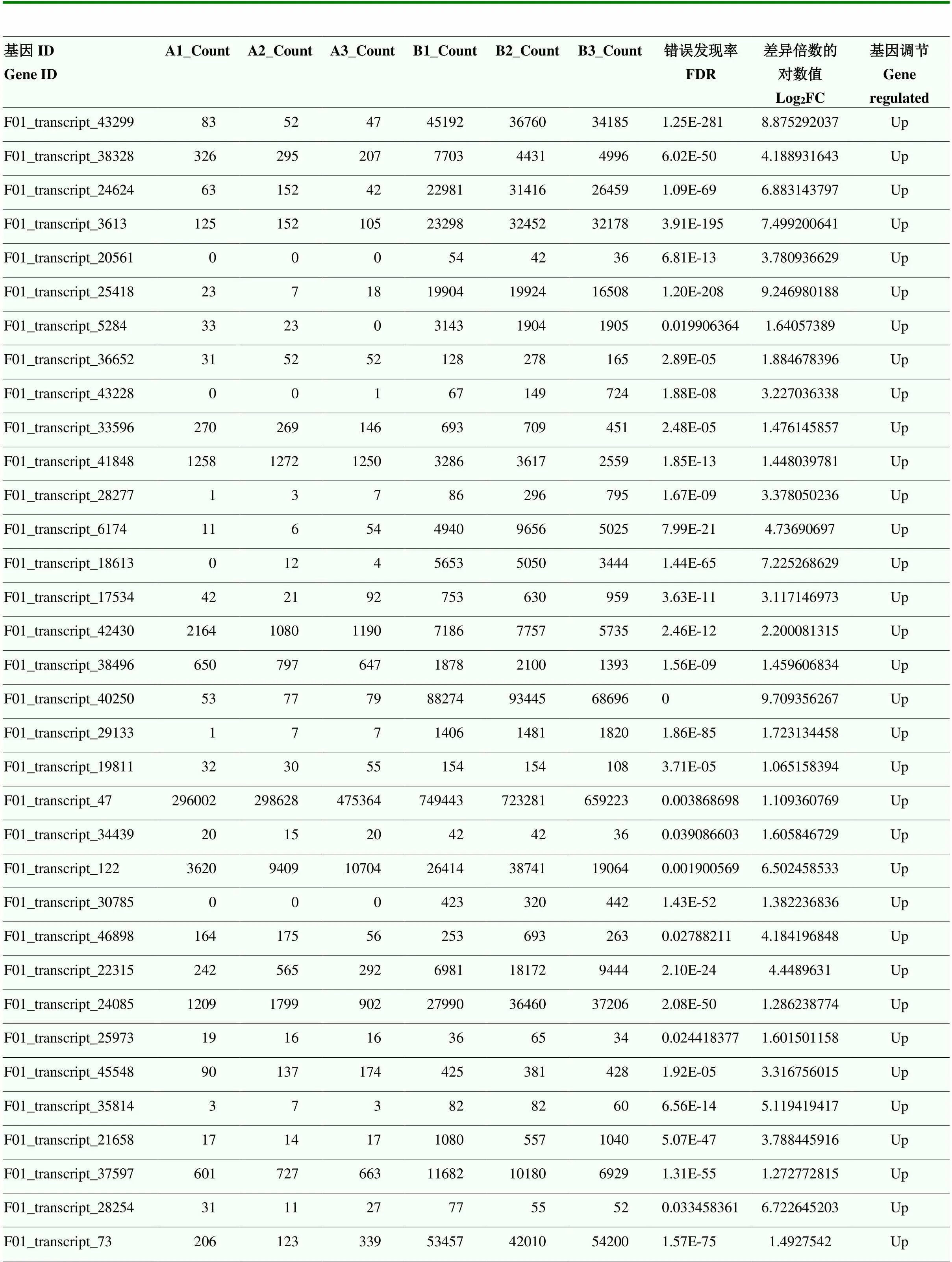
表4 高、低温胁迫转录组中HSP基因差异表达的统计分析
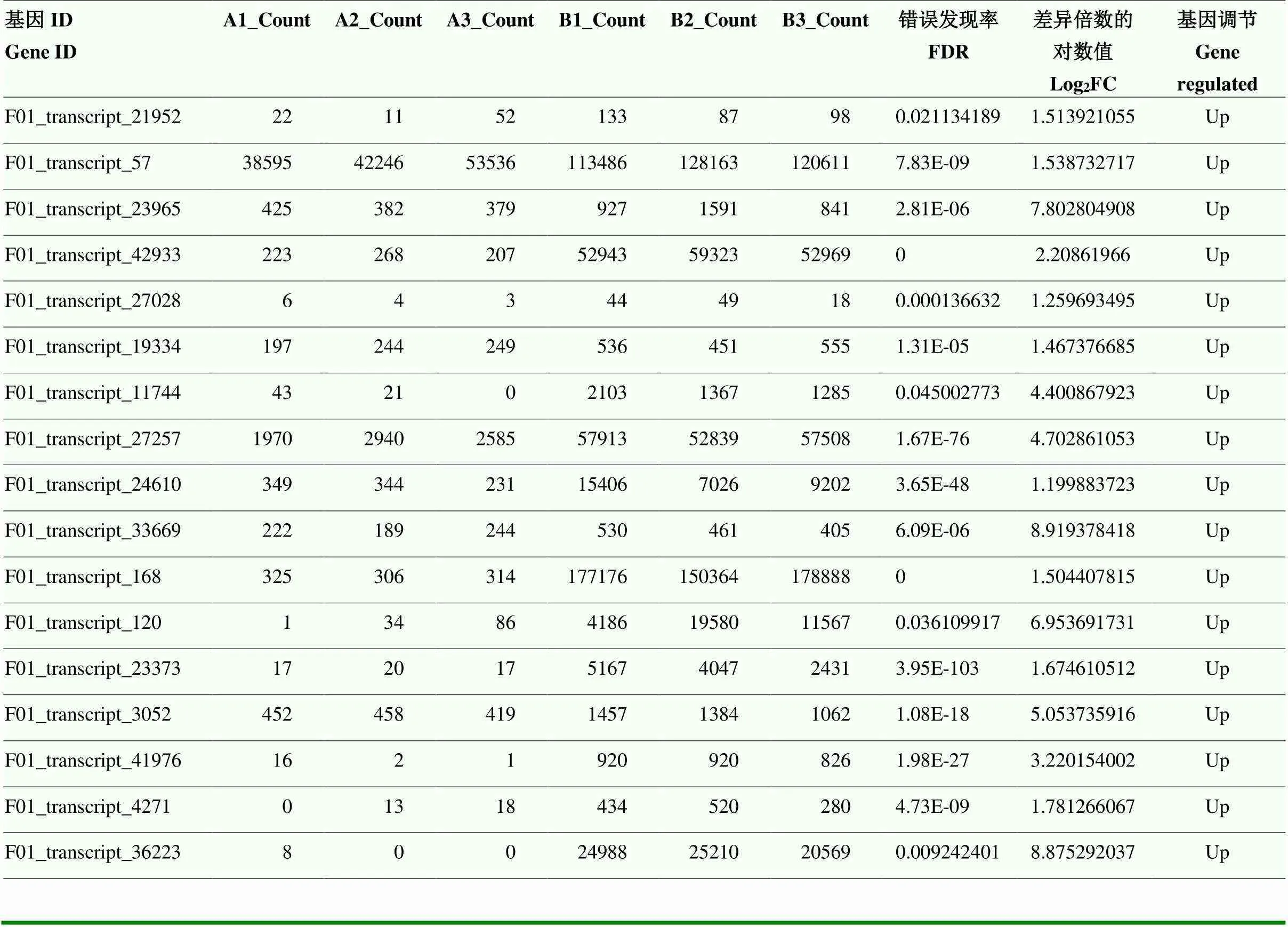
续表4 Continued table 4
Count:比对到基因组上的reads个数the number of reads compared to the genome;FDR:衡量错误发现率的指标,所有检验中假阳性的概率A measure of the false discovery rate, the probability of false positives in all tests;Log2FC:差异倍数以Log2FC的形式展示,FC=实验组表达情况/对照组表达情况The differential multiple was presented as Log2FC, FC=expression of experimental group/expression of Control group
4 结论
绿豆象全长转录组中鉴定出4个亚家族共31个HSP超家族基因成员,不同亚家族间具有不同的蛋白结构及表达模式,其中绿豆象成虫和在经历高温胁迫后,表达量上调显著,推测这两个基因可能在绿豆象成虫抵御高温胁迫中起重要作用。
[1] 仲建锋, 万正煌, 李莉, 陈宏伟, 伍广洪. 低温和高温对仓储绿豆象的防治效果. 中国农业科学, 2013, 46(1): 54-59. doi: 10.3864/ j.issn.0578-1752.2013.01.007.
ZHONG J F, WAN Z H, LI L, CHEN H W, WU G H. Effect of low and high temperatures on controlling Azuki bean beetle (L., Coleoptera: Bruchidae) in storage. Scientia Agricultura Sinica, 2013, 46(1): 54-59. doi: 10.3864/j.issn.0578-1752.2013.01.007. (in Chinese)
[2] XIE J, PENG G, HU X, GU S, BI J, WEI L, TANG J, SONG X, FENG F, LI B. Functional analysis of a novel orthologous small heat shock protein (shsp) hsp21.8a and seven species-specific shsps in. Genomics, 2020, 112(6): 4474-4485.
[3] JIANG X, ZHAI H, WANG L, LUO L, SAPPINGTON T W, ZHANG L. Cloning of the heat shock protein 90 and 70 genes from the beet armyworm,, and expression characteristics in relation to thermal stress and development. Cell Stress and Chaperones, 2012, 17(1): 67-80.
[4] CHEN H, XU X L, LI Y P, WU J X. Characterization of heat shock protein 90, 70 and their transcriptional expression patterns on high temperature in adult of(Busck). Insect Science, 2014, 21(4): 439-448.
[5] BENOIT J B, LOPEZ-MARTINEZ G, PHILLIPS Z P, PATRICK K R, DENLINGER D L. Heat shock proteins contribute to mosquito dehydration tolerance. Journal of Insect Physiology, 2010, 56(2): 151-156.
[6] ZHAO L, JONES W A. Expression of heat shock protein genes in insect stress responses. Invertebrate Survival Journal, 2012, 9(1): 93-101.
[7] KING A M, MACRAE T H. Insect heat shock proteins during stress and diapause. Annual review of entomology, 2015, 60: 59-75.
[8] RINEHART J P, LI A, YOCUM G D, ROBICH R M, HAYWARD S A, DENLINGER D L. Up-regulation of heat shock proteins is essential for cold survival during insect diapause. Proceedings of the National Academy of Sciences of the United States of America, 2007, 104(27): 11130-11137.
[9] CHENG J, SU Q I, XIA J, YANG Z, SHI C, WANG S, WU Q, LI C, ZHANG Y. Comparative transcriptome analysis of differentially expressed genes inYang et Zhang (Diptera: Sciaridae) at different acute stress temperatures. Genomics, 2020, 112(5): 3739-3750.
[10] CHANG Y W, ZHANG X X, LU M X, GONG W R, DU Y Z. Transcriptome analysis of(Diptera: Agromyzidae) in response to temperature stress. Comparative Biochemistry and Physiology Part D: Genomics and Proteomics, 2020, 34: 100677.
[11] XIONG Y, LIU X Q, XIAO P A, TANG G H, LIU S H, LOU B H, WANG J J, JIANG H B. Comparative transcriptome analysis reveals differentially expressed genes in the Asian citrus psyllid () upon heat shock. Comparative Biochemistry and Physiology Part D: Genomics and Proteomics, 2019, 30: 256-261.
[12] LI H, LI S, CHEN J, DAI L, CHEN R, YE J, HAO D. A heat shock 70 kDa protein MaltHSP70-2 contributes to thermal resistance in(Coleoptera: Cerambycidae): quantification, localization, and functional analysis. BMC genomics, 2022, 23(1): 646.
[13] DONG C L, ZHU F, LU M X, DU Y Z. Characterization and functional analysis of Cshsp19.0 encoding a small heat shock protein in(Walker). International Journal of Biological Macromolecules, 2021, 188: 924-931.
[14] XU Y, SHI F, LI Y, ZONG S, TAO J. Genome-wide identification and expression analysis of the Hsp gene superfamily in Asian long-horned beetle (). International Journal of Biological Macromolecules, 2022, 200: 583-592.
[15] WANG X R, WANG C, BAN F X, ZHU D T, LIU S S, WANG X W. Genome-wide identification and characterization of HSP gene superfamily in whitefly () and expression profiling analysis under temperature stress. Insect Science, 2019, 26(1): 44-57.
[16] AKER O, TUNCER C. Use of extreme low temperatures against Adzuki bean weevil (L., Coleoptera: Chrysomelidae) in storage management. KSU Journal of Agriculture and Nature, 2022, 25(3): 511-520.
[17] YIN C, SHEN G, GUO D, WANG S, MA X, XIAO H, LIU J, ZHANG Z, LIU Y, ZHANG Y, YU K, HUANG S, LI F. InsectBase: a resource for insect genomes and transcriptomes. Nucleic Acids Research, 2016, 44(D1): D801-D807.
[18] FINN R D, CLEMENTS J, EDDY S R. HMMER web server: interactive sequence similarity searching. Nucleic Acids Research, 2011, 39: W29-W37.
[19] Kumar S, Stecher G, Li M, KNYAZ C, TAMURA K. MEGA X: molecular evolutionary genetics analysis across computing platforms. Molecular biology and evolution, 2018, 35(6): 1547-1549.
[20] Chen C, Chen H, Zhang Y, THOMAS H R, FRANK M H, HE Y, XIA R. TBtools: an integrative toolkit developed for interactive analyses of big biological data. Molecular plant, 2020, 13(8): 1194-1202.
[21] LIVAK K J, SCHMITTGEN T D. Analysis of relative gene expression data using real-time quantitative PCR and the 2-ΔΔCTmethod. Methods, 2001, 25(4): 402-408.
[22] Tribolium Genome Sequencing Consortium. The genome of the model beetle and pest. Nature, 2008, 452(7190): 949-955.
[23] CANNON S B, MITRA A, BAUMGARTEN A, YOUNG N D, MAY G. The roles of segmental and tandem gene duplication in the evolution of large gene families in. BMC plant biology, 2004, 4: 10.
[24] ZHENG H Y, QIN P H, YANG K, LIU T X, ZHANG Y J, CHU D. Genome-wide identification and analysis of the heat-shock protein gene superfamily inand expression pattern analysis under heat shock. Insects, 2022, 13(7): 570.
[25] WANG Y, LIN J, CHEN Q Z, ZHU N, JIANG D Q, LI M X, WANG Y. Overexpression of mitochondrial Hsp75 protects neural stem cells against microglia-derived soluble factor-induced neurotoxicity by regulating mitochondrial permeability transition pore opening. International Journal of Molecular Medicine, 2015, 36(6): 1487-1496.
[26] HAVIRD J C, SHAH A A, CHICCO A J. Powerhouses in the cold: mitochondrial function during thermal acclimation in montane mayflies. Philosophical Transactions of the Royal Society of London Series B: Biological Sciences, 2020, 375(1790): 20190181.
[27] BOORSTEIN W R, ZIEGELHOFFER T, CRAIG E A. Molecular evolution of the Hsp70 multigene family. Journal of molecular evolution, 1994, 38: 1-17.
[28] XIE J, HU X X, ZHAI M F, YU X J, SONG X W, GAO S S, WU W, LI B. Characterization and functional analysis of hsp18.3 gene in the red flour beetle,. Insect science, 2019, 26(2): 263-273.
[29] SONG J, LU M X, DU Y Z. Molecular cloning and expression patterns of two small heat shock proteins from(Walker). Journal of Integrative Agriculture, 2020, 19(6): 1522-1529.
[30] YANG C L, MENG J Y, ZHOU L, YAO M S, ZHANG C Y. Identification of five small heat shock protein genes inand expression analysis in response to different environmental stressors. Cell Stress and Chaperones, 2021, 26(3): 527-539.
[31] MIAO Z Q, TU Y Q, GUO P Y, HE W, JING T X, WANG J J, WEI D D. Antioxidant enzymes and heat shock protein genes fromare involved in stress defense upon heat shock. Insects, 2020, 11(12): 839.
[32] BAI J, WANG Y C, LIU Y C, CHANG Y W, LIU X N, GONG W R, DU Y Z. Isolation of two new genes encoding heat shock protein 70 inand analysis during thermal stress. International Journal of Biological Macromolecules, 2021, 193: 933-940.
[33] CHANG Y W, ZHANG X X, LU M X, DU Y Z, ZHU-SALZMAN K. Molecular cloning and characterization of small heat shock protein genes in the invasive leaf miner fly,. Genes, 2019, 10(10): 775.
[34] BAI J, LIU X N, LU M X, DU Y Z. Characterization of genes encoding small heat shock proteins fromand expression under thermal stress. PeerJ, 2019, 7: e6992.
Identification and expression analysis of heat shock proteinsuperfamily genes in
ZHANG Xin, YANG XingYu, ZHANG ChaoRan, ZHANG Chong, ZHENG HaiXia, ZHANG XianHong
College of Plant Protection, Shanxi Agricultural University, Taigu 030801, Shanxi
【Objective】The purpose of this study is to identify the gene members of theheat shock protein (HSP) superfamily, and to clarify the expression changes of HSP genes inafter high and low temperature stress, so as to provide a theoretical basis for further exploration of HSP gene function.【Method】The CDS and protein sequences of HSP genes of different insects were downloaded from Insect Base 2.0 and used as a reference for local BLASTp and tBLASTn comparison search in the full-length transcriptome sequencing database of. At the same time, target sequences were screened again by combining HMMER and key words to complete the summary of search results. Bioinformatics analysis of HSP superfamily genes inwas performed using CDD, MEGA, ProtParam, and other online analytical tools. Seven candidate HSP genes were screened out based on high and low temperature transcriptome sequencing data ofadults and the expression characteristics of 7s were compared and analyzed by qRT-PCR technique under different developmental stages and temperature stresses of.【Result】A total of 31 HSP genes were identified, including 3 HSP90s, 8 HSP70s, 8 HSP60s, and 12 sHSPs (small HSP). Physicochemical analysis showed that the proteins encoded bys contain 159-776 amino acid residues (aa), the molecular weights are about 18.4-88.9 kDa, and the theoretical isoelectric points are 4.95-9.17. Subcellular localization results showed that mosts were located in the cytoplasm, while a few genes were located in the mitochondrial matrix, endoplasmic reticulum and nucleus. Phylogenetic analysis showed that different family members of HSPs incould integrate well with HSP in other insects, which indicating their evolutionary conservation. The results of qRT-PCR showed that the 7 candidates were differentially expressed under different temperature stresses. after high temperature stress, the expression level of-in male and female adults was up-regulated by 500 and 450 times. after the larvae undergoing high and low temperature stress, the expression level ofand-was significantly different.【Conclusion】A total of 31 complete HSP superfamily gene members were identified by the full-length transcriptome sequencing data of, which were divided into 4 subfamilies. Different HSP families had different gene structures, protein conserved domains and gene expression characteristics. The differential expression of 7 candidates in different developmental stages and under different temperature stresses indicated that they played different functions and roles. It is speculated thatandmay perform important functions in the adult resistance to high temperature stress, and the high temperature tolerance of larvae may be related to the differential expression ofand.
; full-length transcriptome sequencing; heat shock protein; gene family; bioinformatics analysis; expression analysis
10.3864/j.issn.0578-1752.2023.19.009
2023-06-19;
2023-07-03
国家重点研发计划(2021YFD1600603-01)、山西省现代农业产业技术体系(杂粮)(2022-03)、国家现代农业产业技术体系建设专项(CARS-08-G10)
张昕,E-mail:sxauzx2018@163.com。通信作者张仙红,E-mail:zxh6288@126.com
(责任编辑 岳梅)
