宽皮柑橘褐斑病抗性的全基因组关联分析
2023-10-23杨胜男程莉谈月霞朱延松江东
杨胜男,程莉,谈月霞,朱延松,江东
宽皮柑橘褐斑病抗性的全基因组关联分析
杨胜男,程莉,谈月霞,朱延松,江东
西南大学柑桔研究所,重庆 400712
【目的】挖掘宽皮柑橘褐斑病抗性基因及抗病资源中的相关基因型,为柑橘品种抗褐斑病改良提供依据。【方法】在夏秋两季对136份宽皮柑橘离体叶片进行褐斑病病菌接种试验,将两次试验结果取交集,得到121份宽皮柑橘的褐斑病感抗结果。用121份宽皮柑橘表型对前人开发的CAPS进行验证,再对这121份宽皮柑橘的表型结果与利用简化基因组获得的SNP基因型结果进行主成分分析、GWAS分析和Fst分析,获得宽皮柑橘褐斑病抗性相关SNP位点。对通过GWAS获得的SNP进行基因型分析,在所有候选SNP的上下游25 kb范围内选取候选基因,根据phytozome注释对候选基因进行筛选。在感病资源‘秤砣红橘’和抗病资源‘新克里曼丁’接种褐斑病病菌24、48和72 h后,利用实时荧光定量PCR对筛选出的基因进行表达量分析。【结果】发现在121份宽皮柑橘中,温州蜜柑和克里曼丁类等67份资源表现抗褐斑病,大部分红橘和椪柑等54份资源感褐斑病。本研究发现前人开发的CAPS准确率为76.86%,其相关性为中等程度相关。以-log10(P)>4.5为标准,GWAS筛选出6个强关联SNP;以Fst>0.38为标准,Fst筛选出8个SNP。GWAS筛选出的6个SNP的基因型与宽皮柑橘抗褐斑病表现为强相关,其中位于3号染色体上24 838 146 bp位置处的SNP1基因型对宽皮柑橘抗褐斑病区分能力最强。从14个SNP中筛选出、、、和等5个基因。这5个基因在‘秤砣红橘’接种病原菌后表达量上调,且都在48 h后表达量达到最高,上调倍数高达90倍。【结论】通过GWAS挖掘出3号染色体上24 838 146 bp位置的SNP与宽皮柑橘褐斑病抗病性相关性最显著,相关系数为0.641。并且该位置的基因型能比较有效地将抗性资源和感病资源进行区分。挖掘到、、、、等5个调控宽皮柑橘抗褐斑病的候选基因。
宽皮柑橘;褐斑病;分子标记;全基因组关联分析;群体遗传分化指数分析
0 引言
【研究意义】柑橘作为世界第一大水果,在农业中占据不可替代的作用。许多柑橘品种尤其是宽皮柑橘类品种在栽培过程中易受褐斑病的危害,严重的会导致叶片和果实的脱落以及树枝枯死,影响果实品质和树体生长[1-2]。因此,开发柑橘褐斑病抗性相关分子标记以及挖掘相关基因,对于培育抗褐斑病柑橘品种具有重要意义。【前人研究进展】柑橘褐斑病于1903年首次在澳大利亚的皇帝柑上被发现,之后陆续在多个国家发现[3]。我国于2010年首次报道,之后在各个产区陆续有褐斑病的爆发,对当地柑橘产业发展造成了严重的影响[4]。已有不少研究者对柑橘抗褐斑病进行了研究。在柑橘品种抗性评价方面,国外的研究表明,‘诺瓦橘’‘默科特’‘明尼奥拉’‘Idith’等都是易感品种,克里曼丁和温州蜜柑等属于抗性品种[5-6]。符雨诗等[7]发现‘南丰蜜橘’‘418红橘’‘满头红’‘无核早橘’等都高度感病,而‘爱伦达尔’‘红玉柑’‘清见’‘不知火’‘南香’等则对褐斑病免疫。在柑橘对褐斑病抗性的分子标记以及抗性基因发掘方面,2005年DALKILIC等[8]发现柑橘对褐斑病的抗性由一个隐性等位基因控制,只有这个位点为隐性纯合子才表现为抗病。2013年CUENCA等[9]在3号染色体的着丝粒附近定位到与柑橘褐斑病抗性相关的3.3 Mb基因组区域。2016年对该位点进一步开展精细定位,将含有柑橘褐斑病的抗性基因限制在366 kb的区域内,两侧分别有0.4和0.7 cM的标记。该区域包含9个与病原菌抗性相关的基因,在3号染色体25 862 085 bp处的SNP能够将感病品种和抗病品种进行区分[10]。2020年ARLOTTA等[11]针对此位点设计了CAPS标记,该CAPS能将柑橘抗病品种和感病品种分开。唐科志等[12]通过对红橘感染褐斑病前后转录组比对,发现红橘在受到褐斑病胁迫时,LRR类受体基因、LRK受体激酶基因等能够响应柑橘褐斑病的侵染。【本研究切入点】目前国内对于宽皮柑橘抗褐斑病的基因定位研究较少,本研究基于前人开发的CAPS标记进行验证,发现该CAPS的准确率并未达到100%,目前利用简化基因组测序结合全基因组关联分析(genome-wide association study,GWAS)和群体遗传分化指数(fixation index)分析(Fst)来挖掘抗病候选基因的应用非常广泛[13-14],但对于柑橘褐斑病抗性的GWAS分析和Fst分析还未见报道。【拟解决的关键问题】本研究旨在评估不同宽皮柑橘对于褐斑病的抗性,并利用简化基因组测序结合GWAS和Fst分析,以寻找出更多的SNP位点和抗病基因来辅助柑橘抗病育种。
1 材料与方法
试验于2022年在西南大学柑桔研究所资源室进行。
1.1 试验材料
试验所使用链格孢菌THJ由西南大学柑桔研究所综防课题组提供。试验所用的136份宽柑橘材料,均采自西南大学国家柑桔种质资源圃(重庆),具体材料表型及基因型见附表。
1.2 褐斑病抗性评价
夏季柑橘褐斑病抗性评价:参照唐志科等[12],取THJ病菌于PDA培养基上28 ℃活化,再取活化后的THJ加入PDB培养基,28 ℃,150 r/min摇48 h,用75%酒精将成熟度、大小一致且未感病未受损的柑橘夏季新梢进行表面消毒,再用无菌水清洗,将叶片放入托盘中,喷洒无菌水保持湿润,之后取菌丝球接种于清洗后的离体叶片上,再用保鲜膜将托盘覆盖。每个品种资源接种3片叶,并设置3份叶片不进行接种作为对照,再将托盘放入28 ℃恒温培养箱进行培养,72 h后对感病情况进行记录。若无病斑则记录为抗病,有病斑则记录为感病[10]。
秋季柑橘褐斑病抗性评价:参照刘荣萍等[15],取THJ病菌于PDA培养基上28 ℃培养4 d,再用75%酒精对成熟度一致、未感病、未受损的柑橘秋季新梢进行表面消毒,再用无菌水清洗,放入托盘中,再喷洒无菌水保持湿润,之后在离体叶片背面接种一个4 mm菌饼,用保鲜膜将托盘覆盖,每个品种资源接种15片叶,并设置15片叶不接种作为对照。将托盘放入28 ℃恒温箱进行培养,72 h后对感病情况进行记录,若有两片及两片以上的叶片出现明显的感病现象则记录为感病,反之则抗病。
1.3 CAPS抗性预测
将室内接种与田间调查结果一致的宽皮柑橘作为CAPS基因型验证的材料。用CTAB法提取121份宽皮柑橘DNA。再用SNP08-spare-F/R(表1)对DNA片段进行扩增,具体扩增体系为:2 μL DNA模板,正、反向引物各1 μL,6 μL无菌水,10 μL 2×Hieff Canace® Gold PCR Master Mix酶。扩增体系为98 ℃预变性3 min,98 ℃变性10 s,68 ℃延伸30 s,35次循环,72 ℃终延伸1 min。
用限制内切酶I对扩增片段进行酶切,酶切体系为:4 UI限制性内切酶,1 μL CutSmart Buffer,6 μL扩增产物。置于37 ℃金属浴90 min后65 ℃灭活20 min。酶切产物通过1.5%琼脂糖凝胶电泳检测。
将CAPS预测表型与实际表型进行比较,用SPSS进行肯尔顿相关分析。
1.4 全基因组关联分析和遗传分化分析
以夏季接种和秋季接种结果一致的121份宽皮柑橘进行GWAS以及Fst分析。用PLINK1.9从已进行简化基因组测序的240份宽皮柑橘(https://www. ncbi.nlm.nih.gov/genome/11310)中提取出121份宽皮柑橘的SNP信息[16],获得112 768个高质量SNP。用GEMMA通过混合线性模型进行GWAS分析,基于R语言用ggplot2包绘制曼哈顿图,用qqman绘制QQ图。通过PLINK1.9进行Fst分析,再基于R语言用ggplot2包绘制曼哈顿图。以Bonferroni校正计算阈值,再根据实际情况进行调整,以-log10(P)>4.5为标准,筛选GWAS获得的SNP。WRIGHT研究表明Fst>0.25群体分化极大[17],以Fst>0.38筛选SNP。通过PLINK1.9对GWAS获得的显著SNP进行提取,再用Excel对不同基因型的表型进行统计分析。利用SPSS对基因型与表型进行肯尔顿相关性分析。
1.5 候选基因筛选与表达分析
由于宽皮柑橘LD为25 kb[16],根据GWAS和Fst筛选出来的SNP在其前后25 kb区域内选择候选基因,依据phytozome对这些基因的注释进行筛选,获得5个与抗病相关基因。为了再次验证候选基因,取‘秤砣红橘’和‘新克里曼丁’叶片进行病原接种,测定接种24、48和72 h后基因的表达情况。使用Biospin总RNA提取试剂盒提取其总RNA。根据试剂盒Hifair® III 1st Strand cDNA Synthesis SuperMix for qPCR(g DNA digester plus)说明书进行反转录,再根据Hieff UNICON® Universal Blue qPCR SYBR Master Mix试剂盒对该5个基因进行实时荧光定量分析。以为内参(表2),实时荧光定量PCR反应体系为:10 μL SYBR、1 μL cDNA、0.4 μL正向引物、0.4 μL反向引物、8.2 μL RNas-free水。反应程序为:95 ℃预变性30 s;95 ℃变性10 s,60 ℃退火20 s,95 ℃延伸15 s,39个循环。以2-ΔΔCT计算基因的相对表达量。
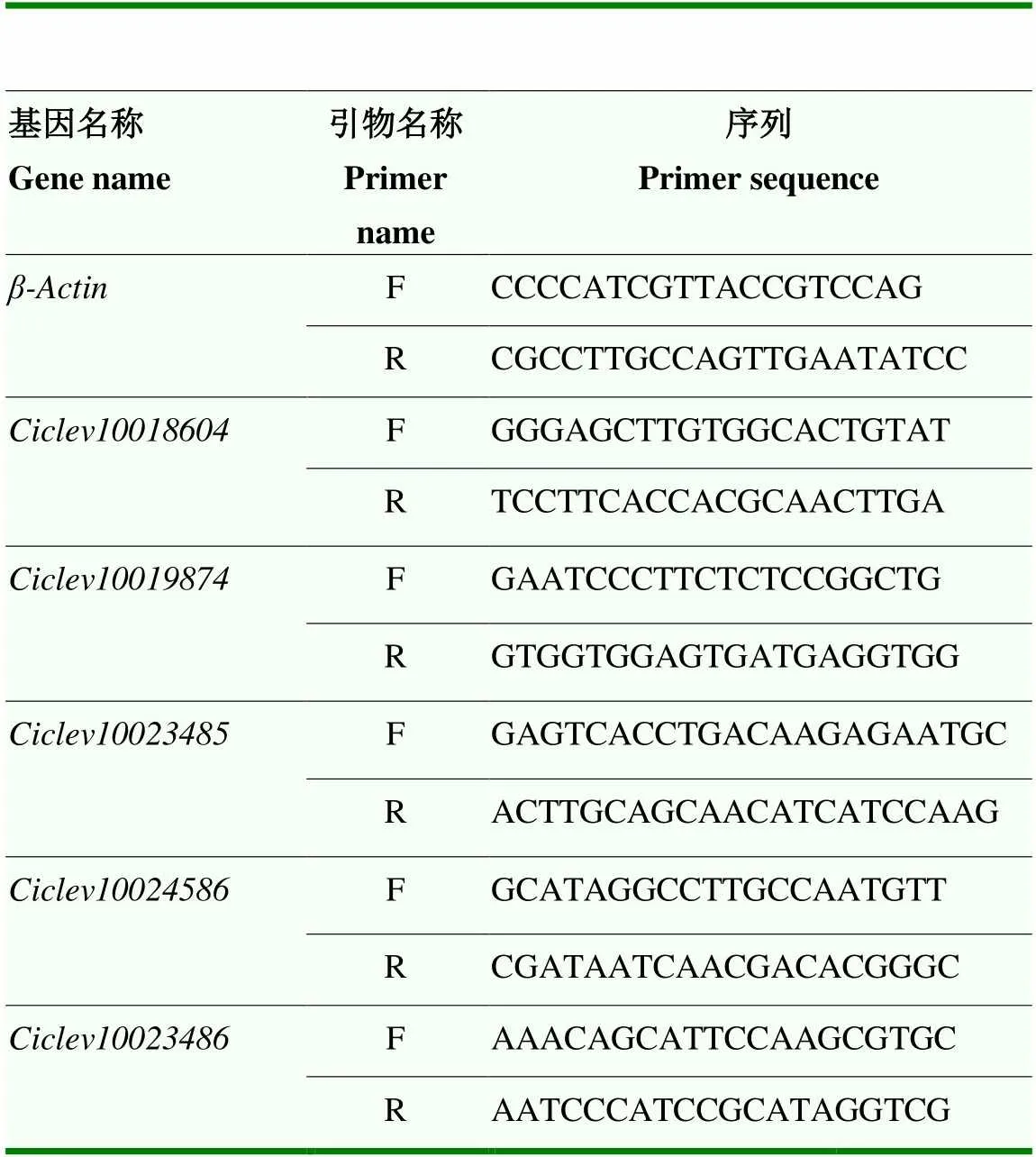
表2 候选基因荧光定量引物
2 结果
2.1 不同宽皮柑橘品种资源对褐斑病的抗性评价
在136份品种资源中,有121份品种资源夏、秋鉴定结果表型一致。其中‘橘红’‘春香’‘春见’‘扁柑’和‘聂都野橘No.1’在夏季接种时,表现出抗病,却在秋季接种时表现为感病。而‘陈皮实生2号’‘摩克sidi’‘王柑’‘爱媛21号’‘本地广橘’‘青岛温州’‘寿太郎温州蜜柑’‘清见’‘帕森特’和‘油皮橘’在夏季接种表现出感病,却在秋季接种时表现出抗病。对夏季和秋季接种结果进行肯德尔相关性分析,发现相关系数为0.78。
在这121夏秋鉴定结果表型一致的宽皮柑橘中,有67份呈现出感病,如图1中‘大香柑’在夏季和秋季接种后都表现为感病。有54份呈现出抗性,如图1中‘桂平朱砂橘’在夏季和秋季接种后均表现为抗病。其中大部分的温州蜜柑(‘宫川温州’‘久能温州’‘石川温州蜜橘’‘国庆一号温州’‘松山’)和克里曼丁类(‘克里曼丁×本地早’‘新克里曼丁’‘克里曼丁(波娜S.R.A 65)’)都呈现出抗病。此外还有像‘南香’‘无核沃柑’‘金橘’‘永顺蜜橘’‘爱媛30’等资源对褐斑病表现出抗性,而大部分椪柑品种,如‘兴春椪柑’‘尼8013’‘尼8010’‘尼8005’‘蜂洞橘’‘新生系三号椪柑’‘乌干达橘’等,大部分的红橘如‘418红橘’‘秤砣红橘’‘和平92号红橘’‘兴义大红袍’等都呈现出感病。这也符合前人研究结果[5,7]。

A:‘大香柑’夏季接种结果;B:‘桂平朱砂橘’夏季接种结果;C:‘大香柑’秋季接种结果;D:‘桂平朱砂橘’秋季接种结果。图A和图B左3片叶片为接种叶片,右3片叶片为对照叶片;图C和图D前15片叶片为接种叶片,后15片叶片为对照叶片
2.2 CAPS抗性预测
CUENCA等发现在3号染色体的25 862 085 bp处的SNP基因型能将感病资源和抗资源进行区分,ARLOTTA等针对此位点设计了CAPS标记,可以通过对特定片段酶切再通过琼脂糖凝胶电泳结果区分感病资源和抗病资源。具体表现为,若在琼脂糖凝胶电泳中产生一条带,则基因型为TT,表现为抗性;若为2条或者3条带则为GG或GT型,表现为感病(图2)。从图2可知,‘明尼奥拉橘柚’‘橘橙22-53’‘圆红香柑’‘安江红橘’‘沙柑’‘奥坦尼科’为感病资源,其余为抗病资源。
用2.1获得的121份宽皮柑橘的抗性评价结果对该CAPS进行验证。在这121份宽皮柑橘中,共有93份验证正确,准确为76.86%。67份感病资源中有52份CAPS验证正确,准确率为77.61%,而54份抗病资源中有41份验证正确,准确率为75.93%(图2)。用肯德尔相关分析对抗性评价结果与CAPS验证结果进行相关性分析,发现相关系数为0.534,表现为中等程度相关。表明该CAPS判断宽皮柑橘对褐斑病是否有抗性判定有一定的参考价值,但还需要其他分子标记辅助判断。

A:20份宽皮柑橘CAPS酶切结果;1:2000 DNA marker;从2-21依次为:‘明尼奥拉橘柚’‘橘橙22-53’‘圆红香柑’‘新克里曼丁’‘安江红橘’‘金橘’‘酸柑子’‘韦尔金橘’‘阿匹热诺’‘沙柑’‘克里曼丁×本地早’‘津之望’‘1-26’‘平阳橘’‘清见×明尼奥拉’‘奥坦尼科’‘四季橘’‘无核w默科特’‘无核早橘’‘滑皮橘’。B:CAPS准确率饼状图
2.3 利用简化基因组测序数据的全基因组关联分析与Fst分析
通过对121份宽皮柑橘进行主成分分析,发现在感病品种资源和抗病品种资源之间并未出现分层(图3-A)。为了鉴定宽皮柑橘对于褐斑病的抗性位点,对前期采用GBS简化基因组测序获得的SNP位点与柑橘褐斑病抗感表型进行全基因关联分析和Fst分析。全基因关联分析表明显著关联的SNP位点主要集中在2号和3号染色体上,QQ图中曲线上翘表明GWAS结果较好(图3-C)。GWAS分析共筛选出6个SNP。其中4个位于3号染色体上,1个位于2号染色体上;最显著的SNP位于3号染色体24 838 146 bp处,其-log10(P)为7.74(图3-B)。Fst分析筛选出的显著关联SNP主要集中在3号、2号以及5号染色体上。本研究以Fst>0.38为标准,共筛选到8个SNP。其中6个位于3号染色体上,1个位于2号染色体上,1个位于5号染色体上;最显著的SNP位于5号染色体处,其Fst值为0.42(图3-D)(表3)。

A:121份柑橘主成分分析图;B:褐斑病抗性性状GWAS曼哈顿图;C:褐斑病抗性性状GWAS QQ图;D:褐斑病抗性性状Fst分析的曼哈顿图
接着对GWAS筛选出的6个SNP(表4)进行基因型分析,发现SNP1、SNP2、SNP4和SNP6能良好地区分出感病资源。SNP1基因型为G/G则全部为感病资源,基因型为A/A则85%的几率为抗病资源。SNP2基因型为C/C时全部为感病资源,基因型为T/T则有75%的几率为抗病资源。SNP4基因型为G/G时全部为感病资源,基因型为T/T则有73%的几率为抗病资源。SNP5基因型为A/A则95%几率为抗病资源。SNP6基因型为C/C时全部为感病资源,基因型为T/T则有74%的几率为抗病资源。另外,这几个SNP的基因型若为杂合型,则有更大的可能性表现为易感褐斑病。之后对这6个SNP基因型与表型进行肯德尔相关性分析,SNP1表现为强相关,相关系数为0.641。其余5个表现为中等程度相关,SNP2、SNP3、SNP4、SNP5和SNP6相关系数分别为0.572、0.583、0.564、0.45和0.549。表明这几个SNP的基因型可以作为判断宽皮柑橘是否感/抗褐斑病的一个参考依据。

表3 GWAS和Fst分析获得的14个SNP的基本信息
为获得宽皮柑橘对于褐斑病抗性的候选基因,本研究在获得的14个SNP上下游25 kb区域内筛选基因,总共筛选到38个基因,根据phytozome的注释,筛选出5个和抗病相关基因(表5)。这5个基因都位于3号染色体上,均含有抗病相关的结构域。对这5个基因在感病资源‘秤砣红橘’和抗病资源‘新克里曼丁’接种病原菌24、48和72 h后,分别进行实时荧光定量分析,发现这5个基因在‘秤砣红橘’接种病原菌后表达量存在显著差异,均表达上调,且在感染48 h时表达量达到最高,72 h时有所下降(图4)。在72 h时下降幅度最低。48 h时,上调倍数最高,约是接种24 h时的97倍,上调倍数最低,约是接种24 h时的40倍。对‘新克里曼丁’而言,5个基因在接种病原菌后表达量都有所上调,但上调幅度远不如感病资源‘秤砣红橘’,尤其是在48 h时。其中和的表达量上调倍数最高,在感病48 h时上调倍数分别为24 h时的7倍和9倍。、和在‘新克里曼丁’接种病原菌后的24、48和72 h表达量均高于‘秤砣红橘’接种24 h时的表达量,但是要低于‘秤砣红橘’接种48和72 h时的表达量。的表达量在接种后3个时间点并未发生较大的改变,表达量与‘秤砣红橘’接种后24 h差异不大。在‘新克里曼丁’接种24、48和72 h时的表达量均低于‘秤砣红橘’接种24 h时的表达量。
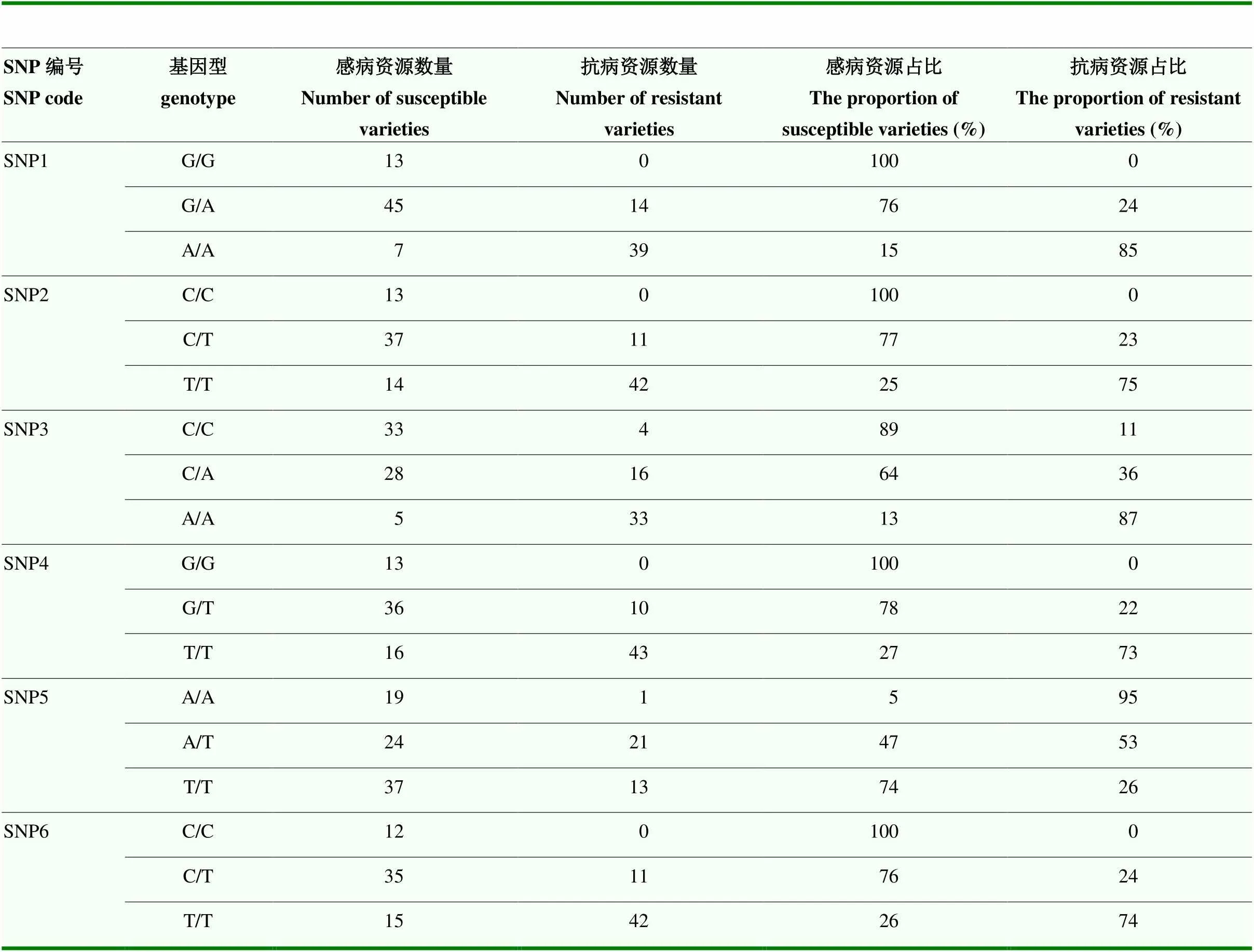
表4 GWAS 分析获得的6个SNP基因型的基本信息
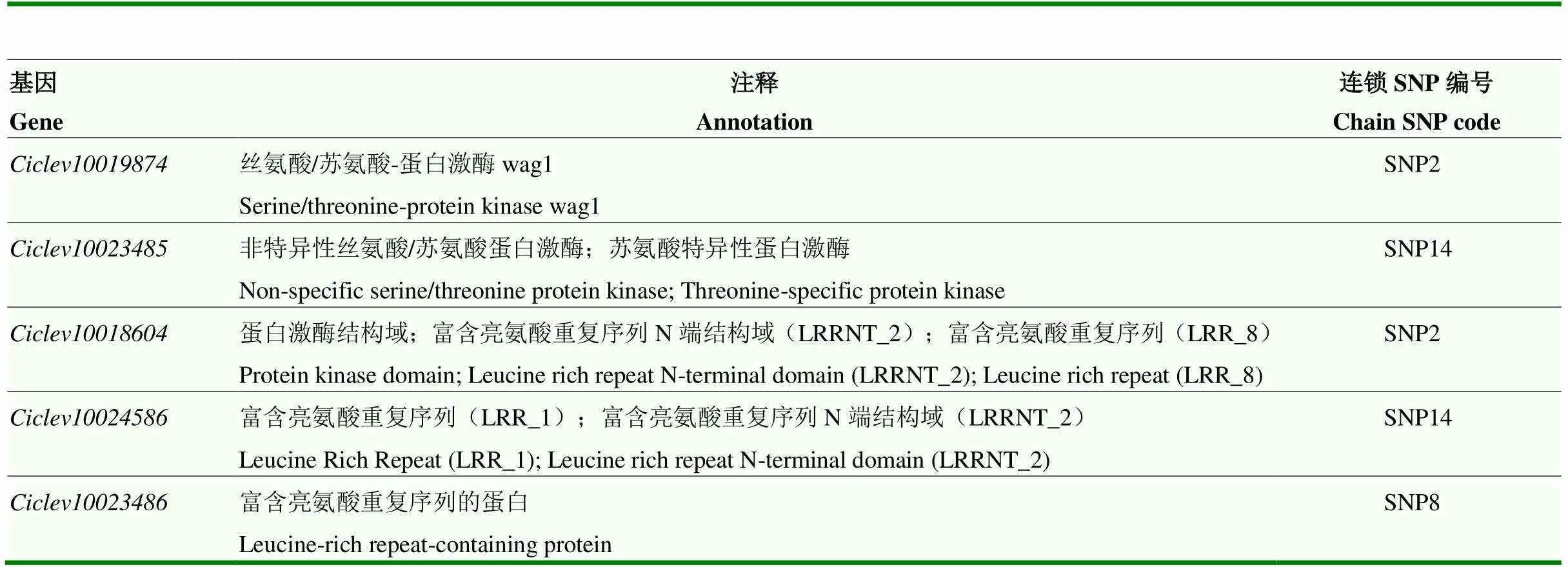
表5 5个候选基因的功能注释

*、**和***分别表示在0.05 、0.01和0.001水平上差异显著。CTHJ-24、CTHJ-48、CTHJ-72分别代表‘秤砣红橘’接种后24、48和72 h。XKLMD-24、XKLMD-48、XKLMD-72分别代表‘新克里曼丁’接种后24、48和72 h
3 讨论
柑橘褐斑病是柑橘中的重要病害之一,危害柑橘产业的发展[18]。柑橘褐斑病主要发生在宽皮柑橘和葡萄柚上,而宽皮柑橘是我国的主栽柑橘类型,探究宽皮柑橘对褐斑病的抗性很有必要。
3.1 宽皮柑橘对褐斑病抗性评价分析
抗褐斑病宽皮柑橘种质的鉴定可为育种家对于抗病亲本的选择提供重要的参考。本研究发现在宽皮柑橘资源中存在许多的抗性资源,有54份资源的抗性稳定,在今后的育种工作中,这些资源可以优先考虑作为亲本。另外,这54份抗性资源中也存在品质优良的品种,例如‘无核沃柑’在夏、秋接种时都表现出对褐斑病的抗性。本研究发现16份品种资源在夏季和秋季接种表型不一致,推测可能受到叶片成熟度影响。研究发现‘Clauselina IVIA-19’‘Okitsu IVIA-195’‘Unshu SRA-529’‘Saigon SRA-227’等一些资源品种在接种叶片长度为2—4 cm时出现症状,而在叶片接种长度为5—7 cm时却没有症状[5]。可能是老叶质地更硬,不利于菌丝入侵。本研究是采取成熟度为50%的叶片进行接种,而叶片成熟度的具体判断是根据笔者的经验进行,因此存在一定的误差,上述16份品种资源可能是采摘的叶片成熟度过高,导致在接种后并未表现为感病。
3.2 宽皮柑橘抗褐斑病相关基因型分析
本研究发现CAPS的准确率并未达到100%,ARLOTTA等[11]对其开发的CAPS进行验证时其准确率达到100%。推测可能是因为在ARLOTTA的试验中用于验证的群体为杂交群体,而本研究验证的是一个自然群体,杂交群体的遗传多样性低于自然群体,所以柑橘抗褐斑病还受其他位点或基因的影响,可能该位点在ARLOTTA研究的杂交群体中并不存在多样性。本研究发现虽然大部分克里曼丁类和温州蜜柑类表现为抗病,但是也有像‘岩崎早生温州’‘宁红73-19温州’‘费尔柴尔德’等表现为感病,但‘岩崎早生温州’的6个SNP基因型与其他抗病“温州系”一致,表明可能还存在其他基因结构层面上或者基因表达层面上的差异导致其对于褐斑病感抗上的差异。对所有椪柑在SNP1位置的基因型进行统计,发现其基因型均为G/A,而红橘的基因型均为G/G,表明在椪柑和红橘中,该位点比较保守,不易发生突变。而‘费尔柴尔德’为克里曼丁与其他柑橘的杂交种。在克里曼丁类别中,发现‘费尔柴尔德’SNP1位置的基因型为G/A,其余的基因型都为A/A。所以‘费尔柴尔德’表现为感病推测是由于其另外一个亲本的感病基因渗入,而该感病基因可能与SNP1连锁。
近年来,随着测序成本的不断降低,GWAS已广泛且有效地应用于植物抗病基因的定位研究,也取得了许多进展[19-21]。本研究通过GWAS发现了6个SNP与宽皮柑橘褐斑病高度相关,这6个SNP中有5个位于3号染色体,其中最显著的SNP位于3号染色体的24 838 146 bp位置,该位置距离CUENCA等[10]发现的SNP08相差1 023.939 kb,推测可能是因为其利用的是杂交群体进行相关SNP定位,杂交群体的多样性更差,而本研究利用的是自然群体,多样性更好。通过Fst筛选出的SNP也主要集中在3号染色体。这表明柑橘褐斑病抗性相关基因主要存在于3号染色体上,这与前人研究相吻合[9]。
在梨中,黑斑病也由链格孢菌引起,而且梨中的链格孢菌所产生的致病因子结构与柑橘所产生的致病因子结构相似。研究人员对梨的黑斑病进行研究,提出梨对黑斑病的易感性由单个显性基因控制,称为A,易感品种具有杂合基因型(A/a)[22]。在苹果中的斑点病也是由链格孢菌引起,研究发现苹果对链格孢菌斑病的易感性由显性基因控制,该基因定位于‘美味’的11号染色体,抗性品种为基因型/,易感品种为/或/[23]。柑橘中的研究也推测抗病品种的抗病基因座为隐性纯合型,而易感品种可能是杂合型或显性纯合型[8-9]。本研究通过对SNP基因型分析,发现若基因型为杂合子时,其表型更有可能表现为感病。
3.3 宽皮柑橘抗褐斑病相关基因分析
R蛋白是植物体内最大的抗病蛋白。R蛋白都具有一些共同的结构域,包括富亮氨酸重复序列蛋白(leucine-rich repeat,LRR)、丝氨酸/苏氨酸蛋白激酶(serine/threonine protein kinase,S/TK)、卷曲螺旋结构(coiled coil,CC)、跨膜结构受体(transmembrane receptor,TM)、核苷酸结合位点(nucleotide binding site,NBS)和Toll/白细胞介素-1受体(toll/interleukin-1 receptor,TIR)等。这些结构域通过不同的组合形成抗病基因参与植物抵抗病原菌[24-26]。例如LRR与S/TK以及TM组合成LRR-RLK主要参与第一层防御[27-30],NBS与LRR以及TIR组合成TIR-NBS-LRR则主要通过识别效应因子参与第二层防御[31-33]。本研究通过GWAS和Fst获得的SNP对基因进行筛选,但并未发现有上述传统上的抗病基因,不过筛选出5个含有上述结构域的基因。其中和都含有丝氨酸/苏氨酸蛋白激酶结构域,而、和都含有LRR结构域。
丝氨酸/苏氨酸蛋白激酶(S/TKs)是生物酶中的一个大家族,其功能包括介导植物防御反应信号的转导。在植物与微生物相互作用过程中,S/TKs主要参与病原菌信号的识别和转导[34]。相较于LRR-RLK,单独的S/TK基因参与植物抗病的报道比较少,不过GAO等[34]发现Nr S/TK基因能够对烟草黑斑病易感品种产生抗性。在泸定百合中,Ls S/TK经百合镰刀菌诱导后表达量会上升[35]。在大麦中,编码含有两个串联激酶结构域的丝氨酸/苏氨酸蛋白激酶的基因为大麦茎锈病的抗性基因[36]。表明单独的S/TK也可以参与植物的抗病过程。本研究中,2个含有S/TKs基因的表达都能被链格孢菌的侵染所诱导,而且和的表达量在感病品种‘秤砣红橘’接种48 h后的表达量达到最高,而抗病品种‘新克里曼丁’的上调表达幅度远不如‘秤砣红橘’,表明和在感病品种中参与了柑橘褐斑病浸染后信号的识别响应和信号的传导。而在抗病品种中24、48和72 h的表达量变幅不大,说明其对外界链格孢菌病原菌的识别和信号响应并不敏感,这可能是‘新克里曼丁’抗病的主要原因,因此,推测可能是前期参与宽皮柑橘对链格孢菌病原菌的识别和响应,由于基因型的不同,表现出不同的感抗性。
LRR结构域是一段含有1—47个LRR的蛋白片段,在感测各种信号以调节植物发育和环境响应方面发挥着核心作用[37-38]。为潜在病原体的存在提供预警系统,并激活植物中的保护性免疫信号[39]。单独含有LRR结构域的基因参与植物抗病报道比较少,不过之前有研究发现,在拟南芥中受体被鉴定为富亮氨酸重复受体样蛋白(responsiveness to botrytis polygalacturonases1,RBPG1)能识别真菌内分泌的内聚半乳糖醛酸酶(endopolygalacturonase,endoPG),而endoPG能够降解细胞壁聚合物从而渗透入细胞内。在柑橘中,之前研究发现链格孢菌的endoPG基因突变体会导致柑橘褐斑病的症状显著降低[24]。本研究中,、和在‘秤砣红橘’感病48 h时其表达量分别上调40倍、90倍和70倍,推测、和可能通过参与识别endoPG来参与褐斑病感病过程。同时发现,和的表达量上调幅度要高于和,因此推测这两个基因可能对于褐斑病的发生更加敏感;尤其是在‘新克里曼丁’感病后的48 h,其表达量也上调了约9倍,猜测可能属于第一层防御系统,通过感知链格孢菌入侵信号并将信号传递下去以激活下游基因参与病原菌的侵染过程。但本研究只针对R基因进行了探索,之前有研究表明柑橘感染褐斑病与乙烯、类黄酮、己酸的代谢有关[2,40-41]。后续可以在3号染色体定位的褐斑病抗性基因区域挖掘与乙烯、类黄酮、己酸代谢相关的基因进行研究,以探索出更多宽皮柑橘抗褐斑病的基因。
4 结论
136份宽皮柑橘中有121份春、秋2次接种结果一致。其中大部分克里曼丁类和温州蜜柑类基本表现为抗褐斑病,而红橘类和椪柑类都表现为感褐斑病。利用CAPS识别柑橘褐斑病抗感性状的准确率为76.86%,表现为中等程度相关,相关系数为0.534。通过GWAS共筛选出6个与褐斑病抗性相关的SNP,通过Fst共筛选出8个与褐斑病抗性相关的SNP。GWAS筛选出在3号染色体24 838 146 bp位置的SNP为最显著的位点,该位置的基因型能比较有效地将抗病资源和感病资源进行区分,表现为强相关。筛选出的候选基因、、、、在感病品种‘秤砣红橘’接种后表达量上调,且在接种48 h后表达量最高,这5个基因为宽皮柑橘抗褐斑病的候选基因。
[1] GAI Y P, MA H J, CHEN Y N, LI L, CAO Y Z, WANG M S, SUN X P, JIAO C, RIELY B K, LI H Y. Chromosome-scale genome sequence ofcausingbrown spot of. Molecular Plant-Microbe Interactions, 2021, 34(7): 726-732.
[2] LLORENS E, FERNÁNDEZ-CRESPO E, VICEDO B, LAPEÑA L, GARCÍA-AGUSTÍN P. Enhancement of the citrus immune system provides effective resistance againstbrown spot disease. Journal of Plant Physiology, 2013, 170(2): 146-154.
[3] 陈昌胜, 黄峰, 程兰, 冯春刚, 黄涛江, 李红叶. 红橘褐斑病病原鉴定. 植物病理学报, 2011, 41(5): 449-455.
CHEN C S, HUANG F, CHENG L, FENG C G, HUANG T J, LI H Y. Identification of the pathogenic fungus causing brown spot on tangerine (CV. Hongjv. Acta Phytopathologica Sinica, 2011, 41(5): 449-455. (in Chinese)
[4] 张玉洁, 杨续旺, 张志信, 李红超, 田洪, 张铁. 柑橘褐斑病的病原分离和药物筛选. 北方园艺, 2010(14): 169-171.
ZHANG Y J, YANG X W, ZHANG Z X, LI H C, TIAN H, ZHANG T. Isolation ofblack patch pathogen and comparison of the sensitivities to eleven kind fungicides. Northern Horticulture, 2010(14): 169-171. (in Chinese)
[5] REIS R F, DE ALMEIDA T F, STUCHI E S, DE GOES A. Susceptibility of citrus species to, the causal agent of thebrown spot. Scientia Horticulturae, 2007, 113(4): 336-342.
[6] SOLEL Z, KIMCHI M. Susceptibility and resistance ofgenotypes topv. citri. Journal of Phytopathology, 1997, 145(8/9): 389-391.
[7] 符雨诗, 罗君琴, 徐建国, 李红叶. 不同柑橘品种对链格孢褐斑病的感病性离体评价. 浙江农业学报, 2016, 28(1): 84-89.
FU Y S, LUO J Q, XU J G, LI H Y.susceptibility evaluation of citrus cultivars topv.citri. Acta Agriculturae Zhejiangensis, 2016, 28(1): 84-89. (in Chinese)
[8] DALKILIC Z, TIMMER L W, GMITTER F G. Linkage of andisease resistance gene in mandarin hybrids with RAPD fragments. Journal of the American Society for Horticultural Science, 2005, 130(2): 191-195.
[9] CUENCA J, ALEZA P, VICENT A, BRUNEL D, OLLITRAULT P, NAVARRO L. Genetically based location from triploid populations and gene ontology of a 3.3-mb genome region linked tobrown spot resistance in citrus reveal clusters of resistance genes. PLoS ONE, 2013, 8(10): e76755.
[10] CUENCA J, ALEZA P, GARCIA-LOR A, OLLITRAULT P, NAVARRO L. Fine mapping for identification ofbrown spot candidate resistance genes and development of new SNP markers for marker-assisted selection. Frontiers in Plant Science, 2016, 7: 1948.
[11] ARLOTTA C, CIACCIULLI A, STRANO M C, CAFARO V, SALONIA F, CARUSO P, LICCIARDELLO C, RUSSO G, SMITH M W, CUENCA J, ALEZA P, CARUSO M. Disease resistantbreeding using newly developed high resolution melting and CAPS protocols forbrown spot marker assisted selection. Agronomy, 2020, 10(9): 1368.
[12] 唐科志, 周常勇. 红橘响应褐斑病菌侵染的转录组学分析. 中国农业科学, 2020, 53(22): 4584-4600.doi: 10.3864/j.issn.0578-1752.2020. 22.006.
TANG K Z, ZHOU C Y. Transcriptome analysis ofblanco, cv.Hongjv infected withtangerine pathotype. Scientia Agricultura Sinica, 2020, 53(22): 4584-4600. doi: 10.3864/j.issn.0578-1752.2020.22.006. (in Chinese)
[13] DEMIRJIAN C, VAILLEAU F, BERTHOMÉ R, ROUX F. Genome-wide association studies in plant pathosystems: Success or failure? Trends in Plant Science, 2023, 28(4): 471-485.
[14] LI Y L, RUPERAO P, BATLEY J, EDWARDS D, DAVIDSON J, HOBSON K, SUTTON T. Genome analysis identified novel candidate genes forblight resistance in chickpea using whole genome re-sequencing data. Frontiers in Plant Science, 2017, 8: 359.
[15] 刘荣萍, 胡军华, 姚廷山, 王雪莲, 左佩佩, 王延杰, 李鸿筠. 柑橘褐斑病室内快速评价方法的研究. 果树学报, 2013, 30(5): 889-892.
LIU R P, HU J H, YAO T S, WANG X L, ZUO P P, WANG Y J, LI H Y. A rapid laboratory evaluation method of citrus brown spot caused byalternate. Journal of Fruit Science, 2013, 30(5): 889-892. (in Chinese)
[16] 王小柯, 江东, 孙珍珠. 利用GBS技术研究240份宽皮柑橘的系统演化. 中国农业科学, 2017, 50(9): 1666-1673.doi: 10.3864/j.issn. 0578-1752.2017.09.012.
WANG X K, JIANG D, SUN Z Z. Study on phylogeny of 240 mandarin accessions with genotyping-by-sequencing technology. Scientia Agricultura Sinica, 2017, 50(9): 1666-1673. doi: 10.3864/ j.issn.0578-1752.2017.09.012. (in Chinese)
[17] WRIGHT S. The genetical structure of populations. Annals of Eugenics, 1951, 15(4): 323-354.
[18] PEEVER T L, OLSEN L, IBAÑEZ A, TIMMER L W. Genetic differentiation and host specificity among populations ofspp. causing brown spot of grapefruit and tangerine × grapefruit hybrids in. Phytopathology, 2000, 90(4): 407-414.
[19] BHATTARAI G, SHI A N, MOU B Q, CORRELL J C. Resequencing worldwide spinach germplasm for identification of field resistance QTLs to downy mildew and assessment of genomic selection methods. Horticulture Research, 2022, 9: uhac205.
[20] ALAVILLI H, LEE J J, YOU C R, POLI Y, KIM H J, JAIN A, SONG K. GWAS reveals a novel candidate gene CmoAP2/ERF in Pumpkin () involved in resistance to powdery mildew. International Journal of Molecular Sciences, 2022, 23(12): 6524.
[21] PHUKE R M, HE X Y, JULIANA P, KABIR M R, ROY K K, MARZA F, ROY C, SINGH G P, CHAWADE A, JOSHI A K, SINGH P K. Identification of genomic regions and sources for wheat blast resistance through GWAS in Indian wheat genotypes. Genes, 2022, 13(4): 596.
[22] TERAKAMI S, ADACHI Y, IKETANI H, SATO Y, SAWAMURA Y, TAKADA N, NISHITANI C, YAMAMOTO T. Genetic mapping of genes for susceptibility to black spot disease in Japanese pears. Genome, 2007, 50(8): 735-741.
[23] MORIYA S, TERAKAMI S, OKADA K, SHIMIZU T, ADACHI Y, KATAYOSE Y, FUJISAWA H, WU J, KANAMORI H, YAMAMOTO T, ABE K. Identification of candidate genes responsible for the susceptibility of apple (× domestica Borkh.) toblotch. BMC Plant Biology, 2019, 19(1): 1-13.
[24] DALIO R J D, MAGALHÃES D M, RODRIGUES C M, ARENA G D, OLIVEIRA T S, SOUZA-NETO R R, PICCHI S C, MARTINS P M M, SANTOS P J C, MAXIMO H J, PACHECO I S, DE SOUZA A A, MACHADO M A. PAMPs, PRRs, effectors and R-genes associated with citrus-pathogen interactions. Annals of Botany, 2017, 119(5): 749-774.
[25] KOURELIS J, VAN DER HOORN R A L. Defended to the nines: 25 years of resistance gene cloning identifies nine mechanisms for R protein function. The Plant Cell, 2018, 30(2): 285-299.
[26] WANG J, TAN S J, ZHANG L, LI P, TIAN D C. Co-variation among major classes of LRR-encoding genes in two pairs of plant species. Journal of Molecular Evolution, 2011, 72(5/6): 498-509.
[27] SCHWESSINGER B, ZIPFEL C. News from the frontline: recent insights into PAMP-triggered immunity in plants. Current Opinion in Plant Biology, 2008, 11(4): 389-395.
[28] CHAPARRO-GARCIA A, WILKINSON R C, GIMENEZ-IBANEZ S, FINDLAY K, COFFEY M D, ZIPFEL C, RATHJEN J P, KAMOUN S, SCHORNACK S. The receptor-like kinase SERK3/ BAK1 is required for basal resistance against the late blight pathogenin. PLoS One, 2011, 6(1): e16608.
[29] DANNA C H, MILLET Y A, KOLLER T, HAN S W, BENT A F, RONALD P C, AUSUBEL F M. Theflagellin receptor FLS2 mediates the perception ofAx21 secreted peptides. Proceedings of the National Academy of Sciences of the United States of America, 2011, 108(22): 9286-9291.
[30] TAKAI R, ISOGAI A, TAKAYAMA S, CHE F S. Analysis of flagellin perception mediated by flg22 receptor OsFLS2 in rice. Molecular Plant-Microbe Interactions, 2008, 21(12): 1635-1642.
[31] ENCISO-RODRÍGUEZ F E, GONZÁLEZ C, RODRÍGUEZ E A, LÓPEZ C E, LANDSMAN D, BARRERO L S, MARIÑO-RAMÍREZ L. Identification of immunity related genes to study the:pathosystem. PLoS One, 2013, 8(7): e68500.
[32] LI J Y, WANG X D, ZHANG L R, MENG Q F, ZHANG N, YANG W X, LIU D Q. A wheat NBS-geneparticipates in-mediated resistance to. Plant Physiology and Biochemistry, 2017, 119: 1-8.
[33] LV L L, LIU Y S, BAI S H, TURAKULOV K S, DONG C H, ZHANG Y G. A TIR-NBS-gene MdTNL1 regulates resistance toleaf spot in apple. International Journal of Molecular Sciences, 2022, 23(11): 6323.
[34] GAO Y L, WANG B W, XU Z L, LI M Y, SONG Z B, LI W Z, LI Y P. Tobacco serine/threonine protein kinase gene NrSTK enhances black shank resistance. Genetics and Molecular Research, 2015, 14(4): 16415-16424.
[35] 马璐琳, 段青, 杜文文, 张艺萍, 崔光芬, 贾文杰, 吴学尉, 王祥宁, 王继华. 泸定百合丝氨酸/苏氨酸蛋白激酶基因(LsS/TK)的克隆与表达分析. 分子植物育种, 2020, 18(18): 5925-5932.
MA L L, DUAN Q, DU W W, ZHANG Y P, CUI G F, JIA W J, WU X W, WANG X N, WANG J H. Cloning and expression analysis of a serine/threonine protein kinase gene (LsS/TK) in. Molecular Plant Breeding, 2020, 18(18): 5925-5932. (in Chinese)
[36] BRUEGGEMAN R, DRADER T, KLEINHOFS A. The barley serine/threonine kinase gene Rpg1 providing resistance to stem rust belongs to a gene family with five other members encoding kinase domains. Theoretical and Applied Genetics, 2006, 113(6): 1147-1158.
[37] CHAKRABORTY S, NGUYEN B, WASTI S D, XU G Z. Plant leucine-rich repeat receptor kinase (LRR-RK): Structure, ligand perception, and activation mechanism. Molecules, 2019, 24(17): 3081.
[38] CHEN T S. Identification and characterization of the LRR repeats in plant LRR-RLKs. BMC Molecular and Cell Biology, 2021, 22(1): 9.
[39] MATSUSHIMA N, MIYASHITA H. Leucine-rich repeat (LRR) domains containing intervening motifs in plants. Biomolecules, 2012, 2(2): 288-311.
[40] DÍAZ L, DEL RÍO J A, PÉREZ-GILABERT M, ORTUÑO A. Involvement of an extracellular fungus laccase in the flavonoid metabolism infruits inoculated with. Plant Physiology and Biochemistry, 2015, 89: 11-17.
[41] ORTUÑO A, NEMSA I, ALVAREZ N, LACASA A, PORRAS I, GARCIA LIDÓN A, DEL RÍO J A. Correlation of ethylene synthesis infruits and their susceptibility topv.. Physiological and Molecular Plant Pathology, 2008, 72(4/5/6): 162-166.
Genome Wide Association Study for Resistance to Citrus Brown Spot Disease
YANG ShengNan, CHENG Li, TAN YueXia, ZHU YanSong, JIANG Dong
Citrus Research Institute of Southwest University, Chongqing 400712
【Objective】The study objective was to explore candidate genes associated with resistance to Alternaria brown spot disease in citrus mandarin, so as to provide the basis for developing suitable molecular markers for further citrus resistance breeding work. 【Method】In summer and autumn of 2022, the young leaves of 136 citrus mandarin accessions were picked and inoculated with the fungus mycelium ofin the laboratory. The combined results of two experiments were used to obtain the reliable results of 121 citrus mandarins responding to Alternaria brown spot. The phenotypes of 121 citrus mandarins were selected to verify the effectiveness of the CAPS marker developed in a previous study. Then, in order to obtain the candidate SNPs related to Alternaria brown spot resistance, the phenotypes of 121 citrus mandarins and corresponding SNP genotype data from GBS sequencing were analyzed with PCA, GWAS, and Fstmethods, respectively. Candidate genes had been selected from the flanking region of 25 kb sequences surrounding the candidate SNPs site, and then they were screened out according to phytozome annotations. The expressions of candidate genes were analyzed after inoculating with the pathogen fungus on leave of Chengtuo hongju and Clementina (Algeria) for 24, 48, and 72 h. 【Result】Among 121 citrus mandarins, 67 varieties such as Clementine and Satsuma were resistant to Alternaria brown spot disease, whereas 54 varieties such as Hong Ju and Ponkan were susceptible. Some varieties resistance to Alternaria brown spot disease could discriminated by CAPS, but its accuracy only accounted for 76.86% in the study. GWAS analysis identified six significant SNPs highly associated with disease resistance, among which SNP1 located inat 24 838 146 bp of chromosome 3 could be used to predicate the resistance of varieties, and their genotype showed a strong correlation with phenotype. Eight significant SNPs highly associated with disease resistance selected by Fstanalysis.Finally, five genes of,,,andwere screened out. The expression levels of these five genes in Chengtuo Hongju were up-regulated extensively after inoculating leaves with the pathogen fungus, and their expression levels reached the highest 48 hours later. 【Conclusion】Through GWAS, the SNP at 24 838 146 bp on chromosome 3 was found to be the most significant one with high resistance correlation to Alternaria brown spot disease, and the genotype at this location could be effectively used to distinguish the resistant varieties. The candidate genes responding to Alternaria brown spot disease in mandarin were discussed, i.e.,,,,, and.
citrus mandarin; Alternaria brown spot; molecular marker; genome-wide association study; fixation index
10.3864/j.issn.0578-1752.2023.18.012
2023-02-14;
2023-05-04
国家重点研发计划(2019YFD1001400)、柑橘种质资源精准鉴定(19211142)
杨胜男,E-mail:y_sn@qq.com。通信作者江东,E-mail:jiangdong@cric.cn
(责任编辑 赵伶俐)
