利用玉米F1 群体进行玉米全株鲜重的全基因组预测分析
2023-10-13杨宗莹肖贵张红伟
杨宗莹 肖贵 张红伟
(1 吉林农业大学农学院,130118,吉林长春;2 定西市农业科学研究院,743000,甘肃定西;3 中国农业科学院作物科学研究所,100193,北京)
玉米是我国第一大粮食作物,在我国乃至全世界都占据着至关重要的地位[1]。当前,由于人口增加和资源短缺,粮食安全面临着严峻的威胁,所以急需开发生物量和产量高的玉米品种[2]。与籽粒玉米相比,青贮玉米具有更高的经济价值。近年来,随着畜牧业的快速发展,青贮玉米的需求也显著提高,选育出更高产的青贮玉米品种尤为重要。植株鲜重即单位面积内地上有机物质总质量代表着全株生物量,玉米鲜重与产量相关性较大,且生物量是青贮玉米品种选育中的重要参考指标[3]。因此,对玉米生物量性状进行研究,可以为选育鲜重高的玉米新品种提供参考。
基因组预测(genomic prediction,GP)是由Meuwissen 最早提出的[4],它可以预测作物品种或种质资源的表现。与其他分子育种工具相比,基因组预测能同时估计标记效应和计算育种值,不需要对标记—性状的相关性进行检测;基因组预测还可加快育种周期、提高遗传增益来提高育种效率[5]。但不同群体间基因组预测准确性较低,导致选择效率降低,制约了该技术在田间的应用[6-7]。
对杂交组合表型进行预测可以减少杂交制种和田间测试的过程,大大减少工作量,节省杂交组合选育和测试的成本,并提高杂交组合的选育效率。Xu 等[8]利用全基因组选择技术以278 份杂交种作为训练群体,预测了21 945 个潜在杂交种;Cui 等[9]以1495 个不同品种的杂交水稻作为训练群体,预测了3000 份水稻种质育成的44 636 个潜在杂交组合的表现,并在此基础上确定了200 个最好的潜在杂交组合[10-11]。这都证明全基因组预测可以在大规模缩减工作量和经费的条件下,选出最好的杂交组合。
本研究构建了4 个玉米杂交组合群体,并对群体的全株鲜重进行评估。然后对所有材料进行亲缘关系分析,设计不同的基因组预测方案,评估不同基因组预测方案的准确度,找到最佳的方案,为杂交组合基因组预测提供参考。
1 材料与方法
1.1 试验材料构建
以青贮玉米材料的骨干自交系中北410 的父母本(SN915×YH-1)以及北农368 的父母本(60271×2193)4 个材料为父本,中北410 和北农368 为专用型青贮玉米,具有生物量大和产量高的优势。以实验室青贮性状表型优异的120 个自交系材料为母本,通过杂交组配出一个4×120 的群体,用于后续的表型鉴定。田间试验期间按照常规农田管理。4 个群体依次命名为群体1(P1,SN915×母本)、群体2(P2,YH-1×母本)、群体3(P3,2193×母本)和群体4(P4,60271×母本)。
1.2 试验材料鉴定
在2019 年,分别在河北廊坊、新疆昌吉和甘肃定西种植480 份F1材料,按照完全随机设计,每个地点设置2 个重复,双行区种植,行长5m,行距60cm,株距25cm,每行种植21 株。全株鲜重鉴定标准为散粉期45d 时,收获2 行材料中生长一致的8 株进行称重,精确到0.05kg,取其平均值代表全株鲜重。3 个种植环境分别用LF(河北廊坊)、CJ(新疆昌吉)和DX(甘肃定西)表示。
1.3 表型数据处理
对于获得的一年多点数据,首先利用R 语言分别计算每个环境的平均数、标准差、变异系数等基本统计量。根据田间试验设计计算材料的BLUE值和遗传力等参数,BLUE 值计算模型[12-13]:yikm=μ+gi+τk+gτik+δ(k)m+εikm。
式中,yikm代表第ith(i=1,2,...,480)个材料在第kth(k=1,2,3)个环境、mth(m=1,2)个重复下的表型值,μ是总体平均数,gi是基因型效应,τk是环境效应,gτik是基因型和环境互作效应,δ(k)m是第k个环境内第m个重复的效应,εikm是随机误差效应,服从正态分布并且相互独立。计算材料的BLUE 值时,除了基因型效应为固定效应外,其他因素均为随机效应,并假设服从正态分布。使用R 语言lm4 包进行拟合[14]计算BLUE 值。
计算广义遗传力(H2)的公式[15]:H2=Vg/[Vg+(VGL/L)+(Ve/RL)]。
式中,Vg是材料的方差组分,VGL是材料与地点的交互方差组分,Ve为残差方差组分,L是环境个数,R是地点内重复数。
1.4 基因分型及基因数据分析
取所有材料亲本叶片,采用改良CTAB 方法提取DNA[16],使用中玉金标记的10k 育种芯片进行基因组鉴定,SNP 质控标准如下:删除亲本有多态性的标记;去除缺失率>20%、杂合率>20%的亲本基因型;删除没有物理位置信息的SNP;去除最小等位基因频率(MAF<0.05),对确实标记利用Beagle 进行填充,获得亲本基因型,包含7120 个SNP 位点。根据双亲的基因型信息,推测出每个杂交组合的基因型[9]。利用过滤后的SNP进行主成分分析。
1.5 全基因组预测模型
用4 个群体的BLUE 值进行全基因组选择。考虑加性和显性遗传效应的基因组BLUP(GBLUP)模型为y=μ+ξa+ξd+ε。
式中,y是F1杂交种的BLUE 值,μ是总体平均值,ξa是服从ξa~N(0,Kaσa2)分布的加性多基因效应向量,ξd是服从ξd~N(0,Kdσd2)分布的显性多基因效应向量,ε是正态分布的残差ε~N(0,Iσε2),其中I是单位矩阵,σε2是残差方差。ξa和ξd是由单位矩阵构成的。利用R 包BGLR 对线性混合模型进行拟合[17](参数nIter 和burnin 分别是15 000 和1000)。分别使用了GBLUP 和Bayes B这2 种全基因组预测模型。
1.6 全基因组预测
通过设计不同的训练群体进行全基因组预测,主要有2 种设计,第1 种是4 个群体中的1个群体分别预测其他3 个群体;第2 种是4 个群体中的3 个群体和剩余1 个群体随机选取一半预测剩余1 个群体的一半。利用BLUE 值进行基因组预测,预测准确性(PA)是预测值与测量值之间的相关系数。基因组预测模型采用5 折交叉验证方案,重复200 次。PA 为200 次的平均值。
2 结果与分析
2.1 表型数据分析
在3 种环境中,甘肃定西的材料全株鲜重最高。在4 个杂交群体中,P2 的全株鲜重最高,其次是P1 和P4,最小的是P3(图1)。结果表明这个群体中YH-1 的一般配合力最好(图1,表1)。对于每个群体来说,3 个环境之间的相关性都显著,这表明遗传基础在3 个环境中发挥了重要的作用(图2)。利用BLUE 值计算,全株鲜重的H2为0.66,虽然3 个环境之间有所差异,但是每个环境都是稳定的。以上结果表明遗传因素对全株鲜重起主要作用。
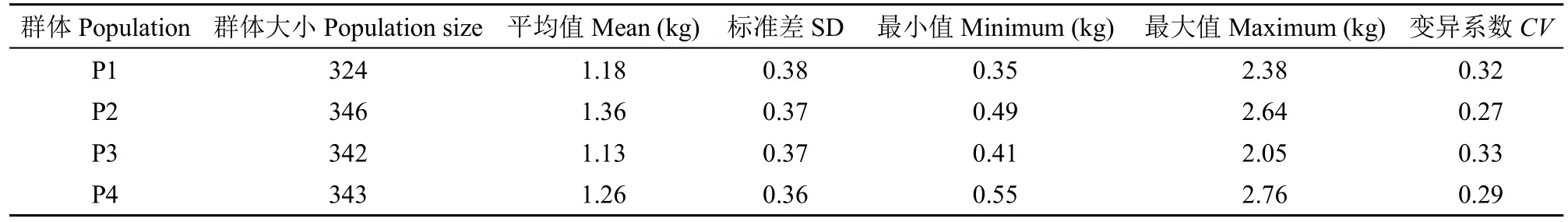
表1 4 个群体鲜重基本统计数据汇总Table 1 Summary of the basic statistics of fresh weight of four groups
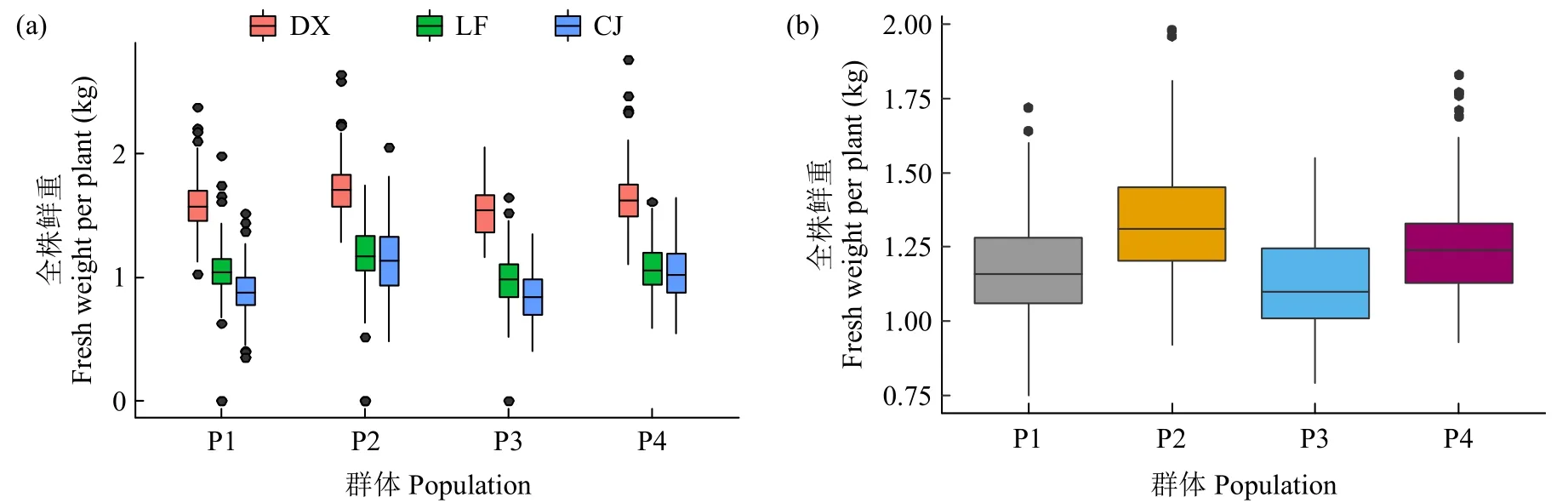
图1 不同环境中材料及不同群体全株鲜重汇总Fig.1 Summary of fresh weight data of different environmental materials and different groups
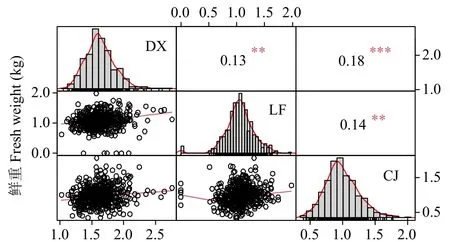
图2 3 个环境中全株鲜重相关性分析Fig.2 Correlation analysis of fresh weight per plant in three environments
2.2 多群体间的遗传关系
经过SNP 筛选和定位后,总共获得7119 个高质量的SNP。这些SNP 均匀分布在10 条染色体上(图3a),而且标记密度足够高,可以用于全基因组预测[18]。对所有亲本的进化树分析结果表明了北农368 父母本(60271,2193)亲缘关系较近,中北410 的亲本(SN915,YH-1)亲缘关系较远,所有材料共分为3 个部分(图3b)。考虑到群体之间存在亲缘关系,利用基因型数据对4 个群体间的遗传关系进行分析。由主成分分析(PCA)结果(图3c)可以看出,4 个群体中P3 和P4 的亲缘关系最近,PC1 和PC2 可以解释总方差的20.91%和14.85%。亲缘关系热图(图3d)显示P3 和P4亲缘关系较为密切,与PCA 的结果是一致的。聚类分析也表明所有材料分为3 个部分,YH-1 为父本组配的一个群体(P1)、SN915 为父本组配的一个群体(P2)以及60271 和2193 为父本组配的一个群体(P3,P4)(图3c)。


图3 测序标记及遗传相似性分析Fig.3 Sequencing markers and genetic similarity analysis
2.3 不同群体的群基因组预测
通过构建不同的训练群体,利用GBLUP 和BayesB 这2 种模型进行预测,发现2 种模型的预测准确性差异不大(图4)。全基因组预测的结果表明,第2 种预测方案的预测准确性较高于第1种,即杂交群体间的基因组预测准确性低于多群体作为训练群体的预测准确性,且当训练群体中包含预测群中的材料时,预期准确性变高。亲缘关系较近的2 个群体间预测准确性更高。

图4 利用GBLUP 和BayesB 模型对不同群体全株鲜重的全基因组预测Fig.4 Genome-wide prediction of fresh weight per plant in different populations using GBLUP and BayesB models
3 讨论
基因组预测是一种重要的分子育种技术,对玉米品种的改良具有重要意义,特别是对复杂性状的改良[19-20]。尽管以前报道过在动植物中基于多种群的基因组预测研究[6-7,21],但这些研究中使用的群体是自然群体或纯合株系组成的群体。然而生产中应用的是杂交种,因此对F1群体的基因组预测具有重要意义。本研究在分析F1群体亲缘关系的基础上,利用F1群体进行全基因组预测,对于开展基于F1群体的全基因组选择育种具有参考价值。
训练群体大小、训练群体与预测群体亲缘关系都会影响基因组预测[22-23],我们也发现不同群体的全基因组预测的准确性与训练和验证群体之间的关系有关。例如,我们通过尝试不同的训练群体,发现训练和验证群体之间的关系是可以影响预测准确性的。因此,训练群体与验证群体之间的遗传关联性是造成群体遗传变异的重要因素之一。此外,4 个群体间性状遗传基础的差异可能是影响全基因组预测准确性的另一个重要因素[17]。训练群体和验证群体之间的遗传关系越密切,它们共同的遗传基础就越多,进行全基因组预测的准确性会增加。基于这一推测,当预测群体的个体包含在训练群体中时,训练群体和验证群体的遗传关系将是密切的。因此我们设计了第2 种预测方案来验证这一假设,发现第2 种方案的预测准确性高于群体间预测准确性,这一结果支持了我们的推测。
4 结论
杂交群体间的基因组预测准确性较低。通过改进训练群体的组成,加入与训练群体有关的群体可以提高杂交群体基因组预测的准确性。
