大豆根瘤菌菌株5873 PCR快速检测方法的建立及应用效果评价
2023-08-15马鸣超姜昕王鹏辉关大伟李俊
马鸣超,姜昕,王鹏辉,关大伟,李俊
大豆根瘤菌菌株5873 PCR快速检测方法的建立及应用效果评价
马鸣超,姜昕,王鹏辉,关大伟,李俊
中国农业科学院农业资源与农业区划研究所,北京 100081
【目的】大豆根瘤菌()是微生物肥料重要的功能菌种之一,可通过生物固氮为大豆生长提供氮素,实现节本增效,菌株5873作为其典型代表,已广泛应用于农业生产。筛选并鉴定其特异引物,建立大豆根瘤菌5873菌株水平的快速检测方法,对微生物肥料生产菌种鉴定、产品质量检测和功能评价至关重要。【方法】以商业化菌株大豆根瘤菌5873为供试材料,基于其全基因组序列和NCBI数据库中大豆根瘤菌种内参比菌株相关序列,以及与其高度同源(基因组ANI值大于99.95%)的大豆根瘤菌USDA 6T差异片段,通过多重序列比对,进行特异引物设计和筛选,获得特异性引物对,并通过对PCR反应条件/体系优化、特异性及灵敏度检测,建立大豆根瘤菌5873的快速检测方法。然后,采用盆栽试验,将大豆根瘤菌5873与其他根瘤菌菌株混合接种于大豆根际,应用上述方法对大豆根瘤菌5873竞争结瘤能力进行评价和方法验证。【结果】筛选获得了一组特异引物4-4和Q1(4-4-F 5′-GATAAGGCCACGGGTGAACA-3′/4-4-R 5′-CACTCGATAAGCTCCGCTGT-3′和Q1-F 5′-CCGGTCGTGACTGGAATGAT-3′/Q1-R 5′-TCGAGGCCTACAAGAACGTC-3′),优化并建立了PCR快速检测方法,即反应体系:Premix TaqTM12.5 μL,引物各1.0 μL,基因组DNA 15 ng左右,加ddH2O补足至25 μL;反应条件:95℃ 预热5 min,94℃ 变性45 s,61℃ 退火45 s,72℃ 延伸60 s,30个循环,再72℃ 延伸10 min。通过凝胶电泳检测目的条带的有无(355和218 bp),即可实现大豆根瘤菌5873的快速检测,该方法检出灵敏度为1 850 CFU/µL。此外,借助该方法可以成功地评价大豆根瘤菌5873竞争结瘤能力,与传统BOX-PCR评价结果一致。【结论】大豆根瘤菌5873快速鉴定方法的建立实现了以菌体发酵液或根瘤破碎提取液为模板,直接进行PCR扩增的鉴定操作,省去了根瘤的分离、根瘤菌分离纯化、培养、DNA提取及测序鉴定等繁琐的环节,大大减少了工作量,只需短短几个小时便可准确检测大豆根瘤菌5873,为微生物肥料中根瘤菌菌剂的产品质量检测和竞争结瘤能力评价提供了参考。
大豆根瘤菌;特异引物;PCR扩增;竞争结瘤
0 引言
【研究意义】随着国家在农业战略上的调整,发展资源节约型、环境友好型的“两型农业”成为现代农业可持续发展的必由之路[1]。其中,包括根瘤菌在内的微生物肥料作为绿色新型投入品和优先发展的生物制品,在减肥减药、节本增效、提高作物品质等方面表现突出[2]。大豆根瘤菌()可与大豆形成高效的共生固氮体系,有效减少化学氮肥的施用[3-4],是微生物肥料中重要的功能菌种之一,大豆根瘤菌5873作为其典型代表,目前已在农业农村部登记并广泛应用于农业生产。筛选并鉴定其特异性引物,建立菌株水平的快速检测方法,对微生物肥料产品质量检测、功能评价及菌株知识产权保护具有重要的研究意义和实践价值。【前人研究进展】目前对于大豆根瘤菌的检测主要依靠传统的形态学分析和Biolog等生理生化鉴定[5-6],基于16S rDNA、、、、、、和基因等保守序列的系统发育学分析等传统多相分类鉴定[7-13],以及新发展起来的全基因组测序[14]、平均核苷酸一致性(ANI)比较[15]、多位点序列分析(MLSA)法[16]、BOX-PCR指纹图谱分析[17]等技术,其结果通常可实现种水平、菌株水平上的鉴别[14-19],但由于操作步骤多、时间长,难以广泛地实施推广。以慢生大豆根瘤菌为例,包括在国内外广泛商业化应用的菌株大豆根瘤菌USDA 110T[20](后改为有效慢生根瘤菌[21])和国内外研究较为常见模式菌株大豆根瘤菌USDA 6T[15,22-23]在内,均未建立菌株水平的快速检测技术。此外,在全基因组水平上,大豆根瘤菌5873和USDA 6T的基因组ANI值等于99.95%,前者与后者相比缺失了一段51 bp的序列,另外,两个菌株间还有3个SNP位点差异,常规技术手段很难区分。在应用层面上,土壤中存在大量竞争结瘤能力强而固氮能力弱的土著根瘤菌群,这直接影响到接种根瘤菌剂的占瘤率,导致其接种效果不理想[24-25]。目前,BOX-PCR被广泛地应用于根瘤菌占瘤率的测定[26-27],但在操作中需要根瘤菌分离纯化培养、DNA提取及需要结合基因测序鉴定等繁琐环节,费时费力,且试验结果重复性差,实验室间结果很难重现。【本研究切入点】鉴于以上问题,亟需建立菌株快速检测和评价技术。因此,本研究选用自主研发且已商业化的大豆根瘤菌5873为供试材料,设计特异性引物,建立PCR快速检测方法。【拟解决的关键问题】在特异性引物设计上,除考虑大豆根瘤菌5873全基因组序列和NCBI数据库大豆根瘤菌参比菌株相关序列外,重点分析与其高度同源的大豆根瘤菌USDA 6T差异片段,设计引物,优化PCR反应条件/体系,并进行特异性及灵敏度检测,实现大豆根瘤菌菌株水平上的快速检测,为微生物肥料中根瘤菌的检测和竞争结瘤能力评价提供技术支撑。
1 材料与方法
试验于2020—2021年在中国农业科学院农业资源与农业区划研究所完成。
1.1 供试菌株
选用商业化菌株大豆根瘤菌5873为供试菌株,以实验室保藏的42株慢生根瘤菌属、2株中华根瘤菌属和1株根瘤菌属的菌株为参比菌株(含模式菌株),结合NCBI数据库中大豆根瘤菌相关序列,进行引物设计和特异性验证。供试菌株及编号见表1。

表1 供试菌株
1.2 主要试剂和仪器
TIANamp Bacteria DNA Kit(天根生化科技北京有限公司);Fast-T1化学感受态细胞、5 minTMTA/ Blunt-Zero Cloning Kit、Fast Pure Gel DNA Extraction Mini Kit、Fast Pure Plasmid Mini Kit等均购自南京诺唯赞生物科技公司;裂解液配制[28](20 mmol·L-1Tris(pH 8.0)、25 mmol·L-1NaCl、2.5 mmoL·L-1EDTA、0.05% SDS、5% chelex-100);Premix Taq(TaKaRa公司);YMA液体培养基(甘露醇10 g、七水合硫酸镁0.2 g、氯化钠0.1 g、磷酸二氢钾0.5 g、酵母膏0.5 g,定容至1 L,pH 6.8—7.2);C1000 PCR仪、凝胶成像系统(Bio-Rad公司);DYY-8C电泳仪(北京市六一仪器厂);NanoDropND-2000C微量紫外分光光度计(Thermo)。
1.3 菌株DNA提取与纯度检测
挑取培养好的单菌落,采用TIANamp Bacteria DNA Kit提取菌株DNA,经1%琼脂糖凝胶电泳检测其完整性,并用NanoDropND-2000C微量紫外分光光度计检测其浓度。
1.4 特异引物设计和筛选
大豆根瘤菌5873基因组总长度为9 160 134 bp(登录号:WWEZ00000000),选取其中scf-1序列(总长度为1 442 588 bp),初步分割成721个2 kb的片段,在GenBank数据库进行序列比对,找出同源性较低的特异序列。在NCBI数据库中,将特异序列进行primer-BLAST,综合考虑引物长度、GC含量、是否产生二聚体及发夹结构等原则,引物设计选择默认值设定为20 bp,GC含量选择为40%—60%,两引物之间不存在互补结构,进行特异性引物设计,然后从16个同源性较低的2 kb的片段中,选择较好的20对引物进行PCR验证。引物由生工生物工程(上海)股份有限公司合成。
分别提取大豆根瘤菌5873和上述参比菌株DNA,进行PCR扩增,产物经1%琼脂糖凝胶电泳检测,以筛选具有特异性的引物,同时以无菌水为阴性对照。
1.5 PCR扩增优化及方法建立
1.5.1 PCR体系/反应条件的建立与优化 以大豆根瘤菌5873基因组DNA为模板,进行PCR扩增。通过优化退火温度(54—61℃)、模板DNA量(3—51 ng)、引物添加量(浓度为10 μmol·L-1,0.5—2.0 μL)等建立最适PCR反应条件/体系。
1.5.2 菌落PCR扩增 菌株用YMA液体培养基于30℃,180 r/min培养5 d。取2 μL作为模板,以最优化的PCR条件进行菌落PCR验证。
1.5.3 灵敏度检测 将大豆根瘤菌5873接种于YMA培养基,180 r/min,30℃,培养5 d,用无菌水对上述培养液进行系列梯度稀释。分别取2 μL为模板,进行最优条件的菌落PCR反应。并将稀释倍数为105、106和107的菌液涂布于添加琼脂的YMA培养基平板上进行菌落计数。每个稀释梯度做3个平行。
1.5.4 目的条带验证 以大豆根瘤菌5873 DNA为模板,以最优PCR反应条件进行PCR扩增,PCR产物经试剂盒纯化后,与pCE2载体连接,转化,培养后,随机挑取阳性克隆,送至生工生物工程(上海)股份有限公司测序。
1.6 方法验证及评价应用
1.6.1 菌剂制备及盆栽试验 将供试菌株大豆根瘤菌5873、实验室保存的菌株大豆根瘤菌5841、5821、5136、5119、5665和商业化菌株有效慢生根瘤菌USDA 110T,分别进行液体发酵培养。然后,将大豆根瘤菌5873分别与其他6株菌两两等体积混合后作为复合菌剂待用。
选择大小相近的大豆种子,在95%的酒精中浸泡1 min,用5%次氯酸钠灭菌5 min,然后用无菌水清洗5—6次。分别将每株菌的发酵液与保护剂共同包衣在灭菌的大豆种子表面,播种在盛有灭菌蛭石的塑料盆(9.5 cm×10 cm)中,作为阳性对照;把两两混合后的复合菌剂与保护剂共同包衣后播种,作为处理组。将植株置于光照培养箱中,光照10 h、28℃,黑暗14 h、25℃。出苗后每盆留苗3棵,并以不接种为阴性对照。每个处理设3个重复,培养40 d后测定大豆根瘤数量及大豆根瘤菌5873占瘤率。
1.6.2 占瘤率测定 分别用本研究建立的PCR快速检测技术及传统BOX-PCR两种方法测定大豆根瘤菌5873占瘤率。测定时,将大豆根部置于自来水中冲洗干净,把根瘤浸泡于蒸馏水中重复3—4次,然后,将清洗的根瘤蘸取95%的酒精,用火焰进行灼烧。挑取充分灭菌的根瘤,放置于1.5 mL的EP管中,加入200 μL的裂解液,用灭菌的竹签将根瘤破碎。破碎的液体置于振荡仪上充分混合,置于沸水浴中5 min,在-20℃进行冰冻。取2.0 μL冻融的样品作模板,进行上述特异PCR扩增和BOX-PCR扩增[22],将产物电泳后统计条带,计算大豆根瘤菌5873占瘤率。
2 结果
2.1 特异引物设计及筛选
2.1.1 基因组序列比对及特异引物设计 通过对大豆根瘤菌5873和USDA 6T的基因组比较发现(图1),大豆根瘤菌5873基因组缺失了51 bp序列,经测序和比对,该缺失序列参与编码YcjX家族蛋白基因。因此,特异引物设计采用如下策略,即在NCBI数据库中进行大豆根瘤菌参比菌株序列比对,找到大豆根瘤菌5873的特异序列进行引物设计,保证其与除大豆根瘤菌USDA 6T之外的菌株有较好的特异性。其次,通过缺失段序列,设计特异性引物,以区分5873与USDA 6T。引物设计示意图见图2,引物部分结果如表2所示。

图1 菌株的序列差异比较

表2 大豆根瘤菌5873的特异序列引物设计结果
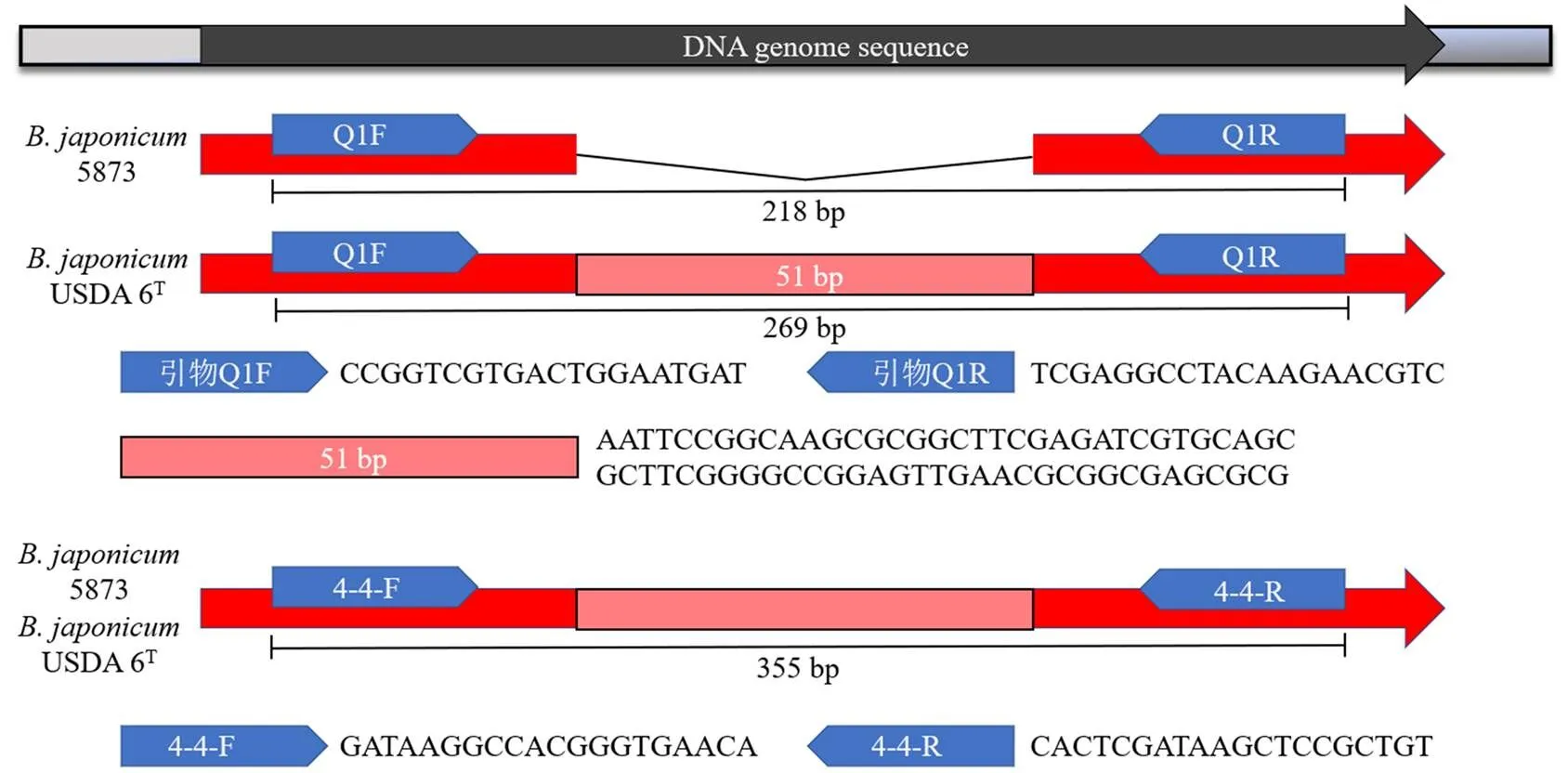
图2 引物设计示意图
2.1.2 特异引物筛选 以大豆根瘤菌5873和上述参比菌株DNA为模板,分别将设计的20对特异性引物进行PCR扩增,供试引物中只有4-4与5873、USDA 6T产生355 bp扩增条带(图3-A、3-B),表明4-4引物扩增的序列与菌株USDA 6T具有高度的同源性,与其他菌株不同源或同源性很低,使用该引物可以将5873和USDA 6T从其他菌株中区分出来。
为进一步区分,利用引物4-4、Q1及上述两对混合引物,同时扩增高度同源的5873和USDA 6T,以有效慢生根瘤菌USDA 110T为对照,结果如图4所示,使用引物4-4时,仅5873和USDA 6T可扩增出355 bp左右的条带;使用引物Q1时,5873可扩增出长度为218 bp左右的条带,而USDA 6T和有效慢生根瘤菌USDA 110T均扩增出269 bp左右的条带。因此,引物4-4和Q1复合使用,可通过检测目的条带的有无(355和218 bp),实现大豆根瘤菌5873的快速检测。
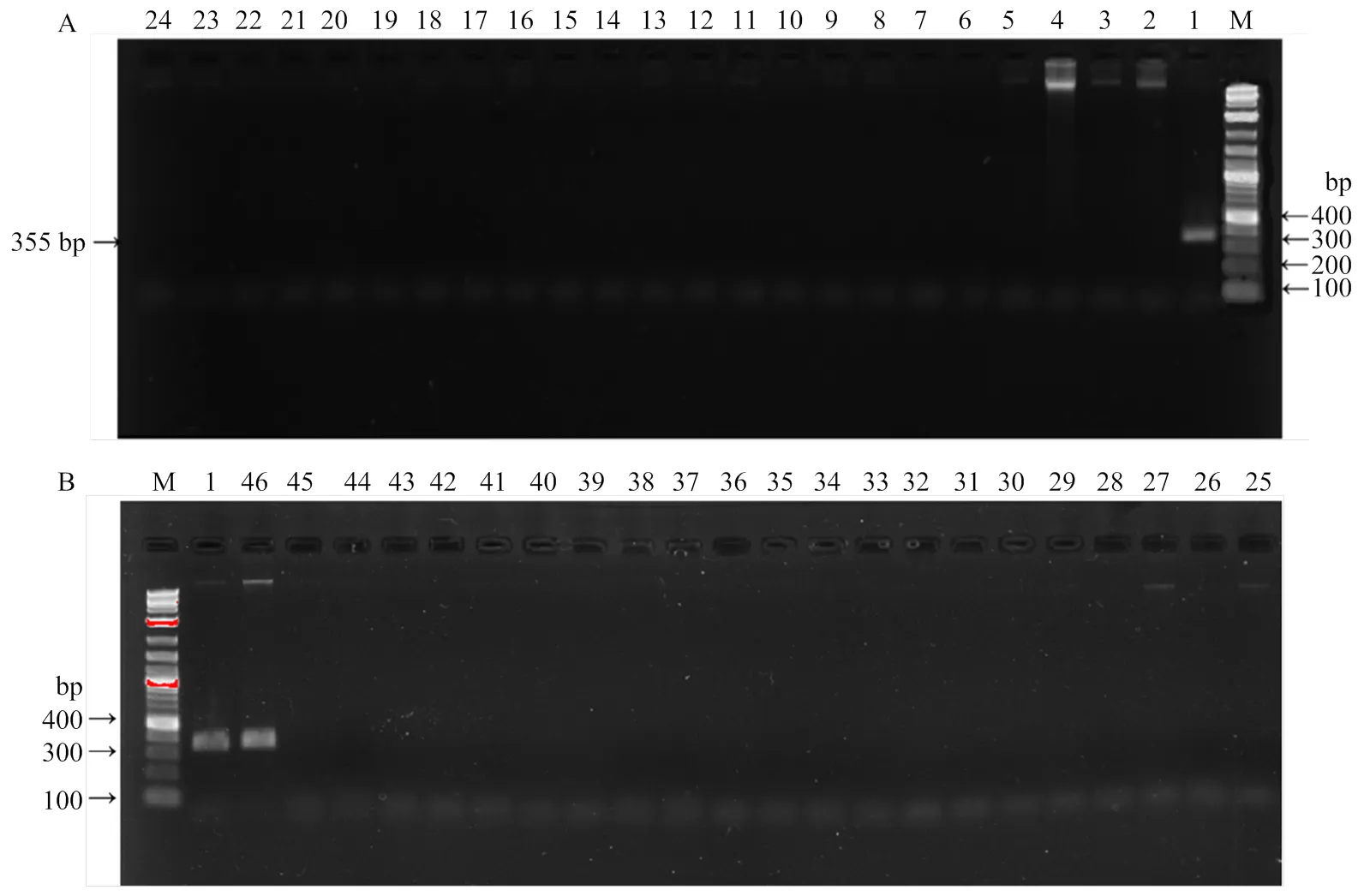
A: M: 2-Log ladder marker. 1: B. japonicum 5873; 2: B. japonicum 5841; 3: B. elkanii 5136; 4: B. diazoefficiens 5119; 5-17: B. japonicum 5821, 5129, 4892, 5452, 5599, 5016, 5437, 4129, 4470, 4530, 4438, 5547, 5248; 18: S. fredii 4475; 19: R. alamii 4370; 20-24: B. japonicum 5876, 4784, 5528, 5412, 5260. B: M: 2-Log ladder marker. 1: B. japonicum 5873; 25: B. diazoefficiens USDA 110T; 26: B. japonicum 5665; 27: S. fredii USDA 205T; 28-33: B. japonicum 5588, 4503, 5039, 4248, 5240, 5190; 34-35: B. elkanii 5109, 5161; 36-45: B. japonicum 5742, 4855, 4794, 4291, 4698, 5128, 4770, 4296, 4207, 4543; 46: B. japonicum USDA 6T

1—3:引物为4-4 The primer is 4-4;4—6:引物为Q1 The primer is Q1;7—9:4-4与Q1混合引物The mixed primers of 4-4 and Q1;M:DL1000 ladder marker;1、4、7:B. japonicum 5873;2、5、8:B. japonicum USDA 6T;3、6、9:B. diazoefficiens USDA 110T
2.2 PCR条件/体系优化和验证
2.2.1 PCR条件的优化 以大豆根瘤菌5873的基因组DNA为模板,建立PCR反应体系并进行优化。结果表明,DNA模板含量为3—51 ng时均有条带产生,但添加量为15 ng时条带开始变亮,所以体系模板DNA需15 ng以上(图5-A);在引物浓度为10 μmol·L-1时,添加均有产物形成,在添加1.0 μL时条带较亮,从成本及引物二聚体问题考虑,选择添加最适引物量为1.0 μL(图5-B);退火温度在54—61℃均有产物形成,但考虑到较高的退火温度有利于PCR反应的特异性,所以选择61℃作为最适的退火温度(图5-C)。因此,通过优化条件,确定4-4引物PCR反应的最优体系为Premix TaqTM12.5 μL,引物各1.0 μL,基因组DNA 15 ng左右,加ddH2O补足至25 μL。扩增程序如图5-D。

M:2-Log ladder Marker。A:不同DNA浓度Different concentrations of DNA templates。1—3:3 ng;4—6:15 ng;7—9:27 ng;10—12:51 ng。B:不同引物浓度Different dosages of primers。1—3:0.5 μL;4—6:1.0 μL;7—9:1.5 μL;10—12:2.0 μL,10 μmol·L-1 of each primer。C:不同退火温度Different annealing temperatures。1—8:54、55、56、57、58、59、60、61℃。D:最适的扩增程序optimized amplification procedure
2.2.2 菌落PCR扩增验证 以大豆根瘤菌5873菌液为模板,用引物4-4进行菌落PCR扩增,可扩增到355 bp长的特异性片段。由此可以说明,以基因组DNA为模板和以菌悬液为模板均可扩增出目的条带。因此,所建立的PCR快速检测方法是可行的。
2.2.3 灵敏度检测 将大豆根瘤菌5873的菌悬液连续稀释10倍,以不同稀释梯度的菌液为模板,以最优PCR体系扩增。结果表明,PCR反应体系含有超过1 850 CFU/µL时能扩增355 bp的特异性条带。因此,所建立的PCR快速检测方法的灵敏限度是每微升反应体系达到1 850 CFU以上。
2.2.4 目的条带验证 以大豆根瘤菌5873的总DNA为模板,进行特异PCR扩增。将PCR产物纯化、连接、转化、筛选后,进行测序。其结果与大豆根瘤菌5873的特异片段相似性为100%。
2.3 特异PCR快速检测评价菌株5873竞争结瘤能力
对生长40 d后的对照组大豆根瘤数进行统计,接种菌株5873的根瘤数与其他菌株无显著差异(>0.05)。应用上述建立的特异PCR快速鉴定方法和BOX-PCR,对处理组的大豆根瘤进行5873占瘤率的测定,根据电泳条带计算占瘤率,结果见表3,本研究建立的PCR快速检测方法和基于BOX-PCR方法测定的大豆根瘤菌5873占瘤率一致,该方法可以成功地评价菌株5873竞争结瘤能力,方法可行。
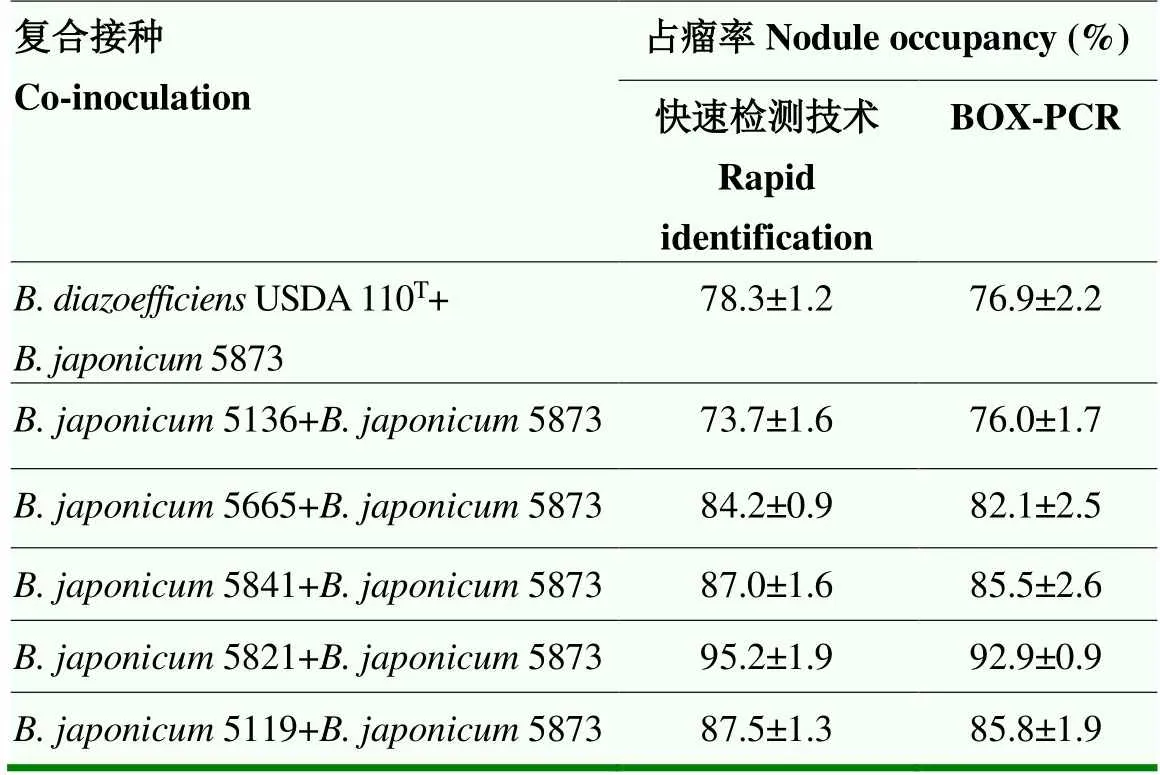
表3 大豆根瘤菌5873占瘤率
3 讨论
3.1 慢生大豆根瘤菌检测和鉴定技术
慢生根瘤菌属是大豆种植中应用最为广泛的菌种,起源于热带酸性土壤,被认为是所有根瘤菌的祖先,由于该属的菌种组成非常复杂,涉及各种类型的生态系统,属内种的确定与分类系统进展较为缓慢,分类仍然很复杂。此外,由于慢生根瘤菌属16S rRNA基因的高度保守性,只有极少的物种进行了明确的鉴定[16]。目前对于慢生大豆根瘤菌的检测和鉴定,主要依靠传统的形态学分析、Biolog生理生化鉴定、保守基因系统发育学分析、全基因组测序、平均核苷酸一致性比较、BOX-PCR指纹图谱分析等多相分类技术[7-17]。但根据形态学、生理生化表型等实验和生物功能特征设计检测方法很难将近源种准确区分;而基于16SrDNA、、基因等保守序列的系统发育学分析方法只能鉴定到种属水平[18-19]。本研究建立的特异PCR快速检测技术是在目标菌株全基因组序列的基础上,根据菌株特异的碱基序列设计引物,并进行特异PCR扩增。通过目的产物的有无及大小,对目标微生物进行准确检测或鉴定。该技术具有特异性强、敏感度高、快速灵敏的优点,为微生物肥料根瘤菌等生产菌株的快速检测提供了重要参考。
3.2 特异引物设计
特异引物的设计是特异PCR技术的关键环节,常见的特异引物设计方法包括:针对研究的特定菌株,从GenBank数据库中获取较常用的种属特异基因或功能基因,通过多重序列比对,进而确定引物序列[29-30]。例如,Wu等[31]基于基因序列设计引物,成功地利用特异引物扩增出胶质类芽孢杆菌519 bp的保守序列。而本试验并没有局限于特异基因或功能基因,而是通过对大豆根瘤菌5873的全基因组序列与NCBI中已报道的大豆根瘤菌菌株序列进行全面的筛选与比对,从而实现在最大范围内检索菌株5873的特异序列,这样可有效促进特异的非编码序列被深度挖掘和发现。在设计的20对特异引物中,包含具有功能的编码基因以及基因间的非编码序列,本研究筛选的特异引物4-4为非编码序列。同样,本课题组王璇等[32]在建立胶质类芽孢杆菌PCR快速检测方法时,也是基于基因间的一段非编码序列进行了特异引物设计。由于在物种进化的过程中,大多数的编码基因具有进化保守性,尤其在与近源种进行比对分析时,其同源性相对较高。因此,在设计种内特异引物时,非编码序列可作为筛选的重要部分。引物设计时,其引物的长度一般为18—30 bp,考虑到引物太短会提高错配率,导致引物缺乏特异性,而引物过长会导致其延伸温度过高,进而影响聚合酶的工作效率等原因,引物设计选择默认值20 bp。引物序列的GC含量选择为40%—60%,过高或过低都会影响PCR反应的发生。此外,为避免产生二聚体及发夹结构,两引物之间不能存在互补结构,否则会降低引物的有效浓度。根据以上过程及原则,从16个同源性较低的2 kb的片段中,选择较好的20对引物进行PCR验证,建立快速检测方法。
3.3 特异PCR方法的建立
虽然特异PCR技术鉴别大豆根瘤菌菌株的效果明显,但在建立PCR技术时,只是尽可能多的对与其相关的菌株进行特异性验证。为提高特异PCR的准确性,特异性验证时,应尽量多地选取近缘菌种作为参比菌株。本研究中,采用软件OrthoANIu(https://www. ezbiocloud.net/tools/ani)进行ANI计算发现,大豆根瘤菌5873的全基因组序列与模式菌株大豆根瘤菌USDA 6T的ANI值为99.95%,上述两株菌亲缘关系很近[33]。引物4-4基本可以区分大部分的菌株,但遇到与其全基因组极其相近的菌株,还需通过全基因组序列比对等手段,找到其差异并将其区分。如通过上述两株菌的全基因组比较发现了51 bp特异性缺失序列,通过这段序列,设计了一段引物,以区分5873与USDA 6T。后续的验证中,使用引物Q1扩增分别得到长度为218和269 bp的扩增条带,正是缺失片段所致。此外,通过灵敏度试验,确定所建立特异PCR反应的灵敏度为1 850 CFU/μL,与其他学者[31-32]特异PCR的检测灵敏度相近。本试验建立的PCR快速检测体系具有较高的敏感性,在较低浓度DNA或者一定浓度的菌体发酵液或根瘤破碎提取液为模板的体系中,可以方便、快捷检测大豆根瘤菌5873。
3.4 基于特异PCR技术评价菌株竞争结瘤能力
在应用层面,接种的根瘤菌剂能否在田间得到推广受诸多因素的影响。其中,与根瘤菌-宿主匹配性及根瘤菌在不同理化性质的土壤适应性相比,根瘤菌的竞争结瘤能力是限制其应用的关键因素[34-35]。通常采用占瘤率来评价根瘤菌和宿主的匹配性以及菌株竞争结瘤能力。然而,在测定根瘤菌的占瘤率时,重要的前提是一株根瘤菌侵染形成一个根瘤。在正常情况下,一个根瘤由一个细胞侵染形成所致,由两个菌株侵染形成的根瘤(即所谓的“双侵染根瘤”)概率不到千分之一。此外,很多文献报道在计算根瘤菌占瘤率时,也以一株根瘤菌侵染一个根瘤为前提。传统方法常借助BOX-PCR指纹图谱研究接种根瘤菌的占瘤率[24,36],但需要从根瘤中分离、纯化根瘤菌,进而进行DNA提取、PCR扩增等繁琐的环节,其过程费时费力。本研究建立的特异PCR鉴定技术,大大减少了传统技术中的繁琐环节,实现了对大豆根瘤菌5873的竞争结瘤能力快速评价,且与传统BOX-PCR检测结果一致。
4 结论
建立了大豆根瘤菌5873菌株的快速检测方法,利用建立的特异PCR检测方法可快速、准确评价大豆根瘤菌5873竞争结瘤能力,与传统BOX-PCR评价结果一致。该方法省去了根瘤的分离、根瘤菌分离纯化、DNA提取及测序鉴定等繁琐的环节,大大提高了工作效率,可为微生物肥料中根瘤菌剂的产品质量检测、菌种鉴定和功能评价提供参考。
[1] 张启发. 资源节约型、环境友好型农业生产体系的理论与实践. 北京: 科学出版社, 2015.
ZHANG Q F. Theory and practice of resource-conservation and environment-friendly agricultural production system. Beijing: Science Press, 2015. (in Chinese)
[2] 李俊, 姜昕, 黄为一, 马鸣超. 微生物肥料生产应用技术百问百答. 北京: 中国农业出版社, 2019: 18-19.
LI J, JIANG X, HUANG W Y, MA M C. Q & A Center for the application technology of biofertilizer. Beijing: China Agriculture Press, 2019: 18-19. (in Chinese)
[3] ZHU J, JIANG X, GUAN D W, KANG Y W, LI L, CAO F M, ZHAO B S, MA M C, ZHAO J, LI J. Effects of rehydration on physiological and transcriptional responses of a water-stressed rhizobium. Journal of Microbiology, 2022, 60: 31-46.
[4] MUS F, CROOK M B, GARCIA K, COSTAS A G, GEDDES B A, KOURI E D, PARAMASIVAN P, RYU M H, OLDROYD G E D, POOLE P S, UDVARDI M K, VOIGT C A, ANE J M, PETERS J W. Symbiotic nitrogen fixation and the challenges to its extension to nonlegumes. Applied and Environmental Microbiology, 2016, 82(13): 3698-3710.
[5] Ormeno-ORRILLO E, Martínez-ROMERO E. Phenotypic tests inspecies description: An opinion and (a sympatric speciation) hypothesis. Systematic and Applied Microbiology, 2013, 36(3): 145-147.
[6] 陈文峰. 根瘤菌系统学研究进展与展望. 微生物学通报, 2016, 43(5): 1095-1100.
CHEN W F. Progress and perspective of systematics of rhizobia. Microbiology China, 2016, 43(5): 1095-1100. (in Chinese)
[7] KALITA M, MALEK W. The ftsA gene as a molecular marker for phylogenetic studies inand identification of. Journal of Applied Genetics, 2019, 60(1): 123-126.
[8] STEPKOWSKI T, ZAK M, MOULIN L, KROLICZAK J, GOLINSKA B, NAROZNA D, SAFRONOVA V I, MADRZAK C J.andare the two dominant rhizobium species in root nodules of lupin and serradella plants growing in Europe. Systematic and Applied Microbiology, 2011, 34(5): 368-375.
[9] VINUESA P, Leon-BARRIOS M, SILVA C, WILLEMS A, Jarabo-LORENZO A, Perez-GALDONA R, WERNER D, Martinez-ROMERO E.sp. nov., an acid-tolerant endosymbiont that nodulates endemic genistoid legumes (Papilionoideae: Genisteae) from the Canary Islands, along withbv.,genospecies alpha andgenospecies beta. International Journal of Systematic and Evolutionary Microbiology, 2005, 55(2): 569-575.
[10] RIVAS R, MARTENS M, LAJUDIE P, WILLEMS A. Multilocus sequence analysis of the genus. Systematic and Applied Microbiology, 2009, 32(2): 101-110.
[11] MENNA P, BARCELLOS F G, HUNGRIA M. Phylogeny and taxonomy of a diverse collection ofstrains based on multilocus sequence analysis of the 16S rRNA gene, ITS region and glnII, recA, atpD and dnaK genes. International Journal of Systematic and Evolutionary Microbiology, 2009, 59(12): 2934-2950.
[12] REHMAN N, ABBAS F, IMRAN M, ALAM I, IMRAN M, ULLAH I, RIAZ M, KHAN F U. Genome wide analysis of DWARF27 genes in soybean and functional characterization of GmD27c reveals eminent role of strigolactones in rhizobia interaction and nodulation in. Molecular Biology Reports, 2022, 49(6): 5405-5417.
[13] JI Z J, LIU T Y, ZHANG J X, YAN H, WANG E T, CUI Q G, CHEN W X, CHEN W F. Genetic divergence amongstrains nodulating wild and cultivatedspp. in China. Systematic and Applied Microbiology, 2019, 42: 223-231.
[14] TORRES D, REVALE S, OBANDO M, MARONICHE G, PARIS G, PERTICARI A, VAZQUEZ M, WISNIEWSKI-DYE F, MARTINEZ- ABARCA F, CASSAN F. Genome sequence ofE109, one of the most agronomically used nitrogen-fixing rhizobacteria in argentina. Genome Announcements, 2015, 3(1): e01566-14.
[15] SIQUEIRA A F, ORMENO-ORRILLO E, SOUZA R C, RODRIGUES E P, ALMEIDA L G P, BARCELLOS F G, BATISTA J S S, NAKATANI A S, MARTINEZ-ROMERO E, VASCONCELOS A T R, Hungria M. Comparative genomics ofCPAC 15 andCPAC 7: elite model strains for understanding symbiotic performance with soybean. BMC Genomics, 2014, 15: 420.
[16] AZEVEDO H, LOPES F M, SILLA P R, HUNGRIA M. A database for the taxonomic and phylogenetic identification of the genususing multilocus sequence analysis. BMC Genomics, 2015, 16(Suppl. 5): 10.
[17] KALITA M, MALEK W. Root nodules ofharborandbacteria exchanging nodC and nodZ genes. Systematic and Applied Microbiology, 2020, 43: 126026.
[18] KALITA M, MALEK W. Molecular phylogeny ofbacteria isolated from root nodules of tribe genisteae plants growing in southeast Poland. Systematic and applied microbiology, 2017, 40(8): 482-491.
[19] VOS M, QUINCE C, PIJL A S, HOLLANDER M, KOWALCHUK G A. A comparison of rpoB and 16S rRNA as markers in pyrosequencing studies of bacterial diversity. PloS One, 2012, 7(2): e30600.
[20] KANEKO T, NAKAMURA Y, SATO S, MINAMISAWA K, UCHIUMI T, SASAMOTO S, WATANABE A, IDESAWA K, IRIGUCHI M, KAWASHIMA K,. Complete genomic sequence of nitrogen-fixing symbiotic bacteriumUSDA110. DNA Research, 2002, 9(6): 189-197.
[21] DELAMUTA J R, RIBEIRO R A, ORMENO-ORRILLO E, MELO I S, MARTINEZ-ROMERO E, HUNGRIA M. Polyphasic evidence supporting the reclassification ofgroup Ia strains assp. nov. International Journal of Systematic and Evolutionary Microbiology, 2013, 63(9): 3342-3351.
[22] ZHU S Z, GONG C Y, SONG D W, GAO S H, ZHENG G J. Discovery of a novel (+)--lactamase fromUSDA 6 by rational genome mining. Applied and Environmental Microbiology, 2012, 78(20): 7492-7495.
[23] NAROZNA D, PUDELKO K, KROLICZAK J, GOLINSKA B, SUGAWARA M, MADRZAK C, SADOWSKY M. Survival and competitiveness ofstrains 20 years after introduction into field locations in Poland. Applied and Environmental Microbiology, 2015, 81(16): 5552-5559.
[24] ITURRALDE E T, COVELLI J M, ALVAREZ F, PEREZ-GIMENEZ J, ARRESE-IGOR C, LODEIRO A R. Soybean-nodulating strains with low intrinsic competitiveness for nodulation, good symbiotic performance, and stress-tolerance isolated from soybean-cropped soils in Argentina. Frontiers in Microbiology, 2019, 10: 1061.
[25] XING P F, ZHAO Y B, GUAN D W, LI L, ZHAO B S, MA M C, JIANG X, TIAN C F, CAO F M, LI J. Effects ofco-inoculated withandon the structure and functional genes of soybean rhizobacteria community. Genes, 2022, 13: 1922.
[26] 全紫曼, 陈远学, 刘明, 彭丹, 蒋帆, 周元, 邹兰, 徐开未. 成都平原蚕豆高效根瘤菌的筛选及其促生功能初步评价. 植物营养与肥料学报, 2019, 25(6): 943-952.
QUAN Z M, CHEN Y X, LIU M, PENG D, JIANG F, ZHOU Y, ZOU L, XU K W. Screening of high efficiency symbiotic rhizobia to faba bean and a preliminary investigation on their promotion ability in Chengdu Plain. Journal of Plant Nutrition and Fertilizers, 2019, 25(6): 943-952. (in Chinese)
[27] WANG E T, CHEN W F, TIAN C F, YOUNG J P W, CHEN W X. Working on the taxonomy, biodiversity, ecology and evolution of rhizobia//Ecology and Evolution of rhizobia. Singapore: Springer, 2019: 251-273.
[28] ZOU Y P, MASON M G, WANG Y L, WEE E, TURNI C, BLACKALL P J, TRAU M, BOTELLA J R. Nucleic acid purification from plants, animals and microbes in under 30 seconds. PLoS Biology, 2017, 15(11): e2003916.
[29] 曹凤明, 沈德龙, 李俊, 关大伟, 姜昕, 李力, 冯瑞华, 杨小红, 陈慧君, 葛一凡. 应用多重PCR鉴定微生物肥料常用芽孢杆菌. 微生物学报, 2008, 48(5): 651-656.
CAO F M, SHEN D L, LI J, GUAN D W, JIANG X, LI L, FENG R H, YANG X H, CHEN H J, GE Y F. Multiplex-PCR approach to identifyspecies applied in microbial fertilizers. Acta Microbiologica Sinica, 2008, 48(5): 651-656. (in Chinese)
[30] SHAMS M, VIAL L, CHAPULLIOT D, NESME X, LAVIRE C. Rapid and accurate species and genomic species identification and exhaustive population diversity assessment ofspp. using recA-based PCR. Systematic and Applied Microbiology, 2013, 36(5): 351-358.
[31] WU J G, WANG J F, ZHANG X H, ZHANG S S, HU X F, CHEN J S. A gyrB-targeted PCR for rapid identification of. Applied Microbiology and Biotechnology, 2010, 87(2): 739-747.
[32] 王璇, 马鸣超, 关大伟, 姜昕, 李力, 丁延芹, 李俊. 胶质类芽胞杆菌PCR快速检测方法. 微生物学报, 2011, 51(11): 1485-1493.
WANG X, MA M C, GUAN D W, JIANG X, LI L, DING Y Q, LI J. Rapid identification forby PCR. Acta Microbiologica Sinica, 2011, 51(11): 1485-1493. (in Chinese)
[33] 王鹏辉, 姜昕, 马鸣超, 关大伟, 曹凤明, 刘尧, 康耀卫, 李俊. 一株耐干燥大豆根瘤菌菌株的筛选与固氮效果评价. 大豆科学, 2020, 39(1): 90-96.
WANG P H, JIANG X, MA M C, GUAN D W, CAO F M, LIU Y, KANG Y W, LI J. Screening of drought-tolerant soybeanand its symbiotic compatibility verification. Soybean Science, 2020, 39(1): 90-96. (in Chinese)
[34] LIU Y, JIANG X, GUAN D W, ZHOU W, MA M C, ZHAO B S, CAO F M, LI L, LI J. Transcriptional analysis of genes involved in competitive nodulation inat the presence of soybean root exudates. Scientific Reports, 2017, 7(1): 10946.
[35] AMBROSINI V G, FONTOURA S M V, MORAES R P, TAMAGNO S, CIAMPITTI I A, BAYER C. Soybean yield response tostrains in fields with inoculation history in Southern Brazil. Journal of Plant Nutrition, 2019, 42(16): 1941-1951.
[36] 陈文新, 汪恩涛. 中国根瘤菌. 北京: 科学出版社, 2011: 114-115.
CHEN W X, WANG E T. Chinese rhizobia. Beijing: Science Press, 2011: 114-115. (in Chinese)
Rapid identification for5873 by PCR and its evaluation in application
MA MingChao, JIANG Xin, WANG PengHui, GUAN DaWei, LI Jun
Institute of Agricultural Resources and Agricultural Regional Planning, Chinese Academy of Agricultural Sciences, Beijing 100081
【Objective】is one of the important functional bacteria in microbial fertilizer products. As a commercialized strain,5873 was wildly used in agricultural production and played important roles in symbiotic nitrogen fixation. Therefore, it isnecessary to establish a rapid identification method at the strain level by screening and identifying specific primers for rhizobia, and is of great importance in the aspects of microbial fertilizer product quality inspection, strain identification and function evaluation.【Method】According to the whole genome sequencing of5873 and other strain related sequences from NCBI, as well as the differential fragments ofUSDA 6Twhich is highly homologous to5873 (the ANI value of the genome is greater than 99.95%), specific PCR primers were designed and obtained. After optimization of PCR reaction conditions and the detection of sensitivity and specificity, a rapid detection method for5873 was established. Then, using pot experiment,5873 was mixed with other rhizobium strains and inoculated in the soybean rhizosphere, and the above method was used to evaluate the competitive nodulation ability of5873.【Result】A set of species-specific primers 4-4 and Q1 (4-4-F 5′-GATAAGGCCACGGGTGAACA-3′/4-4-R 5′-CACTCGATAAGCTCCGCTGT-3′ and Q1-F 5′-CCGGTCGTGACTGGAATGAT-3′/Q1-R 5′-TCGAGGCCTACAAGAACGTC -3′), were successfully designed to selectively amplify 355 and 218 bp amplicon from5873. Well specificity was demonstrated by reference strains, from which only the targeted bands of5873 were observed. Amplifications were performed in a 25 μl reaction mixture, which contained Premix TaqTM12.5 μL, template DNA (15 ng of genomic DNA), primers 4-4 and Q1 (1.0 μL, respectively). The PCR cycling consisted of one cycle of 5 min at 95 ℃and 30 cycles of 45 s at 94 ℃, 45 s at 61 ℃, and 60 s at 72 ℃. A final extension step was run for 10 min at 72 ℃. The sensitivity was 1 850 CFU/µL. In addition, the method mentioned above was successfully used to evaluate the competitive nodulation ability of5873, which was consistent with the results of traditional BOX-PCR identification.【Conclusion】The established rapid detection method can directly use the bacterial solution or squeezed nodules as templates, which will eliminate laborious tasks, such as processes of nodule isolation, purification, culture, DNA extraction, sequencing and sequence comparison. It only takes a few hours to accurately detect5873, which provides a reference for product quality detection and competitive nodulation ability evaluation of microbial fertilizers.
; strain-specific primer pair; PCR amplification; competitive nodulation

10.3864/j.issn.0578-1752.2023.14.010
2022-11-13;
2022-12-21
国家重点研发计划(2021YFD1700200)、云南省重大科技专项计划(202202AE090025)、国家现代农业产业技术体系建设专项(CARS-04)
马鸣超,E-mail:mamingchao@caas.cn。通信作者姜昕,E-mail:jiangxin@caas.cn
(责任编辑 岳梅)
