稻瘟病菌类SRRM1转录因子的功能分析
2023-08-07邱凯华方淑梅梁喜龙
邱凯华 方淑梅 梁喜龙
(黑龙江八一农垦大学,163319,黑龙江大庆)
真核生物的结构基因为断裂基因,其转录生成的mRNA 前体(pre-mRNA)必须经过5'端加帽、剪接(去除内含子,连接外显子)、3'端加polyA 尾这一系列高度整合和偶联的步骤形成成熟的mRNA,随后从细胞核穿过核孔,运送至细胞质进行下一步反应[1-4]。pre-mRNA 剪接是由一个大复合物——剪接体来执行,剪接体由U1、U2、U4、U5和U6 5 种核小核糖核蛋白( small nuclear ribonucleoproteins,snRNP)和大量非核小核糖核蛋白(non-snRNP)组成[5-6],首先,U1snRNP 识别内含子的5'剪接位点并与其结合,SF1(splicing factor 1)与分支点结合,从而促进U2AF 因子在3'剪接位点的结合,形成复合体E;随后U2snRNP 取代SF1参与剪接,产生复合体A;接着预成型的U4/U6-U5三聚体加入复合体A,形成复合体B;再经过大量蛋白的交换和募集最终转化为具有催化活力的复合体C,至此剪接体形成[7]。pre-mRNA 剪接过程涉及2 个连续的转酯反应,首先,分支点腺苷的2'-OH基团对内含子5'剪接位点的磷酸二酯键进行亲核攻击,产生裂解的上游外显子,第2 步,由上游外显子的3'-OH 基团攻击3'剪接位点的磷酸二酯键,使外显子连接,内含子形成套索结构释放[5,8]。
SR(Serine/Arginine)家族及其相关蛋白是众多非snRNP 剪接因子中表征最广泛的一类,在剪接位点的选择和剪接的其他过程中发挥重要作用[9-12]。SRRM1(Serine/arginine repetitive matrix protein 1)/SRm160(the SR-related nuclear matrix protein of 160kDa)是其中的一个成员,是一种剪接辅激活因子,通过结合5'剪接位点、分支点和3'剪接位点的剪接因子发挥作用[13],其N 端含有一个高度保守的PWI(Pro-Trp-Ile)结构域。PWI 结构域是一类新型的RNA 结合域,其可促进剪接过程中pre-mRNA的3'末端切割,有利于mRNA 向细胞质释放[13-15]。研究[16-21]发现,SRm160 在转录后仍与mRNA 结合,作为EJC(exon junction complex)的组成成分,通过NMD(nonsense-mediated mRNA decay)途径参与mRNA 从细胞核到细胞质的输出和转运。
研究[22]发现,SRRM1/SRm160 在调节生物生长发育和行为中发挥重要作用。对果蝇的研究[23]显示,SRm160 可以通过可变剪接影响果蝇的节律行为;在秀丽隐杆线虫中SRm160 和CstF-50 基因同步缺失会导致晚期胚胎发育停滞。本研究以稻瘟菌类SRRM1 蛋白为研究对象,首先对其进行生物信息学分析,进一步采用以基因重组原理为基础的基因定点敲除技术对该基因功能进行研究,为稻瘟病菌中转录因子调控机制的研究提供参考。
1 材料与方法
1.1 试验材料
稻瘟病菌菌株Y34、遗传转化所用土壤农杆菌AGL-1、敲除载体pXEH20 为本实验室保存;真菌DNA 提取试剂盒为OMEGA 公司产品;细菌质粒提取试剂盒、DNA 凝胶回收试剂盒为AXYGEN 公司产品;基因克隆所用试剂与大肠杆菌(DH-5α)感受态细胞购自TaKaRa 公司;基因克隆载体pMD18-T 与引物购于生工生物工程(上海)股份有限公司,具体引物信息如表1 所示;其他药品和试剂均为分析纯。

表1 敲除载体构建及突变体验证所用引物信息Table 1 Primer information for knockout vector construction and mutant verification
1.2 试验方法
1.2.1 生物信息学分析 在NCBI 数据库(https://www.ncbi.nlm.nih.gov/gene)搜索“SRRM1”,获取人的SRRM1 蛋白序列,再通过NCBI Blast(https://blast.ncbi.nlm.nih.gov/),物种选择为“Pyricularia oryzae”,获取稻瘟病菌中的类SRRM1 蛋白序列。利用ProtParam tool(https://web.expasy.org/protparam/)预测理化性质;利用Prot Comp 9.0(http://linux1.Softberry.com/berry.phtml?topic=protcompan&group=programs&subgroup=prol oc)预测蛋白质亚细胞定位;利用SignalP-5.0(https://services.healthtech.dtu.dk/service.php?Signa lP-5.0)预测信号肽;利用TMHMM-2.0(https://services.healthtech.dtu.dk/service.php?TMHMM-2.0)预测跨膜螺旋;利用SOPMA(https://npsa-prabi.ibcp.fr/cgi-bin/npsa_automat.pl?page=/NPSA/npsa_sopm a.html)预测二级结构;利用NCBI Conserved Domain Search ( https://www.ncbi.nlm.nih.gov/Structure/cdd/wrpsb.cgi)预测结构域,通过Dog 2.0画图;在SWISS-MODEL(https://swissmodel.expasy.org/interactive)获取三级结构。通过NCBI 数据库(https://blast.ncbi.nlm.nih.gov/Blast.cgi)同源比对得到人(Homo sapiens)、小鼠(Mus musculus)、拟南芥(Arabidopsis thaliana)、棉花(Gossypium hirsutum)、大豆(Glycine max)、稻瘟病菌(Pyricularia oryzae) 、白僵菌(Beauveria bassiana)、炭疽病菌(Colletotrichum truncatum)、白粉病菌(Erysiphe necator)、白纹羽病菌(Rosellinia necatrix)中同源蛋白的氨基酸序列,利用MEGA 7.0.26 软件中的Neighbor-Joining 法绘制系统进化树,参数默认,生成系统进化树。
1.2.2 敲除载体的构建及土壤农杆菌转化 (1)扩增M-0 左、右臂:以野生型稻瘟病菌菌株Y34 基因组DNA 为模板,PCR 扩增基因左、右臂,电泳检测。然后分别将左、右臂与pMD18-T 载体连接,转化大肠杆菌DH-5α感受态细胞,进行测序,确认无误后保存菌液。(2)敲除载体连接:将敲除载体pXEH20 与连接到T 载体的基因左、右臂菌液进行活化,提取质粒。利用相应内切酶先后将左、右臂连接于pXEH20 载体,形成重组的敲除载体。将敲除载体转化到大肠杆菌DH-5α感受态细胞,进行菌液PCR 验证,能同时扩增基因左、右臂,证明成功转入,然后提取质粒,转化到土壤农杆菌感受态AGL-1 中,菌液保存备用。
1.2.3 稻瘟病菌的遗传转化(土壤农杆菌介导法)(1)稻瘟病菌分生孢子的准备:将稻瘟病菌接种于燕麦片培养基(含浓度为100μg/mL 的链霉素),28℃黑暗培养7d,去除培养基表面的气生菌丝,盖一层纱布,连续光照培养3d,使之产生孢子。取2mL IM 液体培养基加入平板,洗出分生孢子,通过3 层擦镜纸折成的无菌漏斗过滤,显微镜调整孢子浓度至1×106个/mL。(2)土壤农杆菌AGL-1的活化与诱导:在LB 培养基加50μg/mL 利福平和50μg/mL 卡那霉素划线培养含有敲除载体的土壤农杆菌AGL-1 约2d,用接种环刮取适量菌体于10mL IM 液体培养基,用移液器反复吸吹至菌体充分悬浮,而后28℃恒温、180 转/min 黑暗诱导培养6h。(3)分生孢子与土壤农杆菌AGL-1 的共培养:将诱导的土壤农杆菌菌液和孢悬液1:1 混匀,加入终浓度为500μmol/L 的AS 试剂。将上述混合液涂抹于铺有玻璃纸的IM 固体培养基,每个平板200μL,28℃恒温黑暗培养48h。(4)转化子的筛选与纯化:取下玻璃纸反向贴于含400μg/mL 头孢霉素、200μg/mL 羧苄青霉素、200μg/mL 潮霉素的PDA 筛选培养基,同样条件下继续培养48h 后取下玻璃纸,将可扩展菌落接于新的潮霉素筛选平板,连续筛选3 代,用滤纸片保存。
1.2.4 敲除突变体的获得与鉴定 活化初筛转化子,用CM 液体培养基摇培约3d 后取菌丝,微波炉法提取转化子的基因组DNA,以其为模板,用特异性引物PCR 扩增Hyg 抗性基因片段,确认敲除载体是否转入,再扩增敲除掉的基因片段,初步确认转化子是否为敲除转化子。进一步以上述转化子基因组DNA 为模板,在敲除基因的左臂上游序列设计正向引物,在转入的Hyg 抗性基因序列设计反向引物,同理,在Hyg 抗性基因序列设计正向引物,在敲除基因的右臂下游设计反向引物,进行PCR 扩增反应,若均可扩增相应片段,确认转化子为敲除突变体。
1.2.5 生物学特性分析 在CM 培养基和PDA 培养基进行营养生长分析;细胞壁的完整性分析,在终浓度为200μg/mL 刚果红(Congo-Red)和0.01%十二烷基硫酸钠(SDS)的CM 固体培养基进行;渗透逆境,在分别含有1mol/L 山梨醇和NaCl 的CM固体培养基进行;杀菌剂逆境,在含有0.0074μg/mL吡唑醚菌酯的CM 固体培养基进行。上述试验条件均为28℃恒温培养7d,十字交叉法测量菌落直径并拍照记录。
1.3 数据处理
利用Microsoft Excel 统计数据与绘图,采用SPSS 22.0 软件进行ANOVA 单因素方差分析。
2 结果与分析
2.1 生物信息学分析
类SRRM1 蛋白ID 为XP_003713560,对应基因名为MGG_04513,含有氨基酸402 个,分子量为43 408.33,等电点为10.48,是一种碱性亲水蛋白,该蛋白不含信号肽,为非分泌蛋白,无跨膜螺旋,亚细胞定位存在于细胞核。如图1a 所示,二级结构以α-螺旋为主,占比31.09%。如图1b 所示,该蛋白仅在5'端含有一个高度保守的结构域,即PWI 结构域。如图1c 所示,三级结构预测模板为1mp1.1.A,序列与模板一致度为49.02%,GMQE值为0.12,QMEAN 值为-4.08,数值均在可信范围内。如图2 所示,进化关系中,棉花、大豆和拟南芥构成一个分支,人和小鼠构成一个分支,真菌中炭疽病菌和白纹羽病菌亲缘关系最近,二者与白粉菌构成一个分支,而白僵菌与动植物构成一个大的分支,稻瘟病菌则独立构成一个分支,由此我们推测,SRm160 同源物在动物和植物中进化较保守,而在真菌中进化差异较大。Motif 1、2、4 为10 个物种共有基序,PWI 为共有结构域,且均在N 端。

图1 类SRRM1 蛋白的空间结构Fig.1 The spatial structure of SRRM1-like protein
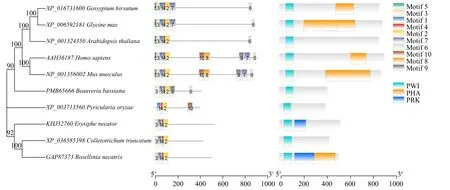
图2 类SRRM1 蛋白的进化关系与结构分析Fig.2 Evolutionary relationship and structural analysis of SRRM1-like protein
2.2 敲除载体的构建与验证
以Y34 基因组DNA 为模板,利用特异性引物扩增出MGG_04513(以下简称M-0)基因的左、右臂(M-0-L、M-0-R),大小分别为1152 和1305bp,电泳检测结果见图3,可见条带正确。然后分别将M-0-L 和M-0-R 与pMD18-T 载体连接,转化到大肠杆菌进行测序,确认后保存。
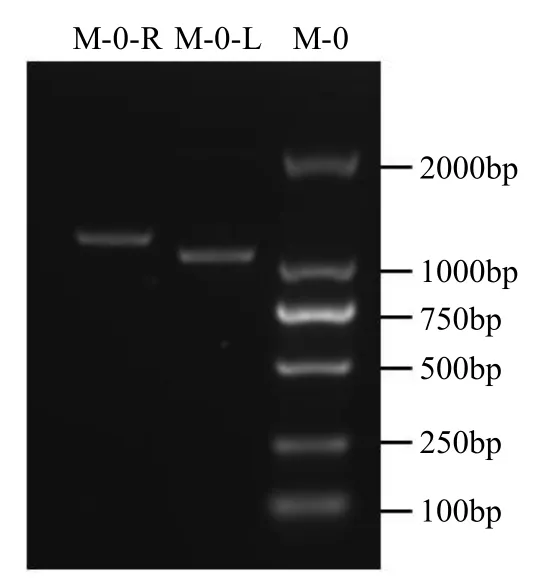
图3 M-0 基因左、右臂的PCR 扩增Fig.3 PCR amplification of left and right arms of M-0 gene
取出pXEH20,连接于T 载体的M-0-L 和M-0-R,进行菌株活化,提取质粒,利用内切酶XhoI 和EcoR I 分别对连接于T 载体的M-0-L 与pXEH20 载体双酶切,酶切产物M-0-L 与pXEH20胶回收纯化,再用T4DNA 连接酶将二者连接转化为pXEH20-L,简写为P-L,再利用内切酶BamH I和Hind III 分别对连接于T 载体的M-0-R 与P-L重组载体进行双酶切,酶切产物M-0-R 与P-L 胶回收纯化,然后用T4DNA 连接酶将其连接为重组载体pXEH20-LR,简写为P-LR。对pXEH20、P-L、P-LR 质粒进行电泳检测,结果见图4,条带大小均正确。

图4 pXEH20、P-L、P-LR 质粒电泳图Fig.4 Electropherogram of pXEH20,P-L,P-LR plasmid
构建敲除载体pXEH20-LR 的示意图如图5a 所示,将重组载体pXEH20-LR 转化到大肠杆菌DH-5α感受态细胞,菌液PCR 验证(扩增M-0-L与M-0-R 片段),结果见图5b,条带正确。然后提取大肠杆菌中重组敲除载体pXEH20-LR 质粒转化到土壤农杆菌感受态AGL-1 中,菌液-80℃保存备用。

图5 M-0 基因敲除载体的构建示意图及验证Fig.5 Schematic diagram and verification of construction of knockout vector of M-0 gene
2.3 敲除突变体的获得与鉴定
以遗传转化过程中筛选出的转化子基因组DNA 为模板,扩增Hyg 抗性基因(753bp)与敲除片段基因(653bp),结果见图6,初步确认转化子为敲除转化子。
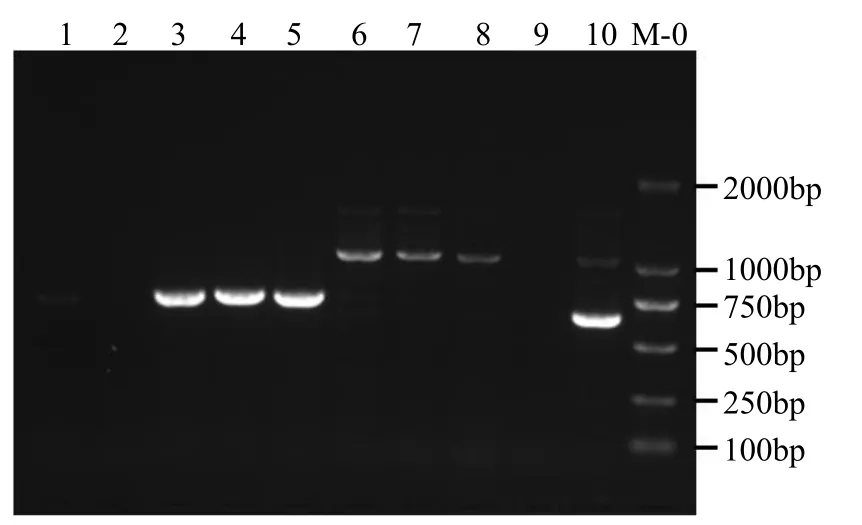
图6 M-0 转化子目的基因验证电泳图Fig.6 Electropherogram of the verified M-0 transformant target genes
再利用特异性引物M-0-L-H-S、M-0-L-H-A与M-0-H-R-S、M-0-H-R-A 进行扩增,电泳检测结果见图7,皆能成功扩增相应片段,说明敲除载体上的同源基因与稻瘟病菌基因组中的基因正确交换,转化子为敲除突变体。

图7 M-0 转化子侧翼序列验证电泳图Fig.7 Electropherogram of the verified M-0 transformant flank sequences
2.4 生物学特性分析
营养生长分析在CM 和PDA 培养基进行,结果见图8,野生型菌株Y34 及突变体M9、M11、M17 的菌落生长速率相近,无明显差异。为探究其是否参与稻瘟病菌对外界胁迫的应答,将野生型菌株和敲除突变体菌株分别接种于含有Congo-Red、SDS、山梨醇、NaCl 以及吡唑醚菌酯的CM固体培养基,结果显示,在几种逆境下敲除菌株与野生型菌株相比受到不同程度的抑制,均达到显著差异水平,其中,Congo-Red 条件下敲除菌株直径减小最少,敲除菌株M9、M11 和M17 相比野生型菌株Y34 的菌落直径分别减小5.37%、5.38%和6.80%;山梨醇条件下敲除菌株M9、M11和M17 相比野生型菌株Y34 的菌落直径分别减小7.39%、8.36%和8.04%;SDS 条件下敲除菌株M9、M11 和M17 相比野生型菌株Y34 的菌落直径分别减小11.56%、9.25%和10.98%;NaCl 条件下敲除菌株受影响最大,敲除菌株M9、M11 和M17 相比野生型菌株Y34 的菌落直径分别减小13.27%、11.85%和11.37%。另外,在吡唑醚菌脂杀菌剂逆境下敲除突变体菌株直径分别减小了9.83%、9.25%和12.14%,也表现出较强的敏感性。

图8 基因敲除对稻瘟病菌营养生长及逆境适应的影响Fig.8 Effects of gene knockout on the vegetative growth and stress adaptation of Magnaporthe grisea
3 讨论
SR 蛋白在N 端含有1 或2 个RRM(RNA 识别基序),使其具有RNA 结合特性,在C 端有一个RS(富含精氨酸和丝氨酸)结构域,用于促进蛋白质-蛋白质相互作用[24-25]。因为SRm160 缺少RRM 基序,但在功能上满足SR 蛋白的定义,它可以通过PWI 结构域直接结合核酸,所以它不是典型的SR 家族成员,而被定义为SR 相关蛋白[15,26]。类SRRM1 是哺乳动物剪接因子SRRM1 在稻瘟病菌中的同源物,本研究通过生物信息学分析与其他物种中同源物的进化关系,白僵菌与动植物在一个分支,稻瘟病菌独立构成一个分支,其他真菌在一个分支,与其他9 个物种共有基序为Motif 1、2、4,缺少其他物种都具有的Motif 3,比真菌物种多一个Motif 10,仅在N 端含有一个PWI 结构域,由此我们认为该基因在真菌中进化差异较大。
Beckwith 等[22]研究表明,SRm160 通过剪接调控果蝇大脑起搏器神经元的基因表达来确保分子钟的适当振荡,进而影响果蝇的节律行为;在秀丽隐杆线虫中,McCracken 等[23]通过RNA 干扰技术研究发现,SRm160 和卵裂因子CstF-50 存在特定的相互作用,2 种基因共同缺失会导致线虫的晚期胚胎发育停滞;其后McCracken 等[27]又发现,SRm160 与染色质调节和姐妹染色单体结合有关;Cheng 等[28]发现,SRm160 通过与Sam68 相互作用来调节人体内源跨膜糖蛋白CD44 的选择性剪接,进而影响肿瘤细胞的侵袭。由此可见,SRm160 同源基因在不同生物体中发挥的作用以及重要程度不同。本研究发现,在稻瘟病菌中敲除类SRRM1不会致死,对于稻瘟病菌而言,CM 与PDA 培养基以及28℃都是最适的培养条件,此条件下野生型与敲除突变体菌落直径相近,无明显差异,由此推测在适当条件下,类SRRM1 对稻瘟病菌营养生长不是必需的;然而经过Congo-Red、SDS(两者均抑制细胞壁的形成)和渗透逆境(山梨醇、NaCl)以及杀菌剂吡唑醚菌酯处理后,敲除突变体的菌丝生长均受到明显影响,表现出抗逆性减弱。
本研究结果可以为了解稻瘟病菌中类SRRM1功能提供参考,我们将继续设计基因缺失与致病性相关的试验方案,进一步研究类SRRM1 与稻瘟病菌致病力的关系,为明确pre-mRNA 剪接与稻瘟病菌应对寄主逆境和致病性的深层关系提供基础,为稻瘟病有效防控提供新思路。
4 结论
生物信息学分析表明,稻瘟菌中类SRRM1 是存在于细胞核的碱性亲水蛋白,不含信号肽,无跨膜螺旋,在N 端含有一个PWI 结构域。类SRRM1基因缺失不影响稻瘟病菌的营养生长,但对Congo-Red、SDS、山梨醇、NaCl 及杀菌剂吡唑醚菌酯逆境胁迫敏感,表现为抗逆性减弱。因此推测,类SRRM1 与稻瘟病菌细胞壁形成有关,参与渗透逆境应答,也与稻瘟病菌应对杀菌剂吡唑醚菌酯的过程相关,类SRRM1 在稻瘟病菌逆境适应过程中发挥重要作用。
