基于代谢组学和转录组学筛选丙泊酚致神经毒性关键基因的研究
2023-08-01李慎风庄曌刁玉晶曹宏王寿世
李慎风 庄曌 刁玉晶 曹宏 王寿世
(青岛大学附属青岛市中心医院麻醉与围术期医学科,山东 青岛 266042)
丙泊酚因为麻醉起效快、患者苏醒迅速及副作用少而在临床上被广泛使用[1]。近年来,研究表明,长期或多次使用丙泊酚,特别是对发育期动物,具有诱发严重的神经毒性并影响其大脑发育的可能[2-3]。2016年12月美国食品和药物管理局发布警告称,孕晚期的孕妇以及3岁以下儿童长时间(超过3 h)或频繁使用全身麻醉药,可能导致胎儿或儿童脑发育异常,批准丙泊酚仅用于≥2个月儿童的麻醉维持和≥3岁儿童的麻醉诱导[4]。但目前关于丙泊酚致神经毒性的具体机制并不完全明确。本研究通过代谢组学和转录组学联合分析,筛选丙泊酚致神经毒性的关键基因,为后续丙泊酚致神经毒性的分子机制研究提供理论基础,也为丙泊酚的神经毒性预防用药和治疗用药的开发提供研究方向。
1 材料和方法
1.1 不同浓度丙泊酚对HT22细胞活性的影响
将小鼠海马神经细胞HT22细胞(上海雅吉生物科技有限公司)放置于含体积分数0.10的FBS和100 kU/L青霉素、100 mg/L链霉素的DMEM中, 37 ℃下于含体积分数0.05的CO2环境中进行培养。当细胞密度达80%~90%时接种于96孔板中,每孔约5 000个细胞。培养24 h细胞贴壁后,弃去原培养液,分别加入含有0、25、50、75、100 mg/L丙泊酚(Ctrl组、P25组、P50组、P75组、P100组)的DMEM。丙泊酚货号:KM7Q-3CMN。每个浓度设置3个复孔,同时再设置一组无细胞孔板作为空白组。将各组细胞继续培养24 h,换液为含有10%的CCK-8溶液培养基,37 ℃下孵育2.5 h,用酶标仪检测波长450 nm处各孔吸光度值,以吸光度值表示细胞活性。细胞活性=(实验组吸光度值-空白组吸光度值)/(对照组吸光度值-空白组吸光度值)×100%。
1.2 各组HT22细胞中代谢物和RNA的提取
将HT22细胞接种于直径10 cm细胞培养皿中,使用含有体积分数0.10的FBS和100 kU/L青霉素、100 mg/L链霉素的DMEM培养基,37 ℃下于含体积分数0.05 CO2的细胞培养箱中培养。当细胞密度达到60%~70%时,弃去原培养基,更换为含不同浓度丙泊酚(Ctrl组、P25组、P50组、P75组、P100组)的新鲜DMEM培养基。移入培养箱内继续培养24 h。实验重复3次,收集3次实验的HT22细胞样本,转移至1.5 mL EP管中,-80 ℃冰箱保存。委托广州基迪奥生物技术有限公司提取各组细胞样本中的代谢物和RNA,用于后续分析。
1.3 各组HT22细胞中代谢物的代谢组学分析
1.3.1液相色谱-质谱(LC-MS)分析 使用QTOF/MS-6550质谱仪(美国Aglient公司)、1290 Infinity LC超高效液相色谱仪(美国Aglient公司)对各组HT22细胞中提取出的代谢物进行检测。使用Proteo Wizard软件对LC-MS检测到的代谢物的种类和数量进行分析,结果以环形图表示。
1.3.2差异代谢物分析 使用R软件的limma包对各组HT22细胞代谢物进行差异代谢物分析,获得差异代谢物的种类和数量,绘制火山图。
1.3.3代谢物含量的趋势分析 使用OmicShare Tools (https://www.omicshare.com/tools)对各组HT22细胞代谢物含量进行趋势分析。获得同趋势代谢物,即各浓度组与Ctrl组相比,细胞代谢物均有上升趋势或均有下降趋势的代谢物。将差异代谢物和同趋势代谢物取交集,得到关键代谢物。
1.4 各组HT22细胞RNA的转录组学分析
1.4.1序列比对分析 对提取出的各组HT22细胞中的RNA构建cDNA文库,使用Illumina HiSeq Xten高通量测序仪上机测序。使用HISAT2软件以小鼠基因组作为参考系,进行测序序列比对分析,获得各组HT22细胞中的测序序列数量,并计算总映射(定位到基因组上全部的序列数占有效序列)及其百分比和多个映射(在参考序列上有多个比对位置的序列)及其百分比。
1.4.2差异基因分析 使用Stringtie重构转录本,对基因表达水平进行定量分析。基于各组HT22细胞中基因表达量,使用R软件的limma包分析获得各浓度组与Ctrl组的差异基因,并绘制火山图。
1.5 代谢组学和转录组学的联合分析
1.5.1加权基因共表达分析(WGCNA) 将小鼠基因组按照基因表达相似性分为29个模块,同一模块内的基因表达相似性接近。使用R软件的WGCNA包以小鼠基因组为参考系,以关键代谢物的二级分类为表型,使用Pearson相关分析探究小鼠基因组各模块和HT22细胞关键代谢物之间的相关性,以聚类热图形式表示。提取与关键代谢物有显著相关的小鼠基因组模块中全部基因,即相关基因。
1.5.2蛋白质-蛋白质相互作用网络(PPI)分析 使用R软件将上面WGCNA分析获得的相关基因与转录组学分析获得的差异基因取交集,得到差异相关基因。为了解基因之间的功能关系,将差异相关基因所编码的蛋白通过STRING数据库(https://string-db.org/)和Cytoscape软件(3.4.0版)进行PPI可视化分析,筛选出基因功能关系密切的关键基因。
1.6 统计学方法
采用GraphPad Prism 9.0软件进行数据分析,呈正态分布的计量资料两组简单比较采用t检验,多组间比较采用单因素方差分析,进一步两两比较采用Bonferroni方法,以P<0.05为有统计学意义。基因及代谢物表达量的计算采用FPKM法。采用R软件的limma包进行两组间代谢物含量和基因表达水平差异分析,将错误发现率P<0.05和差异倍数绝对值(|Fold Change|)≥1作为差异代谢物和差异基因的筛选标准。
2 结 果
2.1 各组HT22细胞活性比较
Ctrl组、P25组、P50组、P75组、P100组HT22细胞的活性分别达到(100.70±1.25)%、(81.36±1.43)%、(70.76±1.27)%、(30.37±0.72)%、(10.27±0.86)%,各组间比较差异有显著性(F=3 221.00,P<0.05),进一步行各组间HT22细胞活性两两比较,差异均具有显著性,并呈现剂量依赖性下降(t=11.40~97.25,P<0.05)。
2.2 各组HT22细胞中代谢物的代谢组学分析
2.2.1LC-MS分析 LC-MS分析结果显示,从各组HT22细胞代谢物中共检测到2 701种代谢物,其中属于二级分类的有620种,一级分类的有23种,占比最多的是苯及其取代衍生物,见图1。环形图中不同颜色代表不同代谢物一级分类,色块长度表示其所占比例;Class代表所检测到的代谢物的一级分类。
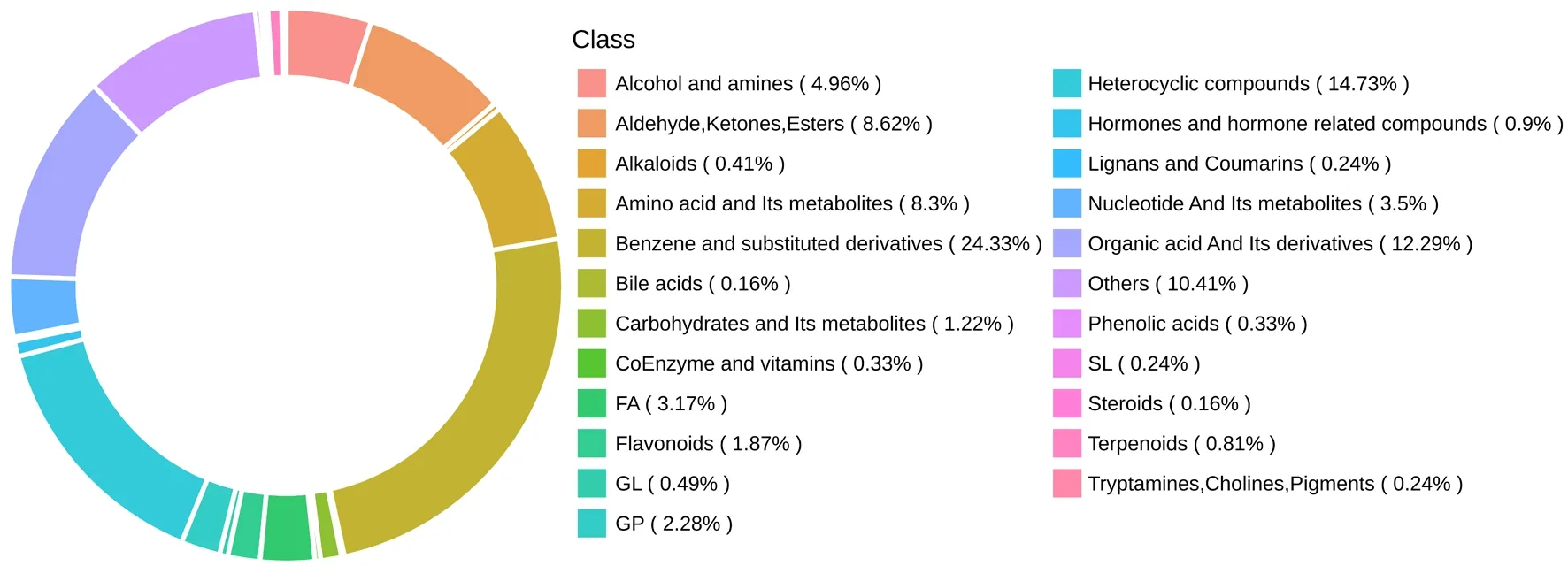
图1 代谢物种类和数量的环形图
2.2.2差异代谢物分析 Ctrl组与P25组间的差异代谢物共有9个,其中上调的2个,下调的7个(图2A);Ctrl组与P50组间的差异代谢物共有21个,其中上调的6个,下调的15个(图2B);Ctrl组与P75组间的差异代谢物共有27个,其中上调的6个,下调的21个(图2C);Ctrl组与P100组间的差异代谢物共有67个,其中上调的55个,下调的12个(图2D)。

A、B、C、D分别为Ctrl组与P25组、P50组、P75组、P100组的差异代谢物分析;绿色表示下调,灰色表示无统计学差异,红色表示上调
2.2.3趋势分析 各浓度组与Ctrl组相比,随丙泊酚浓度升高均持续上升的代谢物共有208种,没有发现随丙泊酚浓度升高均持续下降的代谢物。与上面获得的所有差异代谢物取交集,共得到49种关键代谢物。
2.3 各组HT22细胞中RNA的转录组学分析
2.3.1序列比对分析 RNA序列比对分析结果示,Ctrl组、P25组、P50组、P75组及P100组总有效序列数分别为46 308 970、42 145 574、39 362 782、45 367 077、46 692 981;总映射及其百分比分别为42 232 951(91.20%)、39 633 550(94.04%)、35 152 896(89.30%)、42 430 229(93.53%)、44 139 716(94.53%),多个映射及其百分比分别为3 537 096(7.64%)、2 459 613(5.84%)、3 369 032(8.56%)、2 685 366(5.92%)、2 271 619(4.86%)。各组总映射百分比>70%,多个映射百分比均<10%,说明各组HT22细胞的测序序列数量符合要求,能够满足数据分析需要。
2.3.2差异基因分析 Ctrl组与P25组的差异基因共有1 254个,其中上调的1 130个,下调的124个(图3A);Ctrl组与P50组的差异基因共有4 695个,其中上调的2 544个,下调的2 151个(图3B);Ctrl组与P75组的差异基因共有4 923个,其中上调的2 324个,下调的2 599个(图3C);Ctrl组与P100组间的差异基因共有5 031个,其中上调的2 704个,下调的2 327个(图3D)。

A、B、C、D分别为Ctrl组与P25、P50组、P75组、P100组的差异基因分析;红色表示上调,蓝色表示下调
2.4 代谢组学和转录组学联合分析
2.4.1小鼠基因组和HT22细胞关键代谢物的WGCNA分析 WGCNA分析结果显示,小鼠的基因组模块当中navajowhite2、darkolivegreen模块与HT22细胞关键代谢物呈正相关,floralwhite、white模块与HT22细胞关键代谢物呈负相关(图4)。上面4个小鼠基因组模块当中共包含有5 698个相关基因。

横坐标代表HT22细胞代谢物的2级分类,纵坐标为小鼠基因组的29个模块;图中的括号外数值代表Pearson相关系数,绝对值越接近1代表相关性越强;括号内数值代表P值,P<0.05有统计学差异。红色代表模块基因与各类代谢物呈正相关,蓝色代表各类模块基因与代谢物呈负相关
2.4.2差异相关基因的PPI分析 相关基因与所有差异基因取交集,共得到88个差异相关基因。PPI分析结果显示,有15种蛋白之间关系密切,其所对应的基因分别为NTNG2、ENG、SEMA4G、JAG2、FGF11、SERPINE1、GDF15、GADD45G、F3、NGF、FGF21、PGF、EGFL7、SEMA6D、LRFN1,即为关键基因。
3 讨 论
丙泊酚已有30余年的临床应用史,目前是临床中最常用的静脉麻醉药物,主要用于患者的麻醉诱导和维持。然而丙泊酚应用于患儿时,对处于发育期患儿大脑的安全性一直存在争议[5]。近年多项研究表明丙泊酚对啮齿类、斑马鱼、恒河猴等发育期动物可产生神经毒性,导致神经元死亡和长期的神经行为损伤[3,6-7]。然而关于丙泊酚致神经毒性的关键基因以及分子机制尚不明确,因此了解丙泊酚致神经毒性的关键基因及其相关分子机制至关重要。
本研究中CCK-8实验检测结果显示,随丙泊酚浓度升高,HT22细胞活性显著下降,提示丙泊酚对HT22细胞存在神经毒性,且其毒性作用随丙泊酚浓度的升高而升高。对不同浓度丙泊酚处理HT22细胞24 h后,提取各组细胞中的代谢物进行代谢组学分析,LC-MS分析显示,共检测到2 701种代谢物;进一步对这2 701种代谢物进行各组间差异分析和趋势分析,最终获得了49种关键代谢物。对不同浓度丙泊酚处理HT22细胞24 h后,提取细胞中的RNA进行转录组学分析,获得Ctrl组和各浓度组之间的差异基因。最后进行代谢组学和转录组学的联合分析,以小鼠基因组为参考系,以关键代谢物为表型,进行WGCNA分析,筛选出相关基因;将差异基因与相关基因取交集,得到88个差异相关基因。为进一步了解基因之间功能联系,筛选出关键基因,对这88个基因所编码的蛋白进行PPI分析,发现15个基因(NTNG2、ENG、SEMA4G、JAG2、FGF11、SERPINE1、GDF15、GADD45G、F3、NGF、FGF21、PGF、EGFL7、SEMA6D、LRFN1)所编码蛋白之间关系密切,即为本研究最终获得的关键基因。
NGF对神经元的发育、分化和存活起着关键作用。为了探索NGF在丙泊酚致神经毒性中的作用,研究发现,NGF可通过Rac1信号通路对体外培养的新生大鼠海马神经元发挥神经保护作用,从而减轻丙泊酚诱导的海马神经元凋亡[8]。NTNG2编码膜锚定蛋白netrin-G2,敲低小鼠NTNG2基因会导致小鼠神经元形态严重受损以及皮质神经元迁移障碍[9],使小鼠在学习、记忆、视觉和运动功能方面存在明显缺陷[10]。FGF11属于成纤维细胞生长因子信号蛋白家族,可能参与神经系统的发育和功能的维持[11-12]。研究显示,在有机磷导致的中国珍稀米诺鱼的神经毒性研究中,FGF11发挥了重要作用[13]。F3是一种糖基磷脂酰肌醇锚定的糖蛋白,参与神经元发育、突触维持和神经元网络的形成,可能影响神经元凋亡和β-淀粉样蛋白(Aβ)的产生[14]。EGFL7是表皮生长因子样蛋白家族的成员,在神经系统中,EGFL7通过Notch通路促进神经干细胞形成神经元[15]。用分泌EGFL7的间充质干细胞治疗新生小鼠缺血缺氧性脑损伤,能够改善其运动功能[16]。SERPINE1是纤溶酶原激活抑制剂,能够阻断纤溶酶原向纤溶酶的转化,在中枢神经系统中对于神经元的迁移、成熟突触的连接等过程起着重要作用[17]。纤维蛋白溶解系统能够调节中枢神经系统内的炎症和退行性事件。小鼠大脑中长期升高的SERPINE1水平可能通过抑制纤溶酶依赖性的Aβ的降解,引起Aβ的累积,进而促进其阿尔兹海默症的进展[18]。GDF15是转化生长因子-β超家族的一员,研究证明GDF15可通过p62-Keap1-Nrf2信号通路抑制脊髓损伤后神经元氧化应激依赖性铁死亡,减轻神经损伤,从而促进脊髓损伤小鼠运动功能恢复[19]。FGF21能够通过其多种代谢调节作用和抗炎作用改善肥胖诱导的小鼠认知功能障碍和焦虑样行为[20]。PGF是一种血管生成因子,在病理条件下可发挥神经营养和神经保护的作用[21]。以上的研究均表明,NTNG2、FGF11、F3、EGFL7、SERPINE1、GDF15、FGF21、PGF基因均在神经损伤的过程中发挥着重要作用,但这些基因在丙泊酚致神经毒性的研究中尚未见相关报道,结合本研究的筛选结果,它们可能是丙泊酚致神经毒性的关键基因。
除此之外,既往的研究发现,SEMA4G、JAG2、GADD45G、SEMA6D、LRFN、ENG基因在轴突引导[22-23]、神经系统发育[24-25]、新皮质形成[26]等过程中均发挥重要作用,但尚未见这些基因与神经毒性或者神经损伤有关联的报道,结合本研究的筛选结果,这些基因可能是丙泊酚导致神经毒性的潜在靶点。
综上所述,通过代谢组学和转录组学联合分析,丙泊酚致神经毒性的关键基因为NTNG2、ENG、SEMA4G、JAG2、FGF11、SERPINE1、GDF15、GADD45G、F3、NGF、FGF21、PGF、EGFL7、SEMA6D、LRFN1,为后续丙泊酚致神经毒性的分子机制研究提供了理论基础,同时也为丙泊酚的神经毒性预防和治疗用药的开发提供了研究方向。
作者声明:李慎风、庄曌参与了研究设计;李慎风、王寿世、刁玉晶、曹宏参与了论文的写作和修改。所有作者均阅读并同意发表该论文,且均声明不存在利益冲突。
