基于SNP标记的单种属野鸦椿遗传变异和分化分析
2023-07-25陈志峰蔡丽娟孙维红邹双全
陈志峰, 倪 辉, 冯 翯, 陈 啸 , 蔡丽娟, 孙维红,3, 邹双全
(1.遵义师范学院,贵州 遵义 563000;2.福建农林大学林学院,福建 福州 350002;3.自然生物资源保育利用福建省高校工程研究中心,福建 福州 350002;4.福州外语外贸学院,福建 福州 350202)
野鸦椿属(Euscapis)为省沽油科(Staphyleaceae)落叶灌木或小乔木,仅野鸦椿(E.japonica)单个种[1],主要分布在中国南部、日本和朝鲜,是我国近年来快速发展的观赏植物和药用植物。野鸦椿挂果期长、果色鲜红、果成熟开裂似展翅蝴蝶,在园林上可盆栽、孤植、列植或片植,在我国福建、江西、贵州等地作为行道树、观赏树、药用林而被广泛种植[2]。野鸦椿枝、叶和果均富含酚酸类、黄酮类、总三萜、生物碱等成分,可用于治疗感冒、头晕、头痛等,是我国民间的传统药材[1-3]。目前,对野鸦椿的研究主要集中于种质资源鉴定、种苗繁育、栽培、化合物分离与鉴定、药理活性等方面,而忽略了野生资源的保护[4-5]。了解物种的遗传变异是制定科学保护策略的理论基础。野鸦椿自然群体主要分布于海拔100~1 200 m的山谷或林缘,因受到人类活动的干扰,导致天然群体面积和分布逐渐缩小,再加上自然更新能力弱,野鸦椿种质资源和遗传变异正在遭受威胁[6]。因此,了解野鸦椿的遗传变异可为其分类以及种质资源利用和制定保护策略等提供参考。
遗传变异是物种适应和进化的基础[7],也是抵抗环境变化的重要保障。林木的遗传多样性越丰富,则越能适应不断变化的环境和恶劣条件[8]。单核苷酸多态性(single nucleotide polymorphism, SNP)是全基因组水平上由单个核苷酸碱基变异引起的DNA序列多态性,可以检测基因组序列中每个核苷酸的变化[9],具有数量多、分布广、代表性强、遗传稳定性好等特点,是评价物种遗传背景最为理想的分子标记[10]。特异性位点扩增片段测序技术(specific-locus amplified fragment sequencing, SLAF-seq)能够大规模、快速、有效、准确地开发物种中的SNP[11],尤其适用于没有参考基因组的物种。本研究利用SLAF测序技术开发野鸦椿群体的SNP标记,旨在从分子水平上揭示其遗传变异和分化,了解现存野生种群的遗传背景及其对种群结构的影响,为野鸦椿种群现状的评价和科学管理提供遗传学依据。
1 材料与方法
1.1 取样与群体特征
2019年8月—2020年8月,在野鸦椿果期开展野外调查,收集了自然分布的12个种源,共94份样品。2020年4月,采集所有样品的嫩叶,用体积分数为95%酒精和纯水洗净,经液氮处理后,置于-80 ℃冰箱中保存。样品取样信息详见表1。2019—2022年,对全部样本进行物候观测,详细记录各种源果和叶的表型。
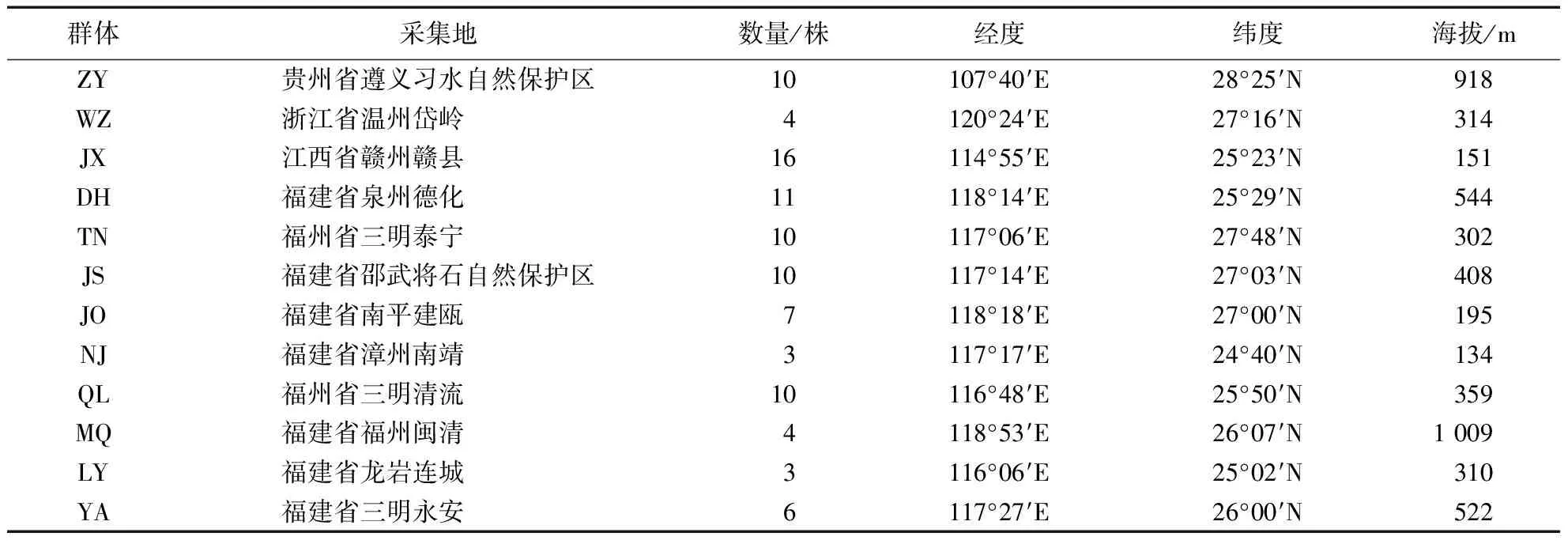
表1 野鸦椿的采样信息Table 1 Sampling information of E.japonica
1.2 SLAF文库的构建和测序
SLAF测序包括样品质量检测、文库构建、文库质量检测和文库测序。使用改良的十六烷基三甲基溴化铵法(acetyltrimethylammonium bromide, CTAB法)提取叶片DNA,以琼脂糖凝胶电泳检测和Nano-drop 2000紫外分光光度计(Thermo,美国)分别检测DNA的纯度和浓度。质检合格的DNA用于SLAF文库的构建。
利用选定的RsaⅠ+HaeⅢ对检测合格的每个样品DNA进行酶切;对得到的酶切片段进行3′端加多聚腺苷酸[polyadenylic acid, poly(A)]处理、连接Dual-index测序接头、PCR扩增、纯化、混样、切胶,选取314~364 bp的目标序列定义为野鸦椿的SLAF标签。文库质检合格后在华大基因Illumina HiSeq 2500平台进行测序,下机的原始数据用于开发SLAF标签和SNP分子标记。
1.3 群体遗传结构和变异分析
使用MEGA X软件中的邻接法(neighbor-joining method, NJ),采用p-distance模型,bootstrap重复1 000次,以省沽油(Staphyleabumalda)作为外类群,构建进化树;利用Admixture(V1.3.0,http//software.genetics.ucla.edu/admixture/index.html)进行群体结构分析,假设样本的分群数(K值)为1~10,当交叉验证错误率(cross-validation errors, CV)最小时为最佳分类群数。EIGENSOFT(V7.2.1,https://www.hsph.harvard.edu/alkes-price/software/)用于主成分分析。利用GenAlEx 6.5软件计算各群体和每对引物的观测等位基因数(observed number of alleles,Na)、有效等位基因数(effective number of alleles,Ne)、观测杂合度(observed heterozygosity,Ho)、期望杂合度(expected heterozygosity,He)、Nei′s多样性指数(Nei′s gene diversity,H)、香农多样性指数(Shannon′s information index,I)、多态性信息含量(polymorphic information content, PIC)、遗传分化指数(fixation index,FST)等遗传多样性参数。由于浙江省温州岱岭(WZ)和福建省漳州南靖(NJ)、福州闽清(MQ)、龙岩连城(LY)等地采集到的野生野鸦椿个体数量过少,不进行遗传参数估算分析。
2 结果与分析
2.1 SLAF标签和SNP开发
对测序后的数据进行过滤,共获得329.73 Mb 干净数据,平均测序深度为16.00×,Q30的平均质量为92.80%,表明测序数据质量好,可用于SLAF标签的开发。对获取的数据进行聚类分析,序列相似的定义为同一个SLAF标签。在此基础上,筛选样本间存在差异的SLAF标签为多态性的SLAF标签,以深度最高的多态性SLAF标签作为参考序列进而开发全基因组范围的SNP标记。根据次要等位基因频率(minor allele frequency, MAF)大于0.05和完整性大于0.8的原则,筛选出高质量的SNP标记。本研究中,基于SLAF测序共获得19 111 187个SLAF标签,每个样品平均获得607 458个多态SLAF标签和238 850个高质量的特异性SNP,可用于野鸦椿遗传变异和分化的分析(表2)。

表2 野鸦椿群体的SLAF标签统计1)Table 2 Statistics of SLAF tags for different populations of E.japonica
2.2 群体变异
由表3可知,在物种水平上,野鸦椿的H和I分别为0.373 4和0.546 8,Ho(0.069 2)小于He(0.371 2)。群体间的H和I分别在0.201 2(JX)~0.449 7(TN)和0.334 2(JX)~0.607 8(TN)之间,除了JO群体,其他群体的Ho均小于He(表3)。此外,分子方差分析表明,种群间的遗传变异占总变异的40.36%(P≤0.001),而群体内的遗传变异仅占总变异的27.65%(P≤0.001)(表4)。

表3 野鸦椿群体的遗传多样性指数分析1)Table 3 Analysis on genetic diversity index of different E.japonica populations

表4 分子方差分析Table 4 Analysis of molecular variance
2.3 遗传结构
基于SNP构建的系统发育树表明,以省沽油科的省沽油(SXY)为外类群,所有野鸦椿样品分为两大类。聚类Ⅰ包含36个样本,主要来自ZY、WZ、MQ、YA、LY等群体以及DH9~DH11、TN6~TN10、JS10(图1A);其余JX、JO、QL、NJ等群体以及DH1~DH8、TN1~TN5、JS1~JS9则为聚类Ⅱ。结合2019—2022年的物候和表型观察,发现聚类Ⅰ为落叶类,花期4—5月,果实期7—10月,6月果皮变色,7月果皮全红,10—11月开始落叶掉果;叶纸质或厚纸质,叶缘具细锯齿,果软革质,果表皮肋脉明显(图1E、1F、1G)。而聚类Ⅱ为常绿类,花期5—6月,果实期9—3月,8月果皮变色,9月果皮全红,果实可在枝头宿存直到次年3月;叶膜质,叶缘具钝锯齿,果革质,果表皮肋脉不明显(图1B、1C、1D)。
使用Admixture软件构建了野鸦椿种群的群体结构(图2)。从图2B可见,当K=2~6时,可以明显地看到落叶类和常绿类群体之间几乎不存在基因交流,当K=7~10时,落叶类和常绿类群体之间存在少量的基因渐渗。但在图2A中,K=3时,交叉验证错误率最低,意味所有群体的最优分类为3组,第1组和第2组样本为常绿类,第1组样本(绿色)共有42个,主要来自福建省的群体,如JO、QL、JS和NJ等群体;第2组样本(黄色)有16个,主要来自江西省JX群体;第3组(灰色)为落叶类,包含36个样本,且这些样品与系统发育树中聚类Ⅰ的样本一致(图2B)。

A.群体结构点图;B.种群遗传的聚类图。图2 野鸦椿的遗传聚类Figure 2 Genetic clustering analysis of E.japonica
主成分(PCA)分析发现,省沽油(SXY)与野鸦椿明显分开,野鸦椿所有样品主要分为3类,与群体结构的结果完全相同。A组中的样本与系统发育树中聚类Ⅰ和群体结构中第3组的一样;B组和C组中的样本分别与群体结构中第2组和第1组的一致。由此可见,A组是落叶类,B组和C组是常绿类(图3)。
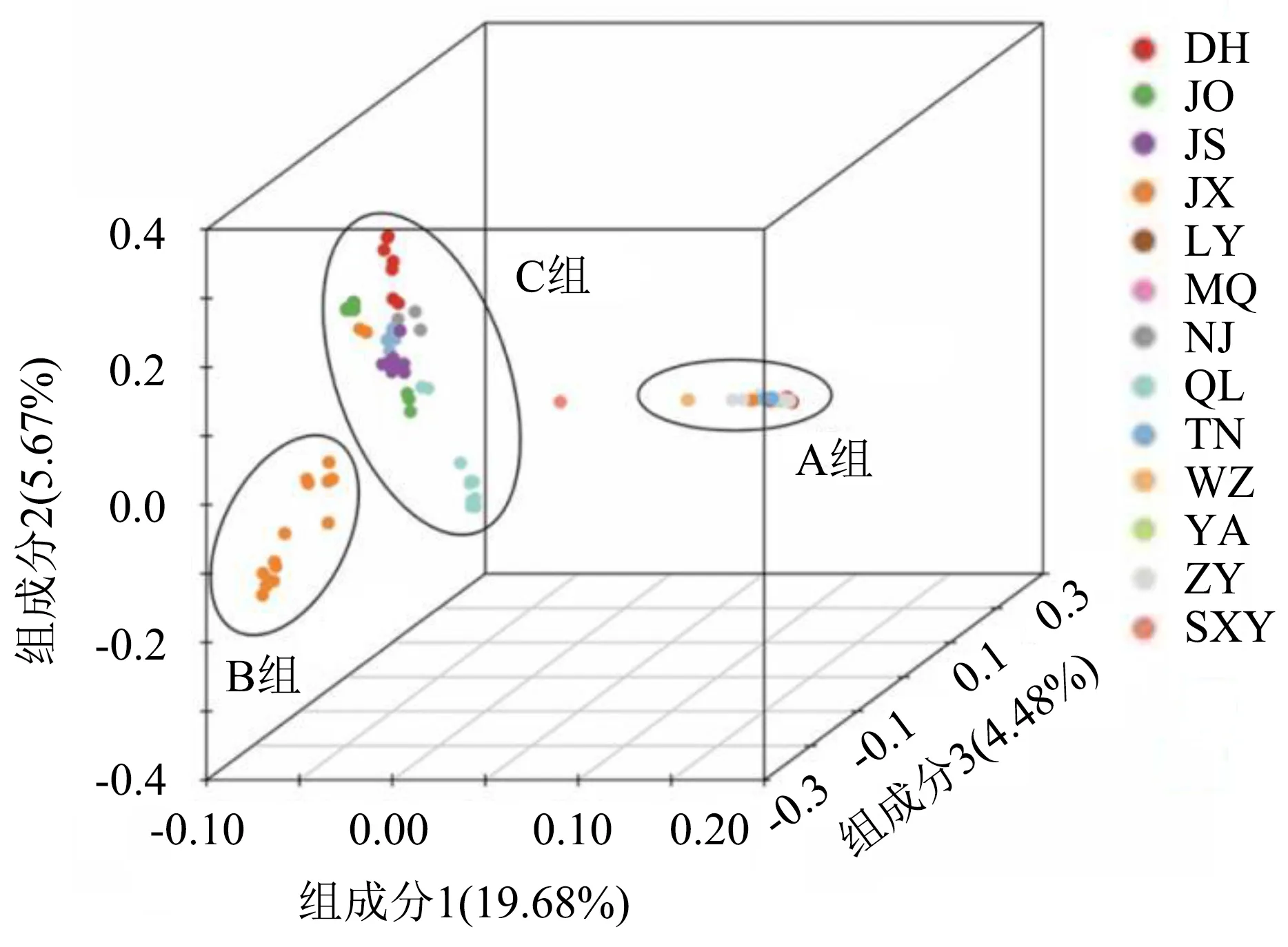
图3 基于SNP位点的主成分分析Figure 3 Principal component analysis based on SNP sites
2.4 遗传分化
FST是衡量群体间分化程度的一个指标。Wright[12]认为,FST为0~0.05,群体间遗传分化很小,可以不考虑;FST为0.05~0.15,群体间存在中等程度的遗传分化;FST为0.15~0.25,群体间遗传分化较大;FST为0.25以上,群体间有很大的遗传分化。分析各个群体的FST值发现,本研究群体间存在很大遗传分化的群体有TN(0.79)、DH(0.76)、JS(0.53)、JX(0.44);群体间存在中等程度遗传分化的群体有ZY(0.08)、QL(0.05)群体;YA(0.01)、JO(0.02)群体的分化程度小(表5)。另外,综合表型观察、系统发育树、PCA和群体结构的分析结果,对野鸦椿落叶类和常绿类的样品进行FST值分析发现,落叶树和常绿树的FST值分别为0.28和0.36。

表5 各个群体的遗传分化指数分析1)Table 5 Genetic differentiation index of each population
3 讨论
利用简化基因组测序技术SLAF-seq,获取大量多态性SLAF标签,进而开发特异性的SNP标记,被广泛应用于遗传进化分析[13]、GWAS关联分析[14]、遗传图谱构建[15]。本研究对野鸦椿自然分布区12个种源共94个样品进行SLAF测序,每个样品平均获得了238 850个多态性SNP标记,与栎树(Quercusshumardii)群体(200 735)、古茶树(Camelliasinensis)群体(283 376)的平均SNP标记相近[16-17]。基于高质量的SNP进行了野鸦椿遗传变异分析发现,野鸦椿在物种水平上的Nei′s遗传多样性(H)和Shannon多样性指数(I)分别为0.373 4和0.546 8,比野生省沽油群体的遗传多样性指数(H=0.265 3,I=0.411 8[18];H=0.219 0,I=0.349 3)[19]高,但这并不能说明野鸦椿与同科的树种相比在物种水平上的遗传多样性更为丰富。因为本研究中野鸦椿的观测杂合度(0.069 2)远小于期望杂合度(0.371 2),这意味着野鸦椿中杂合子缺失,群体遗传多样性水平较低。
据《Flora of China》[1]记载,野鸦椿属仅1种落叶灌木或小乔木树种,即野鸦椿。通过对12个群体长达3 a的物候观察发现,野鸦椿群体主要有落叶类和常绿类两大类型,且它们的物候、叶和果的表型差异明显。本研究中,系统发育分析、遗传结构分析和主成分分析结果基本一致,将所有野鸦椿样品主要分为落叶类和常绿类。地理隔离、栖息地破碎化、基因交流不频繁可以改变物种进化模式的方向,影响其遗传变异,导致种群内的遗传多样性低以及不同种群之间的分化程度高[20-24]。对大多数木本植物而言,群体内的遗传变异比群体间的遗传变异丰富[20],而本研究中野鸦椿的群体间遗传变异(40.36%)比群体内的丰富(21.99%),且野鸦椿显著分化为落叶类和常绿类,这暗示了在长期进化过程中某些因素推动了野鸦椿的分化。
地理隔离使得同一物种因地理障碍难以发生基因交流,导致生殖隔离,形成独立的种群[21]。生境破碎化会导致遗传侵蚀、遗传漂变和近交,引起杂合度降低,使个体适应性下降,最终抑制种群的持续[21,24]。野外调查发现,NJ、LY、MQ、WZ等群体位于自然村落,受到人类活动的干扰,其群体数量小;贵州省的ZY群体、浙江省的WZ群体和江西省的JX群体与福建省内的群体地理相隔远,群体间难以发生基因交流。野鸦椿为兼性异交,蜜蜂为主要传粉者[25],但野外观测发现,即使在同一群体内落叶类和常绿类野鸦椿的花期至少相差1个月,导致落叶类和常绿类之间不可能存在基因交流;遗传结构分析也证实落叶类和常绿类间几乎没有基因渗透。TN、DH、JS等群体的FST值较高,说明了落叶类和常绿类的同时存在是野鸦椿群体高度分化的结果。综上分析,本研究推测地理隔离、栖息地破碎化、基因交流受阻或缺乏是野鸦椿群体内遗传多样性低、群体间分化程度高的原因。
4 结论
本研究观测了自然分布区12个野鸦椿种源的物候、花和果的表型;并首次利用SLAF-seq对这些野生种质进行测序,挖掘了238 850个高质量的多态性SNP位点,从分子层面分析了野鸦椿的遗传变异和分化。野鸦椿群体内遗传多样性低、群体间遗传分化程度高。多种分析方法表明,单种属野鸦椿已经分化为落叶类和常绿类。落叶类的叶纸质或厚纸质、叶缘具细锯齿,果软革质、果表皮肋脉明显;而常绿类的叶膜质、叶缘具钝锯齿,果革质、果表皮肋脉不明显。
