MRI序列以及注意力门、残差网络对U-Net脑肿瘤分割模型的影响
2023-07-24张巨朱文珍张顺朱虹全吴迪刘栋
张巨,朱文珍,张顺,朱虹全,吴迪,刘栋
兴趣区(region of interest,ROI)勾画是脑肿瘤影像研究及临床定量分析的基础。手动勾画兴趣区工作量大,效率低,受人员经验及主观判断影响较大,具有明显的局限性。一些简单的算法如阈值分割、区域生长[1,2]等在勾画边界分明、特征简单、体素值差异明显的目标时可以显著提高效率,是传统分割方法中常用的辅助方式。然而医学图像往往具有极高的复杂性和多样性,传统手段在包括脑肿瘤分割等多种使用场景下效果欠佳,常需大量手工调整。深度学习技术实现了特征工程的自动化,相对传统技术具有明显优势。语义分割(semantic segmentation)是目前使用深度学习对医学图像进行分割的常用的方式[3]。其中相较传统全卷积网络(fully convolutional networks,FCN),U-Net结构包含了一个与编码路径对称的解码路径并通过相应层级拼接的方式提供了更为精确的定位[4],该结构因为简单可靠被广泛运用于图形分割领域[5,6]。部分研究认为对U-Net的结构调整可能可以进一步提高模型的准确性,Oktay等[7]提出的注意力门机制可以增加目标区域的权重,He等[8]提出的残差网络有效的应对了网络退化的问题。深度学习模型的训练、使用及数据的储存、传输均需要一定的硬件及时间成本,该成本可通过降低数据量来有效控制,然而充足的数据量是充分训练模型的基础。识别并剔除无法显著提升模型分割准确率的冗余数据则具有实用意义。本研究使用BraTS 2021的数据,探索了不同序列组合对U-Net模型的影响,并对比了基线U-Net以及添加了注意力门、残差网络模块的模型在分割脑肿瘤及亚区方面的差异。
材料与方法
1.图像数据处理
使用BraTS 2021脑胶质瘤数据1251例,每个病例包括T1、T1增强、T2、T2FLAIR四个序列的图像以及一个标签图像,标签包括:0,背景(非肿瘤区域);1,肿瘤坏死区域;2,水肿、浸润区域;4,肿瘤强化区域。将所有MRI图像归一化处理,标签值4改为3。将MRI图像使用7种序列组合(4通道组、无T1的3通道组、无T2的3通道组、无T2FLAIR的3通道组、无T1增强的3通道组、无T1及T2的2通道组,无T2及T2FLAIR的2通道组)堆叠为多通道图像,对图像进行裁剪,保留图像中心范围为128×128×128的体素。由于部分病例经裁剪后目标体素较少且缺乏肿瘤图像特征,这部分病例被排除。剩余的病例以7:2:1的比例分为训练组、验证组、和测试组。
2.模型构建
基线U-Net模型:使用3D U-Net模型,结构如图1,编码器包括5层子模块,每层子模块包括2个卷积层(首个子模块filters=16,后续子模块filters依次乘以2;kernel_size=3)、2个激活函数层(activation=LeakyReLU,alpha=0.01)、以及一个随机失活层,前4个子模块包括一个最大池化层(pool_size=2)。解码器包括4层子模块,每层子模块包括一个反卷积层(首个子模块filters=128,后续子模块filters依次除以2;kernel_size=2)、2个卷积层(首个子模块filters=128,后续子模块filters依次除以2;kernel_size=3)、2个激活函数层(activation=LeakyReLU,alpha=0.01)、以及一个随机失活层,其中反卷积层的输出接受来自相应编码层的跳跃连接。

图1 基线U-Net模型。
注意力U-Net模型:编码器与基线模型一致。解码器与基线模型间的区别在于卷积层前的注意力门结构(图2):来自深层的信号经卷积(首个注意力门结构filters=128,余依次除以2,kernel_size=1)、激活函数(ReLU)、卷积(同前)后,与来自相应编码层的跳跃连接的信号经卷积层(首个注意力门结构filters=128,余依次除以2,kernel_size=1,strides=2)后相加,经过激活函数ReLU、卷积层(filters=1,kernel_size=1)、激活函数Sigmoid后,上采样至前述跳跃连接的大小并与之相乘,经卷积层(首个注意力门结构filters=128,余依次除以2,kernel_size=1)、批量归一化后输出。
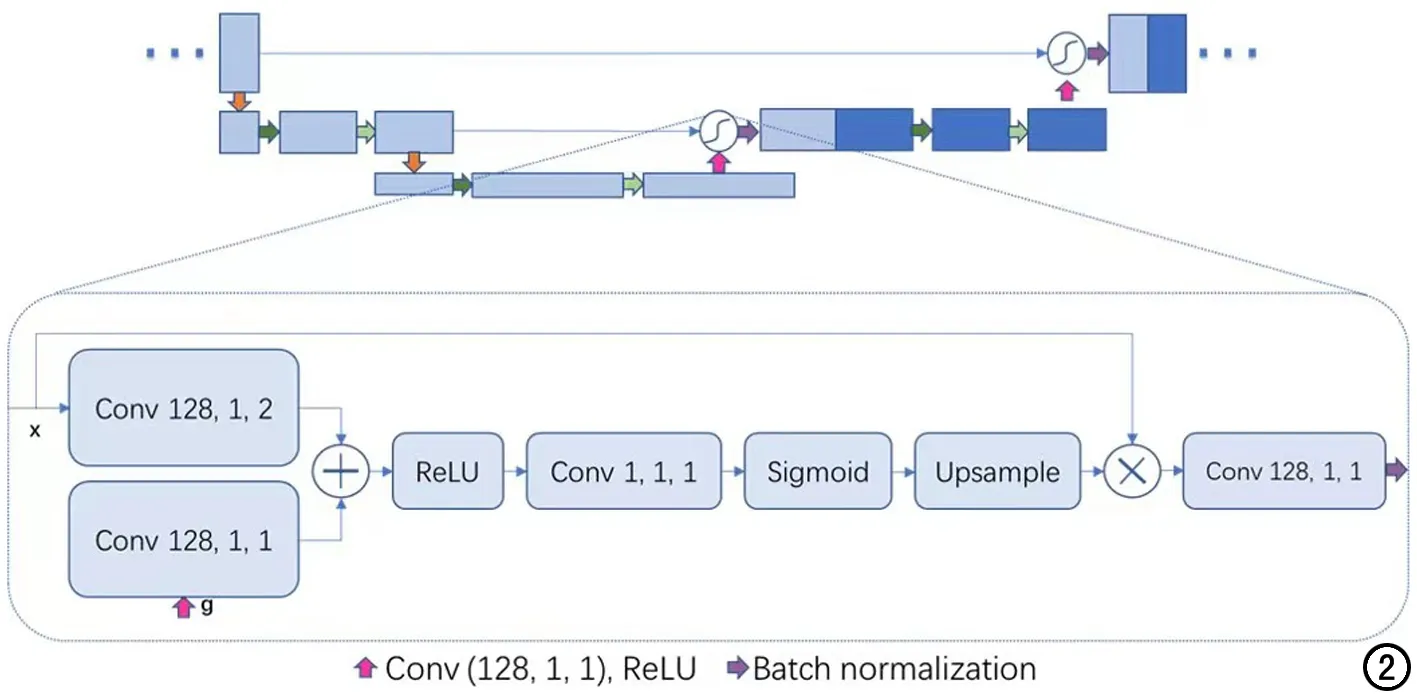
图2 注意力门的结构(图中显示第1个注意力门的参数,后续注意力门filters依次减半)。
残差U-Net模型:与基线模型的区别在于各子模块第2次卷积后的输出接受第1次卷积前的输入逐点卷积(filters与相应子模块卷积层一致,kernel_size=1)后的跳跃连接(图3)。

图3 残差网络的跳跃连接。
残差注意力U-Net模型:即结合上述注意力门及残差模块至基线U-Net的模型。
3.模型训练
使用前述7种不同多通道图像的训练组和验证组训练基线U-Net,使用4通道图像的训练组和验证组训练残差U-Net、注意力U-Net、残差注意力U-Net。损失函数使用Categorical Focal Loss结合Dice Loss, 优化器使用自适应矩估计(Adaptive Moment Estimation,Adam),初始Learning rate为0.0001,Batch size为4,每种序列组合、模型至少训练1000个Epoch,保存每次验证IOU分数提高的模型。训练使用一块NVIDIA GeForce RTX 4090显卡,在TensorFlow框架(2.10.1)/Keras API(2.10.0)下完成。
4.模型对比
选取每次训练1000个Epoch中验证IOU分数最高的模型进行对比,使用各模型对测试组(n=116)进行图像分割,计算分割结果的Dice系数。使用Friedman检验和成对比较(经Bonferroni校正法调整显著性值)对比不同序列组合以及不同模型间Dice系数的差异,采用IBM SPSS Statistics 26进行统计学分析。
结 果
经过裁剪,最终1151例患者被纳入研究,其中训练组805例,验证组230例,测试组116例。
使用不同序列组合训练基线U-Net模型分割肿瘤各区域的Dice系数均值、标准差及秩均值见表1。在分割肿瘤强化区域(Gd-enhancing tumor,ET)时,无增强序列的3通道组Dice系数低于其他6组(P<0.001),余组间差异无统计学意义;在分割肿瘤核心区域(tumor core,TC)时,同样是无增强序列的3通道组Dice系数低于其他6组(P<0.001),余组间差异无统计学意义;在分割全肿瘤(whole tumor,WT)时,无T2和T2FLAIR的2通道组Dice系数显著低于其他组(P<0.001),无T2FLAIR的3通道组Dice系数低于除前述2通道组以外的其他组(P<0.001),4通道组和无T1的3通道组的Dice系数高于其他组(P<0.05),余组间无差异无统计学意义。

表1 各序列组合Dice系数均值、标准差及秩均值
基线U-Net、注意力U-Net、残差U-Net、注意力残差U-Net模型对一例颞叶脑肿瘤的分割结果见图4。
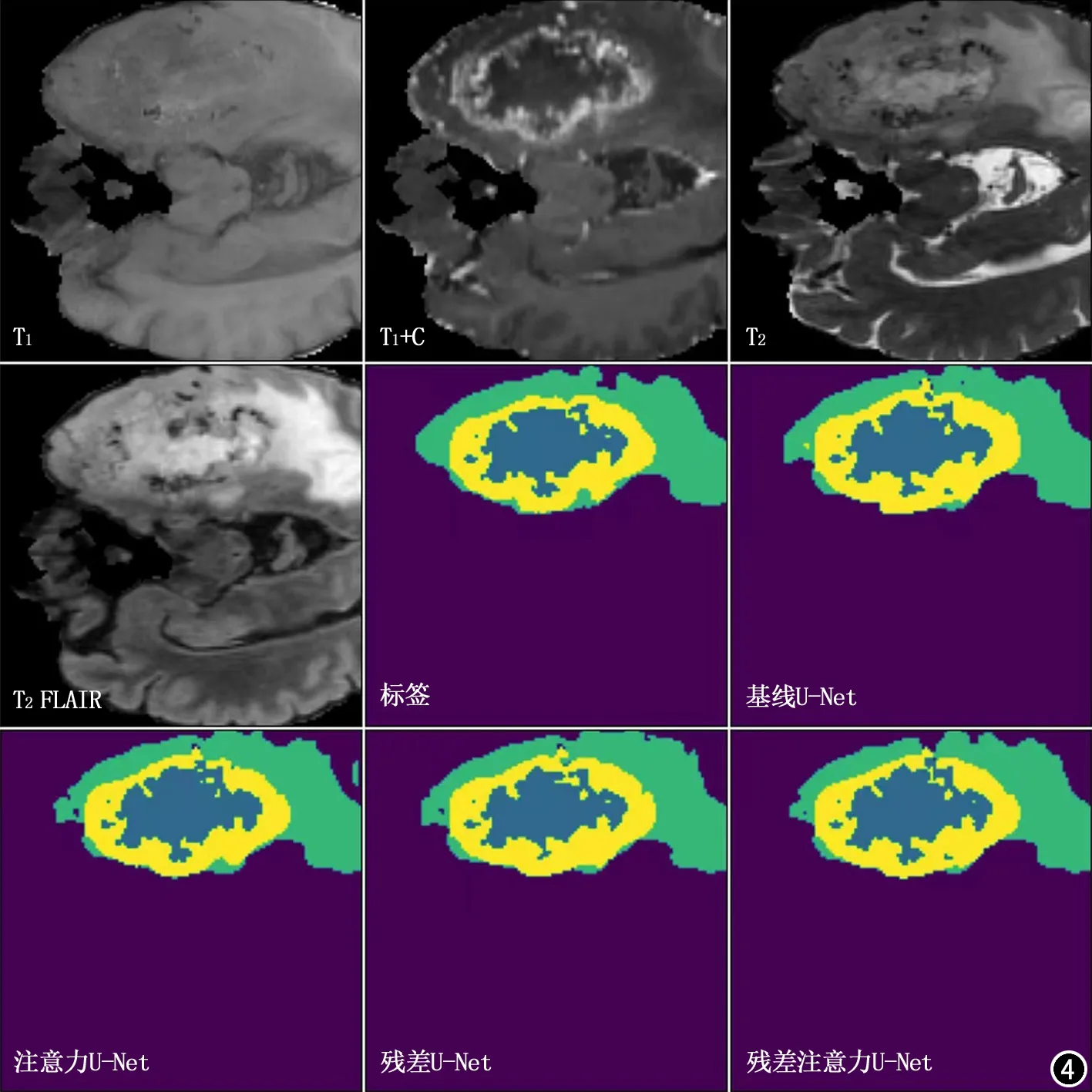
图4 一个颞叶肿瘤的T1、T1增强、T2、T2 FLAIR图像,以及预先标注的标签、4种U-Net模型的分割结果。
基线U-Net、注意力U-Net、残差U-Net、残差注意力U-Net的Dice系数均值、标准差及秩均值见表2。在分割肿瘤强化区域和核心区域时,4种模型间的Dice系数差异无统计学意义。在分割全肿瘤时,Friedman检验中4种模型间差异有统计学意义(P<0.05),但在进一步的成对比较中,经Bonferroni校正法调整显著性值后,两两间差异均无统计学意义。

表2 各序列组合Dice系数均值、标准差及秩均值
讨 论
本研究结果显示剔除T1增强序列会显著降低模型分割肿瘤强化区域及核心区域的准确性,而剔除T2FLAIR、尤其是同时剔除T2FLAIR和T2会显著降低模型分割全肿瘤的准确度,以上结果可能与BraTS的标注原则有关。BraTS中对肿瘤强化区域的定义是相对于T1平扫和影像表现正常的白质,增强后高信号的区域。肿瘤的核心区域则包括肿瘤的强化区域和囊变、坏死区。囊变、坏死区的典型表现为T1增强图像上的低信号。全肿瘤的定义则进一步包括了肿瘤周围的水肿及肿瘤浸润区域,通常表现为T2FLAIR上异常的高信号[9]。T1增强和T2FLAIR序列可以提供较为明确的组织对比度和肿瘤边缘信息,剔除这些序列后,可能会导致模型对特征的提取不充分,降低准确率的同时提高误检率。且这两种序列可以提供不同的肿瘤信息,去掉一种或两种,则可能导致模型的鲁棒性降低,难以处理相对复杂的分割任务。在剔除T2FLAIR的基础上进一步剔除T2后,模型对全肿瘤分割的准确性进一步显著下降可能是因为T2序列同样对肿瘤的水肿或浸润区较为敏感,图像存在较好的对比度,将其剔除后,剩余序列(T1、增强)在部分病例相应区域的图像特征不足导致的。本研究中4通道组和无T1的3通道组在分割全肿瘤时Dice系数显著高于其他组,结合分割其他区域的结果,这两组具有较好的综合表现,且二者间差异无统计学意义。该结果一方面提示我们训练和使用模型时,保证信息的丰富度对模型分割的准确率具有重要意义,另一方面也提示我们序列并非越多越好。本研究中,相对于4通道组,剔除T1平扫序列未对模型的分割准确性产生显著性的影响,可能是由于T1序列在上述标注区域常无明显的对比度或清晰的边界,可能所蕴含的相应图像特征较为稀少且不稳定,T1的加入可能无法提供足够提升模型表现的有用信息,反而增加了噪声和冗余信息(针对标注区域),提高了模型的复杂度和过拟合风险。影像组学的研究认为医学图像包含大量人眼不易直接捕捉的图像特征[10,11],且这部分图像特征同样包含来源于组织的结构或功能信息,具有分析和诊断的价值。由于深度学习特征工程的自动化,本研究的结果可能提示,由T2及T2FLAIR图像特征所反映的脑肿瘤组织和边界信息,并未被T1图像所完整和可靠地蕴含,或至少不能被本研究所使用模型捕捉到。
本研究中对比的4种U-Net模型仅在分割全肿瘤时表现出显著差异,且在随后的成对比较中,若经Bonferroni校正法调整显著性值后则两两间差异无统计学意义。部分研究认为对U-Net的结构调整可能不如非结构调整有效,如Isensee等提出的nnU-Net(“no-new-Net”),仅围绕基础的U-Net模型调整,将重点集中于数据处理、模型训练、推理、后处理上,其自适应的特点令该框架具有良好的泛用性和优秀分割准确性[12]。本研究的结果可能提示部分对U-Net结构的调整可以提高其在特定领域的分割效果,但也可能在其他领域增加过拟合风险以及降低泛用性。本研究中残差模型在分割全肿瘤时具有最大的Dice系数均值和秩均值,而注意力模型则具有最小的Dice系数均值和秩均值,结合Friedman检验结果,笔者推测在分割全肿瘤时,残差网络可能较注意力门更有优势。
残差网络通过跳跃连接可以实现恒等映射的效果,有效应对了深层网络的退化问题[8]。在本研究中,模型可能通过残差模块减少了信号的损失,保证了网络中梯度信号的强度。此外,医学图像的像素之间是存在局部相关的,则反向传播时的梯度也具有相关性,但随着网络深度的增加,该梯度间的相关性会越来越弱,难残差网络则极大的缓解了该相关性的衰减[13],保留了更多的梯度空间结构信息。
注意力门的加入会令模型逐步增加对图像兴趣区的相对权重,理论上可以增加模型的敏感性和准确性。而本研究中的注意力模型表现并未显著高于其他模型,笔者推测可能由于脑肿瘤形态、结构、大小、位置、信号等的异质性较大,测试集中可能存在的一些与训练集差异较大的病例,注意力门机制由于缺乏相应的学习从而导致关注区域的错误和分类能力的下降。
综上所述,在脑肿瘤分割模型的构建与使用过程中,输入图像的选择需要结合具体使用场景,在保证信息丰富度的同时也要避免冗余。多余的序列无法提高模型表现,反而可能引入噪声和冗余信息,提高模型的复杂度和过拟合风险,此外还会增加数据的储存、传输、运算的时间及硬件成本。在分割模型的选择方面,残差网络可能减少了模型的信号损失,保留了更多的原始图像特征信息,确保了梯度信号的强度,且减少了梯度间相关性信息的损失,从而提高了模型的表现。
