油茶闽43叶绿体基因组序列特征及其近缘物种亲缘关系分析
2023-07-04王小薇陈世品
王小薇 陈世品
摘 要:為了探究油茶闽43与山茶属近缘物种间叶绿体基因组的差异以及进化关系,利用基因组学分析方法,对油茶闽43与其他近缘山茶属物种叶绿体基因组进行比较分析。叶绿体基因组结构特征分析结果显示:9种山茶属物种的叶绿体基因组大小为156 902~157 567 bp,有130~136个基因。REPuter分析发现,这9种山茶属物种的长重复序列类型均为P和F,通过MISA软件确定9种山茶属物种的SSR数量为52~71 个,其中闽43的SSR数量为65 个。通过IR边界分析,白毛茶和油茶闽43与其他3种属内物种的边界基因有较为明显差异,同时通过核苷酸多态性分析,检测出叶绿体基因组共有5个高变区,分别为psbK~atpA、rpoB~psbD、rps4~ndhJ、ycf1、ndhF~rps12~exon1。并从系统发育树的结果得出,油茶闽43与落瓣油茶的亲缘关系最近。该研究可为普通油茶种群遗传结构提供基础,为提高普通油茶的产油量以及优良品种的选育提供理论依据。
关键词:油茶;叶绿体基因组;长重复序列;系统发育树
中图分类号:S 794.4 文献标志码:A 文章编号:0253-2301(2023)01-0009-09
DOI: 10.13651/j.cnki.fjnykj.2023.01.002
Analysis of the Sequence Characteristics of Chloroplast Genome and theGenetic Relationship of Closely Related Species of Camellia oleifera Min 43
WANG Xiao-wei, CHEN Shi-pin*
(College of Forestry, Fujian Agriculture and Forestry University, Fuzhou, Fujian 350002, China)
Abstract: In order to explore the differences and evolutionary relationships of chloroplast genomes between Camellia oleifera Min 43 and the closely related species of Camellia L., the chloroplast genomes of Camellia oleifera Min 43 and other closely related species we compared and analyzed by using the genomics analysis. The results of the structure characteristic analysis of chloroplast genome showed that the chloroplast genome size of 9 Camellia L. species ranged from 156 902 to 157 567 bp, with 130-136 genes. The REPuter analysis showed that the long repetitive sequence types of these 9 Camellia L. species were P and F. The SSR number of 9 Camellia L. species was determined to be 52-71 by using the MISA software, among which the SSR number of Min 43 was 65. Through the IR boundary analysis, the boundary genes of Camellia sinensis and Camellia oleifera Min 43 were significantly different from those of the other three genus species. At the same time, by using the nucleotide polymorphism analysis, five hypervariable regions were detected in the chloroplast genome, which were psbK-atpA, rpoB-psbD, rps4-ndhJ, ycf1, and ndhF-rps12-exon1. According to the results of phylogenetic tree, the relationship between Camellia oleifera Min 43 and Camellia kissii was the closest. This study could provide a basis for the genetic structure of Camellia oleifera population, and also provide a theoretical basis for improving the oil production of Camellia oleifera and the breeding of excellent varieties.
Key words: Camellia oleifera; Chloroplast genome; Long repetitive sequence; Phylogenetic tree
叶绿体是植物进行光合作用的细胞器,光合作用的全部过程都发生在叶绿体内。此外, 叶绿体中还发生着多种与植物生命活动相关的代谢过程,在植物的能量代谢和物质循环中起着非常重要的作用
[1]。叶绿体含有自己的基因组和一套独特的遗传系统,其基因表达调节非常复杂[2]。叶绿体的遗传方式大多是母系遗传[3],基因组结构组成相对保守,适用于植物系统发育方面的研究。目前已经广泛应用于植物的遗传多样性分析,构建和分析系统发育树,进行不同物种间的比较研究[4-5]。
油茶C.oleifera属于山茶科山茶属常绿小乔木或灌木,与油棕、油橄榄、椰子并称为“世界四大木本食用油料植物” [6-7]。福建是油茶的中心产区之一,目前拥有闽43、闽48和闽60等优良无性系,为全省推广的主栽品种,其产油量超过国内优良无性系标准[8]。目前,油茶虽已选育出一批优良无性系或家系良种,但具有产量稳定、抗性强、广适性好等优良性状的品种仍旧不多。当前,油茶在常规育种及优良栽培技术等方面的研究已处于瓶颈期,很难有新的突破。现阶段可以将分子育种技术应用于油茶育种研究[9]。同时,近几年,有关油茶亲缘关系的报道文献迅速增加。刘佳洁[10]对75份油茶优良种质资源材料的亲缘关系进行了分析;蒋莺[11]通过ISSR、SIRP两种分子标记揭示了不同油茶材料间的亲缘关系。但是,关于山茶属物种间的亲缘关系分析的相关报道较少。山茶属植物形状相似,通过形态鉴定十分困难,而植物DNA中的一些序列具有种间差异,因此可以利用山茶属不同物种有特定的DNA序列这一性质作为油茶闽43相似种的鉴定依据。本研究通过分析油茶闽43的叶绿体基因组序列特征及与近缘种的关系,可为普通油茶种群遗传结构提供基础,为研究提高普通油茶的产油量以及优良品种的选育提供理论基础。
1 材料与方法
1.1 试验材料
普通油茶无性系闽43是福建省审定的优良品种。试验材料种植于福建省闽侯白沙国有林场(桐口工区)(119.17°E,26.05°N),采集叶片,-80℃储存在福建农林大学植物标本室备用(FJFC)。
闽43的叶片样品送至华大基因股份有限公司,采用illumina高通量测序方法提取叶绿体基因组,测序仪为MGI2000。闽43叶绿体基因组的序列已经上传至NCBI数据库,并释放数据,获得序列号OP036120.1。
1.2 试验方法
1.2.1 闽43叶绿体基因组组装与注释
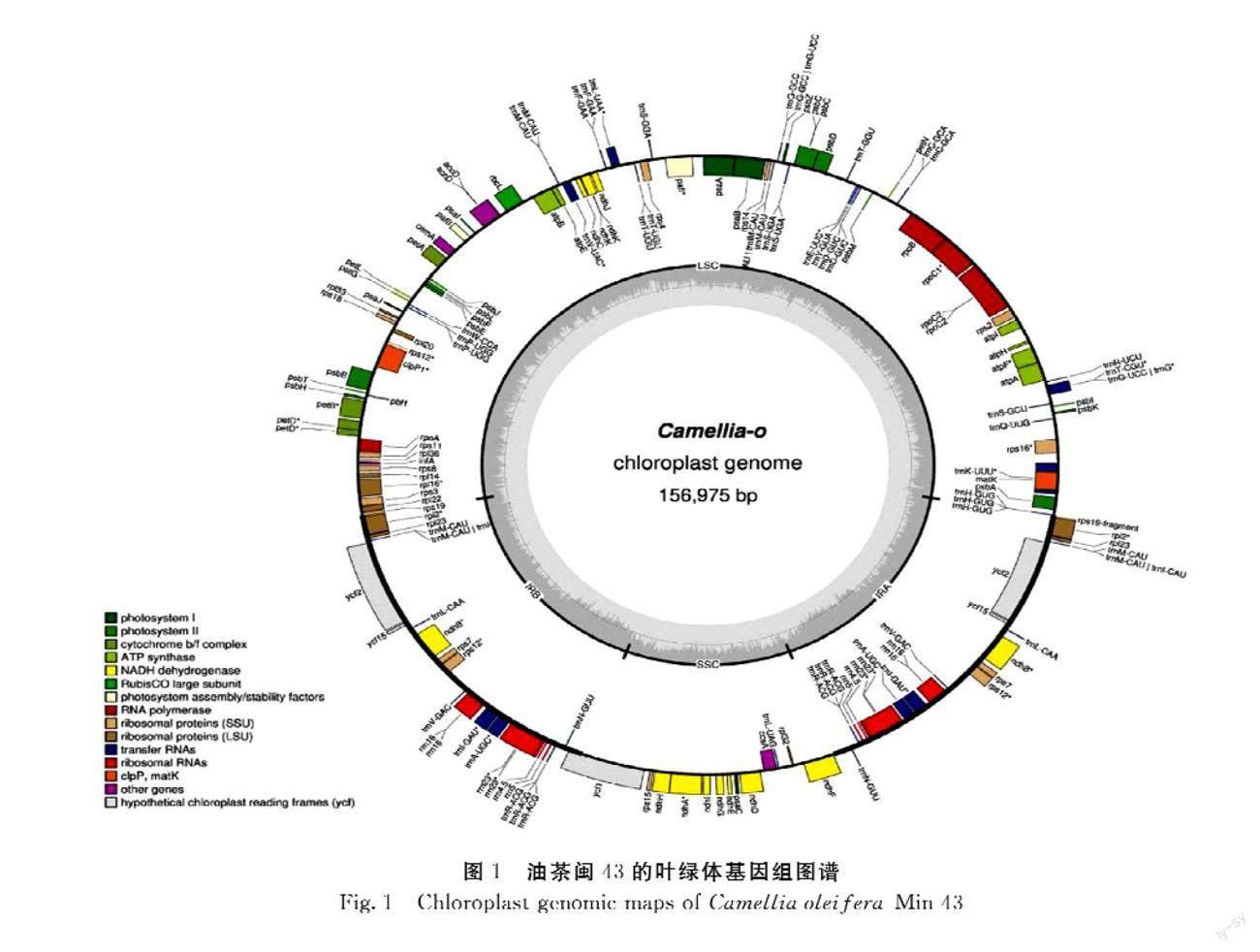
通過GetOrangelle软件对闽43叶绿体基因组进行组装,使用CPGAVAS2软件完成叶绿体基因组的注释工作,并通过Geneious Basic软件对注释后的数据进行校正。再将闽43叶绿体基因组的GenBank格式文件用DRAW Organel Genome Maps(OGDRAW)在线绘图工具绘制出叶绿体基因组物理图谱。
1.2.2 9种山茶属物种叶绿体结构特征比较
从NCBI上下载已公布的8种山茶属物种的叶绿体基因组序列用作结构特征比较分析。其中4种主要分布于广东、福建等地区,分别为:白毛茶Camellia sinensis var.Pubilimba(KJ806280.1)、落瓣油茶Camellia kissii(NC_053915.1)、红皮糙果茶Camellia crapnelliana(KF753632.1)、毛柄连蕊茶Camellia fraterna(NC_050388.1)。另外4种主要分布于广西等地区,分别为:多齿山红茶Camellia polyodonta(NC_060777.1)、金花茶Camellia nitidissima var. Nitidissima(MT157618.1)、龙州金花茶Camellia lungzhouensis(MN579509.2)、南山茶Camellia semiserrata(MT317096.1)。
1.2.3 9种山茶属物种叶绿体长重复序列和微卫星分析
长重复序列有4种类型,即正向(forward)、反向(reverse)、互补 (complement)、回文(palindromic),利用REPuter 软件对9种山茶属物种的长重复序列进行分析,并利用Perl语言在软件 MISA(http://pgrc.ipk~gatersleben.de/misa/)中对叶绿体基因组的SSR(simple~sequence~repeat)序列进行微卫星分析,设置单核苷酸、二核苷酸、三核苷酸、四核苷酸、五核苷酸和六核苷酸的重复参数分别为10、6、5、5、5和5。
1.2.4 5种山茶属物种叶绿体核苷酸多态性分析
采用mVISTA软件以注释过的闽43叶绿体基因组为参考,对4种主要分布于福建、广东等地区的山茶属物种白毛茶、红皮糙果茶、落瓣油茶、毛柄连蕊茶进行叶绿体基因组序列同源性的比较,以及对这5种物种进行核苷酸多态性分析。
1.2.5 5种山茶属物种叶绿体基因组共线性分析
通过Mauve软件对闽43以及4种主要分布于福建、广东等地区的山茶属物种白毛茶、红皮糙果茶、落瓣油茶、毛柄连蕊茶的叶绿体基因组进行共线性比较。
1.2.6 5种山茶属物种IR边界分析
通过JSHYCloud在线工具集分析闽43和4种主要分布于福建、广东等地区的山茶属物种白毛茶、红皮糙果茶、落瓣油茶、毛柄连蕊茶的叶绿体基因组IR区边界结构差异。
1.2.7 9种山茶属物种系统发育树构建
选用9种物种的叶绿体基因组序列,在Geneious中校正这9个目标序列,以木荷属的木荷Schima superba作为外类群,于MAFFT软件中进行序列比对,再进行手工校对。使用软件MEGA11对齐9条叶绿体基因组序列并构建ML系统发育树,Bootstrap support(BS)值设置为1000。
2 结果与分析
2.1 闽43的叶绿体基因组注释结果
由图1可知,闽43叶绿体基因组结构为双链环状结构,具有典型的四分体结构,包括2个反向重复区IRs(inverted repeats)、大的单拷贝区LSC(large single copy)和小的单拷贝区SSC(small single copy)。叶绿体基因组长度为156 975 bp,IR长度为51 908 bp,LSC长度为86 659 bp,SSC长度为18 408 bp。基因组GC含量37.29%。注释基因有132个,其中包括87个蛋白编码基因、37个tRNA基因和8个rRNA基因。
2.2 闽43与8种山茶属物种叶绿体基因组特征比较分析
2.2.1 9种山茶属物种叶绿体结构特征比较
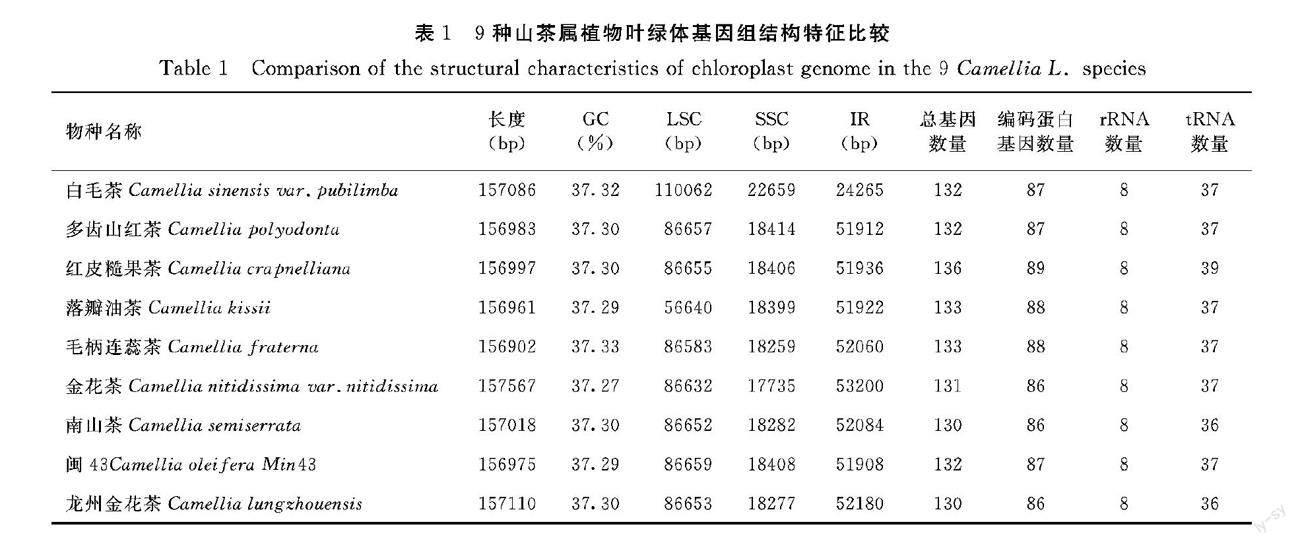
由表1可知,山茶属9种物种的基因组长度为156 902~157 567 bp,LSC、SSC、IR区长度分别为56 640~110 062 、17 735~22 659、24 265~53 200 bp。其中金花茶的叶绿体基因组序列最长,毛柄连蕊茶的最短。且9种物种的GC含量变化不大,差异不超过0.05%,AT含量为62.67%~62.71%,具有明显的AT偏向性。
这9种山茶属物种的基因数量差异较小,数量都在130~136个,其中红皮糙果茶的蛋白编码基因和tRNA最多,分别为89、39个,其余8种物种的蛋白编码基因数量为86~88个,tRNA数量为37个。9种物种的rRNA数量都一样,都为8个,说明这9种山茶属近缘物种叶绿体进化较为保守。
2.2.2 9种山茶属物种叶绿体长重复序列和微卫星分析
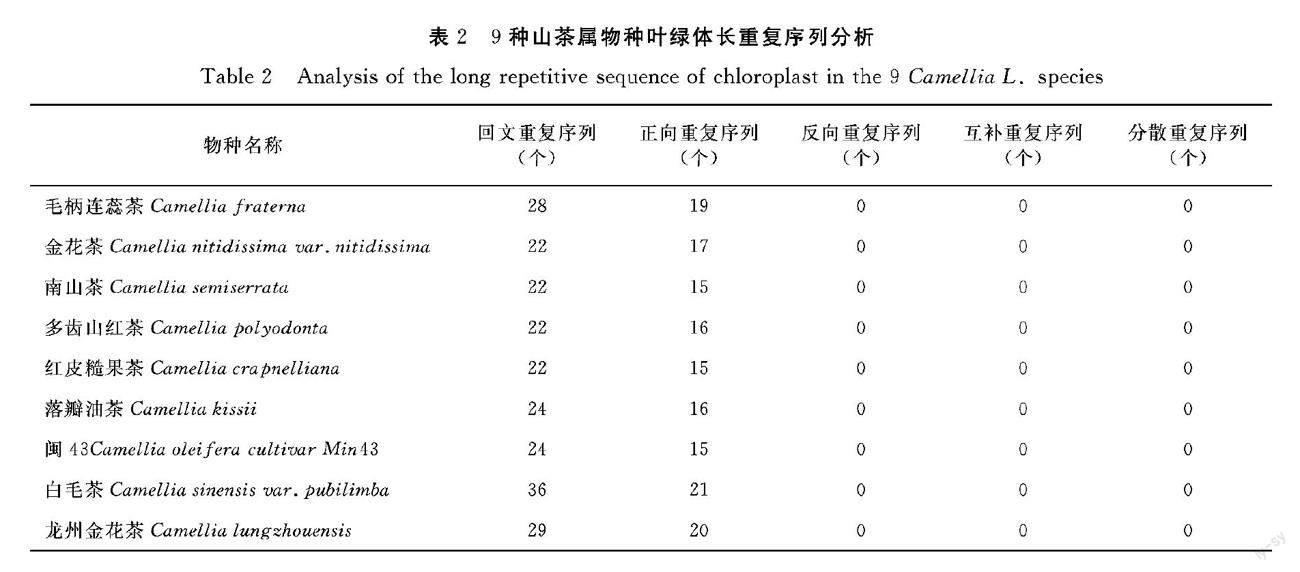
由表2可知,9种山茶属物种长重复序列均只存在正向重复、回文重复,总数为37~57个。其中白毛茶的长重复序列最多,为57个,回文重复有36个,正向重复有21个。
由表3可知,9种山茶属物种的叶绿体基因组中,占比最多的是单核苷酸重复,其次是四核苷酸重复。闽43和落瓣油茶、白毛茶的SSR总数一致,均为65个,并且闽43的微卫星序列情况与落瓣油茶的完全一致,说明油茶闽43 与落瓣油茶这两个物种叶绿体的进化关系可能更为相近。
2.2.3 5种山茶属物种叶绿体核苷酸多态性分析
由图2可知,5种山茶属物种的基因组相似度很高,其中4种物种与闽43中基因有较大差异的是rps16,基因相似度仅在70%左右。同时,闽43与落瓣油茶的psbM基因的相似度极低,仅在50%左右。说明闽43与这4种近缘物种的叶绿体基因组进化程度虽然相似,但是可以通过个别基因的特性,鉴别闽43。
计算以闽43为参考序列的核苷酸多态性值。从图3可知,这5种物种的叶绿体基因组有5个高变区,分别为psbK~atpA、rpoB~psbD、rps4~ndhJ、ycf1、ndhF~rps12~exon1。其中3个热点高变区位于LSC区,2个位于SSC区,说明IR区的变异低于LSC区和SSC区。这些变异较高的区域可用于设计特定的DNA条形码。
2.2.4 5种山茶属物种叶绿体基因组共线性分析
由图4可知,5种山茶属物种叶绿体基因组之间具有高度的相似性,没有发生重排或倒置现象。但在区域(87 000~110 000 bp)中观察到高频变异,其中闽43的基因组序列高度保守。
2.2.5 5种山茶属物种IR边界分析
由图5可知,IR/LSC和IR/SSC边界分布的基因包括rps19、rpl22、rpl2、trnN、ycf1、rps12、ndhF、rpl23、trnH、psbA。JSA(SSC~IRa)区包括基因rps12、trnH、rpl23、psbA。以上分析结果表明,闽43 IR边界基因位置和种类与另外4种近缘物种均有较明显差异。说明不同分布地区同属不同物种的叶绿体基因组IR边界区的基因类型和分布情况存在一定差异,但相对比较保守。
2.2.6 9种山茶属物种系统发育关系分析
由图6可知,9种物种共有7个节点。总体聚为一支,从上到下分别为南山茶、红皮糙果茶、油茶闽43、落瓣油茶、多齿山红茶、白毛茶、金花茶、龙州金花茶、毛柄连蕊茶。在系统发育树中,油茶闽43与落瓣油茶聚为一支,支持率为100,亲缘关系最近。
3 结论与讨论
通过对9种山茶属物种的叶绿体基因组结构的比较发现,闽43的叶绿体基因组序列长度、GC含量以及基因数量与同属的8种物种的差异较小,说明同属物种间的叶绿体基因组相似度较高,这与葉绿体进化相对保守有关。简单重复序列(SSR)在植物叶绿体基因组中分布广泛,信息含量大,具有高多态性,叶绿体基因组SSR以其独特的母系遗传优势被利用于鉴定亲缘关系和系统发育研究中[12-13]。近年来,SSR分子标记在植物系统进化[14]、遗传多样性[15-16]、群体遗传结构[17]及动态进化历史[18]等领域应用广泛。通过微卫星分析发现,9种山茶属物种中,占比最多的是单核苷酸重复,其次是四核苷酸重复,均未检测到五核苷酸的重复类型。
叶绿体基因组是由SSC、LSC、IRa、IRb 4个区间构成的环状结构。其中IR区域相对保守,其收缩和扩张的现象在植物演化过程中起重要作用[19],代表着植物的进化。同时也会引起叶绿体基因的GC含量变化,不同的植物的叶绿体基因组大小也与其有关[20]。根据IR边界分析结果表明,闽43与同分布区的4种属内物种的IR边界基因有较为明显差异,闽43的ycf1基因出现在IRb~SSC(JSB)区,而其余4种属内物种的ycf1基因都仅出现在SSC~IRa(JSA)区,且油茶闽43在IRa~LSC(JLA)和IRb~LSC(JLB)均有与其余4种对比物种不同的基因,说明闽43的IR边界出现了扩张或收缩,可以为物种分类提供良好依据。
叶绿体基因组构建的进化树显示,油茶闽43与落瓣油茶聚为一支,二者亲缘关系最近,但二者的叶绿体基因组长度有明显差异。且通过IR边界分析可以得知,闽43与落瓣油茶在JSA区的基因种类不尽相同,说明二者的叶绿体基因组存在相对较大差异,可以反映出种间差异。且从mVISTA结果来看,闽43和落瓣油茶的psbM基因相似度很低,这可以作为闽43与落瓣油茶之间的分类与鉴定依据。
从核苷酸多样性值结果得出,共有5个高变区,分别为psbK~atpA、rpoB~psbD、rps4~ndhJ、ycf1、ndhF~rps12~exon1。其中3个热点高变区位于LSC区,2个位于SSC区,IR区没有,说明IR区的变异程度低于LSC区和SSC区,这可能与IR区域进化的保守性有关[21]。有一个热点高变区为ycf1,ycf1基因是具有未知功能的开放阅读框[22-23],烟草基因敲除试验证明ycf1基因编码对细胞存活至关重要的产物[24],这可能是油茶闽43的产油量较高[25]的原因。与其他基因相比,ycf1基因進化速度快[26],能够揭示低分类水平上的系统发育关系。因此对于油茶闽43的鉴定,可以通过个别特殊基因进行快速准确的鉴定。
参考文献:
[1]于惠敏,石竹,邵洪伟.植物细胞核与叶绿体基因的双向调控作用[J].生物学通报,2011,46(6):11-14.
[2]于惠敏.植物的叶绿体基因组[J].植物生理学通讯,2001(5):483-488.
[3]原晓龙,刘音,康洪梅,等.蒜头果叶绿体基因组密码子偏好性分析[J].西南林业大学学报(自然科学版),2021,41(3):15-22.
[4]张庆滢,陈璇,郭孟璧,等.野生大麻叶绿体基因组分子多态标记的筛选与开发[J].分子植物育种,2017,15(3):979-985.
[5]NIE X J,LV S Z,ZHANG Y X.Complete chloroplast genome sequence of a major invasive species, crofton weed (Ageratina adenophora)[J].PLoS ONE,2018,7(5):e36869.
[6]马小焕.油茶的研究现状及其应用前景[J].绿色科技,2013,(12):116-117.
[7]庄瑞林.中国油茶(第2版) [M].北京:中国林业出版社,2008.
[8]陈永忠,王湘南,彭邵锋,等.植物生长调节剂对油茶果实含油率的影响[J].中南林业科技大学学报(自然科学版),2007,27(1):25-29.
[9]田仟仟,黄建建,温强,等.油茶分子育种现状与趋势[J].南方林业科学,2021,49(5):53-59,73.
[10]刘佳洁.油茶优良种质资源的收集保存与遗传关系的分子鉴别[D].福州:福建农林大学,2010.
[11]蒋莺.油茶杂交后代及其亲本的遗传关系分析与鉴别选育[D].福州:福建农林大学,2011.
[12]曹瑞兰,胡冬南,周增亮,等.芳樟叶片转录组测序及萜类合成调控相关基因表达分析[J].西南林业大学学报(自然科学),2021,41(5):49-57.
[13]魏潇,章秋平,刘威生,等.基于叶绿体SSR单倍型分析普通杏演化关系[J].植物遗传资源学报,2018,19(4):705-712.
[14]尹航,李冰,张明智,等.短芒大麦草转录组SSR序列特征分析[J].分子植物育种,2021(8):1-9.
[15]李正玲,胡琳,王会伟,等.河南省同名小麦地方品种SSR遗传多样性分析[J].麦类作物学报,2016,36(5):564-570.
[16]黄海燕,杜红岩,乌云塔娜,等.基于杜仲转录组序列的SSR分子标记的开发[J].林业科学,2013,49(5):176-181.
[17]徐金青,夏腾飞,王蕾,等.青稞转录组SSR位点及其基因功能分析[J].麦类作物学报,2017,37(2):175-184.
[18]王恒波,祁舒婷,陈姝琦,等.甘蔗栽培种单倍体基因组SSR位点的发掘与应用[J].作物学报,2020,46(4):631-642.
[19]LI X,LI Y F,ZANG M Y,et al.Complete Chloroplast Genome Sequence and Phylogenetic Analysis of Quercus acutissima[J].International Journal of Molecular Sciences,2018,19(8):2443-2443.
[20]LIU Y.Thirteen Camellia chloroplast genome sequences determined by high-throughput sequencing:Genome structure and phylogenetic relationships[J].BMC Evolutionary Biology,2014,14:151.
[21]沈立群.唇形科三种药用植物叶绿体全基因组及科内的比较与进化分析[D].杭州:浙江大学,2018.
[22]RAINER M M,KAI N,GABOR L I,et al.Complete Sequence of the Maize Chloroplast Genome:Gene Content,Hotspots of Divergence and Fine Tuning of Genetic Information by Transcript Editing[J].Journal of Molecular Biology,1995,251(5):614-628.
[23]DOUGLAS S E,PENNY S L.The plastid genome of the cryptophyte alga,Guillardia theta: complete sequence and conserved synteny groups confirm its common ancestry with red algae[J].Journal of Molecular Evolution,1999,48(2),236-244.
[24]DRESCHER A,RUF S,CALSA T,et al.The two largest chloroplast genome-encoded open reading frames of higher plants are essential genes[J].The Plant Journal,2000,22(2):97-104.
[25]熊年康,任恢康,陈祥平.油茶闽43、闽48、闽60三个优良无性系的选育[J].福建林业科技,1986,1(1):1-6.
[26]吴劲松,张宇思,刘薇,等.白及属药用植物DNA条形码的确立及其应用[J].药学学报,2014,49(10):1466-1474.
(责任编辑:柯文辉)
