基于关系图卷积网络的长链非编码RNA与疾病关联预测
2023-06-25杜晓昕罗金琦金梅王振飞周薇
杜晓昕 罗金琦 金梅 王振飞 周薇

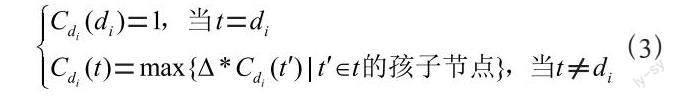


摘 要:针对当前长链非编码RNA(lncRNA)与疾病关联预测研究中存在的异质网络构建不完善、网络节点信息挖掘不充分问题,提出一种基于关系图卷积网络(Relational Graph Convolutional Network, R-GCN)的方法(RGCNLDA)。首先,构建lncRNA-miRNA-疾病异质图,随后在异质图上训练R-GCN获取节点嵌入向量,最后使用多层感知机预测lncRNA-疾病关联。5折交叉验证结果显示,RGCNLDA的受试者工作特征曲线下面积(AUROC)为0.934,表明其具有良好的预测性能。
关键词:lncRNA;关系图卷积网络;异质图;关联预测
中图分类号:TP311 文献标识码:A 文章编号:2096-4706(2023)07-0086-04
Abstract: Aiming at the problems of imperfect heterogeneous network construction and insufficient network node information mining in the current research on association prediction of long non-coding RNA (lncRNA) and disease, a method based on Relational Graph Convolutional Network (R-GCN) is proposed (RGCNLDA). Firstly, a lncRNA-miRNA-disease heterogeneous graph is constructed, and then R-GCN is trained on the heterogeneous graph to obtain node embedding vectors. Finally, a multi-layer perceptron is used to get lncRNA-disease associations. The results of 5-fold cross validation show that the Area Under Receiver Operating Characteristic curve (AUROC) of RGCNLDA is 0.934, indicating a good predictive performance.
Keywords: lncRNA; R-GCN; heterogeneous graph; association prediction
0 引 言
長链非编码RNA(lncRNA)是一种含有200多个核苷酸的非编码RNA[1]。越来越多的研究表明,lncRNA的突变和调控异常与各种复杂人类疾病的发生和发展密切相关,如糖尿病、心血管疾病、神经系统疾病和包括肺癌、乳腺癌和前列腺癌在内的癌症[2]。因此,急需开发高效、准确的计算模型来预测潜在的lncRNA-疾病关联。
预测lncRNA-疾病关联的方法大致可以分为两类:基于机器学习的方法和基于网络随机游走的方法。在基于机器学习的方法中,Chen等人[3]提出了一种称为LRLSLDA的半监督学习方法,该方法使用拉普拉斯正则化最小二乘来识别lncRNA与疾病之间的潜在关联。近年来兴起的深度学习技术也被广泛应用于lncRNA-疾病关联预测中,如Yang等人[4]提出了一个名为BiGAN的双向生成对抗网络模型,由一个编码器、一个生成器和一个鉴别器组成,通过编码器和生成器学习高级特征,并通过鉴别器预测lncRNA-疾病关联。在基于网络随机游走的方法中,Wang等人[5]构建与疾病关联的lncRNA功能网络,进行重启随机游走(Random Walk with Restart, RWR),从而预测与疾病关联的lncRNA;Hu等人[6]提出一种BiWalkLDA方法,在lncRNA-疾病网络上进行双随机游走,整合相互作用谱和基因本体信息,预测lncRNA-疾病关联。
上述方法虽然在lncRNA-疾病关联预测中取得了一定的成果,但也存在一些弊端。例如,没有充分考虑到多源数据整合对于提取节点信息的重要性,着重于提取节点的线性特征而忽略了节点在网络中的拓扑结构信息。因此,本文提出一种基于关系图卷积网络的方法RGCNLDA,整合lncRNA、miRNA以及疾病信息构建异质图,并根据节点与节点之间的不同关系分别提取节点的非线性特征,从而充分挖掘节点信息,提高预测性能。
1 相关概念
1.1 异质图
异质图是指图中节点类型与边类型之和大于1的图。将一个无向异质图定义为G=(V,E,R)。其中,V表示节点集合,任意节点vi ∈ V。E表示边的集合,节点vi与vj之间的边(vi,r,vj) ∈ E。r表示节点vi与vj之间的关系,r ∈ R。
1.2 关系图卷积网络R-GCN
图卷积网络(Graph Convolutional Network, GCN)只能作用于同质图,即图中只有一种类型的节点或边。对于具有多种类型的节点和边的图,GCN将节点和边都视作同一种类型,无法挖掘不同节点类型和边类型的节点之间的信息。关系图卷积网络(R-GCN)能够克服这种弊端。R-GCN根据节点之间的不同关系,分别聚合邻居节点信息。
R-GCN通过式(1)计算节点vi在第l+1层神经网络上的向量表示:
其中,W表示权重矩阵,cir表示归一化常数,Nir表示与节点vi具有r关系的邻居节点的集合。
1.3 链接预测
异质图上的链接预测是指判断给定类型的节点之间是否存在边。计算两两节点之间存在链接可能性的得分如式(2)所示:
其中,hi(k)与hj(k)表示节点vi与vj经过多层图神经网络的向量表示,ϕ表示计算节点vi与vj之间链接可能性得分的函数。
2 基于R-GCN的lncRNA-疾病关联预测
2.1 LMD异质图构建
构建LncRNA-MiRNA-Disease异质图,简称LMD异质图。LMD异质图由lncRNA、miRNA和疾病三种类型的节点和描述这三种类型节点之间相似关系、关联关系和相互作用关系的六种类型的边组成。
2.1.1 数据准备
为构建LMD异质图,从LncRNADisease v2.0数据库[7]和Lnc2Cancer v3.0数据库[8]下载了实验证实的lncRNA-疾病关联数据。将所有疾病名称转换为标准MESH疾病术语,过滤重复数据,删除只有一个或没有关联的lncRNA以避免可能的噪声。已知的lncRNA-miRNA關联数据来自Encori数据库[9]和NPInter v4.0数据库[10]。类似地,从lncRNA-miRNA关联中消除冗余数据,仅保留lncRNA -疾病关联数据中的lncRNA和miRNA-疾病关联数据中的miRNA。从HMDD v3.2数据库[11]中获得了miRNA-疾病关联数据,筛选出了在生物学研究中更有价值的与疾病有因果关系的miRNA。
2.1.2 相似性计算
构建LMD异质图时,计算了相同类型节点之间的相似性分数,从而确定相同类型节点在LMD异质图中是否有边。首先,计算疾病节点之间的语义相似性。根据Wang等人[12]提出的方法,任何疾病都可以用唯一的有向无环图(DAG)来表示。DAG中的节点代表疾病术语,具有层次关系。通过计算疾病术语的语义值,以及两个DAG中常见术语的语义值,可以计算疾病节点之间的相似性分数。计算疾病术语t对疾病A的贡献值如式(3)所示:
其中,Δ表示语义衰减因子,表明在疾病di的DAG图中,当疾病di的某个祖先疾病节点距离di越远时,其对di的语义贡献值越小。随后,由式(4)计算出疾病di的语义贡献值,式(5)根据疾病di和疾病dj的DAG图,计算出di与dj的语义相似性:
对于疾病di与dj,如果通过上述方法计算出的语义相似度大于0,则在LMD异质图中为它们添加一条边。
基于功能相似的lncRNA通常与相似的疾病相关的理论,Chen等人[13]计算了lncRNA之间的功能相似性。将D(li)和D(lj)分别定义为与lncRNAli和lncRNAlj相互关联的疾病组,计算D(li)和D(lj)之间的相似性,作为lncRNAli和lncRNAlj之间的功能相似性。假设疾病d与li关联,则d与疾病组D(lj)之间的相似性计算如式(6)所示,由此,lncRNAli和lncRNAlj之间的功能相似性计算如式(7)所示:
如果两两lncRNA的相似性分数大于0,则在LMD异质图中添加一条li与lj之间的边。
按照上述类似的方法,可以计算出两两miRNA之间的功能相似性。假设miRNAmi与miRNAmj分别与疾病组D(mi)、D(mj)关联,则mi与mj的功能相似性可由式(8)和式(9)计算得到:
类似地,如果两个miRNA之间的相似性大于0,则在LMD异质图添加一条(mi,mj)无向边。
2.1.3 数据整合
整合从数据库中下载的已知lncRNA-疾病关联、lncRNA-miRNA相互作用、miRNA-疾病关联,以及计算出的lncRNA-lncRNA相似性、miRNA-miRNA相似性、疾病-疾病相似性,构建LMD异质图。图中各类型节点的数量如表1所示,各类型的边的数量如表2所示。
2.2 LMD异质图节点特征获取
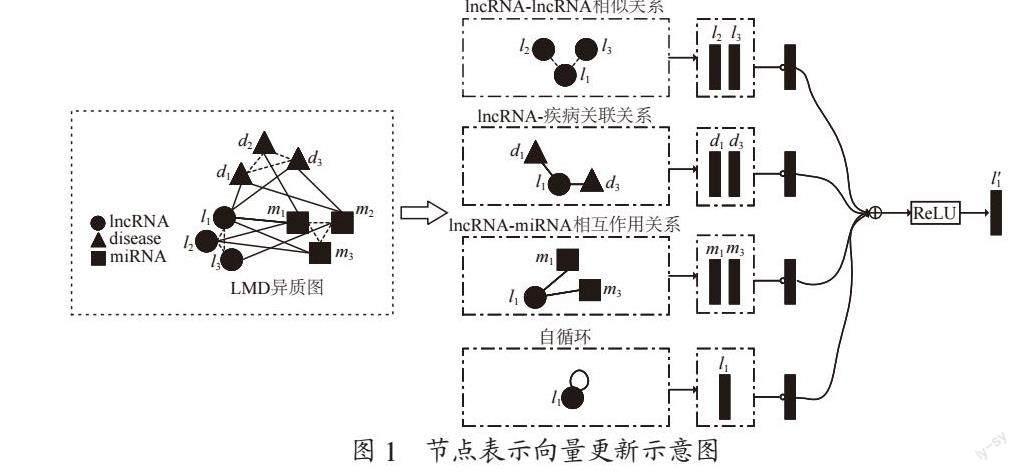
将构建的LMD异质图输入利用R-GCN模型,从而获取的节点表示向量。如图1所示,以节点lncRNAl1为例,根据lncRNA-lncRNA相似关系,聚合节点l2和l3的表示向量;根据lncRNA-疾病关联关系,聚合节点d2和d3的表示向量;根据lncRNA-miRNA的相互作用关系,聚合节点m2和m3的表示向量;最后,将节点自连接作为一种特殊的关系,整合l1自身的表示向量。将上述l1的邻居节点以及l1自身的表示向量经过激活函数得到更新后的表示向量。同理,在LMD异质图上更新其他lncRNA节点、miRNA节点、疾病节点的表示向量。
2.3 lncRNA-疾病关联预测
将R-GCN获取的LMD异质图中的lncRNA节点、疾病节点拼接形成lncRNA-疾病节点对向量;同时,根据已知的lncRNA-疾病关联进行负采样(如:随机采样k个不与lncRN l1关联的疾病节点),将负采样得到的节点绘制成子图,由R-GCN获取节点表示向量后,拼接形成lncRNA-疾病节点对向量。分别将正负样本的lncRNA-疾病节点对向量输入多层感知机,得到lncRNA-疾病关联的得分。
3 实验结果与分析
3.1 评价指标与评估方法
以受试者工作特征曲线下面积(Area Under Receiver Operating Characteristic curve, AUROC)和PR曲线下面积(Area Under Precision–Recall curve, AUPR)作为评估指标,采用五折交叉验证方法对RGCNLDA模型的性能进行评估。将样本集平均分为5份,依次选取一份样本集用于验证,剩余4份样本集用于训练,取5次实验的平均结果作为该分类器的性能指标。
3.2 參数设置
本文基于PyTorch框架实现RGCNLDA模型,训练节点维度为64维,使用Adam优化器,R-GCN网络的层数为2层,负采样节点数为5,学习率为0.001,训练150轮次。
3.3 对比实验
将RGCNLDA模型与其他3种预测lncRNA-疾病关联的先进模型MFLDA[14]、TPGLDA[15]、SDLDA[16]在同一数据集上进行对比实验,五折交叉验证下的ROC曲线和AUC值如图2所示。
由图2可知,RGCNLDA模型在4种方法中具有最优的预测性能。五折交叉验证下的平均AUC值对比TPGLDA模型、MFLDA模型、SDLDA模型分别提升了7.11%、16.31%、5.06%。MFLDA模型和TPGLDA模型虽然整合了异构生物源的数据,但没有充分挖掘lncRNA节点和疾病节点的非线性特征;SDLDA模型虽然利用了矩阵分解和深度学习提取节点非线性特征,但没有考虑到多源数据整合。由此可见,RGCNLDA模型具有良好的预测性能。
4 结 论
研究表明,lncRNA在疾病的产生和发展过程中发挥着至关重要的作用,因此,设计高效的预测模型研究潜在的lncRNA-疾病关联有助于理解疾病的产生原理并辅助医护人员有针对性地开展疾病预防和治疗工作。本文提出RGCNLDA模型,首先整合lncRNA、疾病、miRNA多源数据构建异质图,并根据节点类型不同以及节点之间的不同关系训练R-GCN网络,充分挖掘异质图中节点特征,最后使用多层感知机为lncRNA-疾病节点对进行打分,得到潜在的lncRNA-疾病关联。五折交叉验证的AUC值表明本模型具有良好的预测性能。
参考文献:
[1] TAFT R J,PANG K C,MERCER T R, et al. Non-coding RNAs: regulators of disease [J]. J Pathol,2010,220(2):126-139.
[2] JOHNSON R. Long non-coding RNAs in Huntington's disease neurodegeneration [J] Neurobiol Dis,2012,46:245-254.
[3] CHEN X,YAN G Y. Novel human lncRNA-disease association inference based on lncRNA expression profiles [J].Bioinformatics,2013,29(20):2617-2624.
[4] YANG Q,LI X K. BiGAN: LncRNA-disease association prediction based on bidirectional generative adversarial network [J/OL].BMC Bioinformatics,2021,22[2022-11-26].https://bmcbioinformatics.biomedcentral.com/articles/10.1186/s12859-021-04273-7.
[5] WANG Y T,JUAN L R,PENG J J,et al. LncDisAP: a computation model for LncRNA-disease association prediction based on multiple biological datasets [J/OL].BMC Bioinformatics,2019,20[2022-11-22].https://bmcbioinformatics.biomedcentral.com/articles/10.1186/s12859-019-3081-1.
[6] HU J L,GAO Y Q,LI J, et al. A novel algorithm based on bi-random walks to identify disease-related lncRNAs [J/OL].BMC Bioinformatics,2019,20[2022-11-22].https://pubmed.ncbi.nlm.nih.gov/31760932/.
[7] BAO Z Y,YANG Z,HUANG Z,et al. LncRNADisease 2.0: an updated database of long non-coding RNA-associated disease [J].Nucleic Acids Res,2019,47(D1):D1034-D1037.
[8] GAO Y,SHANG S P,GUO S,et al. Lnc2Cancer 3.0: an updated resource for experimentally supported lncRNA/circRNA cancer associations and web tools based on RNA-seq and scRNA-seq data[J]. Nucleic Acids Res,2021,49(D1):D1251-D1258.
[9] LI J H,LIU S,ZHOU H,et al. starBase v2.0: decoding miRNA-ceRNA, miRNA-ncRNA and protein-RNA interaction networks from large-scale CLIP-Seq data [J]. Nucleic Acids Res,2014,42(Database issue):D92-D97.
[10] TENG X Y,CHEN X M,XUE H,et al. NPInter v4.0: an integrated database of ncRNA interactions [J].Nucleic Acids Res,2020,48(D1):D160–D165.
[11] HUANG Z,SHI J C,GAO Y X,et al. HMDD v3.0: a database for experimentally supported human microRNA-disease associations [J].Nucleic Acids Res,2019,47(D1):D1013-D1017.
[12] WANG D,WANG J,LU M,et al. Inferring the human microRNA functional similarity and functional network based on microRNA-associated diseases [J].Bioinformatics,2010,26:1644-1650.
[13] CHEN X,YAN C C,LUO C,et al. Constructing lncRNA functional similarity network based on lncRNA-disease associations and disease semantic similarity [J/OL]. Scientific Reports, 2015, 5[2022-11-22].https://www.nature.com/articles/srep11338.
[14] Fu GY, Wang J, LUO C, et al. Matrix factorization-based data fusion for the prediction of lncRNA–disease associations [J]. Bioinformatics,2018,34(9):1529-1537.
[15] FU G Y,WANG J,LUO C,et al. TPGLDA: Novel prediction of associations between lncRNAs and diseases via lncRNA-disease-gene tripartite graph [J].Scientific Reports,2018,8(1):1-11.
[16] ZENG M,LU C Q,ZHANG F H,et al. SDLDA: lncRNA-disease association prediction based on singular value decomposition and deep learning [J].Methods,2020,179:73-80.
作者簡介:杜晓昕(1983—),女,汉族,江苏徐州人,教授,硕士研究生,研究方向:生物医学大数据分析与处理;罗金琦(1997—),女,汉族,四川绵阳人,硕士在读,研究方向:临床医学大数据挖掘;金梅(1977—),女,汉族,辽宁鞍山人,讲师,硕士研究生,研究方向:机器学习;王振飞(1999—),男,汉族,山东省潍坊人,硕士在读,研究方向:机器学习与群智能优化算法;周薇(1999—),女,汉族,河北定州人,硕士在读,研究方向:群智能优化算法。
