基于绿豆发状根的快速CRISPR/Cas9基因编辑方法
2023-06-17闫强胡亚群薛冬周琰琰丁佩韦雅雯袁星星陈新
闫强 胡亚群 薛冬 周琰琰 丁佩 韦雅雯 袁星星 陈新


摘要:由于缺乏有效稳定的遗传转化体系,导致基因编辑技术在绿豆中的应用受到极大限制,也使绿豆基因功能研究受到极大阻碍。因此,建立一套基于发根农杆菌的操作简便、快速且高效的绿豆嵌合植株转化技术,可以为绿豆基因功能研究提供技术支撑。首先在CRISPR/Cas9载体骨架中插入1个绿色荧光蛋白(greenfluorescentprotein,GFP)表达框用于阳性发状根的快速筛选,然后将含有靶标基因gRNA的CRISPR/Cas9质粒导入发根农杆菌K599。菌液注射侵染绿豆幼苗植株下胚轴,评价K599在绿豆中诱导发状根发生及CRISPR/Cas9系统在绿豆转基因发状根组织中的效率。结果显示,K599菌液侵染种植7d的绿豆幼苗植株并在保湿条件下培养3周可在侵染点诱导发状根产生,此嵌合植株在切除原生根后可在霍格兰培养液中正常生长且培养1周后即可用于后续检测。用GFP筛选的结果显示,阳性发状根占比约为(57.8±10.0)%。随机选择15个荧光检测阳性的转基因发状根组织,利用测序检测靶位点的编辑效果,结果显示有10个靶位点的序列发生了突变,突变比例达到67%,其中碱基缺失突变9个,碱基转换突变1个。由本研究结果可知,利用该方法可以在无菌组织培养的条件下于4周内获得目的基因编辑的转基因组织,极大地便利了绿豆分子水平上的功能研究,也为其他尚缺乏稳定遗传转化体系的作物进行基因功能研究提供了参考。
关键词:绿豆;CRISPR/Cas9;基因编辑;發状根转化;遗传转化;绿色荧光蛋白
中图分类号:S522.01文献标志码:A
文章编号:1002-1302(2023)10-0048-06
绿豆是我国传统的杂粮作物,其种子含有丰富的人体易消化蛋白、纤维、维生素B和多种矿物微量元素等营养成分,同时还具有清热解毒、消炎杀菌等药理作用,是一种备受消费者喜欢的药食同源类食用豆类作物[1-2]。绿豆由于其生育期短(60~75d)、抗逆性强及共生固氮的能力,是轮作和救灾备荒的优良作物。近年来,随着我国农业供给侧结构调整和人民生活水平的不断提高,绿豆在满足人们日益增长的健康多元化饮食的需求及精准扶贫、产业结构调整、乡村振兴中发挥着重要作用。
基于序列特异性核酸酶的基因组编辑技术在过去10年内发展迅速,并彻底改变了生命科学研究的各个领域[3]。与早期开发的锌指核酸酶(ZFNs)技术和类转录因子效应物核酸酶(TALENs)技术相比,规律成簇的间隔短回文重复相关蛋白技术(CRISPR/Cas9)利用1个向导RNA(gRNA)对靶基因位点进行特异性识别,并在Cas9蛋白作用下对靶点DNA进行切割,断裂的DNA双链通过非同源末端连接、同源重组方式进行修饰,在此过程中实现对靶点序列的编辑[4]。CRISPR/Cas9系统由于载体构建简单、编辑效率高等优点,已被作为一种便捷有效的工具而广泛应用,目前在几十种模式植物和作物中的应用均有相关报道[5-7]。在豆科植物中,CRISPR/Cas9系统已经成功用于百脉根、大豆、苜蓿、豇豆的基因编辑[8-11]。但在绿豆中,CRISPR/Cas9介导的基因编辑目前尚没有相关报道。
目前,主流方案是利用农杆菌将包含CRISPR/Cas9元件的质粒通过遗传转化的方法导入宿主细胞[3]。虽然已有研究发现,用绿豆的初生叶片、下胚轴等作为外植体成功获得了转基因株系,并对侵染条件、培养基激素配方进行了积极探索[12-14],但是这些报道的方法仍存在效率低、可重复性差的问题,绿豆的遗传转化在国内尚没有成功的报道。由此可见,缺乏成熟高效的遗传转化方法是限制CRISPR/Cas9基因编辑技术在绿豆中应用的关键限制因素。
为了建立一套快速便捷的绿豆基因编辑技术体系,本研究拟探索发根农杆菌介导的嵌合植株转化技术在绿豆中的适用性,并对CRISPR/Cas9系统在转基因发状根组织中的编辑效率进行评价,从而为绿豆基因功能研究提供技术支撑。
1材料与方法
1.1试验材料
苏绿1号绿豆种植在装有蛭石和营养土混合物(体积比为2∶1)的小花盆中,浇水后放置在温室中培养,培养条件设置为光—暗周期16h—8h,温度为26℃。种植第3天浇水,培养7d待真叶刚刚展开时用于农杆菌侵染。
本氏烟草(Nicotianabenthamiana)育苗后移栽到上述同样营养土中,并在相同条件下培养5~6周用于外源基因的瞬时表达。试验材料在2021年3—7月分批播种于江苏省农业科学院植物智能生长室内。
1.2载体构建
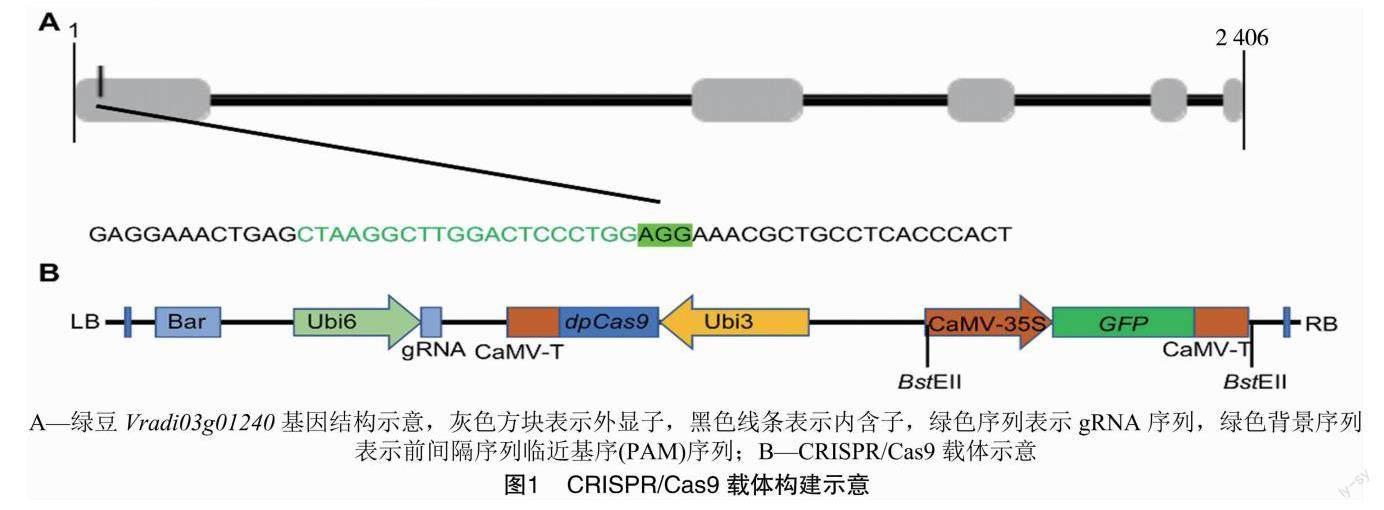
利用在线软件CRISPR(http://crispor.tefor.net/crispor.py)在Vradi03g01240基因第1个外显子区域选择gRNA。首先将gRNA及其反向互补序列分别融合在引物U6-gRNA-F、U6-gRNA-R的5′端,并在引物pSCM-U6-F、pSCM-U6-R的5′端分别融合质粒pSCNcoⅠ上游、XbaⅠ下游同源序列。然后以pGmU6质粒为模板,用pSC-U6-F/U6-gRNA-R、U6-gRNA-F/pSC-U6-R2对引物搭配扩增,获得大小分别为500、200bp的片段,最后利用诺维赞多片段重组试剂盒将片段与NcoⅠ、XbaⅠ双酶切线性化的pSC质粒重组,获得载体pSC-gRNA。为便于阳性发状根的筛选,以pBIN-GFP4载体为模板,用引物GFP-F/GFP-R扩增包含CaMV35S启动子、绿色荧光蛋白(greenfluorescentprotein,GFP)阅读框、终止子的完整表达框,并通过同源重组的方法分别插入pSC-gRNA的BstEⅡ位点处,至此完成载体pSC-GFP-gRNA、pSC-GFP的构建,其中pSC-GFP作为空载体对照。载体构建过程中所用的引物序列见表1。
1.3绿豆发状根的转化
将质粒pSC-GFP-gRNA、pSC-GFP通过冻融法转入发根农杆菌K599感受态细胞中,利用引物pSC-seq-F/RPCR扩增验证阳性克隆。将阳性克隆在含有50mg/L卡那霉素的LB液体培养基中于28℃、200r/min培养36h;待菌液浑浊后,吸取200μL菌液均匀涂布在加有卡那霉素的LB平板上并在28℃培养箱中过夜培养;第2天用涂布棒收集菌体至10mmol/LMgCl2溶液(含有40mg/L乙酰丁香酮)中,并调节D600nm至0.5~0.6。
绿豆幼苗培养7d时,待真叶完全展开后用于发根农杆菌侵染。首先用手术刀在子叶节处往下划1个长约1cm的伤口,然后用移液器往伤口处滴加适量侵染菌液。将侵染处理完毕的植株放置于用透明塑料布做成的密封罩中,并保持罩内湿度在90%以上。每日检查罩内湿度情况直至发状根长出2~5cm,从侵染伤口处往下约1cm处用剪刀将植株茎剪断,将剪下的植株放置于霍格兰营养液中继续培养。此时得到的嵌合植株由非转基因的茎、叶片及含有部分转基因发状根组织组成。待根长至5~7cm时,在宏观荧光变倍显微镜下进行荧光筛选,并用镊子小心去除无GFP的根,然后将此嵌合植株放回培养基中继续培养用于后续试验。
1.4DNA的提取和编辑位点的序列分析
利用基因组提取试剂盒[天根生化科技(北京)有限公司]提取转pSC-GFP-gRNA质粒发状根组织的基因组。用特異性引物Test-F/R扩增包含gRNA在内的附近基因组片段,将获得的片段电泳验证后送擎科生物技术有限公司进行测序。获得相应序列后利用ClustalX软件进行多序列比对并利用GeneDoc进行可视化处理。
1.5烟草叶片的瞬时表达和荧光观察
将质粒pSC-GFP-gRNA、pSC-GFP通过冻融法转入根癌农杆菌GV3101感受态细胞中,经菌落PCR验证后在含有50mg/L卡那霉素、30mg/L利福平的液体LB培养基中于28℃、200r/min条件下培养48h;在4000r/min离心5min收集菌体,用10mmol/LMgCl2溶液(含有40mg/L乙酰丁香酮)清洗3遍后调节D600nm至0.5~0.6。用1mL注射器将侵染液注射进烟草叶片中,将处理过的植物放回培养室继续培养48h后用于荧光观察。切取注射区域烟草叶片置于共聚焦显微镜下,在488nm波长激发光下观察荧光并拍照。
2结果与分析
2.1GFP标记的绿豆基因CRISPR/Cas9载体的构建
用在线软件CRISPR在Vradi03g01240基因第1个外显子区域49~68个核苷酸处设计gRNA(图1-A),将gRNA片段插入载体pSC-IAA14上并进一步完成pSC-GFP-IAA14载体的构建,此载体由大豆GmU6启动子驱动gRNA表达,同时含有1个由大豆GmUbi3启动子驱动的dpCas9表达框及用于荧光筛选的CaMV-35S驱动的GFP表达框(图1-B)。
为了验证插入CRISPR载体中的GFP表达框是否能够正常表达,用烟草瞬时表达系统对GFP信号进行检测。结果显示,将pSC-GFP-gRNA、pSC-GFP在烟草叶片中瞬时表达后,均能够观察到强烈的荧光信号(图2),说明改造的载体中的GFP能够在植物体内正常表达。
2.2绿豆嵌合植株的获得
从图3-A至图3-I可以看出,用含有pSC-GFP-gRNA载体的发根农杆菌K599在绿豆幼苗子叶节处进行侵染,结果显示,诱导产生了发状根组织。保湿处理1周后,侵染点组织变得明显膨大(图3-E);保湿处理2周后,侵染位点继续膨大并形成瘤状的愈伤组织(图3-F);保湿处理3周后,侵染点处陆续有发整根形成(图3-G);待大部分根长至2~5cm时,从侵染伤口处往下约1cm处用剪刀将植株茎剪断,剪下的植株置于霍格兰营养液
中继续培养(图3-H)。待根长至5~7cm时(图3-I),在宏观荧光变倍显微镜下进行荧光筛选,并用镊子小心去除无GFP的根(图4)。经统计,发出绿色荧光的发状根比例约为(57.8±10.0)%,然后将处理完毕的植株放回培养基中继续培养,进行进一步的检测,此植株具有野生型的茎、叶器官,发状根组织为转基因部分。
2.3转基因发状根组织基因编辑效果检测
为了检测获得的嵌合植株发状根组织中Vradi03g01240基因是否被成功编辑。随机选择15个荧光检测阳性的转化pSC-GFP-gRNA发状根组织提取基因组DNA。用特异性引物Test-F/R扩增gRNA附近序列,结果显示,对照和敲除的转基因组织均扩增出了符合预期大小为706bp的条带(图5)。对条带进行测序的结果显示,15个独立的转基因事件中,有10个gRNA附近序列发生了突变,突变比例约为67%(图6)。突变类型表现如下:小片段的碱基缺失9个,占突变总数的90%;碱基转换突变1个,占突变总数的10%。以上结果表明,本研究中使用的CRISPR/Cas9系统可以对绿豆基因进行有效的编辑,创制多样性的突变类型。
3讨论与结论
CRISPR/Cas9基因编辑技术是目前应用广泛的基因编辑技术,为基因功能研究和作物改良提供了大量突变材料[15-17]。本研究在前人基础上,将应用于大豆基因编辑的遗传转化载体进行了进一步优化[18],添加了GFP筛选标记,以便后续的阳性转基因材料筛选鉴定。本研究以绿豆Vradi03g01240基因为目的基因进行基因编辑载体的构建,通过对荧光筛选过的发状根组织进行测序分析,发现CRISPR/Cas9基因编辑的效率达到67%,其中90%的靶标基因gRNA区域发生了缺失突变。本研究结果充分证明了该CRISPR/Cas9系统在绿豆发状根
系统中的适用性。
基于发根农杆菌介导的根毛转化体系目前已被广泛用于难以转化或尚缺乏有效稳定转化技术手段的植物进行基因功能验证[19]。发根农杆菌K599介导的发状根转化技术在豆科作物大豆、菜豆、豇豆中都得到了成功的利用[11,20-21]。发状根瞬时转化技术能够对候选基因进行快速的功能验证,本系统在4周内即可获得用于试验的转基因材料,而且荧光筛选结果表明阳性转基因发状根比例达到50%以上。稳定转化虽然具有更加稳定的遗传背景和一致的表型,但往往需要一年甚至更久才能拿到转基因材料,尤其目前绿豆尚缺乏行之有效的稳定转化技术体系。本研究证明了CRISPR/Cas9系统能够在绿豆体内进行有效的基因编辑,创制大量的目的基因突变类型,发状根转化技术目前可作为研究绿豆基因功能的一个优异替代方案。本研究的方案能够大大提高绿豆根系发育、根瘤形成、盐碱非生物胁迫以及根部病虫害等相关分子机制研究的速度和质量。
致谢:感谢南京农业大学喻徳跃教授惠赠pGmU6、pSC基因编辑载体,南京农业大学窦道龙教授惠赠pBIN-GFP4载体。
参考文献:
[1]程须珍.绿豆生产技术[M].北京:北京教育出版社,2016:11-12.
[2]KeatingeJDH,EasdownWJ,YangRY,etal.Overcomingchronicmalnutritioninafuturewarmingworld:thekeyimportanceofmungbeanandvegetablesoybean[J].Euphytica,2011,180(1):129-141.
[3]刘耀光,李构思,张雅玲,等.CRISPR/Cas植物基因组编辑技术研究进展[J].华南农业大学学报,2019,40(5):46-57.
[4]XieKB,YangYN.RNA-guidedgenomeeditinginplantsusingaCRISPR-Cassystem[J].MolecularPlant,2013,6(6):1975-1983.
[5]DemirciY,ZhangBH,UnverT.CRISPR/Cas9:anRNA-guidedhighlyprecisesynthetictoolforplantgenomeediting[J].JournalofCellularPhysiology,2018,233(3):1844-1859.
[6]ShahT,AndleebT,LateefS,etal.Genomeeditinginplants:advancingcroptransformationandoverviewoftools[J].PlantPhysiologyandBiochemistry,2018,131:12-21.
[7]张瑞,高彩霞.基于双碱基编辑系统的植物基因靶向随机突变技术[J].植物学报,2021,56(1):50-55.
[8]LiZS,LiuZB,XingAQ,etal.Cas9-guideRNAdirectedgenomeeditinginsoybean[J].PlantPhysiology,2015,169(2):960-970.
[9]WangLX,WangLL,TanQ,etal.EfficientinactivationofsymbioticnitrogenfixationrelatedgenesinLotusjaponicususingCRISPR-Cas9[J].FrontiersinPlantScience,2016,7:1333.
[10]JiJ,ZhangCY,SunZF,etal.GenomeeditingincowpeaVignaunguiculatausingCRISPR-Cas9[J].InternationalJournalofMolecularSciences,2019,20(10):2471.
[11]CaasLA,BeltránJP.FunctionalgenomicsinMedicagotruncatula[M]NewYork:HumanaPress,2018,1822:161-174.
[12]JaiwalPK,KumariR,IgnacimuthuS,etal.Agrobacteriumtumefaciens-mediatedgenetictransformationofmungbean(VignaradiataL.Wilczek)-arecalcitrantgrainlegume[J].PlantScience,2001,161(2):239-247.
[13]MahalakshmiLS,LeelaT,KumarSM,etal.Enhancedgenetictransformationefficiencyofmungbeanbyuseofprimaryleafexplants[J].CurrentScience,2006,91(1):93-99.
[14]SahooDP,KumarS,MishraS,etal.EnhancedsalinitytoleranceintransgenicmungbeanoverexpressingArabidopsisantiporter(NHX1)gene[J].MolecularBreeding,2016,36(10):144.
[15]蘇钺凯,邱镜仁,张晗,等.CRISPR/Cas9系统在植物基因组编辑中技术改进与创新的研究进展[J].植物学报,2019,54(3):385-395.
[16]谢先荣,曾栋昌,谭健涛,等.基于CRISPR编辑系统的DNA片段删除技术[J].植物学报,2021,56(1):44-49.
[17]朱丽珍,王芳,王娅丽,等.基因编辑技术及CRISPR/Cas系统在草地植物开发中的应用[J].江苏农业科学,2021,49(20):22-30.
[18]DuHY,ZengXR,ZhaoM,etal.EfficienttargetedmutagenesisinsoybeanbyTALENsandCRISPR/Cas9[J].JournalofBiotechnology,2016,217:90-97.
[19]MakhzoumAB,SharmaP,BernardsMA,etal.Hairyroots:anidealplatformfortransgenicplantproductionandotherpromisingapplications[M]//GangD.Phytochemicals,plantgrowth,andtheenvironment.NewYork:Springer,2012:95-142.
[20]KeresztA,LiDX,IndrasumunarA,etal.Agrobacteriumrhizogenes-mediatedtransformationofsoybeantostudyrootbiology[J].NatureProtocols,2007,2(4):948-952.
[21]Estrada-NavarreteG,Alvarado-AffantrangerX,OlivaresJE,etal.Fast,efficientandreproduciblegenetictransformationofPhaseolusspp.byAgrobacteriumrhizogenes[J].NatureProtocols,2007,2(7):1819-1824.
