基于SSR标记检测与重测序技术的3个李品种鉴定与遗传背景简析
2023-06-15宋海岩孙淑霞涂美艳王玲利徐子鸿江国良
宋海岩,孙淑霞,李 靖,涂美艳,王玲利,徐子鸿,陈 栋,江国良
(1 四川省农业科学院园艺研究所/农业农村部西南地区园艺作物生物学与种质创制重点实验室,成都,610066;2 四川农业大学园艺学院,成都,611130)
我国西南地区李种质资源十分丰富[1],近年来大面积推广的“青脆李[2]”“蜂糖李[3]”“脆红李[4]”“茵红李[5]”等新品种多由实生选育或芽变选种而得。通常果树新品种选育首先需要经过精准鉴定和评价来筛选优株,然后对优株进行DUS测试[6]。但由于李种质资源分子标记开发[7]、基因分型、指纹图谱构建等分子生物学鉴定方法研究滞后[8],许多育成品种的遗传背景不清晰[9],容易导致苗木市场混乱,育种者权益无法得到保障。此外,李育种材料的遗传背景高度相似,还可能导致杂交育种过程中出现返祖现象。
“玉带李”是四川省农业科学院园艺研究所从龙泉驿区李种质资源中选育出的晚熟李新品种(川认果2022001),其成熟期晚于“蜂糖李”和“青脆李”,外观又与“青脆李”相似,市场上常被果农和果商称作“晚熟蜂糖李”或“晚熟青脆李”,这严重影响了“玉带李”的推广和保护。本试验以“玉带李”等3个四川省主栽青皮李品种叶片DNA为模板,根据SSR标记分型结果,结合果实成熟期与外观特征,探索快速区分“玉带李”等3个熟期或外观相近的青皮李品种的方法,并基于重测序技术开展SNP和InDel变异检测及可视化分析,简析3个李品种的遗传背景和亲缘关系,以期为李配套授粉品种的选择和杂交育种亲本的组配提供参考。
1 材料与方法
1.1 供试材料
材料为5~8年生“玉带李”“青脆李”“蜂糖李”的嫁接树,均定植于成都市龙泉驿区柏合镇(N30°31′44.55″,E104°17′49.46″)。常规栽培管理措施,无明显病虫害。在成都市龙泉驿区,“玉带李”的成熟期最晚且果实缝合线处水渍状条带十分明显,“蜂糖李”的成熟期最早且果实缝合线凹陷,“青脆李”的成熟期介于“玉带李”和“蜂糖李”之间,以上物候期和果实性状的差异可以作为初步鉴定这3个青皮李品种的依据(见表1和图1)。
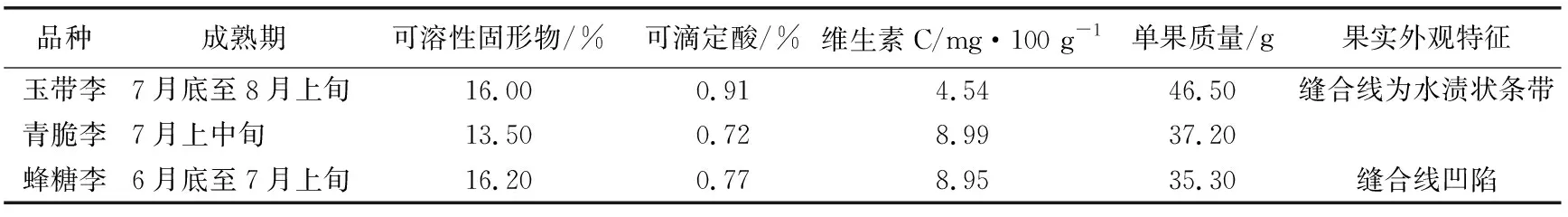
表1 “玉带李”“青脆李”“蜂糖李”3个李品种在成都市龙泉驿区栽培的主要差异特征

图1 “玉带李”“青脆李”“蜂糖李”3个李品种的果实
1.2 方法
1.2.1 SSR标记检测与基因分型 参照前人对李属种质资源多样性研究结果[10-11],设计77对SSR引物用于3个青皮李品种的基因分型。于2021年8月10日采集无病虫害叶片于锡箔纸中,随即置于液氮中,采集完毕后放于-80 ℃超低温冰箱中保存。使用CTAB法提取DNA,随后使用NanoDrop one微量紫外可见分光光度计检测DNA浓度和质量,备用。PCR反应体系:DNA模板(50 ng/μL)1.0 μL,I-5 PCR Mix(TSINGKE)15 μL,正反向引物(10 μmol/L)1.0 μL,加ddH2O至30 μL。PCR扩增程序:98 ℃预变性3 min;98 ℃变性10 s,53~59 ℃退火20 s,72 ℃延伸30 s,循环40次;最后72 ℃延伸5 min。使用1%高纯度低电渗琼脂糖凝胶(TSINGKE)电泳对PCR产物进行基因分型。
1.2.2 DNA提取与文库构建 采用CTAB法提取叶片DNA,琼脂糖凝胶电泳分析DNA降解程度以及是否有RNA污染;随后使用NanoDrop one微量紫外可见分光光度计检测DNA纯度;使用Qubit 2.0荧光仪对DNA浓度进行精确定量。其中,OD260/OD280值在1.8~2.0之间,含量在80 μg/mL以上的DNA样品用于建库。检验合格的DNA样品通过Covaris破碎机随机打断成长度为350 bp的片段。采用TruSeq Library Construction Kit进行建库。DNA片段经末端修复、加ployA尾、加测序接头、纯化、PCR扩增等步骤完成整个文库制备。构建好的文库通过Illumina HiSeq测序仪进行测序。
1.2.3 文库质量检测与上机测序 文库构建完成后,先使用Qubit 2.0荧光仪进行初步定量,稀释文库至1 ng/μL,随后使用Agilent 2100生物分析仪对文库的插入片段大小(insert size)进行检测,insert size符合预期后,使用qPCR方法对文库的有效浓度进行准确定量(文库有效浓度>2 nmol/L),以保证文库质量。库检合格后,根据文库的有效浓度及数据产出需求进行 Illumina HiSeq测序。
1.2.4 重测序数据分析 对下机得到的原始数据(raw data)进行过滤,得到有效数据(clean data)。将clean data与“中李6号”参考基因组(https://www.rosaceae.org/Analysis/9019655)数据[12]进行比对,比对工具为BWA软件[13](参数:mem -t 4 -k 32 -M),比对结果经SAMTOOLS[14]去除重复(参数:rmdup)。最后使用SAMTOOLS和ANNOVAR软件进行单核苷酸多态性(SNP)和得失位(InDel)检测及注释[15]。
2 结果与分析
2.1 SSR标记检测与基因分型
试验结果表明,在已公布的77对李属种质资源SSR引物中,没有任何一对引物能够同时区分出这3个李品种。但29号引物和UDP98-021可以作为区分“蜂糖李”的分子标记:“玉带李”和“青脆李”的29号引物PCR扩增产物为100~250 bp的单条带,而“蜂糖李”的29号引物PCR扩增产物为1 000~2 000 bp的单条带;“蜂糖李”的UDP98-021引物PCR扩增产物为100~250 bp的单条带,“玉带李”和“青脆李”未能扩增出条带。ASS R71引物可以作为区分“青脆李”的分子标记:“玉带李”和“蜂糖李”的ASS R71引物PCR扩增产物为100~250 bp的单条带,“青脆李”未能扩增出条带。通过组合不同引物的基因分型结果能够区分出这3个李品种(见图2、图3和表2)。
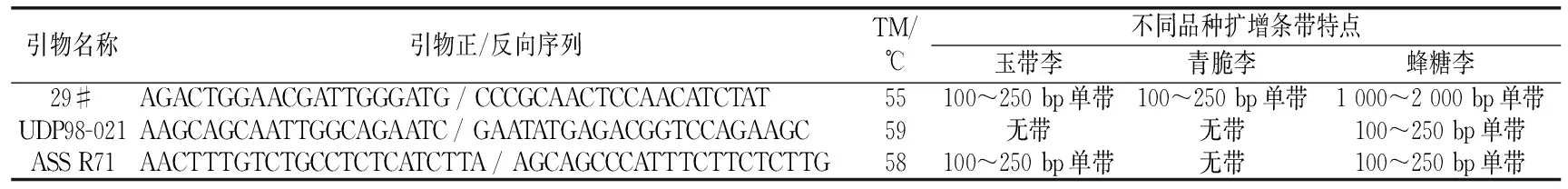
表2 可区分“玉带李”“青脆李”“蜂糖李”的3对SSR引物信息及分型结果
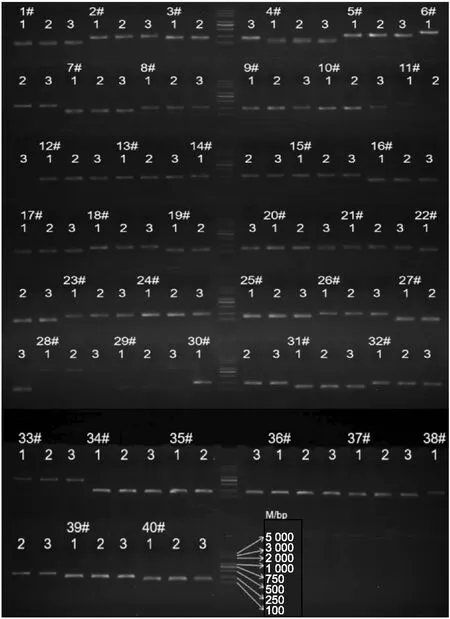
注:1、2和3分别代表“玉带李”“青脆李”“蜂糖李”;1#至40#指引物,M指Marker。
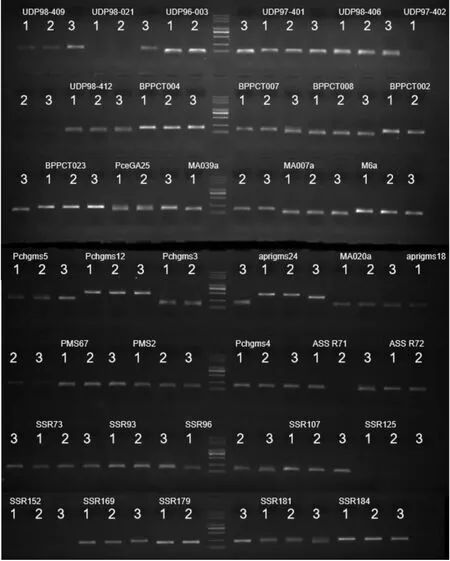
注:1、2和3分别代表“玉带李”“青脆李”“蜂糖李”;中间的Marker,从上至下分别是5 000、3 000、2 000、1 000、750、500、250、100 bp。
2.2 基因组重测序数据质量
以3个品种叶片DNA样本为模板进行基因组重测序,共产生raw data 5.232 Gbp,过滤后的 clean data 5.19 Gbp,各样本的raw data在(1 502.621 ~2 145.653)Mbp之间,测序质量高(Q20≥96.95%,Q30≥91.94%),GC含量在38.8%~40.37%之间,所有样本的数据量足够,测序质量合格,GC分布正常,建库测序结果能够满足基因组之间的比较分析(见表3)。

表3 “玉带李”“青脆李”“蜂糖李”3个李品种基因组重测序数据质量情况

表4 “玉带李”“青脆李”“蜂糖李”3个李品种基因组重测序深度及覆盖度
2.3 基因组重测序数据与参考基因组比对结果
“中李6号”参考基因组大小为318 575 050 bp,所有样本的比对率在88.61%~95.02%之间,对参考基因组(排除N区)的平均覆盖深度在7.13~8.03(×)之间,1(×)覆盖度(至少有一个碱基的覆盖)在76.95%以上。以上比对结果正常,可用于后续的变异检测及相关分析。
2.4 基因组SNP与InDel检测及注释结果
与“中李6号”参考基因组相比,“玉带李”“青脆李”“蜂糖李”基因组检测到的SNP位点数分别为712 529、384 711和373 883个,InDel数量分别为101 979、47 410和48 445个。其中,“玉带李”在基因上下游1 kb区域、内含子区域、外显子区域、剪接位点以及基因间区内的SNP和InDel数量均远高于“青脆李”“蜂糖李”,并且其转换(ts)和颠换(tv)的SNP数量、插入和缺失的InDel数量也为最高值。这表明,“玉带李”的遗传背景与“青脆李”“蜂糖李”有极大差异(见表5和表6)。
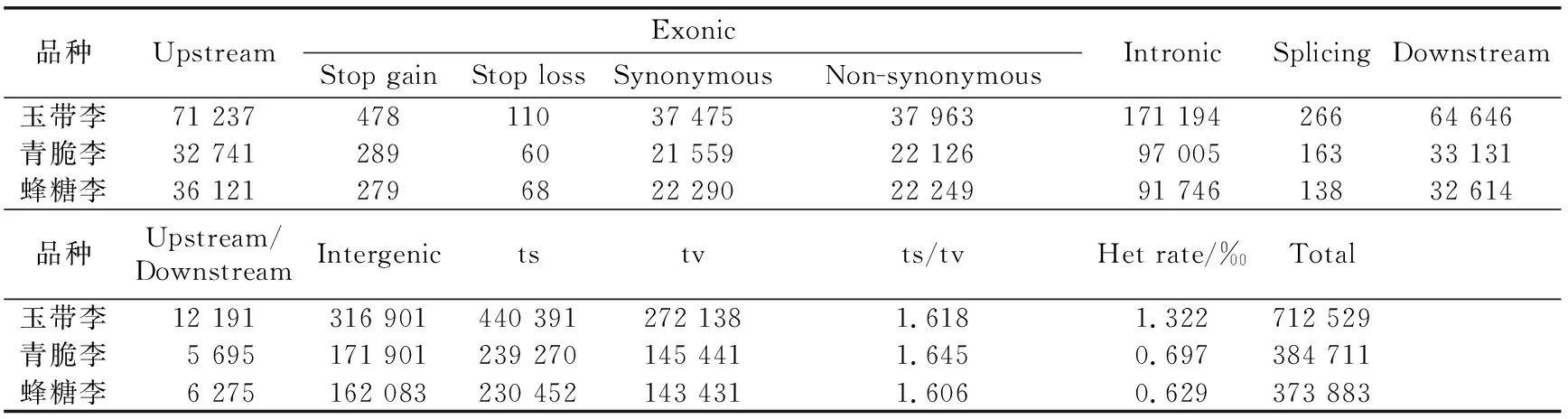
表5 “玉带李”“青脆李”“蜂糖李”3个李品种基因组SNP检测及注释结果统计

表6 “玉带李”“青脆李”“蜂糖李”3个李品种基因组InDel检测及注释结果统计
2.5 SNP和InDel变异检测结果可视化分析
为了更清晰地显示各样品染色体上SNP和InDel的分布情况,以100 kb为窗口,统计每个窗口上的SNP和InDel密度,绘制了3个李品种染色体上的SNP、InDel密度分布热图,详见图4。结果显示,与“中李6号”参考基因组相比,“玉带李”1~8号染色体上SNP和InDel密度分布均明显高于“青脆李”和“蜂糖李”,表明“玉带李”的遗传背景与“青脆李”和“蜂糖李”差异较大。“青脆李”和“蜂糖李”染色体上SNP和InDel密度分布十分相似,表明“青脆李”和“蜂糖李”亲缘关系相对较近。基于重测序技术的SNP和InDel变异检测结果锚定到染色体上后,可以用于初步鉴定这3种青皮李品种的遗传背景和亲缘关系。
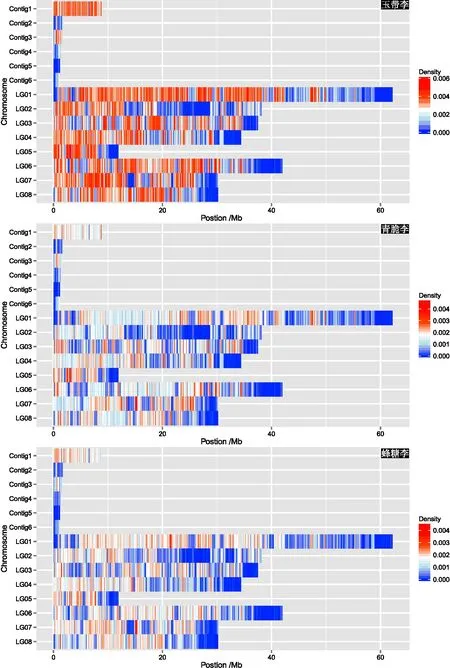
图4 “玉带李”“青脆李”“蜂糖李”3个李品种锚定到染色体上的SNP密度分布
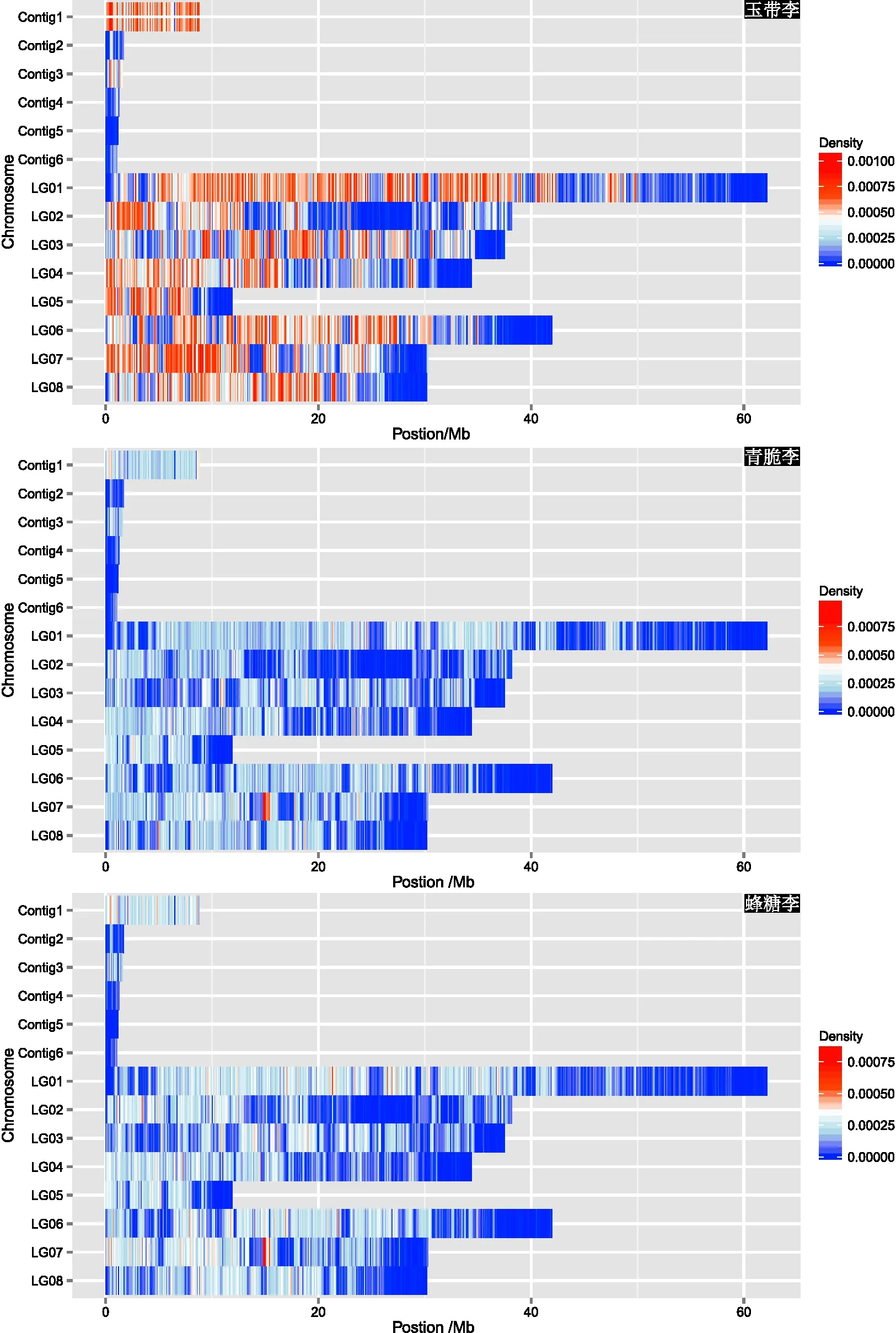
图5 “玉带李”“青脆李”“蜂糖李”3个李品种锚定到染色体上的InDel密度分布
3 讨论与结论
目前四川省主栽青皮李品种为“青脆李”“蜂糖李”“巴山脆李”“玉带李”等选育品种以及“江安李”等少量地方品种。一些青皮李的成熟期相近或果实外观相似,又缺少鉴别方法,常有果农和果商将“玉带李”称作“晚熟青脆李”或“晚熟蜂糖李”进行销售。SSR分子标记是一种具有操作简单、重复性好、共显性高、稳定性强、位点特异等优点的DNA分子标记,是果树新品种鉴定的常用技术。王清明等人[16]在构建观赏桃SSR指纹图谱时,发现选用组合内各引物多态性条带总数最多的组合方式能够为22份观赏桃种质构建指纹图谱。我国李品种选育和分子标记开发相对滞后,因此本试验参照李属近缘作物桃的分子标记方法(《桃品种鉴定SSR分子标记法:NY/T 3642—2020》),从前人研究结果中筛选出77对李属种质资源SSR分子标记对3个四川省主栽青皮李品种进行基因分型,虽然没有任何一对SSR分子标记可以同时区分出这3个李品种,但29号引物和UDP98-021可以作为区分“蜂糖李”的分子标记;ASS R71可以作为区分“青脆李”的分子标记。果实外观特征和成熟期是生产中判断不同李品种的常见指标,再结合“玉带李”的果实成熟期和水渍带状缝合线特征,可以成功区分出这3个李品种。
近年来,由于测序成本的不断降低,对许多果树的认知进入了基因组时代。逐渐丰富的李属种质资源基因组数据库,为鉴定李种质资源的遗传背景和亲缘关系提供了数据支撑。Wang等人[17]对377份分布于海拔2 067~4 498 m的西藏李属种质资源开展精细评价,发现不同李属种质资源之间的SNP数量分布在269 568~4 153 328之间。本试验参照Wang等人[17]对西藏李属种质资源精细评价方法(测序平台、测序深度等所有测试方法保持一致),对成熟期或外观相近的3个李品种进行SNP和InDel变异检测,结果显示3个李品种与参考基因组(中李6号)的SNP位点数均在37万个以上。一般果树基因组的杂合度较高,在杂交组配过程中选择遗传背景差异较大的亲本有利于避免返祖现象。本试验中,“玉带李”染色体上SNP和InDel密度分布热图均明显高于“青脆李”和“蜂糖李”,表明其遗传背景与“青脆李”“蜂糖李”有较大差异。“玉带李”同时具有晚熟、高甜、果形端正和产量高等优势,可以作为“青脆李”“蜂糖李”的配套授粉品种,是李品种改良的优良亲本。值得注意的是,“青脆李”和“蜂糖李”虽然外观有较大差异,但染色体上的SNP和InDel分布密度十分相似,遗传背景相似度极高,因此,可能是研究果实缝合线形成和糖分积累差异机制的适宜材料。
综上所述,利用29号、UDP98-021和ASS R71等3对SSR引物进行分型的结果,结合果实成熟期和外观特征,可以准确区分出“玉带李”“青脆李”“蜂糖李”等3个青皮李品种。基于重测序技术的SNP和InDel检测分析结果表明,“玉带李”的遗传背景与“青脆李”“蜂糖李”有较大差异,“青脆李”“蜂糖李”的遗传背景高度相似。“玉带李”可以作为“青脆李”和“蜂糖李”的配套授粉品种,是李品种改良的优良亲本。
