特异性检测干酪乳杆菌的引物探针设计
2023-05-31陈延宁侯亚茹杜丽霞艾庆蕊张大虎侯少阳
陈延宁 侯亚茹 杜丽霞 艾庆蕊 张大虎 侯少阳


摘 要:目的:基于肠杆菌科基因间重复共有序列聚合酶链反应(Enterobacterialrepetitive Intergenic Consensus PCR,ERIC-PCR)技术,设计特异性引物探针,实现干酪乳杆菌的靶向检测。方法:以干酪乳杆菌(Lactobacillus casei)HP-B1142为研究对象,通过不同实验,优化干酪乳杆菌(Lactobacillus casei)的ERIC-PCR反应体系。通过凝胶电泳获得特定的条带,对其进行测序和分析,设计出特异性的引物探针,达到对干酪乳杆菌靶向鉴别的目的。结果:通过不同实验,得到干酪乳杆菌的最佳ERIC-PCR反應体系;基于干酪乳杆菌ERIC基因片段,获得两对特异性引物,可在多种DNA的混合溶液中快速靶向鉴别出干酪乳杆菌。结论:基于ERIC-PCR技术获得的特异性引物探针,可高效、灵敏地实现复杂生境中干酪乳杆菌检出。
关键词:聚合酶链反应;干酪乳杆菌;靶向检测;引物探针
Abstract: Objective: To design specific primer probes based on ERIC-PCR technology to achieve targeted detection of Lactobacillus casei. Method: Using Lactobacillus casei HP-B1142 as the research object, the ERIC-PCR reaction system of Lactobacillus casei was optimized through different experiments. Specific bands were obtained by gel electrophoresis, sequenced and analyzed, and specific primer probes were designed to achieve the purpose of targeted identification of Lactobacillus casei. Result: Through different experiments, the optimal ERIC-PCR reaction system for Lactobacillus casei was obtained. Based on the ERIC gene fragment of Lactobacillus casei, two pairs of specific primers were obtained, which can quickly target and identify Lactobacillus casei in a mixture of multiple DNA solutions. Conclusion: The specific primer probe obtained based on ERIC-PCR technology can efficiently and sensitively detect Lactobacillus casei in complex habitats.
Keywords: polymerase chain reaction; Lactobacillus casei; targeted detection; primer probes
益生菌(Probiotics)于1907年由梅切尼科夫首次提出。益生菌广泛存在于人体内,种类多样,具有促进营养物质吸收、提高机体免疫力等功能。此外,研究发现益生菌用于发酵乳制品能够有效缓解乳糖不耐受症状[1]。此外,益生菌对维持宿主肠道菌群平衡有非常重要的作用[2-3],同时,益生菌还可用于治疗儿童急性腹泻[4]。干酪乳杆菌属于乳杆菌属,为革兰氏阳性菌,作为益生菌的一种,具有降血压、降胆固醇、抑制肿瘤生长的功能,同时可以促进细胞分裂,具有增强人体免疫以及预防癌症等作用[5]。目前针对肠道细菌的鉴定技术主要有生化反应实验、16S rRNA测序比对和细菌特异性基因检测、全基因组测序[6]等分子生物学方法[7]。近些年来发展的基质辅助激光解析飞行质谱技术,可以通过检测菌体蛋白质指纹图谱达到对不同细菌属、种的鉴定[8],同时基于MALDI-TOF的分型方法也在快速发展当中[9]。但是上述方法均存在操作复杂等问题,急需一种简便、高效的干酪乳杆菌鉴别方法,实现干酪乳杆菌的痕量检出。
ERIC序列,即肠杆菌基因间重复序列,首先由Sharples等[10]于1990年发现于大肠杆菌,并将其命名为基因间重复单位[11]。肠杆菌科基因间重复共有序列聚合酶链反应(Enterobacterialrepetitive Intergenic Consensus PCR,ERIC-PCR)是根据ERIC的核心序列设计引物,PCR扩增两个ERIC之间序列,不同细菌的ERIC序列在染色体的位置不一样,因此可以根据扩增条带位置和数量来判断是否为同一基因型多位点序列分型[12],ERIC序列具有高度保守性,因此通过普通PCR方法扩增片段可以获得DNA指纹图谱[13]。ERIC-PCR技术在DNA指纹图谱技术中的广泛应用使得细菌在亚种层次上的鉴别分类有了更好的依据[14-15]。由于此方法较传统鉴别方法更为便利,目前被广泛应用于微生物学中的菌株鉴别分类方面。由于细菌基因组的复杂性,不同的细菌会有不同的反应条件,通过不断优化反应体系,利用ERIC-PCR技术设计出特异性引物,可在不同的细菌DNA中靶向鉴别出特定的菌株。
本实验通过利用ERIC-PCR技术设计出特异性引物,首先将目的菌株进行分离纯化,再运用改良的SDS碱裂解法提取目的菌株的DNA,通过对比不同反应条件下的结果,对ERIC-PCR反应体系进行优化,再将产物进行凝胶电泳,并将目的片段进行回收测序,得到测序结果后,运用DNAMAN V6软件进行特异性引物的设计,最终得到特异性引物并进行验证。本实验所设计的特异性引物可以在菌株混合基因组DNA中通过PCR技术检测出干酪乳杆菌,提高了菌种的特异性鉴别效率。
1 材料与方法
1.1 仪器与设备
S1010掌上离心机,美国赛洛捷克;DYY-6C型电泳仪,北京六一生物科技有限公司;YXQ-75G全自动数显立式高压蒸汽灭菌器,上海博迅实业有限公司;DHP-9052型电热恒温培养箱,上海皓庄仪器有限公司;T100 Thermal PCR仪,美国BIO-RAD公司。
1.2 材料与试剂
琼脂糖凝胶DNA回收试剂盒,康为世纪生物科技有限公司;2K plus Ⅱ DNA marker,北京全式金生物科技有限公司;纯净水,杭州娃哈哈集团有限公司;Tris碱、十二烷基硫酸钠(Sodium Dodecyl Sulfate,SDS),德国Sigma公司;溴化乙锭(Ethidium Bromide,EB),美国Amresco公司;Taq DNA聚合酶、T5单克隆载体,北京全式金生物技术有限公司。
1.3 实验方法
1.3.1 目的基因DNA的提取纯化
将干酪乳杆菌HP-B1142接种至MRS固体培养基,37 ℃培养48 h,获得纯化的目的菌株。目前有许多提取DNA的方法,为了提高实验效率,本实验使用改良的SDS碱裂解法,用50 μL含有RNase A酶(10 mg·L-1)的1×TE溶液重悬,于-20 ℃中保存备用。
1.3.2 设计特异性引物探针
使用ERIC-PCR技术对HP-B1142基因组DNA进行PCR,对长度约为2 700 bp和500 bp的两条DNA片段进行回收,连接至T5载体,送至金唯智生物科技有限公司进行测序。
将得到的两组测序结果利用BLAST在线比对工具进行比对分析,同时将序列上传至GenBank数据库,接收序列号分别为OP700792、OP700793。利用DNAMAN V6软件设计出对应片段的特异性引物,最后进行引物的特异性检验。
1.3.3 引物探针的含量限度检验
从实验室自建微生物资源菌库中选取5株常用益生菌菌株,分别为植物乳杆菌HP-B1098、鼠李糖乳杆菌HP-B1083、副干酪乳杆菌HP-B1145、嗜酸乳杆菌HP-B1079、瑞士乳杆菌HP-B1126,将其作为阴性对照。取以上5种细菌等浓度的混合DNA溶液,等量混合,使混合基因组DNA浓度与干酪乳杆菌基因组DNA浓度相等。将干酪乳杆菌基因组DNA与5种细菌的混合DNA溶液相混合,配制成干酪乳杆菌基因组DNA相对质量分数为100%(绝对含量900 ng·mL-1)、30%(绝对含量270 ng·mL-1)、3%(绝对含量27 ng·mL-1)、0%(绝对含量0 ng·mL-1)的混合溶液作为模板,使用设计的两对特异性引物进行PCR反应。
1.3.4 引物的特异性检验
为了检测本实验中的两组特异性引物是否可以特异性鉴别干酪乳杆菌,特从实验室中选取了其他不同的5种细菌进行检验。5种细菌分别为植物乳杆菌、嗜酸乳杆菌、鼠李糖乳杆菌、副干酪乳杆菌、瑞士乳杆菌。以本实验所设计的1142-a-F/1142-a-R为一对引物,1142-2-F/1142-2-R为一对引物进行PCR反应。
1.3.5 引物的普适性检验
为了确定该引物探针是否可以通用地检测出干酪乳杆菌这一菌种,现选取干酪乳杆菌的近缘菌株进行普适性检验,验证其通用性。将Lactobacillus casei DSM 20011、Lactobacillus case JCM 8129、Lactobacillus case IDCC 3451、Lactobacillus case JX 20225、Lactobacillus case ATCC 11443共5株近缘干酪乳杆菌作为引物探针普适性检验的对照,分别对上述菌株及本实验所研究的干酪乳杆菌HP-B1142进行PCR,以所设计的1142-a-F/1142-a-R为一对引物,1142-2-F/1142-2-R为一对引物,进行PCR检测。
1.3.6 运用两对特异性引物检验市售公示配方中含有干酪乳杆菌的产品
通过选取3种市售产品来进行检验,可以用来确定本实验设计的两对特异性引物是否可以精准检验出产品中的干酪乳杆菌,检测其实用效果。选取3种市售公示配方中含有干酪乳杆菌的产品,提取产品基因组DNA为模板,以所设计的1142-a-F/1142-a-R为一对引物,1142-2-F/1142-2-R为一对引物,进行PCR反应。
2 结果与分析
2.1 DNA测序及特异性引物的设计
以干酪乳杆菌DNA作为模板进行ERIC-PCR操作,获得长度约为2 000 bp和500 bp的两条DNA条带,通过测序获得其DNA序列,长度分别为1 815 bp和428 bp。使用DNAMAN V6软件分别设计出针对两条DNA片段的特异性引物探针。引物探针命名及序列分别为1142-a-F:5'-CTAAGGTAGCTGATCGGTGGCACGATC-3',1142-a-R:5'-CACTCTTGCCAATGGTCATCGCTG-3';1142-2-F:5'-CAATCCATCAGTCAGAATGTGGAAGC-3',1142-2-R:5'-CGAATGCACATGAGGATATCATTTCAGC-3'。同时使用两对特异性引物以干酪乳杆菌HP-B1142基因组DNA为模板进行PCR,可以得到條带清晰长度约为400 bp和1 800 bp的目标片段,实验结果如图1。
M:2K plus Ⅱ DNA marker;A图中1以1142-2-F/1142-2-R为引物、干酪乳杆菌(Lactobacillus casei)HP-B1142基因组DNA为模板进行验证。B图中1以1142-a-F/1142-a-R为引物、干酪乳杆菌(Lactobacillus casei)HP-B1142基因组DNA为模板进行验证。
2.2 引物的特异性检验
为了确定本特异性引物是否具有特异性,是否能达到特异性鉴别干酪乳杆菌的目的,采用PCR的方式验证两对引物探针的特异性。对所得PCR产物进行0.8%琼脂糖凝胶电泳验证,由图2可知,以干酪乳杆菌基因组DNA为模板的1、2泳道可成功获得目标条带,而以其他菌株基因组DNA为模板的泳道均无法得到目标条带。由此,该特异性引物探针可特异性检出干酪乳杆菌。
M:2K plus Ⅱ DNA marker;图中1、3、5、7、9、11皆选用1142-a-F/1142-a-R为引物,所选用模板分别为干酪乳杆菌HP-B1142、植物乳杆菌、嗜酸乳杆菌、鼠李糖乳杆菌、副干酪乳杆菌、瑞士乳杆菌基因组DNA。图中2、4、6、8、10、12皆选用1142-2-F/1142-2-R为引物,所选用模板分别为干酪乳杆菌HP-B1142、植物乳杆菌、嗜酸乳杆菌、鼠李糖乳杆菌、副干酪乳杆菌、瑞士乳杆菌基因组DNA。
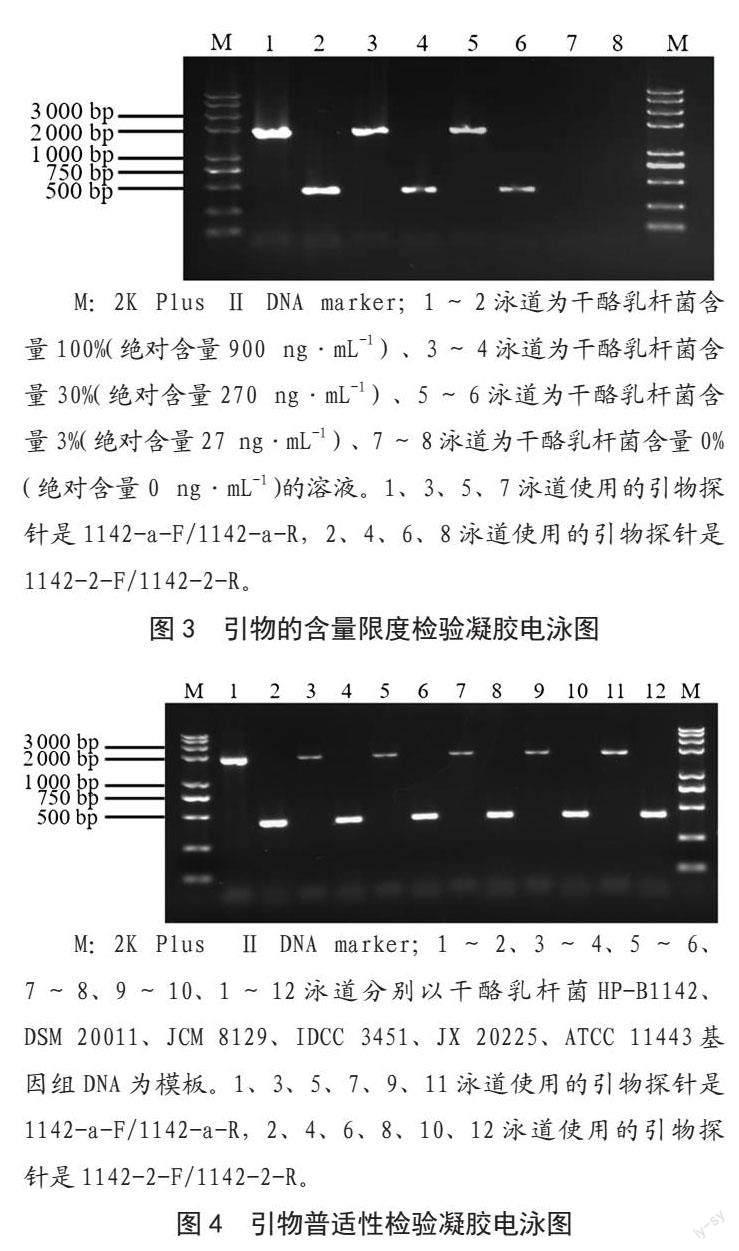
2.3 引物探针可检测基因组DNA的含量限度检验
为确定本研究所设计的两对特异性引物探针可检测的基因组DNA含量限度,以混合DNA溶液为模板进行PCR反应。对所得PCR产物进行0.8%琼脂糖凝胶电泳验证。由图3中可知,在干酪乳杆菌基因组DNA浓度为27 ng·mL-1时,本实验的两对引物探针依然可完成干酪乳杆菌的特异性检出。
M:2K Plus Ⅱ DNA marker;1~2泳道为干酪乳杆菌含量100%(绝对含量900 ng·mL-1)、3~4泳道为干酪乳杆菌含量30%(绝对含量270 ng·mL-1)、5~6泳道为干酪乳杆菌含量3%(绝对含量27 ng·mL-1)、7~8泳道为干酪乳杆菌含量0%(绝对含量0 ng·mL-1)的溶液。1、3、5、7泳道使用的引物探针是1142-a-F/1142-a-R,2、4、6、8泳道使用的引物探针是1142-2-F/1142-2-R。
2.4 引物探针普适性检验
由于干酪乳杆菌作为益生菌被广泛应用于生产应用和研究中,因此,准确鉴别干酪乳杆菌种及其近缘种也同样重要。采用本实验中的两对引物探针,对与HP-B1142近缘的5株干酪乳杆菌进行PCR验证,图4实验结果表明两对引物探针均可扩增出目标条带从而实现近缘干酪乳杆菌检出。
M:2K Plus Ⅱ DNA marker;1~2、3~4、5~6、7~8、9~10、1~12泳道分别以干酪乳杆菌HP-B1142、DSM 20011、JCM 8129、IDCC 3451、JX 20225、ATCC 11443基因组DNA为模板。1、3、5、7、9、11泳道使用的引物探针是1142-a-F/1142-a-R,2、4、6、8、10、12泳道使用的引物探针是1142-2-F/1142-2-R。
2.5 运用两对特异性引物检验市售公示配方中含有干酪乳杆菌的产品
选取3种市售产品来进行检验,可以用来确定本实验设计的两对特异性引物是否可以达到精准检验产品中的干酪乳杆菌,检测其实用效果。对所得PCR产物进行0.8%琼脂糖凝胶电泳验证。图5中泳道均可得到目标条带,进而判断市售产品中含有干酪乳杆菌。由此可知,本实验涉及的两对特异性引物探针可以用于市售产品中干酪乳杆菌的检验,具有廣阔的市场应用前景。
M:2K Plus Ⅱ DNA marker;1~2、3~4、5~6分别以市售混合益生菌产品一、市售混合益生菌产品二及市售益生菌产品三的基因组DNA为模板;1、3、5泳道使用的引物探针是1142-a-F/1142-a-R,2、4、6泳道使用的引物探针是1142-2-F/1142-2-R。
3 结论
ERIC-PCR技术在DNA指纹图谱技术中的广泛应用,使得细菌在亚种层次上的鉴别分类有了更好的依据,本实验通过使用ERIC-PCR技术设计得到的两对特异性引物,已将引物序列上传至GenBank数据库,接收序列号为OP700792、OP700793。获得两对引物探针1142-a-F/1142-a-R,1142-2-F/1142-2-R,并验证其特异性、普适性、含量限度及是否可鉴别市售产品中的干酪乳杆菌,大大增强了该引物探针的实用性及可靠性。上述两对引物探针可以在微量DNA溶液(绝对含量27 ng·mL-1)中高效实现干酪乳杆菌的痕量检出,并可在复杂生境中实现市售产品中干酪乳杆菌的特异性检出。
本实验设计的两对特异性引物探针较现有干酪乳杆菌的鉴别方法有了较大优化,克服了现有方法中操作复杂、易被污染的缺点,可简单、快速检出复杂生境样本中干酪乳杆菌;在医疗保健、科研活动等方面有着良好的实用性和广阔的市场前景。
参考文献
[1]钟燕.益生菌与乳糖不耐受研究进展[J].国外医学(卫生学分册),2003,30(2):101-105.
[2]AFRC R F.Probiotics in man and animals[J].Journal of Applied Microbiology,1989,66(5):365-378.
[3]SALMINEN S,OUWEHAND A,BENNO Y,et al.Probiotics: how should they be defined?[J].Trends in Food Science & Technology,1999,10(3):107-110.
[4]LAI H H,CHIU C H,KONG M S,et al.Probiotic Lactobacillus casei: effective for managing childhood diarrhea by altering gut microbiota and attenuating fecal inflammatory markers[J].Nutrients,2019,11(5):1150.
[5]曹瑞博,汪建明.干酪乳杆菌的功能性研究及其应用[J].中国食品添加剂,2009(增刊1):169-172.
[6]高芸,轩慧勇,姚晓慧,等.MALDI-TOF质谱技术对猪肠道菌群中携带mcr-1细菌的鉴定与聚类分型[J/OL].中国动物传染病学报:1-10[2023-03-30].http://kns.cnki.net/kcms/detail/31.2031.S.20210430.1402.024.html.
[7]罗力文,鄢明辉,游春苹,等.细菌分型方法研究进展[J].中国微生态学杂志,2020,32(7):836-841.
[8]FENSELAU C, DEMIREV P A.Characterization of intact microorganisms by MALDI mass spectrometry[J].Mass Spectrometry Reviews,2001,20(4):157-171.
[9]SAUGET M,VALOT B,BERTRAND X,et al.Can MALDI-TOF mass spectrometry reasonably type bacteria?[J].Trends in Microbiology,2017,25(6):447-455.
[10]SHARPLES G J,LLOYD R G.A novel repeated DNA sequence located in the intergenic regions of bacterial chromosomes[J].Nucleic Acids Research,1990,18(22):6503.
[11]高卫科,王川庆,吴高锋,等.ERIC-PCR及在肠道菌群分析中的应用[C]//河南省畜牧兽医学会第七届暨2008年学术研讨会理事会第二次会议论文集.郑州:河南省畜牧兽医学会,2008:580-582.
[12]王方,吴新明,王彦,等.ERIC-PCR技术与MLST技术對30株耐碳青霉烯类肺炎克雷伯菌的基因分型结果观察[J].山东医药,2020,60(30):35-39.
[13]INTARAPANICH A,KAEWKAMNERD S,SHAW P J,et al.Automatic DNA diagnosis for 1D gel electrophoresis images using bio-image processing technique[J].BMC genomics,2015,16(增12):15.
[14]HULTON C S J,HIGGINS C F,SHARP P M.ERIC sequences: a novel family of repetitive elements in the genomes of Escherichia coli,Salmonella typhimurium and other enterobacteria[J].Molecular Microbiology,1991,5(4):825-834.
[15]JUDD A K,SCHNEIDER M,SADOWSKY M J,et al.Use of repetitive sequences and the polymerase chain reaction technique to classify genetically related Bradyrhizobium japonicaum serocluster 123 strains[J].Applied and Environment Microbiology,1993,59(6):1702-1708.
