苜蓿花叶病毒ELISA检测方法的建立及其在大豆病毒调查和抗性资源鉴定中的应用
2023-05-30战笑蕾王馨玥李小宇张春雨刘建青王晨王斌梁雨欣王永志程晓非
战笑蕾 王馨玥 李小宇 张春雨 刘建青 王晨 王斌 梁雨欣 王永志 程晓非

摘要 苜蓿花葉病毒(alfalfa mosaic virus, AMV)是一种世界性分布、宿主范围广、具有严重危害性的植物病毒,能引起大豆的严重病害。本研究利用原核表达的AMV CP蛋白制备的抗血清,建立了高效、准确的AMV间接ELISA检测方法,并应用于病害调查和抗性鉴定,结果表明制备的3份抗血清对重组蛋白和AMV感染的大豆植物粗提液的效价均达到256 000倍,血清特异性分析结果显示3份抗血清仅识别感染AMV的大豆叶片,不识别感染大豆花叶病毒(soybean mosaic virus, SMV)的大豆叶片。通过建立的AMV间接ELISA与常规RT-PCR同时对采集的50份疑似感染AMV的大豆样品进行检测,有46份样品检测结果一致,符合率达92%。利用建立的AMV ELISA方法和课题组已建立的SMV ELISA方法对吉林省大豆主产区的大豆样品进行病毒检测的结果表明,病毒检出率为38.30%,SMV的检出率达30.85%,AMV的检出率达17.06%,复合侵染率为9.61%。对接种AMV的40个大豆品种进行抗性鉴定,结果显示40份大豆全部感染AMV,但是病毒载量存在差异,部分品种表现出AMV抗性,其中大豆抗性资源11份,首次发现AMV造成的大豆病害已经成为吉林省大豆的主要病害之一。
关键词 大豆; 苜蓿花叶病毒; 抗性鉴定; 间接ELISA
中图分类号: S 431.14
文献标识码: A
DOI: 10.16688/j.zwbh.2022011
Abstract Alfalfa mosaic virus (AMV) is a plant virus with worldwide distribution, wide host range, and serious harmfulness, which causes serious disease in soybean. In this study, the antiserum prepared by prokaryotic expression of AMV CP protein was used to establish an efficient and accurate indirect ELISA method for detection of AMV, which was applied to disease investigation and resistance identification. The results showed that the titers of the prepared three antiserums against recombinant protein and crude extract of soybean plants infected with AMV reached 256 000 times. Serum specificity analysis showed that the three antiserums only recognized AMV infected soybean leaves and did not recognize soybean mosaic virus (SMV) infected soybean leaves. A total of 50 soybean samples suspected to be infected with AMV were simultaneously detected by the established AMV indirect ELISA and conventional RT-PCR methods. The detection results of 46 samples were consistent, and the coincidence rate was 92%. The established AMV ELISA method and SMV ELISA method were used to detect the virus in soybean samples from the main soybean producing areas in Jilin province. The results showed that the detection rate of virus was 38.30%, the positive rate of SMV was 30.85%, the positive rate of AMV was 17.06%, and the composite infection rate was 9.61%. Resistance identification of 40 soybean varieties inoculated with AMV showed that all 40 soybean varieties were infected with AMV, but the viral load was significantly different. Some varieties showed AMV resistance, including 11 soybean resistant resources. The soybean disease caused by AMV had become one of the main diseases of soybean in Jilin province for the first time.
Key words soybean; alfalfa mosaic virus; resistance identification; indirect ELISA
大豆是我国重要的粮食作物,2019年中央1号文件正式提出实施大豆振兴计划[1]。吉林地区的气候与土壤为大豆提供了良好的生长发育环境,一直以来都是我国大豆种植的主产区。已知能够侵染大豆的病毒有50种以上,其中大豆花叶病毒(soybean mosaic virus, SMV)和苜蓿花叶病毒(alfalfa mosaic virus, AMV)是危害吉林省大豆质量与产量的主要病毒。SMV隶属于马铃薯Y病毒科Potyviridae马铃薯Y病毒属Potyvirus,是引起大豆花叶病的主要病原,在世界各地的大豆产区均有发生。该病毒于1915年在美国康涅狄格州首次被发现,自然条件下可造成10%到50%的减产,损失最高可达94%。AMV属于雀麦花叶病毒科Bromoviridae苜蓿花叶病毒属Alfamovirus,是一种世界性分布的病毒[2]。该病毒于1931年在美国的紫花苜蓿中首次被发现,此后在亚洲、非洲、南美、北美相继被报道[3]。AMV能够侵染51科430多种双子叶植物,其中包括豆科和茄科等许多重要经济作物,主要通过蚜虫以非持久方式进行传播,也可以汁液摩擦传播,在某些植物中还能通过种子传毒[4]。AMV造成的症状从隐蔽到严重不等,主要表现为花叶、皱缩、黄化和环斑畸形等,是一种具有严重危害性的植物病毒[5-8]。目前尚无有效治疗病毒病的药剂,高效、精准的病毒检测技术是该病害防治的前提。
AMV的基因组全长约为8 300 nt,由3条正义单链RNA组成,根据长度分别命名为RNA1、RNA2和RNA3[9]。RNA1编码的P1蛋白的N-末端有类似转甲基酶的区域,C-末端有类似解旋酶的区域;RNA2编码的P2蛋白有依赖于RNA复制的RNA聚合酶(RNA-dependent RNA polymerase, RdRp),两者共同构成了AMV的复制酶。RNA3含有2个开放阅读框,编码病毒的运动蛋白(movement protein,MP)和24 kD的病毒衣壳蛋白(coat protein, CP)。MP位于RNA3的5′末端,由RNA3直接翻译,CP位于RNA3的3′末端,通过亚基因组RNA4翻译[10-12]。研究发现,除了病毒粒子组装外,CP还在细胞间运动、复制和促进病毒翻译中起着重要的作用[13-14]。目前植物病毒检测技术主要以分子生物学检测和血清学检测为主。对AMV的检测主要以CP为靶标,序列比对表明,不同AMV株系的CP在核苷酸水平上的一致性为93%~99%,在氨基酸水平上的一致性为92%~100%[2,15-16]。具有高特异性、高灵敏度的RT-PCR是近年來应用于AMV研究的分子生物学检测技术,有学者通过CP保守区设计特异性引物建立不同作物AMV的检测方法[5,17-19]。RT-PCR需要专业人员与设备,成本较高且操作复杂,不适用于基层高通量的田间样品检测[17]。基于血清学的ELISA(enzyme linked immunosorbent assay, ELISA)检测技术,具有成本低、操作简单等优点。韩俊丽等利用美国生产的AMV三抗夹心ELISA法(TAS-ELISA)成功检测到AMV[20]。Ahoonmanesh等利用兔和鸡的抗体建立了检测AMV的双抗夹心ELISA法,该方法还可用于蚜虫传播病毒的研究[21]。
目前国内AMV的ELISA检测主要依赖于进口试剂盒,本研究首次以AMV的大豆中国分离物衣壳蛋白为抗原,通过免疫动物,获得AMV大豆分离物的抗体,建立了大豆AMV间接ELISA检测方法,首次对吉林省大豆进行AMV流行病学调查,发现AMV已经成为吉林省大豆的主要病害之一。初步应用于大豆种质资源的AMV抗性分析,鉴定大豆抗性资源11份,为抗病育种和抗病机理的研究提供技术支持。
1 材料与方法
试验于2020年至2021年在吉林省农业科学院植物保护研究所微生物重点实验室和东北农业大学农学院完成。
1.1 材料
供试材料:感染AMV的大豆及苜蓿叶片、pET-28a载体、Top10感受态菌株、BL21(Plyss)表达菌株均由吉林省农业科学院实验室保存;40份黑龙江大豆品种由东北农业大学农学院保存。
1.2 主要试剂及仪器
主要试剂:6×His标签单克隆抗体购自生工生物工程(上海)股份有限公司;限制性内切酶、DNA聚合酶购自安诺伦(北京)生物科技有限公司;TRIzol试剂盒、1st Strand cDNA Synthesis Kit购自大连宝生物公司(TaKaRa);DAB显色试剂盒、TMB显色试剂盒购自北京索莱宝科技有限公司;EasyFusion Assembly Master Mix同源重组克隆试剂盒购自上海信裕生物科技有限公司;Ni2+离子亲和层析柱购自美国GE公司;无内毒素质粒小提中量试剂盒购自天根生化科技 (北京)有限公司;HRP标记的兔抗鼠IgG抗体和弗氏佐剂购自美国Sigma-Aldrich公司。
主要仪器:洗板机、酶标仪购自美国伯腾仪器有限公司;高速冷冻台式离心机(CT15RE型)购自日本日立公司。
1.3 AMV CP基因克隆及同源性分析
采用TRIzol法从感染AMV的大豆叶片及苜蓿叶片中提取总RNA,根据1st Strand cDNA Synthesis Kit说明书合成cDNA,并以其为模板,根据AMV CP基因保守序列设计特异性引物:AMV-CPs(5′-AGG-AGATATACCATGGGAAGTTCTTCACAAAA-GAAAGCTGGTG-3′)和AMV-CPa(5′-GGTGGTGGTGCTCGAGATGACGATCAAGATCGTCAGC-3′);以cDNA为模板,利用合成的特异性引物进行PCR扩增,PCR扩增采用10 μL反应体系:Premix Ex TaqTM 5 μL、cDNA 1 μL、10 μmol/L正反向引物各0.5 μL、ddH2O 3 μL。PCR反应体系:94℃预变性5 min;94℃变性30 s,55℃退火30 s,72℃延伸1 min,35个循环;72℃延伸10 min。PCR产物经1.5%琼脂糖凝胶电泳鉴定后送往吉林省库美科技有限公司测序验证。利用Clustal X软件对2种不同来源的AMV CP的氨基酸序列进行比对分析。
1.4 重组蛋白的表达与纯化
通过同源重组法将AMV CP基因与pET-28a载体连接,构建pET-28a-AMV重组载体,转化DH5α感受态细胞,挑取单菌落后扩大培养,提取重组质粒测序验证。将构建成功的pET-28a-AMV转入大肠杆菌BL21 Rossetta表达感受态细胞,挑取单菌落扩大培养,当OD600值为0.5~0.6时,IPTG诱导3 h,12 000 r/min离心20 min,弃上清,去离子水重悬菌体,超声裂解后分别收集上清和菌体,SDS-PAGE电泳检测,鉴定重组蛋白表达情况,通过8 mol/L尿素溶解包涵体蛋白,经Ni2+亲和层析柱得到纯化的AMV CP蛋白。纯化后的蛋白进行Western blot鉴定:重组蛋白进行SDS-PAGE电泳,一抗采用6×His标签单克隆抗体,二抗采用辣根过氧化物酶标记的兔抗鼠IgG,DAB显色液显色。
1.5 抗血清的制备及特异性分析
纯化后的AMV CP蛋白与等体积弗氏完全佐剂混合,充分乳化后对昆明鼠进行基础免疫,采用肌肉注射,每次每只小鼠免疫50 μg的重组蛋白,每隔2周以蛋白加等体积弗氏不完全佐剂加强免疫3次后尾部采血,通过间接ELISA对抗血清效价进行检测。
通过Western blot方法对制备的AMV抗血清进行特异性分析,将检测的大豆叶片用10 mL 0.01 mol/L PBS研磨后提取上清,进行SDS-PAGE电泳,一抗使用制备的AMV 抗血清,二抗采用辣根过氧化物酶标记的兔抗鼠IgG,DAB显色液显色。
1.6 间接ELISA检测方法的建立及应用
1.6.1 间接ELISA检测方法的建立
碳酸盐缓冲液(pH 9.6)稀释AMV CP蛋白至2 μg/mL后100 μL/孔包被酶标板,4℃静置过夜;加入5%脱脂奶280 μL/孔,37℃封闭1 h;检测抗体用5%脱脂奶稀释后加入酶标板,100 μL/孔,摇床65 r/min,37℃孵育1 h;酶标抗体用5%脱脂奶稀释后加入酶标板,100 μL/孔,摇床65 r/min,37℃孵育1 h,以上每一步结束后,PBST洗涤6次;加入TMB显色液100 μL/孔,避光显色8~10 min,以2 mol/L H2SO4溶液为终止液50 μL/孔终止显色;酶标仪测定OD450處吸光度值,根据公式:检测样品OD450(P值)/阴性对照OD450(N值)≥ 2.1判定检测结果为阳性。
以上试验每孔设置3个平行,同时设置阳性(感染AMV的大豆叶片)、阴性(健康的大豆叶片)、空白(去离子水)3个对照。
1.6.2 灵敏度检测
将AMV CP蛋白从1 000 ng/mL等比稀释至7.812 5 ng/mL,运用建立的检测方法对其进行检测,建立间接ELISA灵敏度曲线,并得出最低检测限。
1.6.3 样品检测
对田间采集的50份疑似感染AMV的大豆样品进行检测,分别将健康的大豆叶片、疑似感染AMV的大豆叶片按1∶10(m/V,g/mL)比例加入碳酸盐缓冲液(pH 9.6)研磨后取上清100 μL/孔,包被酶标板,运用建立的间接ELISA方法和RT-PCR方法进行验证。AMV特异性引物:AMV CPs(5′-ATGAGTTCTTCACAAAAGAAAGCTGG-3′)和AMV CPa(5′-ATGACGATCAAGATCGTCAGCTTC-3′),按1.2中步骤进行PCR扩增,对比两种方法的检测结果,验证其准确性。
1.7 吉林省大豆SMV病毒和AMV病毒感染情况调查
2021年8月,在吉林省公主岭市、辽源市、抚松市、敦化市和资源区5个地区选择连片地块面积在1 hm2以上的生产田为代表,采用五点取样法随机采集大豆样品,经液氮速冻后置于-80℃冰箱保存。利用建立的AMV间接ELISA检测方法和实验室已建立的SMV间接ELISA检测方法[22]对大豆样品进行检测,分析不同地区大豆植株的病毒感染情况。
1.8 不同大豆品种的AMV抗性评价
取感染AMV的大豆叶片加入PBS(pH 7.4)进行研磨,12 000 r/min 离心3 min,取上清液。在种植的40个不同品种的大豆叶片上喷洒500目金刚砂,吸取100 μL病毒液滴在喷洒金刚砂的叶片上,用手指轻轻涂抹3~5次,摩擦接种3 min后用清水冲洗接种叶,再覆盖一层湿纸巾保湿。观察叶片发病情况,接种21 d后,取新生叶片进行间接ELISA检测,分析不同品种大豆的感染情况。
以上试验每孔设置3个平行,同时设置阳性(感染AMV的大豆叶片)、阴性(健康的‘威廉姆斯10号大豆)、空白(去离子水)3个对照。
2 结果与分析
2.1 AMV CP蛋白的表达与鉴定
PCR产物经1.5%琼脂糖凝胶电泳检测,发现在694 bp左右有一条特异性条带,与AMV CP基因大小相符合,经测序后确定为AMV CP基因,CP蛋白序列对比结果表明(图1),来源于大豆的AMV与来源于苜蓿的AMV CP序列同源性为96%,有4个氨基酸残基不同。
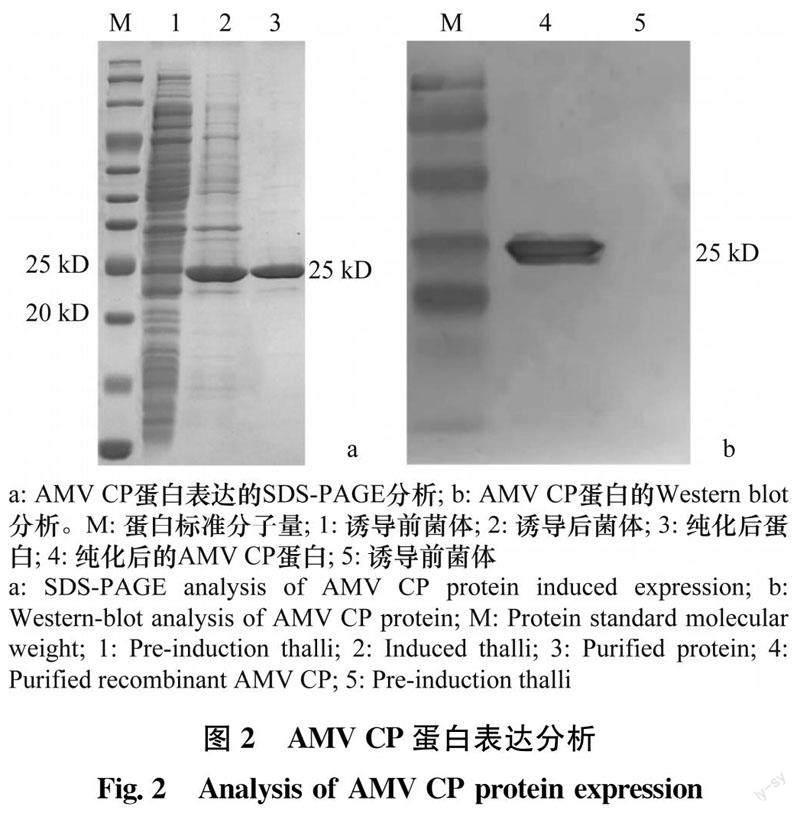
将成功构建的pET-28a-AMV转入表达菌Rossetta中,扩大培养后经IPTG诱导,分别取诱导前、后的菌体及纯化后的AMV CP蛋白进行SDS-PAGE电泳鉴定,如图2a所示,在25 kD左右有一条特异性的条带,与预期大小一致,以包涵体形式表达。Ni2+离子亲和层析柱对目的蛋白纯化后进行SDS-PAGE电泳鉴定,在25 kD左右观察到单一的目的条带,证明已得到高纯度的目的蛋白。纯化后的蛋白进行Western blot鉴定,在25 kD左右有一条清晰的特异性条带(图2b),证明纯化后的重组蛋白为AMV CP蛋白。
2.2 抗血清效价及特异性分析
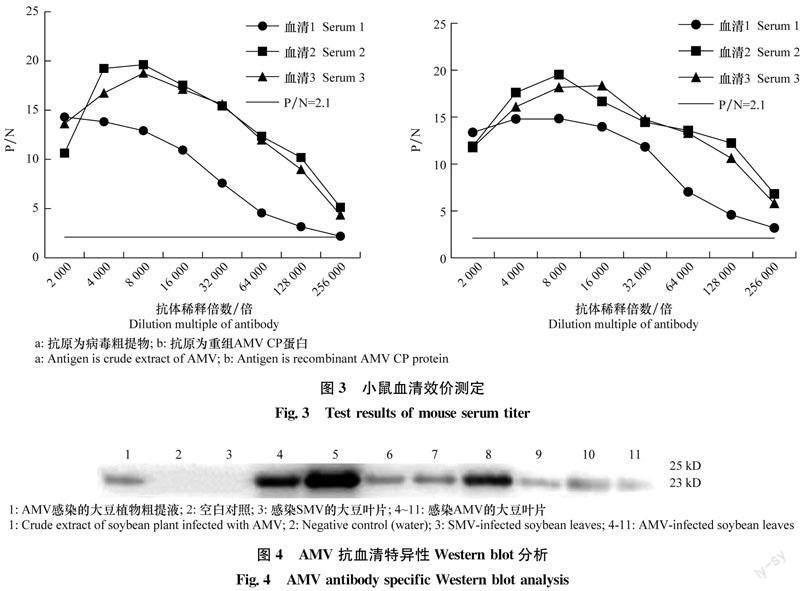
分别以AMV CP重组蛋白和感染AMV的大豆叶片粗提物为抗原包被酶标板,将免疫4次后获得的抗血清梯度稀释,采用间接ELISA检测方法对抗血清的效价进行检测,结果如图3所示,制备的3份AMV抗血清检测AMV CP蛋白和AMV感染大豆叶片蛋白粗提物,效价均能达到256 000倍。
通过Western blot检测对制备的AMV抗血清进行特异性分析,如图4所示,制备的AMV抗血清能够特异性识别感染AMV的不同品种的大豆叶片;不识别感染大豆花叶病毒soybean mosaic virus(SMV)的大豆叶片,说明制备的AMV抗血清特异性良好。
2.3 间接ELISA检测方法灵敏度
将AMV CP蛋白等比稀释,运用建立的间接ELISA检测方法进行检测,如图5所示,以AMV CP蛋白浓度为横坐标,P/N值为纵坐标建立标准曲线,得出线性方程y=0.022 2x+0.964 5,R2=0.998 5,当P/N=2.1时,AMV CP蛋白的最低检测限为46.7 ng/mL。
2.4 检测方法准确性分析
利用本研究建立的AMV间接ELISA检测方法和RT-PCR方法对50份大豆样品进行检测,两种方法检测结果均为阳性的样品数为30份,均为阴性的样品16份,ELISA检测结果为阴性而RT-PCR检测结果为阳性的样品4份,可得:一致率=(共同阳性结果份数+共同阴性结果份数)/样品总数,即92%。
2.5 吉林省大豆SMV病毒和AMV病毒感染情况调查
对采集的1 342份大豆样品进行间接ELISA检测,病毒检出率为38.30%,SMV的阳性检出率达30.85%,AMV的阳性检出率达17.06%,双病毒复合侵染的阳性检出率达9.61%,表明这两种病毒在吉林省大豆种植区普遍存在。
对不同大豆种植区病毒感染情况的调查分析表明(表1),各个产区大豆样品的感染率普遍偏高。其中敦化地区阳性检出率最高,达42.70%,公主岭地区阳性检出率相对较低,为35.75%。此次调查的5个地区中,只有辽源地区不存在病毒复合侵染的情况,其他4个产区均存在双病毒复合侵染的情况,阳性检出率分别为3.62%、9.11%、12.00%、17.88%。其中敦化地区复合侵染率最高。
2.6 大豆品种的AMV抗性评价
AMV接种40个大豆品种,接种21 d后,发现‘垦丰22等29个大豆品种出现出典型AMV感染症状,叶片大面积黄化、叶片边缘明显卷曲皱缩,植株生长受到严重影响(图6a),推测这些品种可能对AMV没有抗性;‘黑农23等11个供试大豆品种的幼苗表现出较轻微的发病症状,主要表现为叶片有黄色斑点或者并无明显的黄斑,无法直接观察到AMV感染的特征(图6b),说明这些品种可能对AMV侵染具有抗性或耐病性。运用建立的AMV间接ELISA检测方法对其进行病毒检测,结果如图7所示,40个大豆样品全部感染AMV病毒,其中‘垦丰22的OD450值最高而且高于阳性对照,结合田间症状表现,该品种是高感AMV品种;‘黑农23‘黑农24‘黑农30‘黑农34‘黑农44‘黑农71‘中龙606‘黑农55‘垦丰17‘黑河3号‘绥农53的OD450值在0.2到0.25之间,病毒载量较低,结合田间症状评价,这11个品种具有AMV抗性;其余28个品种OD450在0.25到0.4之间,病毒载量较高。表明不同大豆品种对AMV的抗性有明显差异。
3 结论与讨论
大豆病毒种类较多,仅凭表型难以区分,通常采用核酸和血清学检测技术进行甄别[21-24]。血清学检测技术简单方便、耗时较短、更适合基层人员使用[27-28]。但是AMV的血清学检测技术研究的较少,尤其是感染大豆的AMV研究更是鲜有报道。本研究从表现花叶和叶片坏死症状的大豆植株上获得了AMV分离物,并克隆了大豆AMV分离物的CP基因,不同来源(大豆和苜蓿)的AMV CP蛋白有4个氨基酸残基不同,因此,以AMV大豆分离物CP蛋白制备的抗体对大豆源AMV更具特异性和敏感性。以大肠杆菌重组表达的CP蛋白为免疫原制备的抗血清能特异性识别AMV病毒,而且效价非常高,说明大肠杆菌重组表达的AMV CP蛋白的免疫特性与天然病毒CP蛋白相似,而重组表达蛋白比病毒纯化要简便很多。另外,本研究选择在载体pET-28b的Nco Ⅰ和Xho Ⅰ两个酶切位点之间插入CP基因,这样表达出来的重组CP蛋白除了His标签以外,只有自身的序列,能夠最大程度体现AMV CP蛋白的抗原特性。
以本研究获得的抗体为基础建立的大豆源AMV间接ELISA检测方法为田间样品检测提供了高效、精准的检测技术,经试验证明,准确性达到92%。本研究还证明该ELISA检测方法适用于苜蓿的AMV检测(数据未列出)。利用本研究获得的重组CP蛋白,通过免疫BABL/c小鼠和细胞融合技术,可以制备AMV的单克隆抗体,建立双抗体夹心ELISA,进一步提高AMV检测的灵敏度和准确性。
吉林省是我国大豆种植的主产区,本研究首次对吉林省田间大豆植株进行AMV和SMV感染调查,结果表明,两种病毒在大豆产区普遍存在,且存在复合侵染的情况,占全部样品的9.61%;Malapi-Nelson等研究发现SMV与AMV复合侵染会导致大豆植株出现更为严重的症状[30]。从本次调查获得的吉林省AMV检出率(17%)来看,AMV已经成为影响吉林省大豆生产的主要病毒之一。敦化是吉林省大豆的主产区,来自敦化地区的大豆植株样品中,AMV检出率超过30%,为吉林省最高,并且复合感染率也达到了17.88%,个别田块受AMV影响而大幅减产(产量损失大于35%),因此,针对AMV的防控技术和抗病育种研究显得越来越重要。气候变化和吉林省特殊的地理位置,导致蚜虫等传播介体种群数量逐年增长,可能是大豆AMV病害流行增加的原因之一,针对传播介体的防控也应该受到足够的重视[29]。
抗病育種是防控病毒病危害的有效手段,抗性鉴定是抗病育种的重要环节,国内学者培育和推广了很多抗SMV的大豆品种,但是至今仍未有抗AMV和SMV的大豆双抗品种和抗AMV的大豆单抗品种。应加强抗性资源的挖掘和AMV致病性与大豆抗病性分子机制的研究,选育抗性品种。本研究对黑龙江地区40个大豆主栽品种进行了AMV接种,接种21 d 后运用建立的间接ELISA方法对其进行病毒检测。试验结果显示,40个大豆品种全部感染AMV病毒。本研究还发现部分感染AMV的大豆没有明显症状,属于隐症感染,因此病毒检测有助于提高AMV抗性鉴定的准确性。本研究未发现对AMV免疫的大豆品种,但部分品种表现一定水平的抗性。结合AMV检测技术,进一步对大豆种质资源进行AMV抗性鉴定,筛选抗性资源[31-32],进而为品种选育提供科学依据。
参考文献
[1] 原梓涵, 邵娜. 大豆振兴计划实施背景下中国大豆市场分析及未来展望[J]. 农业展望, 2019, 15(6): 4-9.
[2] 李梅蓉, 孙媛, 卢建霖, 等. 苜蓿花叶病毒的研究进展[J]. 贵州农业科学, 2019, 47(4): 69-73.
[3] HULL R. Alfalfa mosaic virus [J]. Advances in Virus Research, 1969, 15: 365-433.
[4] 金磊磊. 一株苜蓿花叶病毒的全基因组序列及其寄主生物学研究[D]. 杭州: 浙江理工大学, 2011.
[5] 郭志鹏, 严学兵, 冯长松, 等. 30个紫花苜蓿品种对苜蓿花叶病毒病的田间抗性初步研究[J]. 草业学报, 2019, 28(4): 157-167.
[6] 文朝慧, 南志标. 甘肃省张掖地区苜蓿花叶病病原的检测[J]. 草业学报, 2015, 24(4): 121-126.
[7] 赵桂琴, 慕平. 苜蓿花叶病毒外壳蛋白基因对红三叶的遗传转化及转基因植株的抗病性分析[J]. 西北植物学报, 2004, 24(10): 1850-1855.
[8] 张若男. 豆类病毒病害调查及芸豆皱缩矮化病病原鉴定与分析[D]. 哈尔滨: 东北农业大学, 2020.
[9] ROSSUM C M A V, BREDERODE F T, NEELEMAN L, et al. Functional equivalence of common and unique sequences in the 3′ untranslated regions of alfalfa mosaic virus RNAs 1, 2, and 3 [J]. Journal of Virology, 1997, 71(5): 3811-3816.
[10]何宛芹. 凤果花叶病毒和苜蓿花叶病毒单克隆抗体的创制及其应用[D]. 杭州: 浙江大学, 2021.
[11]GAO Shiqi, LIANG Jing, GUO Kai, et al. First report of alfalfa mosaic virus in Cayratia japonica, Justicia procumbens and Veronica persica in China [J]. Journal of Plant Pathology, 2019, 102(3): 267.
[12]ROZANOV M N, KOONIN E V, GORBALENYA A E. Conservation of the putative methyltransferase domain: a hallmark of the ‘Sindbis-like supergroup of positive-strand RNA viruses [J]. Journal of General Virology, 1992, 73(8): 2129-2134.
[13]BOL J F. Alfalfa mosaic virus and ilarviruses: Involvement of coat protein in multiple steps of the replication cycle [J]. Journal of General Virology, 1999, 80(5): 1089-1102.
[14]GUOGAS L M, LAFOREST S M, GEHRKE L. Coat protein activation of alfalfa mosaic virus replication is concentration dependent [J]. Journal of Virology, 2005, 79(9): 5752-5761.
[15]REUSKEN C B, NEELEMAN L, BOL J F. The 3′ untranslated region of alfalfa mosaic virus RNA3 contains at least two independent binding sites for viral coat protein [J]. Nucleic Acids Research, 1994, 22(8): 1346-1353.
[16]HOUSER-SCOTT F, BAER M L, LIEM K F, et al. Nucleotide sequence and structural determinants of specific binding of coat protein or coat protein peptides to the 3′untranslated region of alfalfa mosaic virus RNA 4[J]. Journal of Virology, 1994, 68(4): 2194-2205.
[17]尤晴, 李小宇, 张春雨, 等. 马铃薯Y病毒属三种病毒通用型单克隆抗体的鉴定[J]. 植物保护, 2016, 42(6): 76-79.
[18]张芯伪. 新疆苜蓿丛枝病和花叶病病原分子鉴定与检测[D]. 乌鲁木齐: 新疆农业大学, 2016.
[19]XU H, NIE J. Identification, characterization, and molecular detection of alfalfa mosaic virus in potato [J]. Phytopathology, 2006, 96(11): 1237-1242.
[20]韩俊丽, 郭庆元, 杨知还, 等. 国家种质库保存大豆和菜豆种质的种传病毒检测[J]. 植物遗传资源学报, 2010(3): 284-289.
[21]AHOONMMANESH A, HAJIMORAD M R, INGHAM B J, et al. Indirect double antibody sandwich ELISA for detecting alfalfa mosaic virus in aphids after short probes on infected plants [J]. Journal of Virological Methods, 1990, 30(3): 271-281.
[22]李小宇, 张春雨, 张伟, 等. 大豆花叶病毒间接ELISA检测方法的建立及应用[J]. 东北农业科学, 2019, 44(1): 22-27.
[23]HILL J H, WHITHAM S A. Control of virus diseases in soybeans [J]. Advances in Virus Research, 2014, 90: 355-390.
[24]KIM E K, YOU J W, PARK J S, et al. Detection and identification of a mixed infection of three viruses in Chinese artichoke in Korea [J]. Research in Plant Disease, 2018, 24(1): 81-85.
[25]李小宇, 张春雨, 郭东全, 等. 双抗夹心ELISA检测转Bar基因抗除草剂大豆[J]. 食品科学, 2016, 37(4): 222-225.
[26]梁雨欣, 吴建祥, 李小宇, 等. 马铃薯Y病毒衣壳蛋白抗原表位分析及其快速ELISA检测方法的建立[J]. 中国农业科学, 2021, 54(6): 1154-1162.
[27]张春雨, 李小宇, 萬千, 等. 马铃薯M病毒衣壳蛋白原核表达和抗血清制备[J]. 东北农业科学, 2017, 42(3): 27-30.
[28]王永志, 万千, 李小宇, 等. OYDV衣壳蛋白表达及其单克隆抗体分析[J]. 东北农业科学, 2018, 43(2): 26-29.
[29]刘伟, 郭秀珍. 吉林省大豆病虫害发生情况及防治技术[J]. 现代农业科技, 2019(22): 76.
[30]MALAPI-NELSON M, WEN R H, OWNLEY B H, et al. Co-infection of soybean with soybean mosaic virus and alfalfa mosaic virus results in disease synergism and alteration in accumulation level of both viruses [J]. Plant Disease, 2009, 93(12): 1259-1264.
[31]ABDALLA O A, AL-SHAHWAN I M, ALSALEH M A, et al. Molecular characterization of alfalfa mosaic virus (AMV) isolates in alfalfa and other plant species in different regions in Saudi Arabia [J]. European Journal of Plant Pathology, 2020, 156(2): 603-613.
[32]任锐. 大豆抗大豆花叶病毒病基因的精细定位和候选基因分析[D]. 南京: 南京农业大学, 2017.
(责任编辑:田 喆)
