小麦白粉病抗性的全基因组关联分析
2023-05-19宋茂兴马宏亮吴志会
陆 晴, 宋茂兴, 马宏亮, 吴志会
(唐山市农业科学研究院, 河北 唐山 063000)
小麦(TriticumaestivumL.)是重要的粮食作物之一[1-3]。小麦白粉病是一种危害小麦地上部分的病害,作为小麦生长期的一种主要病害,会对小麦的产量及品质产生严重影响[4]。小麦白粉病主要病原菌是禾谷类白粉菌(Blumeriagraminisf. sp.Tritici),是典型的子囊菌亚门白粉菌目布氏白粉菌属中的一种专性活体寄生菌,湿润环境有利于其病原体的繁殖及传播[5]。我国白粉病最初多发生在四川、云南等气候湿润地区,但是随着近年来气候的变化、小麦株高的普遍降低导致的群体内湿度上升等原因,白粉病的发生区域逐渐北移[6]。农药能在一定程度上抑制白粉病的发生,但防治白粉病的根本、有效、经济的方法仍然是培育抗病品种。
抗病基因的定位、发掘及利用能够有效提高抗病育种的效率,迄今为止已经被正式命名的抗白粉病基因有64个[7],但多数抗病基因随着病原菌小种不断变异而丧失抗性[8],目前国内较为有效的小麦白粉病抗性基因有Pm12,Pm13,Pm16,Pm20,Pm21,Pm30[9]。近年来不断有学者克隆出新的白粉病抗性基因,董振杰[10]利用小片段易位系将Pm57精细定位到了X 67593和X 62492两个分子标记之间5.13 Mb区间内,并对候选基因comp430422_c0、comp4364_c0、eomp62492_c0进行了功能验证;杜习军[4]利用自然群体结合高密度芯片对小麦白粉病抗性进行了全基因组关联分析,关联到34个与苗期白粉病抗性显著相关的SNP,其中位于7 A染色体上的AX-110071156、AX-94416898,3 B染色体上的AX-94473502和1 D染色体上的AX-109436089为新的小麦抗白粉病位点。随着小麦白粉病菌小种的不断变异,现有抗病基因的抗性逐渐降低,需要不断挖掘新的优异等位基因,从而培育出新的抗病品种。
随着基因芯片检测技术逐步完善,基于SNP标记的全基因组关联分析被广泛地应用在小麦各种复杂性状的遗传解析中[11]。本研究以来源于我国华北、华东、华中等地区的215份小麦品种(系)为材料,利用小麦16 K高密度SNP芯片进行小麦成株期白粉病抗性的全基因组关联分析,发掘小麦白粉病抗性位点,以期为克隆小麦白粉病优异等位基因及抗性遗传改良提供理论依据。
1 材料与方法
1.1 试验材料及种植方法
本研究选取215个小麦品种(系)构成自然群体,来源于我国华北、华东、华中等多个地区,其中河北省133个、北京市37个、山东省28个、河南省3个、山西省9个。试验材料于2020—2021年及2021—2022年分别种植于唐山市农业科学研究院试验田(唐山市)和滦州市龙泽农作物研究所(滦州市),共4个不同环境。试验采用随机区组设计,3次重复,1 m行长,20 cm行距,按照河北省区域试验标准进行田间管理及数据调查。
1.2 白粉病抗性鉴定及数据统计
病害诱发方法采用田间自然诱发,每5行材料种植1行白粉病高感材料(7 DL·7 Ag)作为诱发材料,待成株期7 DL·7 Ag材料发病明显时对白粉病发病等级进行调查。病害等级分5级记载,1级:叶片无肉眼可见症状;2级:基部叶片发病;3级:病斑蔓延至中部叶片;4级:病斑蔓延至剑叶;5级:病斑蔓延至穗及芒。
用Excel 2010软件对表型数据进行统计,用SPSS Statistics 19软件对表型数据的均值、方差等进行计算分析。
1.3 全基因组SNP芯片分型
在小麦拔节期前,每个材料取5~7片嫩叶,将取得的叶片迅速冷冻在-80 ℃冰箱中,随后用干冰低温运输,委托石家庄博瑞迪生物技术有限公司进行总DNA提取、使用小麦16 K高密度SNP芯片进行群体DNA基因分型;去除位点检出率<90%的样本及低频基因型频率(MAF)<5%和缺失数据>30%的SNP标记,最终得到共计210个高质量样本及9 501个高质量SNP标记用于小麦白粉病抗性的全基因组关联分析。
1.4 群体结构分析
从筛选的标记中,随机选取2 000个最小等位基因频率大于10%,且在染色体上均匀分布的SNP,利用Structure 2.3.4软件进行群体结构分析;参数设置为:Length of Burnin Period=10 000,Number of MCMC Reps after Burnin=10 000,选择Admixture Model模型,设置K=2~12,每个K值重复5次。将运行结果打包成zip格式文件上传到在线分析软件Structure Harvester(https://taylor 0.biology.ucla.edu/structure Harvester/)计算ΔK,将出现明显峰值的ΔK所对应的K值作为亚群数目。
在Structure 2.3.4软件运行的结果中选择最佳K值对应的5个重复的分析结果,整合成CLUMPP分析软件能够读取的indfile格式文件,用CLUMPP将结果合并为1个Q值矩阵,利用GraphPad Prism软件结合Q值矩阵绘制群体结构图。
1.5 全基因组关联分析
将210个高质量样本及9 501个高质量SNP标记信息整理成TASSEL 5.0软件可读的MAP和PED文件,并使用TASSEL 5.0软件合并成plink数据。在TASSEL 5.0中选取plink基因型数据进行主成分分析(PCA)及亲缘关系矩阵(Kinship)值计算;将PCA、plink、表型值合并后,结合Kinship值,采用混合线性模型(MLM)对白粉病性状及SNP标记之间进行关联分析,设置阈值p<0.001。
2 结果与分析
2.1 SNP标记分布及群体结构分析
通过小麦16 K高密度SNP芯片对群体DNA进行基因分型,共检测出SNP标记总数为37 669个,其中包含核心SNP标记14 868个,其在A、B、D亚基因组中的分布情况分别为5 681个、5 939个和3 248个(图1)。去除位点检出率<90%的样本及低频基因型频率(MAF)<5%和缺失数据>30%的SNP标记,最终得到共计210个高质量样本及9 501个高质量核心SNP标记用于小麦白粉病抗性的全基因组关联分析。

注:横轴表示SNP标记所在物理位置,纵轴表示SNP标记所在染色体。图1 目标SNP位点在基因组上的分布情况Fig.1 Distribution of target SNPS across the genome
利用Structure 2.3.4软件结合Structure Harvester在线分析软件对群体结构进行分析,结果显示,当ΔK取得最大值时,K=3,所以将关联分析群体划分为三个类群(图2)。第一类群含83个品种(系),主要来自于河北及山东,第二类群含42个品种(系),主要来自于河北,第三类群含85个品种(系),主要来自于河北、北京及山西。
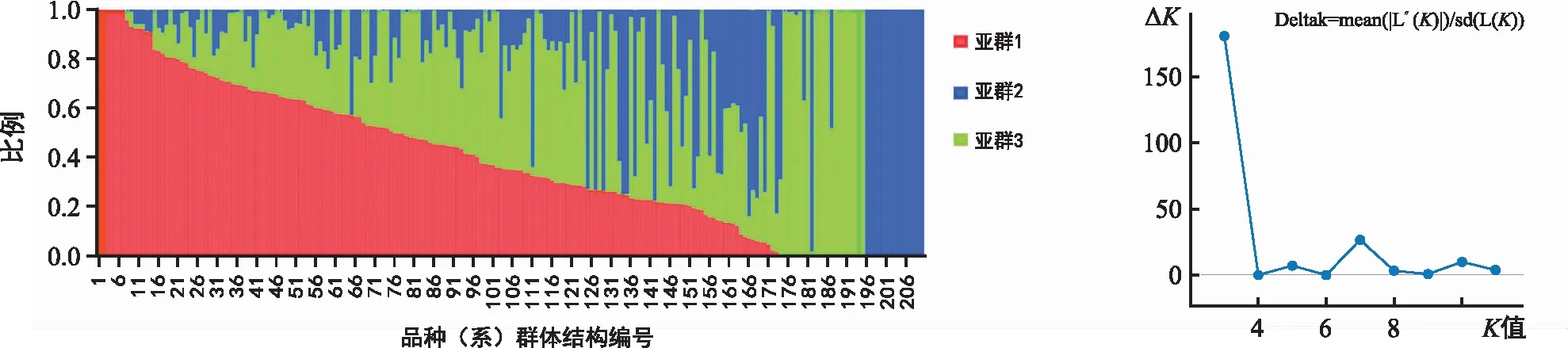
图2 210个小麦品种(系)的群体结构图(左)及K值折线图(右)Fig.2 The population structure map (left) and K value line chart (right) of 210 wheat varieties (lines)
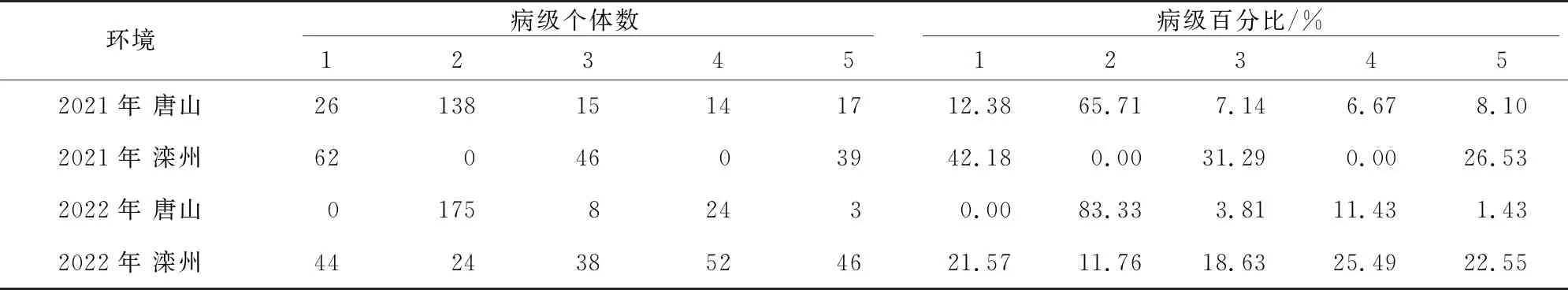
表1 4种环境中各病级个体数分布情况Table1 Distribution of individuals of each disease grade in the four environments
2.2 白粉病抗性鉴定结果
在不同环境条件下,5个病级所对应的个体数有所不同,小麦白粉病表型受环境因素的影响,在不同环境中鉴定结果会有差异(表1)。将病级1~2级规定为抗病表型,病级3~5级为感病表型;在2021年唐山环境和2022年唐山环境中,群体整体发病程度较轻,抗病表型个体数占比分别为78.09%、83.33%,感病个体数占比分别为21.91%、16.67%;在2021年滦州环境和2022年滦州环境中,群体整体发病程度较重,抗病表型个体数占比分别为42.18%、33.33%,感病个体数占比分别为57.82%、66.67%,其中病级为5的个体数占比分别达26.53%、22.55%。环境因素对小麦白粉病表型鉴定有一定影响,多年多点鉴定有利于降低环境造成的误差。
2.3 白粉病抗性全基因组关联分析
试验采用小麦16 K高密度SNP芯片的9 501个高质量核心SNP标记,结合210个高质量样本的白粉病抗性表型,对小麦白粉病抗性显著关联位点进行全基因组关联分析,共检测到分布于1 A(3)、1 D、2 A、2 B(4)、2 D(2)、3 A、3 B(2)、4 A(2)、4 D、5 A、5 B(2)、5 D、6 A、6 B、7 B、7 D染色体上的25个与白粉病抗性显著关联的SNP位点(图3、表2)。
在2021年唐山环境中,位于1 A(3)、1 D、2 A、3 A、4 D、6 B染色体上的8个位点与白粉病抗性显著关联;在2021年滦州环境中,位于3 B、4 A、7 D染色体上的3个位点与白粉病抗性显著关联;在2022年唐山环境中,位于2 B、2 D(2)、4 A、5 A、5 B(2)、5 D、7 B染色体上的9个位点与白粉病抗性显著关联;在2022年滦州环境中,位于2 B(3)、3 B(2)、6 A染色体上的6个位点与白粉病抗性显著关联。山环境中,位于2 B、2 D(2)、4 A、5 A、5 B(2)、5 D、7 B染色体上的9个位点与白粉病抗性显著关联;在2022年滦州环境中,位于2 B(3)、3 B(2)、6 A染色体上的6个位点与白粉病抗性显著关联。
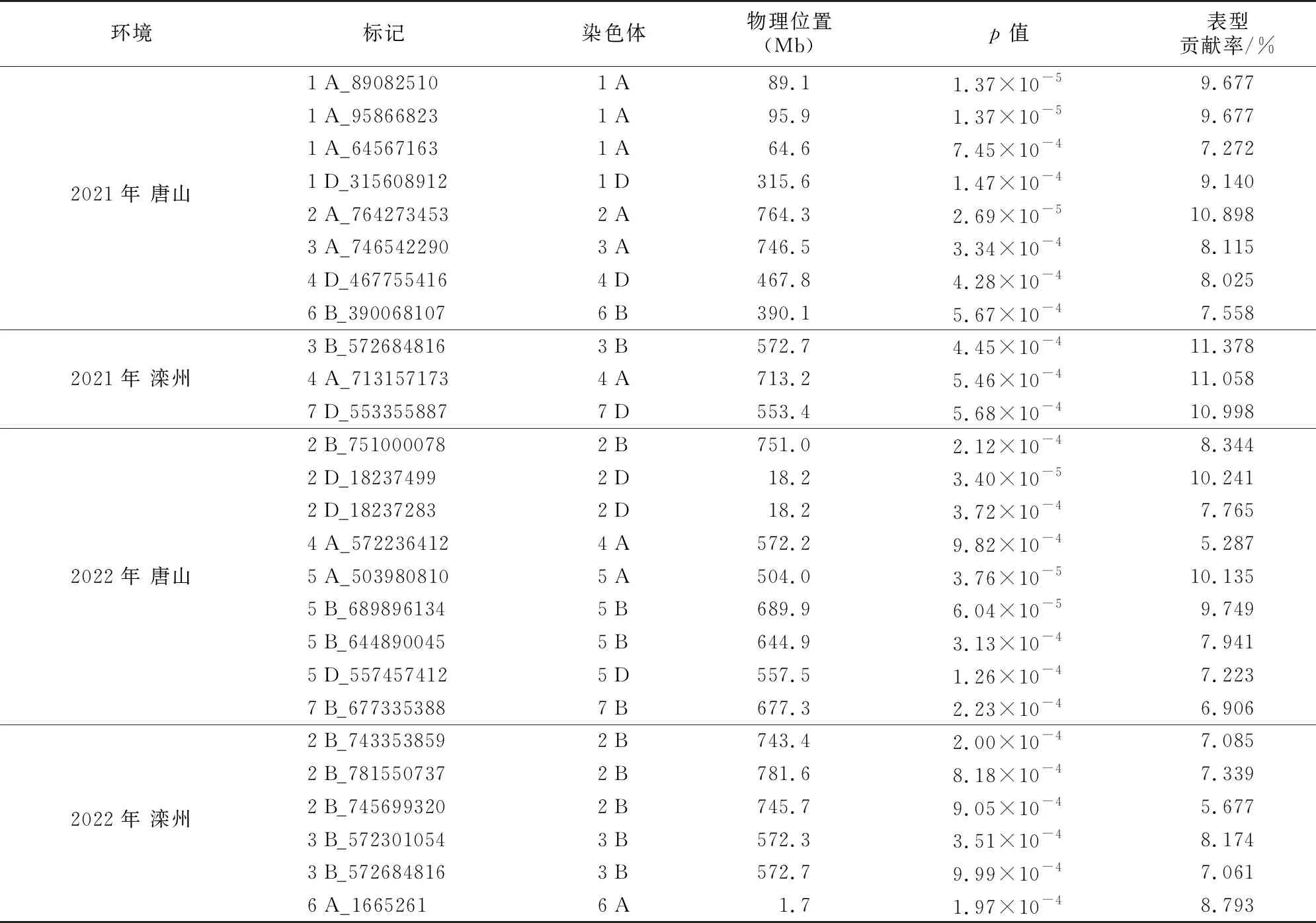
表2 与小麦白粉病抗性显著相关的SNP位点Table 2 SNP markers significantly associated with wheat powdery mildew resistance
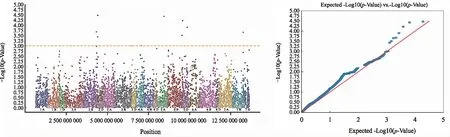
图3 全基因组关联分析曼哈顿图(左)和Q-Q图(右)Fig.3 Manhattan (left) and Quantile-Quantile plot (right) of GWAS
在关联出的25个与白粉病抗性显著关联的SNP位点中,有多个区段(位点)在多个环境中被检测到:位于2 B染色体的743.4~781.6 Mb区段内的2 B_743353859、2 B_745699320、2 B_781550737在2022年滦州环境中被检测到,2 B_751000078在2022年唐山环境中被检测到,该区段表型贡献率为5.677%~8.344%;位于3 B染色体上的3 B_572684816在2021年滦州环境和2022年滦州环境中被检测到,该位点表型贡献率为7.061%~11.378%。
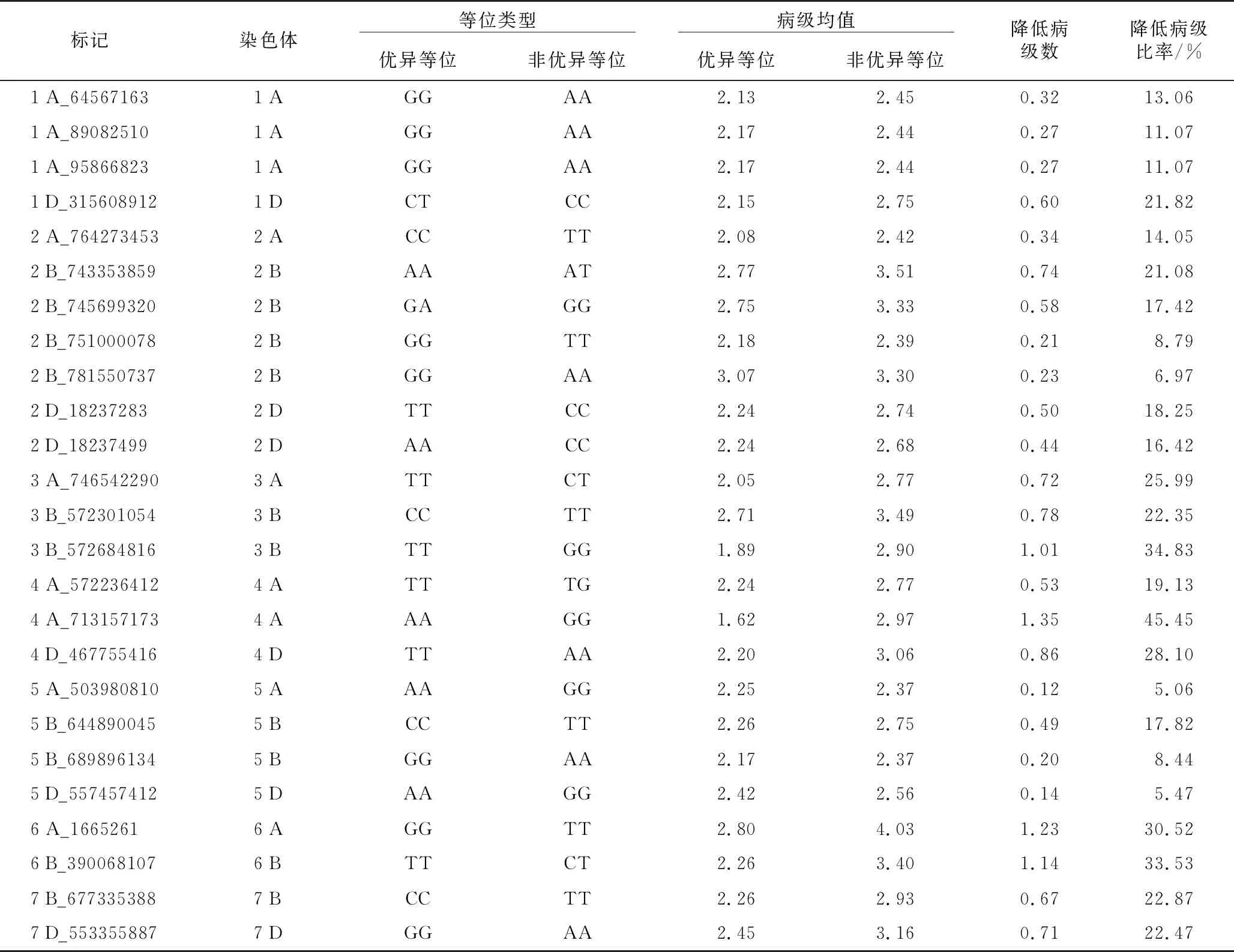
表3 白粉病抗性显著关联位点的优异等位变异Table 3 Superior alleles variation significantly associated with wheat powdery mildew resistance
2.4 白粉病抗性显著关联位点优异等位变异
对白粉病抗性显著关联位点优异等位变异进行分析(表3),以等位基因对应的病级均值为衡量标准,优异等位基因对应的病级均值变化范围为1.62~3.07,非优异等位基因对应的病级均值变化范围为2.37~4.03;优异等位基因比非优异等位基因的病级均值降低0.12~1.35、病级比率降低5.06%~45.45%。从个体角度分析,TM 007多个环境表现抗病,其4个环境的平均病级为1.5,含有14个优异等位变异;TM 124多个环境表现感病,其4个环境的平均病级为4.25,含有9个非优异等位变异。发掘白粉病抗性显著关联位点优异等位变异有利于小麦抗白粉病分子标记的开发,以提高表型选择的准确性。
3 讨 论
3.1 环境因素对白粉病表型鉴定的影响
各种环境因素(气候、地块、管理方式等)均会对小麦白粉病表型鉴定产生较大影响,在2021年唐山环境和2022年唐山环境中,灌溉方式为浅埋滴灌,水肥管理及时,植株生长环境整体较好,外加小麦生长期间的降雨量少、空气湿度小等因素影响,群体整体发病程度较轻,抗病表型个体数占比分别为78.09%、83.33%;而在2021年滦州环境和2022年滦州环境中,采用多次喷灌方式,夏季高温导致空气湿度大,有利于白粉病菌繁殖传播,群体整体发病程度较重,抗病表型个体数占比分别为42.18%、33.33%,而感病个体中病级为5的个体数占比分别达26.53%、22.55%。由此可见,环境因素对小麦白粉病表型影响较大,而多年多点鉴定有利于降低环境造成的误差,在不同环境中均能检测到的小麦白粉病抗性显著关联位点结果可靠、遗传稳定性较高。
3.2 小麦白粉病抗性的全基因组关联分析
在小麦白粉病抗性的全基因组关联分析中,共检测到分布于1 A(3)、1 D、2 A、2 B(4)、2 D(2)、3 A、3 B(2)、4 A(2)、4 D、5 A、5 B(2)、5 D、6 A、6 B、7 B、7 D染色体上的25个与白粉病抗性显著关联的SNP位点,其中在2 B和3 B染色体上各有1个区段(位点)在多个环境中被检测到:位于2 B染色体的743.4~781.6 Mb区段内的2 B_743353859、2 B_745699320、2 B_781550737在2022年滦州环境中被检测到,区段内的2 B_751000078在2022年唐山环境中被检测到;位于3 B染色体上的3 B_572684816在2021年滦州环境和2022年滦州环境中被检测到。
目前在世界范围内报道了一系列与小麦白粉病抗性相关的基因,在2 B染色体上正式命名了多个小麦白粉病抗病基因,包含Pm26、Pm33、Pm42、Pm49、Pm51、Pm52、Pm63、Pm64、Pm68[12-20],但目前均未成功克隆;而目前在3 B染色体上只有1个小麦白粉病抗病基因被正式命名且成功克隆(Pm41)[21]。
本研究中检测到的位于2 B染色体上的区段2 B_743353859~2 B_781550737与上述已命名的抗病基因中的Pm51相近[16],最小距离为3.95 Mb,推测本研究结果与前人结果相似,为同一位点;且该区段包含了杜习军[4]利用小麦660 K芯片检测到的一个位于2 B染色体上的SNP位点AX-110471573(物理位置753837087),也可以认为是同一位点。目前已报道的在3 B染色体上与白粉病抗性相关的基因位点较少,本研究中位于3 B染色体上的显著关联位点3 B_572684816与该染色体上唯一被命名的白粉病抗性基因Pm41(物理位置429382520)[21]相距143.30 Mb,推测不是同一位点,可能是一个新位点。
4 结 论
本试验利用小麦16 K高密度SNP芯片对来源于我国华北、华东、华中等地区共215份小麦种质资源进行白粉病抗性全基因组关联分析(GWAS),检测到25个与小麦白粉病抗性显著相关的SNP位点,其中位于2 B染色体上的1个38.2 Mb的区段及位于3 B染色体上的1个位点在多个环境中被检测到;位于2 B染色体上的区段与已命名的小麦白粉病抗性基因Pm51距离相近,推测为同一位点,而位于3 B染色体上的位点推测可能是一个新位点。以上结果为小麦白粉病抗病育种提供了重要参考和标记资源。
