新型冠状病毒主蛋白酶天然产物抑制剂的筛选
2023-04-29师云凡鞠壮丁显平
师云凡 鞠壮 丁显平

摘 要 :为寻找新型冠状病毒主蛋白酶天然产物抑制剂,本文以SARS-CoV-2主蛋白酶为研究对象,通过计算机辅助药物筛选方法初步筛选出16个潜在抑制剂并进行体外酶活测试.其中,野黄岑素对SARS-CoV-2主蛋白酶的IC 50为(10.79 ± 2.116 ) μmol/L,没食子儿茶素没食子酸酯抑制SARS-CoV-2主蛋白酶的IC 50为(2.104 ± 0.346) μmol/L.最后,将两种天然产物与主蛋白酶进行分子动力学模拟发现没食子儿茶素没食子酸酯和野黄岑素与新型冠状病毒主蛋白酶结合时的复合物RMSD都在0.22 附近,说明两个天然产物与新型冠状病毒主蛋白酶结合较为稳定,可以作为新型冠状病毒主蛋白酶的抑制剂或先导化合物.
关键词 :新型冠状病毒主蛋白酶; 基于结构的虚拟筛选; 没食子儿茶素没食子酸酯; 野黄岺素; 分子动力学模拟
中图分类号 :Q55;R914 文献标识码 :A DOI : 10.19907/j.0490-6756.2023.046001
收稿日期: 2022-11-29
基金项目: 四川省科技厅项目(2020JDS0039)
作者简介: 师云凡(1997-), 女, 河南安阳人, 硕士研究生, 研究方向为计算机辅助药物设计.E-mail: 2811829836@qq.com
通讯作者: 丁显平.brainding@scu.edu.cn
Screening of natural product inhibitors targeting SARS-CoV-2 main protease
SHI Yun-Fan 1,2, JU Zhuang 1,2, DING Xian-Ping 1,2
(1. College of Life Sciences, Sichuan University, Chengdu 610065, China;
2. Joint Key Lab of Sichuan&Chongqing, Bioresource Res & Utilizat, Nanchuan 408400, China)
To screen natural product inhibitors targeting the SARS-CoV-2 main protease, this paper takes the main peoteases of SARS-Cov-2 as the research object, and 16 potential inhibitors were preliminarily screened by computer-aided drug screening method and tested for enzyme activity in vitro. Among them, the IC 50 of scutellarin against SARS-CoV-2 main protease was (10.79 ± 2.116) μmol/L. The IC 50 of (-)-Gallocatechin gallate against SARS-CoV-2 main protease was (2.104 ± 0.346) μmol/L. Finally, molecular dynamics simulations were performed on the two natural products and Mpro. It was found that the RMSD of the complexes of (-)-Gallocatechin gallate and Scutellarein with Mpro was around 0.22 , indicating that the two natural products were stable in binding with Mpro and could be used as inhibitors or lead compounds of Mpro in SARS-CoV-2.
Mpro of SARS-CoV-2; Structure-based virtual screening; (-)-Gallocatechin gallate; Scutellarein; Molecular dynamic simulation
1 引 言
2019年12月底,中国武汉报告了一例不明原因肺炎病例,随后世界上各个国家相继报告类似症状病例,后经研究表明,这是一种新型冠状病毒导致的肺炎疫情,由于波及范围之广,因此被称为大流行病(Pandemic) [1]. 测序结果显示,新型冠状病毒与SARS-CoV具有79%的序列一致性 [2],由此被世界卫生组织命名为SARS-CoV-2,由SARS-CoV-2引起的疾病称为COVID-19 [3].
新型冠状病毒为 β 属类冠状病毒,具有较大的单股正链RNA,其基因组含约30000个核苷酸,从5′→3′端依次编码两种多聚蛋白和四种结构蛋白,中间穿插编码几种辅助蛋白.两种多聚蛋白被病毒中两种主要的蛋白酶类胰凝乳蛋白酶和类木瓜蛋白酶切割成16种非结构蛋白,其中,类木瓜蛋白酶负责nsp1、nsp2和nsp3的切割,而类胰凝乳蛋白酶则负责后13种非结构蛋白的切割,这16种非结构蛋白发挥各自作用从而促进病毒的复制和表达 [4-6].
类胰凝乳蛋白酶也被称作主蛋白酶,由于主蛋白酶在多种冠状病毒中具有较强的保守性,并且目前没有已知的人类半胱氨酸蛋白酶与其具有相似的切割位点,因而成为治疗新型冠状病毒的一个理想靶点 [7,8].
尽管目前全球已经大范围内接种疫苗,但与该病毒导致的疾病相关问题还远远没有解决,许多抗体药物被研发出来也被批准但只能临床应用于危重症患者,也有相关主蛋白酶抑制药物如Paxlovid被FDA批准用于新型冠状病毒患者的治疗,但正有研究表明单一药物使用可能会造成病毒一些突变体耐药,因此新的抑制剂需要被开发以应对此次大流行病 [9,10].
随着结构生物学和生物信息学等学科的不断发展,计算机辅助药物筛选与设计在当下展现了极大的优势,在较短时间内筛选出SARS-CoV-2主蛋白酶的有效抑制剂,将有利于后续药物设计与改进,为新型冠状病毒的治疗提供理论基础和新思路.
2 材料与方法
2.1 材 料
文中所涉及的天然产物购买于上海陶术公司;新型冠状病毒主蛋白酶抑制剂筛选试剂盒(增强型)、DMSO和全黑96孔细胞培养板购自于碧云天.
2.2 方 法
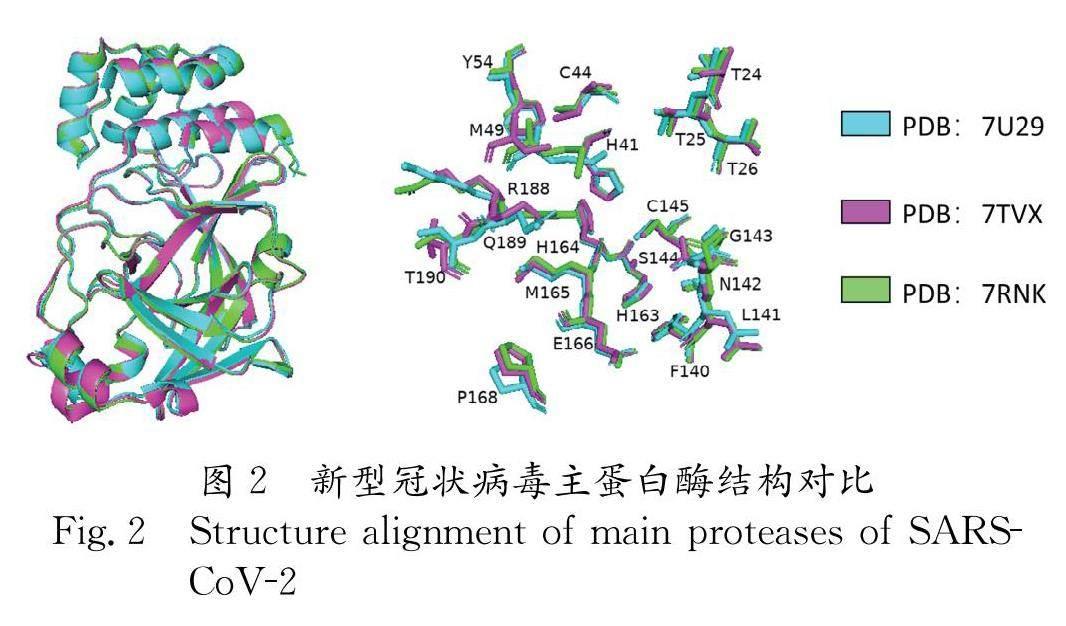
2.2.1 SARS-CoV-2主蛋白酶变种序列和结构比对 于NCBI上下载SARS-CoV-2突变株B.1.1.7、B.1.351、P.1、B.1.617.2、BA.1、BA.1.1、BA.2、BA.3的核苷酸序列;利用ExPASy-Translate Tool将核苷酸序列转化为氨基酸序列;在DNAMAN中载入氨基酸序列,点击Sequence Alignment-Multiple Sequence Alignment,添加氨基酸序列,点击Protein,进行快速比对. 之后利用RCSB PDB数据库中的PairWise Structure Alignment模块进行了结构对比评估野生型主蛋白酶的突变型主蛋白酶差异. 从RCSB PDB数据库中下载7U29(K90R、2.088 )、7TVX(P132H、2.094 )和7RNK(2.1 ),利用Pymol处理蛋白,去除水分子和配体,进行蛋白结构对比,突出展示活性口袋叠合构象.
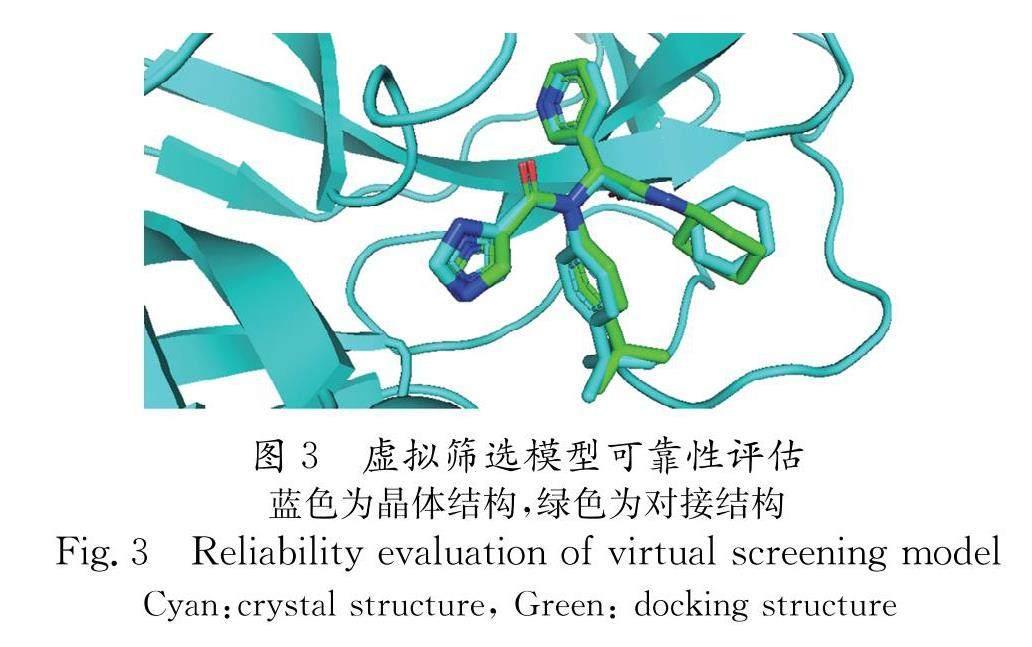
2.2.2 验证基于虚拟筛选模型可靠性 从RCSB PDB数据库下载6LU7结构,用Pymol处理去除水分子和配体,保留蛋白单体,6W63结构则是去除蛋白和水,保留X77配体结构,再用Autodock Tool对蛋白进行加氢计算电荷操作,接着对X77进行探测可旋转键操 作,之后均保存为pdbqt格式,以文献所查阅新型冠状病毒主蛋白酶活性氨基酸作为中心创建对接盒子,盒子具体信息为 center_ x = -10.351、center_ y = 13.549、center_ z = 69.904、 size_ x = 22.5、size_ y = 28.5和size_ z = 26.25. 利用Qvina2.1对X77和主蛋白酶进行分子对接,所选用的exhaustiveness为24,对接模式为9种,能量差为3. 对接完成之后,利用Pymol对比X77打分最高的对接结构和晶体结构,并计算其RMSD [11,12].
2.2.3 天然产物数据库准备 天然产物数据库(L6000)中的小分子配体是由陶术公司用Discovery Studio中的ligprep模块经OPLS3e力场优化后的3D sdf格式,用Openbabel将其转化为pdbqt格式备用.
2.2.4 类药筛选 在基于结构虚拟筛选之后,选出亲和力小于-8 kcal/mol的化合物,合并其sdf结构,将其提交到ADMETlab2.0网站上,根据100 2.2.5 FRET酶活抑制实验 酶活抑制实验是根据试剂盒生产商说明书来执行,初步筛选时化合物终浓度为20 μmol/L. 在添加底物之前,主蛋白酶和化合物先在30 ℃下孵育10 min来证抑制剂和蛋白的结合. 测IC 50时化合物终浓度梯度分别为0.1、 1、 2、 5、 10、 20、 40、 80和160 μmol/L. 2.2.6 分子动力学模拟 利用gromacs中的pdb2gmx处理蛋白结构,力场采用Amber14SB. 利用RESP2_ORCA.sh脚本生成小分子的RESP2电荷,之后利用Sobtop生成小分子拓扑结构 [14-16];合并小分子和蛋白质的拓扑信息,生成十二面体盒子,水模型为TIP3P,添加SPC216水和四个钠离子中和体系;能量最小化收敛在100 kJ/mol/nm内;nvt位置限制性模拟设定为在100ps内从0 K上升到300 K,随后持续100 ps,温度耦合方式为V-rescale;npt位置限制性模拟设定为500 ps,温度耦合方式为V-rescale,压力耦合方式为Berensden;最后放开位置限制,进行正式模拟,温度耦合方式不变,压力耦合方式变为PR [17-19]. 模拟完成之后,提取复合物的RMSD信息,分析天然产物与蛋白酶结合位点. 3 结 果 3.1 SARS-CoV-2突变株主蛋白酶序列和结构对比 首先,笔者对比了SARS-CoV-2突变株主蛋白酶的氨基酸序列,DNAMAN对比结果显示突变株序列一致性为99.8%,如图1所示,主蛋白酶只在两个位置出现了突变,一个突变是K90R,另一个突变是P132H. 在Pymol中叠合三种突变蛋结构,突出展示活性口袋,如图2所示.活性口袋的微小差异几乎可以忽略不计.PairWise Structure Alignment结果显示7RNK和7U29(K90R)之间的RMSD为0.5,TM-score为0.99,而这两个评价值在7RNK和7TVX(P132H)之间分别为0.35和0.98. RMSD结果表征为两个结构之间的距离差异,而TM-score是用来检测两个蛋白质结构相似性的指标,主要考虑蛋白质整体折叠情况. 总之,这些结果在一定程度上反映了SARS-CoV-2突变株主蛋白酶的保守性. 3.2 虚拟筛选模型可靠性评估 在正式虚拟筛选之前,我们利用Pymol对比了X77的晶体结构和用Qvina2.1进行对接的结构,如图3所示.晶体结构和对接结构之间的RMSD为1.263,相当于一个原子大小.因此,基于结构的虚拟筛选模型是准确可靠的. 3.3 基于结构的虚拟筛选和类药筛选结果 在天然产物数据库中,我们从3760种化合物中筛选出295种结合能小于-8 kcal/mol的化合物,接着我们利用ADMETlab2.0软件根 据-4 3.4 酶活抑制结果分析 首先,我们测试了16种天然产物在终浓度为20 μmol/L时对新型冠状病毒主蛋白酶的抑制效果,如图4所示,(-)-Gallocatechin gallate与Scullarein在20 μmol/L时对新型冠状病毒主蛋白酶的抑制率几乎达到了100%,与阳性对照Ebeselen抑制率一致,而其它天然产物在20 μmol/L时对新型冠状病毒主蛋白酶有一定抑制效果,尽管不太明显,也无法排除这些天然产物在其它方面会对新型冠状病毒产生一定抑制效果. 接着我们详细测试了 (-)-Gallocatechin gallate、Scullarein两者对新型冠状病毒主蛋白酶的半数抑制浓度IC 50,数据经Graphpad Prism8.0处理后,显示 (-)-Gallocatechin gallate对新型冠状病毒主蛋白酶的IC 50为(2.104 ± 0.346) μmol/L,而Scullarein的这个值为(10.79 ± 2.116) μmol/L,从结果中还发现,(-)-Gallocatechin gallate在160 μmol/L时对新型冠状病毒主蛋白酶的抑制效果也达不到100%,最大抑制率为97%,而Scullarein在20 μmol/L时对新型冠状病毒主蛋白酶的抑制率就达到了100%. 3.5 分子动力学模拟结果 根据分子动力学模拟轨迹,我们消除了主蛋白酶的平动和转动,以蛋白质骨架为基准,提取了复合物的RMSD,蛋白骨架相对于其本身的RMSD趋势与复合物RMSD趋势一致,因此此处仅展示两个复合物RMSD结果. (-)-Gallocatechin gallate与SARS-CoV-2主蛋白酶复合物在20 ns的分子动力学模拟中RMSD平均值为0.225 647 2,Scutellarein与主蛋白酶复合物RMSD平均值为0.223 724,从结果可以看出,这两个天然产物与新型冠状病毒主蛋白酶结合都较为稳定. 此外,我们分析了新型冠状病毒主蛋白酶与两种天然产物抑制剂的主要结合位点,如图7所示.(-)-Gallocatechin gallate的羟基与主蛋白酶的Asn142残基形成了氢键,其六元杂环上与羧基相连的H与Gln189形成了碳氢键,属于非极性键,其上一个苯环与Glu166形成了Ⅱ-阴离子相互作用,另一个苯环与His41形成了Ⅱ-Ⅱ堆积,与Arg188形成了酰胺-Ⅱ堆叠相互作用.如图8所示,Scutellarein中与六元碳环相连的苯环与Asn142形成了Ⅱ-孤电子对相互作用,而Glu166可能与苯环或其旁边的六元碳环存在Ⅱ-阴离子相互作用,六元碳环可能与Leu141存在酰胺-Ⅱ堆叠作用或者碳氢键相互作用,六元碳环上的一个羰基可能还与Ser144或者His172形成了氢键. 4 讨 论 本研究筛选出了针对新型冠状病毒主蛋白酶的两种天然产物抑制剂,所筛选到的两种天然产物与新型冠状病毒主蛋白酶已知抑制剂黄岑素和黄岑苷相比,提供了氢键受体和配体位置的多样性,而与一些非共价抑制剂X77和ML188相比,其功效范围更广.根据研究结果可知,新型冠状病毒主蛋白酶中的Glu166残基与两种天然产物都形成了Ⅱ-阴离子相互作用,提示我们在药物设计中需要关注芳香环的位置.总之这些结果为新型冠状病毒主蛋白酶抑制剂的发现和设计提供了新的思路. 在上述结果中我们提到筛选到的16种天然产物尽管有些对新型冠状病毒主蛋白酶的抑制效果不是很好,但不能排除它们在其他方面所起的作用.事实上,我们也在虚拟筛选得到的结果中针对新型冠状病毒类木瓜蛋白酶进行了虚筛,结果显示有些天然产物可能对新型冠状病毒类木瓜蛋白酶有潜在的抑制作用. 研究表明(-)-Gallocatechin gallate大多来源于绿茶,为多酚物质,具有抗炎抗氧化作用,而Scutellarein为菊科植物灯盏花中的黄酮类物质,在抗感染抗菌等方面发挥重要作用.由于新型冠状病毒会引起人体炎症因子的释放,而这两种天然产物既是新型冠状病毒主蛋白酶的抑制剂,又是良好的抗炎杀菌药物,因此值得后续研究 [20,21]. 参考文献: [1] Anderson E M, Goodwin E C, Verma A, et al. Seasonal human coronavirus antibodies are boosted upon SARS-CoV-2 infection but not associated with protection [J]. Cell, 2021, 184: 1858. [2] Mousavizadeh L, Ghasemi S. Review article genotype and phenotype of COVID-19: their roles in pathogenesis[J]. J Microbiol Immunol, 2021, 54: 159. [3] WHO. Coronavirus Disease (COVID-19)Dashboard[EB/OL].[2022-10-31].https://covid19.who. int/. [4] Yoshimoto F K. The proteins of severe acute respiratory syndrome coronavirus-2 (SARS CoV-2 or n-COV19), the cause of COVID-19 [J]. Protein J, 2020, 39: 198. [5] Malik J A, Mulla A H, Farooqi T, et al . Targets and strategies for vaccine development against SARS-CoV-2[J]. Biomed Pharmacother, 2021, 137: 111254. [6] Zhu Z, Lian X, Su X, et al . From SARS and MERS to COVID-19: a brief summary and comparison of severe acute respiratory infections caused by three highly pathogenic human coronaviruses[J]. Resp Res, 2020, 21: 224. [7] Wang H, He S, Deng W, et al . Comprehensive insights into the catalytic mechanism of middle east respiratory syndrome 3C-like protease and severe acute respiratory syndrome 3C-like protease [J]. ACS Catal, 2020, 10: 5871. [8] Mody V, Ho J, Wills S, et al . Identification of 3-chymotrypsin like protease (3CLPro) inhibitors as potential anti-SARS-CoV-2 agents [J]. Commun Biol, 2021, 4. [9] Marzi M, Vakil M K, Bahmanyar M , et al . Paxlovid: mechanism of action, synthesis, and in silico study [J]. Biomed Res Int, 2022: 7341493. [10] Heilmann E, Costacurta F, Moghadasi S A, et al . SARS-CoV-2 3CLpro mutations selected in a VSV-based system confer resistance to nirmatrelvir, ensitrelvir, and GC376 [J]. Sci Transl Med, 2022, 15: eabq7360. [11] Trott O, Olson A J. AutoDock Vina: improving the speed and accuracy of docking with a new scoring function, efficient optimization and multithreading[J]. J Comput Chem, 2010, 31: 455. [12] Alhossary A, Handoko S D, Mu Y G, et al . Fast, accurate, and reliable molecular docking with QuickVina 2 [J]. Bioinformatics, 2015, 31: 2214. [13] Xiong G, Wu Z, Yi J, et al . ADMETlab 2.0: an integrated online platform for accurate and comprehensive predictions of ADMET properties [J]. Nucleic Acids Res, 2021, 49: 5. [14] Lu T, Chen F W. Multiwfn: A multifunctional wavefunction analyzer[J]. J Comput Chem 2012, 33: 580. [15] Neese F. Software update: the ORCA program system, version 4.0[J]. WIREs Comput Mol Sci, 2018, 8: e1327. [16] Lu T.Sobtop, Version 1.0(dev3.0)[EB/OL]. [2022-08-09].http://sobereva.com/soft/Sobtop. [17] Weng Y, NaiK S R, Dingelstad, et al . Molecular dynamics and in silico mutagenesis on the reversible inhibitor-bound SARS-CoV-2 main protease complexes reveal the role of lateral pocket in enhancing the ligand affinity [J]. Sci Rep, 2021, 11: 7429. [18] Mahmud S, Uddin M A, Paul G K, et al . Virtual screening and molecular dynamics simulation study of plant-derived compounds to identify potential inhibitors of main protease from SARS-CoV-2[J]. Brief Bioinform, 2021, 22: 1402. [19] Kneller D W, Li H, Galanie S, et al . Structural, electronic, and electrostatic determinants for inhibitor binding to subsites S1 and S2 in SARS-CoV-2 main protease [J]. J Med Chem, 2021, 9: 17366. [20] Koeberle A, Werz O. Multi-target approach for natural products in inflammation [J]. Drug Discov Today, 2014, 19: 1871. [21] Luo L, Yang J, Wang C, et al . Natural products for infectious microbes and diseases: an overview of sources, compounds, and chemical diversities[J]. Sci China Life Sci, 2022, 65: 1123.
