膀胱尿路上皮癌蛋白组学相关个体化预后特征的推导与验证
2023-03-30黄永胜黄彩娜董学岭卓秀丽房娟娟宋文霞张雨露吕仁广
黄永胜 黄彩娜 董学岭 卓秀丽 房娟娟 宋文霞 张雨露 阎 磊 陈 刚 吕仁广
1.山东大学齐鲁医院泌尿外科,山东 济南 250012; 2.青岛市市立医院急诊科,山东 青岛 266000; 3.济南市第七人民医院泌尿外科,山东 济南 250012; 4.山东大学齐鲁医院德州医院日间手术科,山东 德州 254300; 5.山东大学齐鲁医院外科门诊部,山东 济南 250012; 6.山东第一医科大学(山东省医学科学院)临床与基础医学院,山东 济南 250117
膀胱癌在我国泌尿生殖系统恶性肿瘤中发病率居第1 位,是全球第10 大常见恶性肿瘤[1]。据全球癌症数据统计,2020 年约有573 278 例膀胱癌新发病例和212 536 例死亡病例[2]。95%的膀胱癌病理类型为尿路上皮癌(urothelial carcinoma,UC),其中30%为浸润型,70%为浅表型[3]。研究发现,膀胱癌的发病主要与长期接触化学物质有关[4]。目前,经尿道膀胱肿瘤电切术(transurethral resection of bladder tumor,TURBT)是非肌层浸润性膀胱癌的标准治疗手段,但术后复发率极高[5];对于晚期浸润性膀胱癌,根治性膀胱切除术和尿流改道术后患者的生存质量不高[6]。高复发性及持续的化疗使UC成为人均治疗费用最高的癌症[7]。因此寻求新的肿瘤标志物预测患者的生存率尤为重要。
蛋白质组学是在特定条件下研究蛋白质生物学功能的科学[8],癌症蛋白质组学研究旨在识别驱动恶性肿瘤转化的功能性蛋白质,并发现生物标志物以检测早期癌症、预测预后、确定治疗方式、确定新的药物靶点,并最终开发个性化医疗[9]。相比于基因组学,蛋白质能够更直接的反映细胞表型,蛋白质在癌症发展的全程提供生物学信息[10],基于质谱的蛋白质组学可以直接检查基因组畸变的后果[11]。癌症蛋白组学已在三阴乳腺癌[12]、结直肠癌肝转移[13]、胃肠癌[14]、肝内胆管癌[15]的预后中取得了进展。目前关于公共数据库提取UC预后相关蛋白质的研究较少,本研究通过癌症基因组图谱(the Cancer Genome Atlas, TCGA)数据库提取UC的蛋白组学、基因组学、临床性状、泛癌的数据,确定出蛋白组学相关预后特征(proteomics-related prognostic signatures,PRPS)并构建预后模型,以期为寻找UC新的生物标志物提供新思路。
1 材料和方法
1.1 数据准备
UC 蛋白组学数据(tsv 格式,343 例肿瘤样本)、基因组学数据(RNA-seq,正常样本19 例;肿瘤样本411例)、患者的临床性状数据来自于癌症基因组图谱(TCGA)数据库(https://portal.gdc.cancer.gov/)。
1.2 PRPS的筛选及风险评分(risk score, RS)预后模型和列线图的构建
根据多因素Cox 回归模型PRPS 的回归系数与表达值计算UC 患者的风险评分(risk score, RS)。RS 表 达 式:βprotein1× EXPprotein1+βprotein2× EXPprotein2+ …… +βprotein6× EXPprotein6(EXP:蛋白的表达水平,β:回归相关系数)。以RS 的中位数为标准将样本分为高、低风险两组。在构建RS预后模型后,将总体样本随机均分成训练集与测试集互为验证,以训练集RS 的中位值为标准对测试集进行风险分组,并分别进行生存(Kaplan-Meier, KM)分析。采用主成分(principal componet analysis, PCA)分析对风险分组进行可视化;绘制预测患者生存率的临床列线图,并通过校准曲线、受试者工作特征(receive operating characteristic, ROC)曲线与决策曲线分析(decision curve analysis,DCA)评估其可靠性。
1.3 PRPS的基因组学分析及免疫组化验证
基因组学与蛋白组学在癌症的生物学行为上互为补充,通过PubMed 中的Gene 模块检索出表达PRPS 的基因,对这些基因进行泛癌分析、免疫组化分析及KM 分析。PRPS 的免疫组化结果来自于人类蛋白图谱(Human Protein Atlas, HPA)数据库(https://www.proteinatlas.org/)。
1.4 基因集富集分析
通过基因本体(gene ontology,GO)与KEGG 富集分析,分析PRPS在UC中的生物学功能。首先以P <0.05,logFC = 1,错误发现率(false discovery rate, FDR) < 1为过滤标准得到在高、低风险分组中具有统计学意义的差异基因。应用C5 GO(c5.go.symbols.gmt)和C2 KEGG(c2.cp.kegg.symbols.gmt)基因集进行基因富集分析(gene set enrichment analysis,GSEA)(https://www. gsea-msigdb. org/gsea/msigdb/index.jsp)。GO、KEGG 及GSEA分析的筛选标准为P< 0.05,FDR < 0.05。
1.5 免疫细胞浸润性分析
采用免疫细胞浸润分析探索UC患者TME的变化,计算UC 患者TME 中免疫细胞的含量。采用R语言“corrplot”包绘制免疫细胞相关性图、“limma”包分析免疫细胞在高、低风险分组的差异性、“fmsb”包绘制雷达图。
1.6 统计学分析
数据分析与图表绘制均使用R 语言(4.2.1 版本)。Cox回归模型通过“glmnet”包进行;KM分析通过“survival、survminer”包 进行;PCA 分析通过“scatterplot3d”包进行;列线图通过“survival”“regplot”和“rms”包绘制;ROC 曲线通过“timeROC”包进行;用来进行GSEA富集分析的数据集C5和C2来 自 于GSEA 富集分析网站(https://www.gseamsigdb. org/gsea/msigdb/index. jsp);UC 肿瘤微环境免疫细胞的含量通过“MCPcounter”包与CIBERSORT算法提取。检验水准α= 0.05。
2 结 果
2.1 PRPS的筛选
将患者的蛋白质数据与生存数据合并得到用来进行分析的总样本,通过构建单因素Cox 回归模型得到差异蛋白共469个。再对单因素Cox回归结果构建多因素Cox回归模型确定6个具有独立预后意义的蛋白质(EMA、‘PKA-a’、MCT4、ANNEXIN1、Atg4B、‘4EBP1_pT70’)作为PRPS,详见表1。

表1 膀胱尿路上皮癌患者总样本的单因素与多因素Cox回归分析
2.2 RS预后模型及列线图的构建
在计算RS并进行风险分组后,按照1∶1的比例随机将样本分为训练集与测试集。训练集与测试集临床性状相比,差异无统计学意义(P <0.05),见表2。对差异蛋白进行风险分组的差异分析,并对结果进行可视化(图1A)。PCA分析可见,根据PRPS的风险分组聚类(图1B)比总样本(图1C)明显增多。

表2 膀胱尿路上皮癌患者训练集与测试集临床性状差异分析[n(%)]

图1 差异蛋白在风险分组的差异分析及风险分组的PCA结果
进一步对总样本、训练集与测试集进行生存分析。总样本OS 与PFS 分析(图2A,B)、训练集与训练集的OS 分析(图2C,D)均具有统计学意义(P<0.001),表明高风险分组患者的生存时间明显低于低风险分组患者。
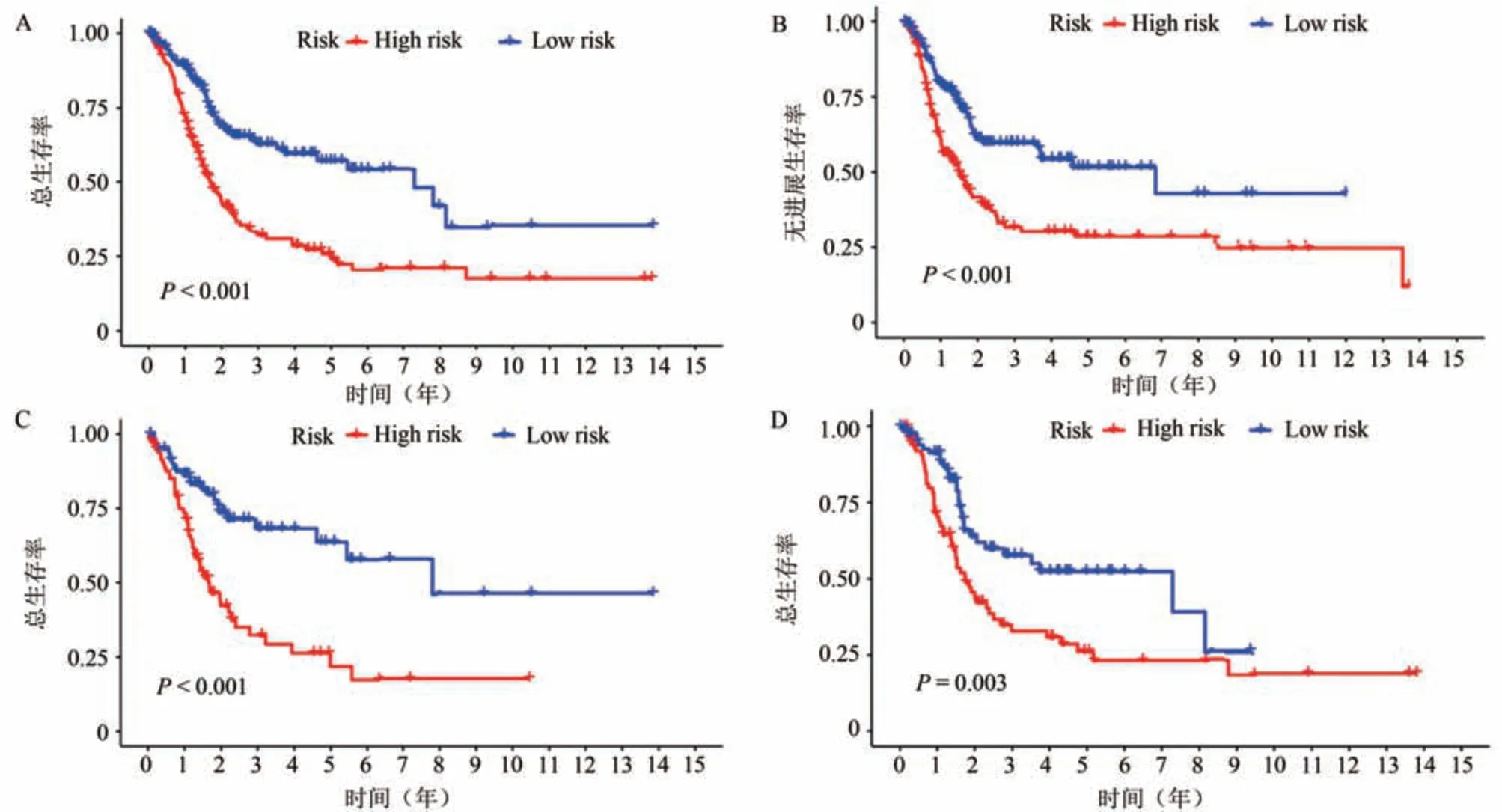
图2 膀胱尿路上皮癌患者总样本、训练集、测试集的KM分析结果
而后对总样本(图3A)、训练集(图3B)、测试集(图3C)分别构建RS 预后模型,ROC 曲线显示预测患者1 年、2 年、3 年生存率的AUC 值分别为0.710、0.709、0.719(总样本),0.684、0.760、0.791(训练集),0.740、0.658、0.663(测试集)。

图3 膀胱尿路上皮癌患者总样本、训练集、测试集的RS预后模型与ROC曲线分析结果
临床性状的单因素与多因素Cox 回归(表3)被用来确定UC 中起独立预后作用的临床性 状(independent prognostic of clinical traits,IPCT)。

表3 膀胱尿路上皮癌患者临床性状的单因素与多因素Cox回归分析
最后,开发一个包括风险分组与IPCT的临床列线图,协助临床医生对UC患者的生存率进行初步预测。临床性状与风险分组的ROC 曲线与DCA 曲线表明,根据PRPS构建的模型其预测能力是可靠的,另外,校准曲线展现出列线图对患者生存率的预测值与实际情况之间具有良好的一致性(图4A ~ C)。
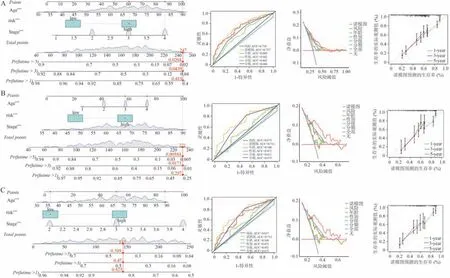
图4 膀胱尿路上皮癌患者总样本、训练集、测试集的列线图、ROC曲线与DCA曲线、校准曲线结果
2.3 PRPS的基因组学验证
通过PubMed 检索出表达PRPS 的基因(表4)。对这6 个基因分别进行泛癌分析、免疫组化分析、KM 分析(图5A ~ F)。发现PRPS 并非完全在基因组学及蛋白组学中表现一致,这也证实了二者在时间上的不平行,蛋白质表达则更加接近细胞表型。

表4 PRPS及对应的基因
2.4 PRPS的分子学机制探讨
差异分析得到在高风险组中上调的基因721 个,低风险组中上调的基因319 个。GO 富集分析表明,体液免疫反应等生物过程、胶原蛋白分子组成、信号受体激活剂活性的分子功能在高风险分组中高表达(图6A);KEGG富集分析表明,细胞因子与受体之间的相互作用、趋化因子信号通路、PI3K-Akt信号通路等在高风险分组中高表达(图6B)。对风险模型进行GCEA富集分析发现,粒细胞、白细胞趋化性、髓系白细胞迁移、T细胞受体复合体等细胞功能在高风险组高表达(图6C);ATP合成耦合的电子运输、胞质核糖体、呼吸系统、核糖体亚基及结构成分等细胞功能在低风险组高表达(图6D)。趋化因子信号通路、细胞因子及受体间的相互作用、焦点粘附、造血细胞系、肌动蛋白细胞骨架调控等功能通路在高风险组中高表达(图6E);药物代谢细胞色素p450、卟啉和叶绿素的代谢、视黄醇代谢、核糖体等功能通路在低风险组中高表达(图6F)。

图6 PRPS的GO、KEGG、GSEA富集分析结果
2.5 风险模型的免疫细胞浸润分析
免疫细胞之间的相关性如图7A 所示;雷达图展现了风险分组中免疫细胞的表达(图7B),并对表现出统计学意义的中性粒细胞与NK细胞进行了可视化(图7C,D)。

图7 PRPS的免疫细胞浸润分析结果
3 讨 论
膀胱癌的发病率占全球癌症的3%,五年生存率为77%,但发生转移的患者五年生存率仅5%[16]。自首次发现蛋白质可作为疾病风险标志物后[17],生物标志物便开始在蛋白组学领域被深入研究[18]。目前临床上组织和血浆[19]、体液[20]、药物及代谢物检测[21-22]是蛋白组学在疾病诊断中应用的典型案例。蛋白组学和基因组学存在着时间差异性,结合基因组学与蛋白组学分析可以帮助人们更深入地了解UC发病机制。
蛋白组学作为表征生物系统的最相关数据集[23],与基因组学互为补充[24],这也在本研究方法中得到了体现。本研究使用Cox回归模型确定出6个PRPS(EMA、`PKA-a`、MCT4、ANNEXIN1、Atg4B、`4EBP1_pT70`)。根据多因素Cox 回归结果计算RS[25-26],高风险组的OS 及PFS 要明显比低风险组低。ROC曲线表明,本研究的风险模型相比于其他临床性状而言,具有较高的准确性(AUC为0.710),提示本研究根据PRPS 构建的模型在UC 患者的生存预后中具有指导意义。功能富集分析揭示了患病高风险人群分组中活跃的生物学功能与通路,免疫细胞浸润分析进一步探索了UC患者免疫治疗的可能性与新思路。而基因组学分析结果印证了基因和蛋白时间表达的差异性,二者的临床意义并非保持前后一致。
目前尚无通过公共数据库中筛选出UC患者的PRPS 作为新的肿瘤标志物来预测患者生存预后的报道。本蛋白组学研究在患者的生存预后中具有一定指导意义,识别出的PRPS 为在蛋白组学层面上寻找癌症预后相关的新标志物及对患者的精准治疗提供了新思路。但本研究具有一定的局限性,数据样本来自于单一数据库,尚未进行细胞生物学实验及人类样本的验证和随访,其外部有效性仍需进一步探究。
利益冲突所有作者均声明不存在利益冲突
