Protein-HVGAE:一种双曲空间中的蛋白质编码方法
2023-03-10王皓白沈昕黄尉健陈可佳
王皓白,沈昕,黄尉健,陈可佳,3+
1.南京邮电大学 计算机学院,南京210023
2.南京邮电大学 理学院,南京210023
3.江苏省大数据安全与智能处理重点实验室(南京邮电大学),南京210023
蛋白质是生命活动的主要承担者,蛋白质相互作用才能发挥个体的功能并参与到生化过程中。由于生物实验的成本较高、实验条件苛刻和结果假阳性概率较大,发现蛋白质之间的潜在交互是一个很大的挑战。因此,研究者构建了以蛋白质为节点、相互作用为边的蛋白质交互(protein-protein interaction,PPI)网络,并设计精确且高通量的算法来识别和预测蛋白质的交互作用[1-2]。这样既能有效指导实验、缩短检测周期,又能辅助检测药物靶标、攻克疑难杂症。
随着PPI数据种类的不断丰富、规模的不断扩大[3],出现了使用机器学习进行蛋白质序列编码[4-7]、PPI 网络表示[8]、蛋白质功能预测[9]和复合物识别[10]的方法。特别在PPI 预测任务中,学习算法旨在捕捉蛋白质节点的拓扑特性,以此推断蛋白质之间是否存在交互。PPI网络分析的早期工作以矩阵分解[11]和随机游走[12-13]为主。图神经网络(如GCN[14]、GAT[2]、Graph-SAGE[1]等模型)随后也应用于PPI 网络,该模型能够有效结合节点自身的生物属性与网络拓扑特性,在许多下游任务中性能显著。
尽管上述模型展现出图表示学习的强大能力,但其嵌入精度仍受限于欧氏空间的维数和计算复杂度[15]。PPI 网络具有无标度属性,满足强幂律分布并呈现类似树状的层次结构。例如,在图1 的Bio-GRID 蛋白质交互网络中,节点的最大和最小度数分别为1 188 和2,平均度数为37.187,满足强幂律分布。以往模型难以学到这一层次结构。
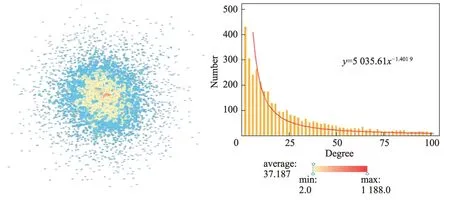
图1 BioGRID 蛋白质交互网络Fig.1 PPI network of BioGRID
最近,以双曲空间嵌入为代表的流形表示学习成为新的发展趋势[16]。它假设高维图数据的分布近似于某个低维流形,可通过学习由距离逆向推断节点间的连接关系。双曲空间可近似为n叉树的连续版本,其空间指数扩张的几何特性与PPI 网络特征高度贴合。因此,本文将双曲空间的图嵌入方法应用于PPI 网络,不仅可以由隐变量的距离判断节点的相似性,还能根据各节点的范数确定网络的潜在层次结构(即节点间的相关性)[15-17]。
本文提出一种在双曲空间中的变分图自编码器,用于蛋白质编码。该模型采用两个双曲图卷积网络(hyperbolic graph convolutional networks,HGCN)[18]作为编码器,计算隐藏层的均值和方差,并利用多个双曲空间的不同曲率捕捉网络的层次结构,区分各节点的低维表示。其中,双曲图卷积操作主要分为三步:(1)通过切平面对节点特征做欧氏-双曲空间的转换;(2)通过注意力机制在双曲空间上进行邻居节点聚合;(3)通过不同曲率构建不同层的HGCN,并据此设计非线性激活函数。模型采用Fermi-Dirac 函数做解码器,在双曲空间上使用内积运算重构网络。最终,在重构的PPI网络上实现PPI预测、蛋白质功能预测等下游任务。
1 相关工作
图表示学习是指将图中的节点转化为能保留原始图结构的低维稠密向量,更好用于节点分类、链接预测等下游任务。与传统的基于矩阵分解和随机游走的方法相比,图神经网络模型能够较好地结合拓扑结构和节点语义特征,广泛应用于图表示学习中。经典的图神经网络包括GCN[14]、GAT[2]和Graph-SAGE[1]模型等。GCN[14]以邻接矩阵和节点特征为输入,并通过聚合一阶邻居节点做图卷积运算,得到节点的低维稠密向量表示。GAT[2]模型在聚合邻居节点上做进一步改进,计算各邻居对中心节点的影响力,以此作为注意力权系数,得到邻节点的加权求和表示。GraphSAGE[1]模型则是将学习单一节点嵌入转化为学习节点聚合函数,并通过抽样指定邻居个数,解决了节点分布各异的问题。后来,Kipf 等人[19]提出变分图自编码器(variational graph auto-encoder,VGAE)模型,以GCN 为编码器,计算网络数据的分布,并通过随机采样作为隐变量,由内积解码得到算法输出。在PPI 网络表示学习研究中,早期方法多致力于解决邻接矩阵的稀疏性,计算蛋白质相似性矩阵[11]。Cho 等人[20]提出Mashup 模型,在整合而成的多物种蛋白质网络上采用带重启机制的随机游走算法,通过不同物种在相同蛋白质上相似的生化属性学习蛋白质节点的特征。近年来,图神经网络开始运用于PPI 网络的研究。Luck 等人[3]比较了LINE、DeepWalk、node2vec 和SDNE 在PPI 预测任务上的优劣。一些基于深度神经网络的模型进一步结合了蛋白质的生物特征与网络结构特征。例如:deepNF 模型[21]采用深度自编码器,将不同物种的异构蛋白质网络整合成通用的低维表示。Yao 等人[10]通过无监督的VGAE 模型,进一步利用无损放缩的自编码器,学习蛋白质网络的低维表示,实现蛋白质复合物的识别。Kulmanov 等人[9]设计DeepGO 算法,将蛋白质的氨基酸序列和其在网络中的结构特征有效结合,通过多层神经网络得到节点的低维嵌入。Hu 等人[22]分别在节点级自监督预训练和图级多任务半监督预训练神经网络,同时捕捉网络的局部和整体表示。由于蛋白质可能参与多个生化过程,存在多种相互作用,Ioannidis 等人[23]在多关系蛋白质网络上构建模型学习其表示。
单个蛋白质一般通过不同的相互作用参与到多种生化反应中,使得PPI 网络具有显著的层次结构,节点间度的差异较大。双曲空间能反映异构拓扑[13,24]特征,即节点数量随着其与根节点的距离呈指数级增长。因此,双曲空间上的节点嵌入可有效区分度数差异较大的节点,突出中枢(hub)节点的主导地位。Krioukov 等人[25]首次从理论上证明双曲模型在复杂网络分析上的有效性,建立双曲表示学习的上游模型。Papadopoulos 等人[26]进一步说明双曲距离在分析复杂网络演化的实用性。不过,上述模型仅学习到数据的浅层嵌入[15,27],而且没有使用节点的自身属性。最近以HGCN 模型[24]为代表的双曲神经网络模型[18,28]验证了结合节点属性和结构的方法在具有层次结构图上的节点分类和图分类任务中表现更加出色。
2 Protein-HVGAE 方法
本文提出了一种蛋白质表示学习方法Protein-HVGAE(hyperbolic graph auto-encoder for protein interaction networks),用于蛋白质链接预测和功能预测,总体框架如图2 所示。该模型在双曲空间上构建VGAE,采用两层HGCN 对蛋白质进行编码,充分学习网络层次结构的特性(节点范数),以及节点间的相似性(节点距离)。这里,A表示PPI 网络的邻接矩阵(adjacency matrix,维数为n×n,其中n为蛋白质节点的数量),X表示网络中蛋白质节点的特征矩阵(feature matrix,维度为n×d,d为节点特征向量的维度),将矩阵A与矩阵X做内积作为HGCN 的输入。

图2 Protein-HVGAE 的框架图Fig.2 Framework of Protein-HVGAE
2.1 HGCN 编码
HGCN[18]是图卷积模型在双曲空间上的实现,其核心在于通过欧氏切平面实现双曲空间上未定义的复杂向量运算,并由不同的曲率区分度数差异较大的节点。其中,双曲空间包含多个等距同构的双曲模型[28],选取内积和度规定义简明、数值计算稳定的洛伦兹模型(又名双曲面模型),学习复杂网络的隐变量表示。
2.1.1 双曲-欧氏变换
2.1.2 隐藏层变换
将节点的欧氏特征映射至双曲空间后,每一层HGCN 中的向量变换均在双曲空间实现,操作如下:
为减少信息损失,以上操作均在各个节点的切平面上实现。为了更好地逼近网络的层次结构,各层HGCN 采用不同的曲率,并通过将当前的欧氏输出转化为下一层的双曲输入,实现层与层间的平滑过渡。由于曲率各异,原点是相邻层所在的两个双曲空间中唯一相同的坐标,在其上做切平面进行特征映射。令-1/Kl-1,-1/Kl分别为l-1和l层上的双曲曲率,则双曲空间中的非线性激活函数为:
2.2 HVGAE 模型
VGAE 是变分贝叶斯和图神经网络的结合。根据变分贝叶斯原理:对于任一观察变量xk都存在一个利用xk得到隐变量的后验分布q(zi|xi)。假设节点满足正态分布,即q(zi|X,A)=N(zi|μi,diag(σ2i)),则对于整个网络而言:
其中,Z为隐变量,A为邻接矩阵,X为特征矩阵。
根据隐变量zi的分布,可通过采样获得其具体数值。再利用隐变量zi的内积进行解码,重构邻接矩阵:
损失函数由重构网络损失和隐变量分布误差构成,通过交叉熵函数以及后验分布与正态分布的KL散度进行度量,即:
其中,Eq(Z|X,A)[lgp(A|Z)]为交叉熵函数,而KL[q(Z|X,A)|p(Z)]则是KL 散度。
算法1HVGAE 的伪代码
3 实验
本文在多个物种的PPI 数据集上进行实验,通过链接预测和节点分类这两个下游任务,观察Protein-HVGAE 的表示能力和泛化能力。
3.1 PPI数据集
本文选取人类和酵母菌的三个蛋白质交互作用数据集:STRING-Human[12]、BioGRID[20]和STRINGYeast[12]。为了减少噪声对数据的影响,仅保留置信度大于0.7 的交互作用。各数据集的统计信息见表1。
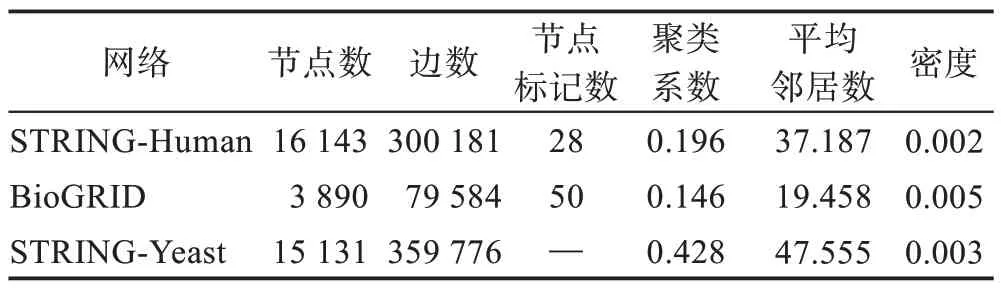
表1 3 个PPI数据集的统计信息Table 1 Statistics of 3 PPI networks
3.2 实验设置
HVGAE 模型使用Adam 方法进行优化,分批次训练,批大小设置为5 000,迭代次数为200 次,学习率设置为0.01。实验使用两层HGCN 嵌入层,每层的嵌入维度大小设置为128。为各对比方法均采用其原始论文中提供的最优参数,为了便于比较,每种方法的嵌入维度也设置为128。实验将每个PPI 网络按照8∶2 的比例将数据集划分为训练集和测试集。
获得蛋白质表示之后,直接用于PPI 预测和蛋白质功能预测的下游任务。实验选择常用的AUC(area under the curve)和AP(average precision)作为PPI 预测性能的评价指标,并采用Macro-F1 作为蛋白质功能预测的评价指标。
3.3 比较方法
本文选取了一系列图表示学习方法进行对比实验,详细介绍如下:
(1)Deepwalk[29]首次提出基于随机游走进行网络表示学习,通过随机游走获取k-hop 领域内的节点对构成节点序列,然后使用skip-gram 算法学习节点的表示。
(2)node2vec[12]是Deepwalk 的改进版本,主要的区别是在随机游走时结合广度优先搜索和深度优先搜索的策略,根据概率转移矩阵进行游走。
(3)Struc2vec[30]与前两个基于近邻相似假设的方法不同,考虑了非近邻的节点也可能拥有很高的结构相似性。
(4)GAE(graph auto-encoders)[19]是一种无监督学习框架,通过编码器学习低维向量,然后通过解码器重构图数据。
(5)VGAE[19]和GAE 不同,编码器学到的不是样本的低维向量表示,而是低维向量表示的分布。
3.4 各模型性能比较
3.4.1 PPI预测
为了公平比较,首先去除网络的节点属性,比较了6 个模型在无节点属性的PPI网络下的链接预测表现,如表2 所示(表中*表示在无节点属性情况下的预测结果)。

表2 PPI预测任务上的结果比较(数据无节点属性)Table 2 Comparison of PPI prediction performance on datasets without node features 单位:%
结果表明,HVGAE 在3 个数据集上的性能均优于其他模型,在BioGRID 数据集的优势最为显著。这表明HVGAE 能够适应不同物种的PPI网络。
随后,恢复PPI 网络的节点属性,观察3 种模型GAE、VGAE 和HVGAE 在STRING-Human 和Bio-GRID 数据集下的预测结果(见表3)。其中,*表示在无节点属性情况下的预测结果。

表3 PPI预测任务上的结果比较(数据包含节点属性)Table 3 Comparison of PPI prediction performance on datasets with node features 单位:%
与预期相同,在添加节点属性后,各个方法的预测性能均有所提高。总体来说,与欧氏空间的图神经网络模型相比,HVGAE 的性能在3 个数据集上均有明显提升;而其余模型之间的性能差异并不显著,且在不同数据集上排名有所波动。进一步说明,合适的几何度量有助于学习准确的低维表示。
3.4.2 蛋白质功能预测
本文还在蛋白质功能预测(即节点分类)任务中观察各方法的嵌入效果。本文将蛋白质的节点属性转化为功能标签用于分类,并选用Marco-F1[2]作为多标签分类的评价指标。表4 列出了在两个数据集上6种方法的蛋白质功能预测结果。

表4 蛋白质功能识别任务上的Marco-F1 比较Table 4 Comparison of Marco-F1 in protein function recognition task 单位:%
3.5 双曲-欧氏模型的对比
为了进一步研究双曲空间是否在层次结构的网络上更具嵌入优势,本文还在异构率和双曲率[12]两方面量化网络,以比较HVGAE 和VGAE 的性能。异构率刻画了网络的无标度属性,节点度数差异较大的强幂律分布的网络异构率较高。由于隐藏的层次结构无法直接提取[15],实验使用Gromov[31]提出的双曲率刻画网络中隐含的类树状结构[15,18,31]。层次结构越显著,双曲率越低。由此可见,异构率和双曲率两个指标呈反比。
使用PPI 预测任务进行比较,通过定义式(15)的优化率来表示HVGAE 对VGAE 在AUC 指标上的提升程度。
其中,AUCH和AUCE分别为HVGAE 和VGAE 在PPI预测任务上的AUC 值。本文使用双坐标图反映HVGAE 在3 个数据集上的优化率,以及各个网络对应的异构率/双曲率(图3)。

图3 HVGAE 对VGAE 的优化率Fig.3 Optimization rate of HVGAE to VGAE
实验结果验证了优化率满足与异构率成正比、与双曲率呈反比的特点。同时,在无标度属性和层次结构最为显著的BioGRID 数据集上,HVGAE 的性能提升最大,符合先验知识。
此外,图表示学习的结果还受到嵌入维数的影响。为了观察VGAE 在不同几何空间下的最优嵌入,本文采用BioGRID 数据集进行实验,设置输出层维数分别为32、16、12、8 和6,比较VGAE 在双曲和欧氏空间上的重构精度(图4)。
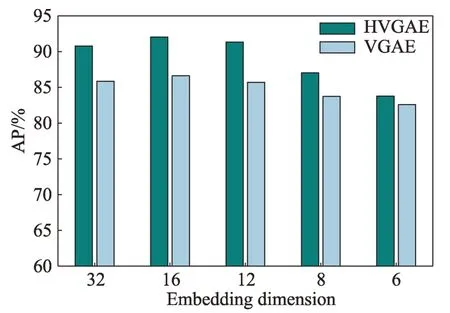
图4 不同嵌入维数下HVGAE 和VGAE 的比较Fig.4 Comparison of HVGAE and VGAE with different embedding dimensions
结果表明,当嵌入维度贴近于数据潜在的真实分布(16 维)时,HVGAE 相较于VGAE 有明显的提升(高达5.4 个百分点)。当对维度进一步压缩,其预测精度始终优于VGAE。这进一步验证了双曲空间在高维数据的表示上信息损失率更低,因此更适用于刻画复杂网络的内在几何空间。
3.6 曲率分析
实验最后分析了HVGAE 中的曲率,以验证模型的稳定性和优化潜力。对于HGCN,不同层曲率的设置有助于在降维过程中逐层逼近蛋白质网络内在的几何拓扑结构[18]。本文通过调整-lgK,便于研究曲率对3 个PPI 网络嵌入精度的影响(见图5)。

图5 曲率分析Fig.5 Curvature analysis
由于人类PPI 网络的层次结构较高,随着曲率降低,PPI 预测的AP 指数呈上升趋势,尤其是层次结构最高的BioGRID 数据集,AP 整体提升达到1 个百分点。对于酵母蛋白网络,曲率下降反而减损了嵌入精度。这一现象与曲率的性质相吻合(即K趋向∞为欧几里德空间)。
4 结束语
本文提出了一种蛋白质表示学习模型,即双曲变分图自编码器Protein-HVGAE,充分发挥HGCN 和VGAE 在具有层次结构网络中的学习能力。在不同物种的PPI 数据集上和多个下游任务中,本文方法均优于现有方法,验证了模型在诸如PPI 网络等具有无标度特性和较高层次结构图上的优势。未来工作中,将进一步研究基于HVGAE 的预训练方法[8]以进一步获得多物种PPI网络的共性特征。
