CA-NHEJ系统对大肠杆菌链霉素耐药基因rpsL的编辑效率及其耐药性研究
2023-02-24谢钰珍覃鸿妮吴凡胡杨晓然薛高旭赵雪张勇
谢钰珍 覃鸿妮 吴凡 胡杨晓然 薛高旭 赵雪 张勇


摘要:以控制质粒拷贝数的pcnB基因与链霉素耐药性rpsL基因为靶标基因,研究CA-NHEJ系统对大肠杆菌的基因编辑效率,及rpsL相关基因与链霉素耐药性的关系。通过CA-NHEJ系统成功实现了对大肠杆菌MG1655的pcnB和rpsL基因编辑,其中,pcnB基因编辑效率达100%,rpsL基因仅有1个克隆缺失第22~24位3个氨基酸,其他4个克隆未编辑成功但具有链霉素抗性;进一步利用CA-HR系统敲除大肠杆菌MG1655的rpsL基因CCTGCGCTG序列(第22~24位氨基酸),编辑效率达100%,验证了CA-HR的高效性,抗性鉴定表明,该序列是影响链霉素抗性的关键序列,是一个未曾报道的新突变;对于未编辑成功但具有链霉素抗性的4个克隆进行基因测序及比对,找出了rhsC和gadB等6个可能与链霉素抗性相关的其他基因。
关键词:CA-NHEJ系统;CA-HR系统;rpsL基因;耐药性
中图分类号:S188 文献标志码:A
文章编号:1002-1302(2023)23-0017-05
CRISPR/Cas9系统因其具有精确识别及特异剪切DNA序列的功能而被开发成为一种高效的基因编辑技术[1-2]。CRISPER系统中的Cas9核酸酶可由sgRNA根据碱基配对定向到任何靶基因组位点,并刺激位点特异性双链DNA断裂(DBS),断裂的DNA链可通过非同源末端连接(NHEJ)和同源性末端连接(HR)2种机制进行修复[3]。其中,NHEJ不仅不依赖于同源臂的设计与合成,而且能实现外源DNA片段随机整合到不同的染色体位点,在基因编辑中显示出显著的优势[4-6]。在真核生物中普遍存在非同源末端连接系统,而大部分的原核生物(如大肠杆菌)缺乏NHEJ修复[7],仅能通过同源重组(HR)和外源引入的人工设计供体DNA来实现基因敲除,编辑不同基因除了构建CRISPR位点外,还需要构建相应的DNA修復模板,操作繁琐,极大限制了该技术应用于基因组规模的基因编辑。现有学者借鉴真核CRISPR-Cas9基因编辑策略,将CRISPR-Cas9介导的DSB与NHEJ引发的易错DSB修复结合起来,在大肠杆菌中开发了一种新型的不依赖于DNA修复模板的基因突变技术CA-NHEJ(CRISPR-Cas9 assisted Non-Homologous End Joining),该技术的应用还在进一步探索当中[8]。
本研究以控制质粒拷贝数的基因pcnB和链霉素耐药性相关基因rpsL为靶标,研究 CA-NHEJ系统对2个基因的编辑效率。同时,采用CA-HR系统编辑rpsL基因,并对编辑后菌株的链霉素耐药性进行研究,以期为CA-NHEJ系统在大肠杆菌中的应用提供参考,同时为细菌耐药性研究提供新思路。
1 材料与方法
1.1 试验材料与地点
MG1655菌株购于ATCC公司,所有质粒由苏州金唯智生物科技有限公司构建;试验用到的抗性平板、缓冲液、测序引物、TaqDNA聚合酶、大肠感受态等均由苏州金唯智生物科技有限公司生产。试验于2021年在苏州金唯智生物科技有限公司开展。
1.2 试验方法
1.2.1 sgRNA设计
利用http://www.rgenome.net/网站设计sgRNA引物,“PAM Type”使用默认选项“spCas9 from Streptococcus pyogenes:5′-NGG-3′”,“Target Genome”选择“Others”,“Genomes”选择“Escherichia coli(K-12,MG1655)”,“Target Sequence”下文本框中输入DNA序列,“crRNA length”默认“20”,设计结果出来后,选择“Out-of-frame-Score”,得出分值最高的sgRNA序列(表1)。
1.2.2 质粒制备
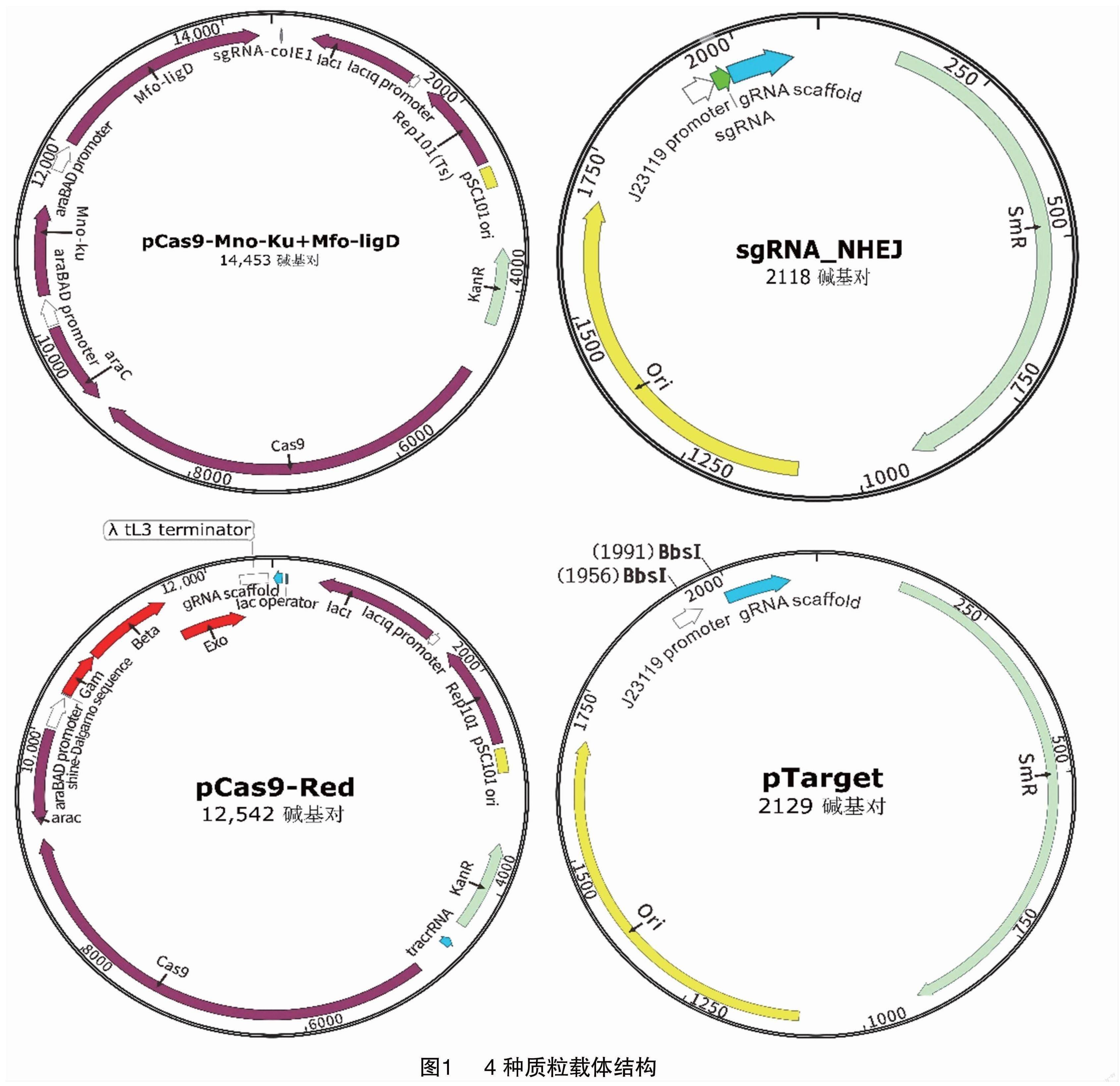
本研究采用CRISPR-Cas9介导的DSB与NHEJ引发的易错DSB修复结合(CA-NHEJ)的系统对pcnB、rpsL基因进行基因编辑。构建表达载体pCas9-Mno-Ku+Mfo-ligD质粒(kan抗性)和靶向目的基因载体sgRNA-NHEJ质粒(Spe抗性),后续采用CRISPR-Cas系统结合CA-HR系统对rpsL基因进行基因编辑,构建表达载体pCas9-Red质粒(Kan抗性)和靶向目的基因载体pTarget质粒(Spe抗性)。4个质粒结构(图1)均交由苏州金唯智生物科技有限公司构建,将酶切测序验证正确的目的质粒菌液取回制备质粒。
1.2.3 CA-NHEJ系统对大肠杆菌基因编辑
将pCas9-Mno-Ku+Mfo-ligD质粒电转至大肠杆菌电转感受态细胞MG1655中,复苏1 h后将菌液离心全涂布至Kan抗性的LB平板,将平板放置烘箱30 ℃培养24 h;挑单克隆并制备相应的电转感受态细胞pMG1655;将sgRNA-NHEJ质粒电转置感受态细胞pMG1655中,复苏1 h后将菌液离心涂布至含Kan+Spe抗性的LB平板,30 ℃培养24 h;从Kan+Spe抗性的LB平板上挑取8~24个单克隆至含200 μL LB的96孔深孔板,将96孔深孔板置于30 ℃摇床培养16 h;通过菌落PCR及Sanger测序鉴定基因编辑情况。
1.2.4 CA-HR系统对大肠杆菌基因编辑
将pCas9-Red质粒电转至大肠杆菌电转感受态细胞MG1655中,复苏1 h后将菌液离心全涂布至Kan抗性的LB平板,将平板放置烘箱30 ℃培养24 h;将pTarget质粒电转置感受态细胞pMG1655中,复苏 1 h 后将菌液离心涂布至含Kan+Spe抗性的LB平板,30 ℃培养24 h;从Kan+Spe抗性的LB平板上挑取8~24个单克隆至含200 μL LB 96孔深孔板,于30 ℃培养16~24 h;通过菌落PCR及Sanger测序鉴定基因编辑情况。
2 结果与分析
2.1 CA-NHEJ系统对pcnB基因的编辑
使用pCas9-Mno-Ku+Mfo-ligD和sgRNA-pcnB-NHEJ质粒,通过CA-NHEJ系统对大肠杆菌pcnB基因进行基因编辑,电转涂布含Kan、Sep抗性平板。从生长良好的平板上挑取16个单克隆,以pcnB-F和pcnB-R为引物进行菌落PCR鉴定结果,由图2可知,所有克隆均显示条带,条带大小不一表明基因序列缺失的大小具有随机性。将PCR产物进行Sanger测序,测序结果显示所有克隆的pcnB均发生基因编辑,其中,15个克隆在Cas9切割位点附近发生不同长度的序列缺失,1个克隆(clone-3)在切割位点附近发生序列缺失,同时插入27 bp序列(ACTGGAAAATGTGCAGCCAAACTCGGC),基因编辑效率达100%,表明CA-NHEJ系统对大肠杆菌pcnB基因编辑效率高。
2.2 CA-NHEJ系统对rpsL基因的编辑
使用pCas9-Mno-Ku+Mfo-ligD和sgRNA-rpsL-NHEJ质粒,通过CA-NHEJ系统对大肠杆菌rpsL基因进行基因编辑,电转化后涂布含卡那霉素、壮观霉素、链霉素LB平板,烘箱过夜培养最终平板形成5个单克隆。从平板挑取这5个单克隆,以rpsL-F和rpsL-R为引物进行菌落PCR鉴定,由图3可知,所有克隆皆显示大小一致的条带,将PCR产物进行Sanger测序,测序结果显示仅有1个克隆的rpsL基因成功编辑,在Cas9切割位点附近发生 9 bp 序列(CCTGCGCTG)缺失导致rpsL蛋白缺失第22~24位氨基酸,即Pro(脯氨酸)、Ala(丙氨酸)、Leu(亮氨酸)3个氨基酸,但序列缺失未引起基因的移码突变;另外4个克隆的rpsL基因未发生编辑,为何没未被编辑的4个克隆具有抗性,后续进一步做测序分析(见“2.4”节)。这里的编辑效率低可能是一种假象,因为rpsL基因为大肠杆菌必需基因,基因编辑导致缺失的片段具有随机性,编辑成功的克隆大部分致死。
2.3 CA-HR系统敲除MG1655大腸杆菌rpsL后的抗性分析
使用pCas9-Red和pTarget-rpsL质粒,通过CA-HR系统敲除MG1655大肠杆菌rpsL基因CCTGCGCTG序列(第22~24位氨基酸),涂布卡那霉素、壮观霉素LB平板,平板克隆形态正常。从平板挑取24个单克隆,以rpsL-F和rpsL-R为引物进行菌落PCR鉴定,由图4可知,所有克隆均显示大小一致的条带,进一步将PCR产物进行Sanger测序,测序结果显示所有克隆rpsL的第22~24位氨基酸序列发生缺失,验证了CA-HR系统的编辑效率高,而且由于编辑位置固定,不致死。
将rpsL基因编辑成功的菌液及野生型MG1655菌液分别涂布含链霉素抗性平板,于37 ℃培养 18 h,次日检查,由图5可知,涂布rpsL基因编辑菌液的平板正常形成菌落,涂布野生型MG1655菌液的平板没有形成菌落,表明大肠杆菌rpsL缺失第 22~24位3个氨基酸会产生链霉素抗性。
2.4 rpsL未编辑的克隆全基因组测序分析
为探究4个rpsL基因未编辑的克隆耐受链霉素抗性的原因,通过二代(Hiseq)和三代(Pacbio)高通量测序平台,将1个编辑成功的克隆和4个未编辑成功的克隆及野生型MG1655大肠杆菌进行全基因组测序,将测序结果与NCBI公布的MG1655基因组序列进行比对,分析序列差异,考虑到转座子等可移动元件基因和链霉素抗性关联度较低,部分RNA基因有序列缺失,但是缺失序列较短,本研究优先探究编码蛋白基因和链霉素抗性的相关性,由测序结果(表2)可知,与NCBI公布的MG1655参考序列比对发现,野生型MG1655部分编码蛋白基因(folD等)发生突变,由于野生型MG1655菌株没有链霉素抗性,因此推测这些基因和链霉素抗性关联性极低;MG1655(rpsLΔ22~24)大肠杆菌rpsL基因有 9 bp 序列缺失,与Sanger测序结果一致;rhsC、gadB、ydfK、pinQ、tfaQ、gadA基因在野生型MG1655菌株均未发生突变,但它们中的1个或多个基因在其他有链霉素康抗性的菌株中发生了突变,推测这些基因中可能存在与链霉素抗性有关的基因。
3 讨论
链霉素是一种广谱氨基糖苷类抗生素,通过提高核糖体和非同源tRNA亲和力、干扰核糖体矫正功能,导致细菌蛋白翻译错误率水平提升[9],细菌有缺陷的突变蛋白的积累会导致细菌死亡,达到杀菌效果[10]。此外,链霉素还会引起氨酰-tRNA从核糖体解离,影响蛋白翻译。rpsL基因编码核糖体蛋白S12,该蛋白对维持翻译水平的精确性有着极为重要的作用,已经有报道表明rpsL基因第43位氨基酸或第88位氨基酸发生突变会产生链霉素抗性,进一步研究发现rpsL更多的突变位点会导致链霉素抗性,这些突变分散在2个区域:第40~43位氨基酸和第87~93位氨基酸[11-13]。本研究使用 pCas9-Mno-Ku+Mfo-ligD和sgRNA-rpsL-NHEJ质粒,通过CA-NHEJ系统对大肠杆菌rpsL基因进行基因编辑,发现1个未有报道的新突变,rpsL缺失CCTGCGCTG(第21~23位氨基酸),即Pro(脯氨酸)、Ala(丙氨酸)、Leu(亮氨酸)3个氨基酸,该突变类型能够产生链霉素抗性。同时,通过对rpsL未编辑但有抗性的4个克隆进行测序和比对,发现了6个可能与链霉素抗性相关的其他基因(rhsC、gadB、ydfK、pinQ、tfaQ、gadA),其作用机理有待进一步研究。
4 结论
通过CA-NHEJ系统成功实现了对大肠杆菌MG1655的pcnB和rpsL的基因编辑,其中,pcnB基因编辑效率达100%,rpsL基因仅有1个克隆rpsL缺失第22~24位3 个氨基酸,其他4个克隆未编辑成功但具有链霉素抗性,编辑效率低可能是因为rpsL基因为必需基因,随机缺失导致大部分编辑成功的克隆致死;进一步利用CA-HR系统敲除大肠杆菌MG1655的rpsL基因CCTGCGCTG序列(第 22~24位氨基酸),编辑效率达100%,验证了CA-HR的高效性,抗性鉴定结果表明该序列是影响链霉素抗性的关键序列;对于未编辑成功但具有链霉素抗性的4个克隆进行基因测序及比对,找出了rhsC和gadB等6个可能与链霉素抗性相关的其他基因,本研究对链霉素抗性机理,核糖体参与的蛋白翻译机制提供新的研究思路。
参考文献:
[1]Mans R,van Rossum H M,Wijsman M,et al. CRISPR/Cas9:a molecular Swiss army knife for simultaneous introduction of multiple genetic modifications in Saccharomyces cerevisiae[J]. FEMS Yeast Research,2015,15(2):fov004.
[2]Ledford H. CRISPR,the disruptor[J]. Nature,2015,522(7554):20-24.
[3]杨丽芸,陈丽娇,李善刚. CRISPR/Cas9系统诱导DNA断裂的修复机制研究进展[J]. 中国细胞生物学学报,2022,44(3):500-511.
[4]李瑞娟,赵晓雨,杨润雨,等. 噬菌体重组酶介导的DNA同源重组工程[J]. 微生物学通报,2021,48(9):3230-3248.
[5]严振鑫,徐冬一. DNA双链断裂的非同源末端连接修复[J]. 生命科学,2014,26(11):1157-1165.[HJ2.15mm]
[6]Burma S,Chen B P C,Chen D J. Role of non-homologous end joining (NHEJ) in maintaining genomic integrity[J]. DNA Repair,2006,5(9/10):1042-1048.
[7]Waters C A,Strande N T,Wyatt D W,et al. Nonhomologousend joining:good solution for bad ends[J]. DNA Repair,2014,17:39-51.
[8]靳海迎. 基于CRISPR-Cas9系统的谷氨酸棒状杆菌基因组编辑技术的研究[D]. 济南:山东大学,2017:24-62.
[9]Ruiz P,Rodríguez-Cano F,Zerolo F J,et al. Investigation of the in vitro activity of streptomycin against Mycobacterium tuberculosis[J]. Microbial Drug Resistance,2002,8(2):147-149.
[10]陳 伊,翁琼琳,孟繁荣. 结核分枝杆菌链霉素耐药基因突变特征分析[J]. 中国热带医学,2018,18(6):534-537,542.
[11]王婉蓉,代 科,马小雨,等. 副猪嗜血杆菌链霉素耐药基因rpsL、rrs分析和关键位点的鉴定[J]. 中国预防兽医学报,2018,40(11):1061-1065.
[12]申秀丽,王兆芬,李 斌,等. 青海地区结核分枝杆菌链霉素耐药基因rpsL和gidB突变特征[J]. 国际药学研究杂志,2018,45(3):205-210.
[13]杨彩虹,曹旭东,史静雪,等. 结核分枝杆菌临床分离株链霉素耐药性的检测及耐药基因突变位点分析[J]. 石河子大学学报(自然科学版),2017,35(2):169-175.
