NEDD4L作为骨肉瘤预后标志物的生物信息学研究
2023-02-13刘懿天
刘懿天,庄 然,丁 勇
(1空军军医大学唐都医院骨科,陕西 西安 710038;2空军军医大学基础医学院免疫学教研室,陕西 西安 710032)
骨肉瘤(osteosarcoma,OS)是一种进展程度快、易发生早期转移的恶性骨肿瘤,好发于青春期以及65岁以上的老年人[1]。OS最常见的病变部位为股骨远端及胫骨近端的干骺端[2],疼痛和肿胀是其早期最常见的症状。由于好发于青春期人群,症状常被误认为是生长痛或剧烈运动导致的疼痛而耽误早期诊断。作为一种恶性程度极高的骨肿瘤,OS具有亲肺转移的特性,在发生转移的患者中,有高达82%的患者存在肺转移[3],并且大约20%的患者在确诊OS时便已发生肺转移[1]。尽管自上世纪80年代以来通过手术切除、化疗等各种治疗方式使未发生肺转移的局灶性患者长期存活率已经保持在60%以上,但对于已经发生肺转移的患者来说,其长期存活率却一直没有改善,仍然低于40%[4]。因此,亟需了解OS发生发展的分子机制,并寻找新的生物标志物用于OS的早期诊断和预后评估。
虽然OS具有高度侵袭性,但其发病率却非常低,在0~19岁的人群中每年每百万人只有不到6人确诊[5]。这导致OS难以像结直肠癌、乳腺癌那样拥有完整的大样本量的转录组图谱,癌症基因组图谱(The Cancer Genome Atlas,TCGA)数据库中31种癌症分类中甚至没有OS,只有肉瘤。尽管既往已有研究采用生物信息学的方法对OS的预后标志物进行了初步筛选,但不是样本量过小就是数据来源单一,只在特定数据集中有效[6]。
本研究从多个不同数据库选取与OS相关的多种不同类型的基因表达谱数据集,分析了OS患者肿瘤组织与正常骨组织的差异表达基因(differentially expressed genes,DEGs),通过功能富集分析、肿瘤微环境(tumor microenvironment,TME)分析以及单因素和多因素Cox回归分析筛选影响患者预后的相关基因,构建预后预测模型并进行了蛋白水平的初步验证,为OS患者的诊断和预后评估提供临床新思路。
1 材料与方法
1.1 材料
1.1.1 生物信息学数据 从TARGET数据库(https://ocg.cancer.gov/programs/target)检索下载TARGET-OS项目的RNA-seq表达矩阵,其表达量单位为TPM。从GEO数据库(http://www.ncbi. nlm.nih.gov/geo)中检索下载OS相关的RNA-seq或Microarray表达矩阵及对应注释信息,其单位为FPKM。从GEO数据库获取单细胞RNA测序(single cell RNA-seq,scRNA-seq)数据,表达量单位为Count。从CCLE数据库(https://sites.broadinstitute.org/ccle)检索下载人OS细胞系的表达矩阵,表达量单位为TPM。31种癌症分类目标基因的TPM表达谱文件获得自GEPIA网站(http://gepia.cancer-pku.cn/)[7]的TCGA数据库。部分需要进行数据处理的数据集及相关信息见表1。

表1 数据集基本信息
1.1.2 主要试剂和仪器 细胞培养箱(Thermo公司,美国);细胞系FOB1.19、MG63、143B、HOS(ATCC中心,美国)、K7M2(普诺赛生命科技有限公司,中国);高糖DMEM培养基、MEM培养基、胎牛血清(Gibco公司,美国);青霉素-链霉素、胰蛋白酶、40 mL/L多聚甲醛溶液(米鼠生物科技有限公司,中国);RIPA裂解液(新赛美生物科技有限公司,中国);聚偏二氟乙烯膜(Kodak公司,美国);50 mL/L牛血清白蛋白溶液、HE染色套装(塞维尔生物科技有限公司,中国);兔抗NEDD4L(博士德生物工程有限公司,中国);兔抗β-actin、鼠抗兔二抗(三鹰生物技术有限公司,中国);化学发光成像系统(Bio-Rad公司,美国);IX73 型倒置相差显微镜(Olympus公司,日本);BALB/c小鼠(空军军医大学实验动物中心,中国)。
1.2 方法
1.2.1 原始数据处理与差异表达基因筛选 使用R语言dplyr包将从GEO数据库下载的FPKM表达矩阵转换为TPM表达矩阵,并进行标准化处理[log2(TPM+1)]。使用sva包的Combat函数进行批次矫正并整合。使用stats包进行主成分分析(principal components analysis,PCA)并用ggplot2包对分析结果进行可视化展示。使用limma包进行DEGs分析,以经本亚明-霍赫贝格法多重检验校正后的P值和差异倍数(fold change,FC)作为判定DEG的标准,|log2(FC)|>2的基因在本文被定义为DEGs,P<0.05表示差异有统计学意义。
1.2.2 功能富集分析 使用GSEA软件进行基因集富集分析(gene set enrichment analysis,GSEA)[8],以目标基因表达量排序,中位数为阈值,将患者分为高表达组和低表达组,使用从MSigDB(http://www.gsea-msigdb.org/gsea/downloads.jsp)下载含有京都基因和基因组百科全书[9]通路信息的c2.cp.kegg.v7.4.symbols. gmt参考子集合,用于评估高、低表达组基因集的变化,P<0.05和FDR<0.25的通路在本文被定义为显著富集的通路。
1.2.3 构建单因素和多因素预后模型 使用survival包和survminer包对上述方法1.2.1合并后数据集的患者年龄、性别以及所有DEGs进行单因素Cox回归分析,筛选P<0.05的变量进行多因素Cox回归分析。使用rms包[10]绘制带有多因素Cox回归分析结果P值的列线图。使用Predict函数对多因素进行风险评分,对评分进行排序并绘制相应基因的热图。
1.2.4 免疫浸润分析 使用ESTIMATE算法[11]对数据集进行分析,并获得每个样本的Stromal评分、Immune评分和肿瘤纯度(tumor purity,TP)。按照TP排序,以中位值为界,将样本分为高纯度组和低纯度组,使用Wilcoxon秩和检验对两组进行统计学检验并用ggplot2包绘制小提琴图。使用ggpubr包计算TP与目标基因的Spearman相关性系数并绘制相关性散点图。P<0.05代表TP与目标基因的表达量存在显著相关性。
1.2.5 单细胞测序分析 使用seurat包[12]对数据集进行数据标准化处理。使用统一流形逼近与投影(uniform manifold approximation and projection,UMAP)算法[13]对数据进行降维分析,使用Louvain算法对数据进行聚类。使用UMAPPlot和FeaturePlot函数分别绘制UMAP降维聚类散点图和目标基因分布特征图。
1.2.6 预后模型的评估 使用生存曲线和受试者工作特征(receiver operating characteristic,ROC)曲线下面积(area under the curve,AUC)评估预后模型的效能。使用survival包和survminer包绘制总生存曲线,并采用Log rank检验评估不同分组之间的差异显著性。使用pROC包绘制单因素或多因素在1、3、5年3个时间点的ROC曲线并计算相应AUC。AUC>0.7代表模型可以有效预测预后。
1.2.7 细胞培养 将冻存细胞置于37 ℃恒温水浴锅中复温至细胞液完全解冻。吸取复温后细胞液至15 mL离心管,加入10 mL含100 mL/L胎牛血清的完全培养基(FOB1.19、143B和K7M2使用高糖DMEM培养基;MG63和HOS使用MEM培养基)稀释,1 000 r/min离心5 min,弃上清,加入对应完全培养基重悬,接种于6 cm培养皿,置于37 ℃、50 mL/L CO2培养箱中,5 h后换一次液。待细胞生长至80%以上融合时,消化并扩增至10 cm培养皿,以后常规传代培养。取第3~5代对数生长期细胞进行后续实验。
1.2.8 蛋白质印迹实验 将处于对数生长期的3种OS细胞系(MG63、HOS和143B)以及人成骨细胞FOB1.19使用PBS洗2次后提取蛋白,BCA法蛋白定量,加入loading buffer后,100 ℃水浴15 min,4℃ 15 000g离心5 min。利用100 g/L SDS-PAGE凝胶进行蛋白电泳。蛋白样品按10 μg/孔上样电泳,恒流湿转法转膜。转膜结束后,50 mL/L牛血清白蛋白溶液室温封闭30 min,孵育一抗:兔抗NEDD4L、β-actin,一抗浓度均为1∶1 000。4 ℃摇床过夜,之后加入山羊抗兔二抗(1∶5 000)室温孵育1 h,最后进行化学发光显影并拍照。使用Image Lab软件进行灰度分析。
1.2.9 小鼠OS肺转移模型的建立 将处于对数生长期的小鼠高转移OS细胞系K7M2消化、离心后用PBS重悬,调整浓度至5×109个/L,按200 μL/只的量尾静脉注射至4周龄雄性BALB/c小鼠,21 d后肺组织取材,40 mL/L多聚甲醛溶液室温固定,石蜡包埋。
1.2.10 切片染色 将小鼠肺转移灶石蜡标本按厚度4 μm切片,脱蜡后进行HE和组化染色。对于HE染色,依次使用苏木精和伊红染色,脱水封片,可使细胞核呈蓝色,细胞质呈红色。对于组化染色,抗原修复后孵育一抗:兔抗NEDD4L(1∶50),4 ℃过夜。之后用50 mL/L牛血清白蛋白溶液封闭2.5 h,并用二氨基联苯胺显色,得到棕色产物。使用IX73型倒置相差显微镜进行拍照记录。
1.2.11 统计学分析 生物信息学分析方法各部分已述。蛋白质印记实验独立重复3次,结果使用GraphPad Prism 9.0软件作图并进行非配对t检验分析。P<0.05表示差异有统计学意义。
2 结果
2.1 DEG分析与单因素Cox回归分析
使用stat包对数据集GSE126209(OS患者12例,健康骨组织11例)进行PCA分析并绘制降维散点图(图1A)。图中点之间的距离越近代表点对应的样本组成越相似,在整体上OS患者与健康骨组织存在显著差异,OS患者组内部也存在较为明显的异质性。
使用limma包对数据集GSE126209进行DEG分析,共得到2 806个DEGs,其中上调基因2 284个,下调基因522个,使用ggplot2包绘制DEGs火山图(图1B)。图中横坐标代表log2处理后的FC,FC越大,横坐标离0越远;纵坐标代表-log10矫正后的P值,差异越显著,纵坐标越大;蓝点代表在OS患者中表达量下调的基因,红点代表上调基因,灰点代表差异不显著的基因。
使用survival包对GSE16091、GSE21257和GSE39055合并后的数据集(OS患者123例)患者年龄、性别以及所有DEGs进行单因素Cox回归分析并绘制森林图(图1C)。筛选到9个基因P<0.05,其中TFPI2和CREB3L1表达水平下调,其余为上调基因,年龄和性别不是患者生存预后相关的独立因素。
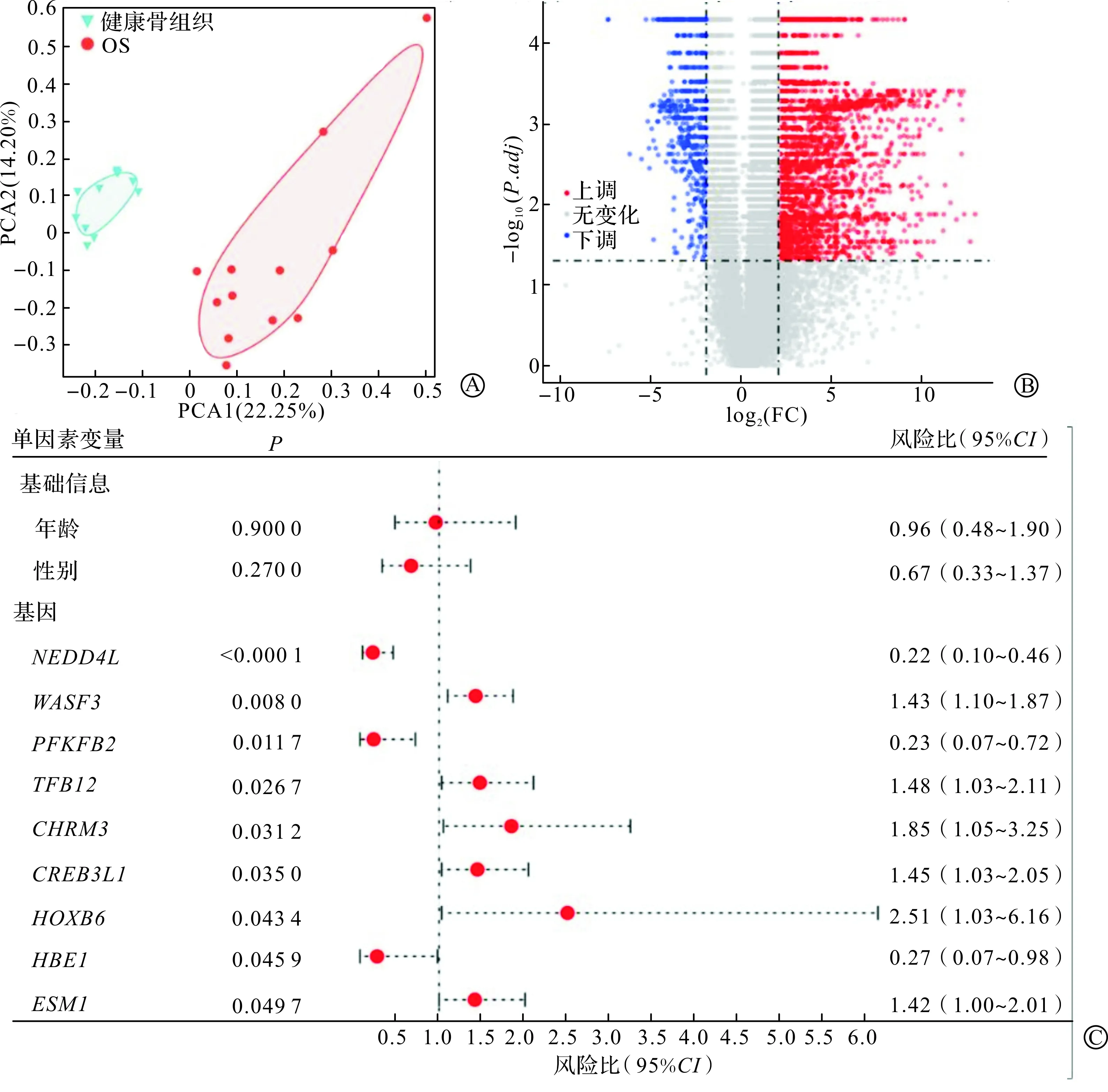
A:OS患者与健康骨组织PCA降维散点图;B:OS患者与健康骨组织DEG火山图;C:DEGs与基础信息单因素Cox回归分析结果森林图。OS:骨肉瘤;DEGs:差异表达基因;FC:差异倍数。
2.2 多因素Cox回归预后模型的构建与评估
对2.1得到的9个基因进行多因素Cox回归分析以构建预后模型,评估这9个基因在123个样本中的预后显著性。采用rms包绘制所有多因素分析P<0.05的基因的列线图(图2A)。结果显示,只有神经前体细胞表达发育下调样基因4(neural precursor cell-expressed developmentally down-regulated 4 like,NEDD4L)、TFBI2和WASF3共同对预后存在显著影响(P<0.05),其中NEDD4L对总分贡献度最大,P值最小(P<0.000 1)。以这3个基因构成的回归模型一致性指数为0.76,提示模型与实际结果较为一致。
使用Predict函数计算多因素风险评分,对评分进行排序并绘制这3个基因的风险评分热图(图2B)。图中上方柱状图纵坐标为患者的风险评分,横坐标按照风险评分由低到高的顺序从左向右排列,中、下两图横坐标均与之对应;中间散点图纵坐标为患者生存时间;下方热图为3个基因Z-score归一化后的表达量。结果显示,NEDD4L表达量越低、WASF3和TFPI2表达量越高,患者风险评分越高、预后越差。
按风险评分排序,以中位数(-0.045 4)为阈值,将患者分为高风险组(62例)和低风险组(61例),绘制总生存曲线(图2C)与ROC曲线(图2D)。结果显示,低风险组与高风险组在总生存率上存在显著差异(P<0.000 1),并且低风险组预后较好,其1、3和5年AUC分别为0.76、0.84、0.86。图2C~D说明以NEDD4L为主的多因素风险评分可以有效预测OS患者的预后。
2.3 NEDD4L TME分析
使用estimate包对数据集TARGET-OS进行免疫浸润分析,获得88例OS患者的TP。按照TP排序,将88个样本分为高纯度组(44例)和低纯度组(44例),对NEDD4L的表达量进行Wilcoxon秩和检验,并绘制小提琴图(图3A)。结果显示,NEDD4L在高纯度组表达量显著高于低纯度组(P=0.005 7)。
使用Spearman相关性系数对TP与NEDD4L进行相关性分析并绘制相关性散点图(图3B)。结果显示,TP与NEDD4L表达量显著正相关(R=0.35,P=0.001 0)。图3A~B共同说明NEDD4L在TME中主要表达于OS细胞。
通过对CCLE数据库和GEPIA网站进行检索获得前文2.1部分筛选到的9个基因在不同OS细胞系以及TCGA肿瘤分类上的表达量并绘制热图(图3C)。结果显示,NEDD4L相较于其他8个基因高表达于多种OS细胞系以及多种其他肿瘤。
使用seurat包对数据集GSE162454进行Louvain聚类后最终获得了5群细胞,通过UMAP算法对数据集进行降维分析并绘制NEDD4L的特征图(图3D)。结果显示,NEDD4L在TME中主要分布在髓样细胞和OS两群细胞。
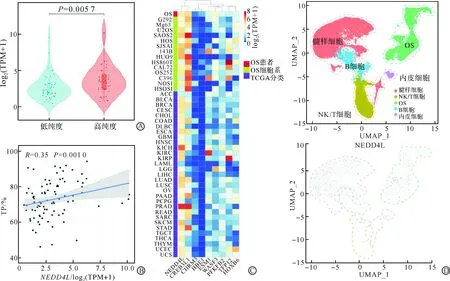
A:免疫浸润分析TME中NEDD4L表达量小提琴图;B:TME中NEDD4L与TP相关性散点图;C:9个DEGs在OS患者、细胞系以及TCGA肿瘤分类的表达量热图;D:OS单细胞测序UMAP降维散点图(上)以及NEDD4L表达特征图(下)。OS:骨肉瘤;TME:肿瘤微环境;TP:肿瘤纯度;DEGs:差异表达基因。
2.4 基于NEDD4L的单因素预测模型及验证
对数据集GSE14359(原发灶10例,转移灶8例)以上9个基因的表达量进行Wilcoxon秩和检验并绘制分边小提琴图(图4A),结果显示NEDD4L在肺转移灶的表达量显著高于原发灶(P=0.005 7)。
对TARGET-OS进行GSEA富集分析,以NEDD4L表达量的中位数为阈值,将88位患者分为高表达组(44例)和低表达组(44例),检验两组之间基因集的差异并用ggplot2包绘制P<0.05的通路(图4B)。图4B分为上、中、下三部分,三部分的横坐标相同:上面部分纵坐标为富集分数(enrichment score,ES),每个基因都有ES,从左至右ES依次相加并连成折线,折线峰值处即为基因集的ES;中间部分每条竖线对应相应通路下的一个基因;下面部分为每个基因的Rank值分布图,纵坐标Rank值即为基因log标准化后的FC;右侧图例为通路名称及对应的ES及P值。结果显示,低表达NEDD4L与包括子宫内膜癌、甲状腺癌、前列腺癌、结肠癌在内的多种癌症通路有关。
绘制NEDD4L的总生存曲线并用Log rank检验进行评估(图4C)。结果显示,高表达组生存预后显著好于低表达组(P=0.002 3)。绘制NEDD4L的ROC曲线(图4D),其1、3和5年的AUC分别为0.78、0.72和0.72。图4C~D说明NEDD4L可以有效预测OS患者的预后。
2.5 NEDD4L表达情况的初步验证
利用蛋白质印迹实验检测人OS细胞系MG63、143B和HOS以及人成骨细胞系FOB1.19 NEDD4L蛋白表达情况(图5A),结果显示,NEDD4L在3种OS细胞系中的表达量均显著低于FOB1.19细胞(MG63:P=0.029 4;143B:P=0.007 1;HOS:P=0.026 0;图5B)。
利用尾静脉注射的方法构建小鼠OS肺转移模型,21 d后鼠肺取材固定并进行HE(图6A)和组化染色(图6B),结果显示,NEDD4L主要定位在肺组织,OS转移灶未见明显染色。蛋白质印迹和组化结果共同说明NEDD4L在OS中的表达水平低于健康组织。
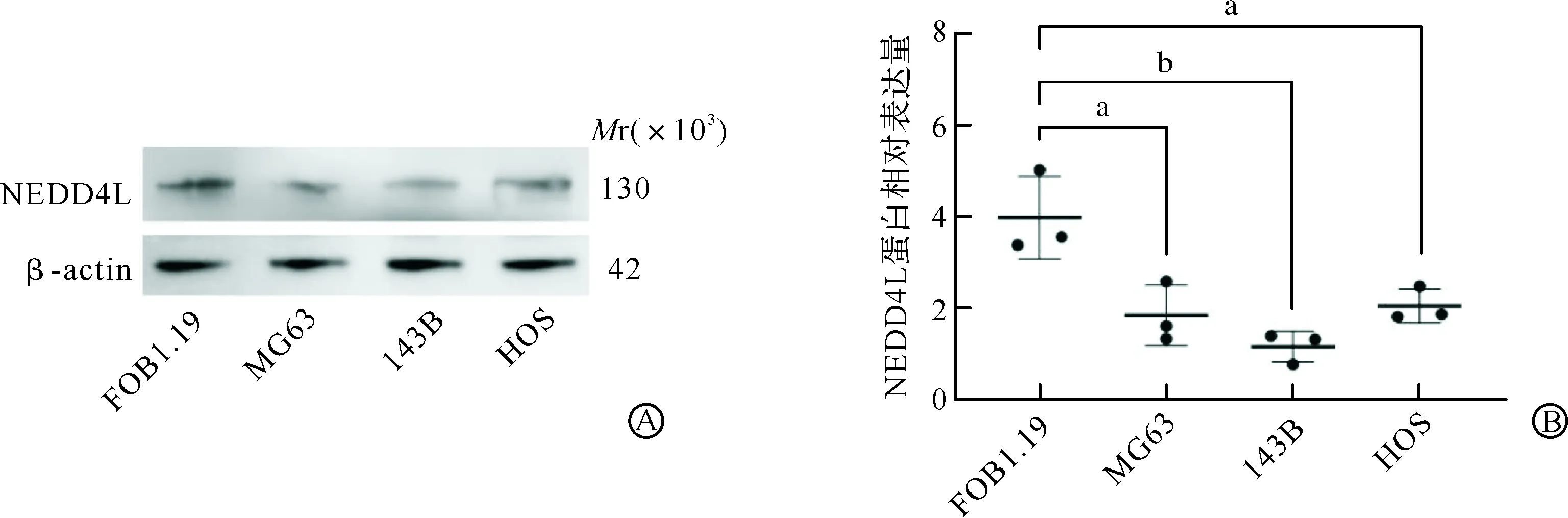
A:不同人OS细胞系NEDD4L表达情况蛋白质印迹条带结果(n=3);B:不同人OS细胞系NEDD4L表达情况统计图。 aP<0.05, bP<0.01。

A:小鼠OS肺转移灶HE染色结果;B:小鼠OS肺转移灶NEDD4L组化染色结果。比例尺为100 μm,黑色箭头为OS肺转移灶。
3 讨论
本研究通过GEO数据库、TARGET数据库、CCLE数据库以及TCGA数据库获得了多个不同测序类型、不同测序对象的OS相关数据集。对RNA-seq数据集GSE126209进行差异表达分析,共获得2 806个DEGs,其中上调基因2 284个,下调基因522个。在Microarray数据集中对DEGs进行单因素Cox回归分析,共得到9个影响预后的基因,对这9个基因进行多因素Cox回归分析并构建预后预测模型,只有NEDD4L、WASF3和TFPI2与预后显著相关,其中NEDD4L对预后的贡献最大。进一步对NEDD4L进行免疫浸润分析、单细胞测序分析发现,在TME中NEDD4L主要表达于OS细胞,且转移灶的表达量显著高于原发灶。使用ROC曲线对NEDD4L的单因素及多因素Cox回归分析预后预测模型进行评估,无论是1年、3年还是5年,其单因素和多因素预后预测模型AUC均大于0.7。这表明本研究所构建的模型在转录组水平可能对OS患者的预后进行有效预测。最后,本研究初步对3种人OS细胞系和人成骨细胞FOB1.19进行了蛋白水平的验证,同时对小鼠OS肺转移灶进行了组化染色,结果均提示与正常组织对比,NEDD4L在OS中的表达减少,与预后预测变化趋势一致。
NEDD4L,又叫NEDD4-2,是一种E3泛素连接酶,能够与多种膜蛋白结合并调节其功能,对于维持细胞内环境稳态是必不可少的[14]。NEDD4L在多种癌症的诊断与预后中已经被证明存在保护作用,如蒋兴旺等[15]利用实时荧光PCR、蛋白质印迹等方法对胃癌组织与癌旁组织进行NEDD4L表达量的检测,发现低表达NEDD4L可能与胃癌的发生密切相关,且可能作为判断胃癌预后的潜在指标;LI等[16]利用生物信息学方法证明低表达NEDD4L促进肺腺癌的进展并进行了实验验证;GUARNIERI等[17]利用miRNA直接抑制NEDD4L,发现这种抑制可以促进乳腺癌的发生。在本研究中,蛋白质印迹实验表明、NEDD4L在OS细胞系中的表达量显著低于成骨细胞对照,而GSEA富集分析结果也显示低表达NEDD4L与多种肿瘤发生通路相关,这提示NEDD4L的作用机制在不同肿瘤之间存在共性,其保护作用可能极为重要。有趣的是,免疫浸润分析和单细胞测序分析结果均表明在TME中NEDD4L主要表达在OS细胞上,其在免疫细胞或间质细胞上的表达量反而更低。尽管NEDD4L在其他肿瘤中的作用已经初步得到研究,但尚无其在OS中相关作用的直接报道。既往研究表明NEDD4L可以下调肿瘤的TGF-β和Wnt信号通路[14,18],而这两个通路在OS的进展中发挥着重要作用[19-20],因此高表达NEDD4L很有可能对OS患者的预后具有保护作用,本研究通过生物信息学方法和初步实验验证推导出了这一点。
本研究具有以下优势:第一,在不同数据库来源的传统RNA-seq测序、Microarray以及单细胞测序数据集上使用多种生物信息学方法初步论证了NEDD4L对OS的预后具有预测价值;第二,首次构建以NEDD4L为主的OS预后预测模型,揭示NEDD4L对于OS可能具有保护作用。同时本研究存也存在局限性:第一,本研究主要结论由生物信息学方法分析得出,仅对NEDD4L的表达情况进行了初步验证,缺少大样本量的预后验证;第二,部分临床信息并非公开,无法纳入分析,而诸如病理类型、分期、化疗、手术等临床自变量与OS预后密切相关,NEDD4L与这些临床自变量之间可能存在不同程度的相关性。因此,在未来的研究中我们将进一步收集OS患者的临床信息和标本,通过体内以及体外实验对NEDD4L的作用进行进一步验证并对其在TME中表达情况相反的原因进行探讨。
综上所述,本研究基于生物信息学方法筛选目标基因,并对其进行大样本分析,对预测OS患者的预后评估提供临床新思路。
