GsERF6基因过表达对水稻耐盐碱性的影响
2023-02-10才晓溪胡冰霜孙明哲贾博为孙晓丽
才晓溪 胡冰霜 沈 阳 王 研 陈 悦 孙明哲 贾博为 孙晓丽
研究简报
基因过表达对水稻耐盐碱性的影响
才晓溪**胡冰霜**沈 阳 王 研 陈 悦 孙明哲 贾博为 孙晓丽*
1黑龙江八一农垦大学农学院 / 作物逆境分子生物学实验室, 黑龙江大庆 163319
乙烯响应因子(ERF)是植物特有的一类转录因子, 在响应非生物胁迫中具有重要作用。本研究通过生物信息学发现, 野生大豆耐盐碱ERF转录因子GsERF6与水稻ERF同源蛋白的氨基酸序列相似性很高, 均包含1个高度保守的AP2结构域。为探究基因在水稻耐盐碱应答中的作用, 通过遗传转化、PCR和半定量RT-PCR鉴定, 获得了2个纯合的过表达转基因水稻株系。表型鉴定表明, 200 mmol L–1NaHCO3处理下转基因水稻的存活率、相对含水量、超氧化物歧化酶、过氧化物酶、过氧化氢酶活性、可溶性糖和脯氨酸含量均显著高于对照, 活性氧积累则反之。实时荧光定量PCR分析表明, 40 mmol L–1NaHCO3处理6 h后渗透调节基因和在转基因水稻中的表达量显著高于对照。本研究表明, 水稻中的过表达可通过提高ROS清除水平、渗透调节能力及胁迫应答基因的表达来提高其耐盐碱性。
水稻; 耐盐碱; 乙烯响应因子; 野生大豆;
土壤盐碱化严重影响作物的产量, 是全球粮食安全所面临的巨大挑战之一。盐胁迫会引发植物细胞的离子毒害、渗透胁迫和氧化伤害, 造成植物体内代谢紊乱[1]。植物遭受碱胁迫时, 除了上述盐胁迫造成的伤害外, 还需应对外界高pH伤害和碳酸盐胁迫。目前, 全球超过8.33×108hm2的土壤已经受到盐碱化的影响, 而我国有盐碱地9.913×108hm2, 位居世界第三。其中, 东北苏打盐碱地面积高达3.73×106hm2[2], 开发利用这些后备土地资源, 对于我国粮食安全具有重大意义。科研人员经过多年研究, 提出了物理方法、化学措施、生物种植及农艺管理措施等盐碱地综合改良技术。其中, 较为成熟的是利用改良盐碱地种植耐盐碱作物, 可有效解决盐碱地改良过程中脱盐和抑制返盐的难题[3]。因此, 挖掘耐盐碱基因, 培育特定的耐盐碱作物新品种, 是盐碱地资源开发利用的重要途径。
乙烯响应因子(ethylene responsive factor, ERF)是植物特有的一类转录因子, 属于AP2/ERF (APETALA2/ ethylene responsive factor)转录因子家族, 包含1个高度保守的AP2 DNA结合结构域, 可以特异地结合GCC-box (AGCCGCC)、DRE/CRT (A/GCCGAC)等顺式作用元件, 在多种非生物逆境响应中扮演重要角色[4-7]。研究表明在烟草(L.)中过表达小黑杨, 可通过抑制丙二醛(malondialdehyde, MDA)和活性氧(reactive oxygen species, ROS)积累, 提高转基因烟草的耐盐性[8]。过表达大豆(L.)可以增强转基因烟草对盐碱和干旱胁迫的耐受性[9]。极耐寒的枳()中3个ERF转录因子基因和可直接调控过氧化物氧化还原酶基因、谷胱甘肽S-转移酶、ACC合成酶基因和棉子糖合酶基因的表达, 有效增强ROS清除和渗透调节能力提高耐寒性[10-12]。通过调节抗坏血酸(ascorbic acid, AsA)的生物合成, 增强拟南芥()的耐盐性[13]。水稻(L.) ERF亚家族的转录抑制因子基因(ethylene-responsive element binding proteins 1/2)正向调控水稻耐盐性[14], 而过表达降低了水稻对盐胁迫的耐受性[15]。
野生大豆(L.)耐盐碱性状突出, 是耐盐碱基因挖掘与大豆新种质创新不可或缺的资源[16]。课题组前期从野生大豆中克隆出多个耐盐碱功能显著的基因, 包括2个乙烯响应因子基因和[17-18], 利用过表达技术确定了上述2个基因可以增强拟南芥对苏打盐碱胁迫(NaHCO3)的耐受性。鉴于优异的耐苏打盐碱功能, 本研究将其在水稻中过表达, 通过对比转基因植株及其对照在盐碱胁迫下表型、形态、生理指标以及渗透调节相关基因的表达, 为下一步培育耐盐碱水稻提供材料储备。
1 材料与方法
1.1 试验材料
选用粳型常规水稻‘农丰1701’为试验材料, 生育期138 d左右, 由黑龙江八一农垦大学自主培育。大肠杆菌DH5α、农杆菌EHA105菌株和植物载体pCAMBIA230035SU, 由黑龙江八一农垦大学作物逆境分子生物学实验室保存。
1.2 ERF亚家族蛋白生物信息学分析
通过文献检索结合国家水稻数据中心数据库, 查找已具有耐逆功能报道的水稻ERF转录因子。结合Blastp比对从水稻中鉴定相似性较高的同源蛋白(值4e-26), 下载其氨基酸序列, 并通过Pfam、SMART软件分析确定蛋白保守结构域。采用Clustal X软件(Ver.1.81, European Bioinformatics Institute)分析蛋白序列相似性, 利用MEGA 6.0软件的邻接法(Neighbor-Joining, NJ)分析进化关系。
1.3 GsERF6植物过表达载体构建
采用USER克隆[19]技术, 将具有新霉素抗性基因的pCAMBIA230035SU载体与GsERF6完整CDS区构建重组质粒。以前期获得的pGEM-T-GsERF6质粒为模板进行PCR扩增, 所用引物为GsERF6-F/R (表1)。PCR总反应体系50 μL, 包括2×EasyPCR Super Mix 25 μL, 上下游引物(10 μmol L–1)各1.5 μL, ddH2O 21 μL以及质粒pGEM-T-GsERF6 1 μL。反应条件为: 95°C预变性5 min; 95℃变性30 s, 60℃退火30 s, 72°C延伸60 s, 30个循环; 72℃终延伸10 min。扩增产物与I及I消化的pCAMBIA230035SU片段连接, 转化大肠杆菌DH5α, PCR鉴定阳性菌落并送交测序。

表1 本研究所用的引物
1.3 水稻遗传转化
将上述构建的过表达载体转化农杆菌EHA105, 并利用农杆菌介导法对水稻愈伤组织进行遗传转化[20]。对饱满和无菌斑的‘农丰1701’水稻种子消毒后, 平铺于水稻愈伤诱导培养基上(营养琼脂, nutrient broth, NB, pH 5.8), 28℃暗培养4周左右诱导愈伤组织。挑选黄色颗粒状愈伤组织依次转接入继代培养基(NB, pH 5.8)、共培养培养基(NB+20 mg L–1乙酰丁香酮, pH 5.2)、筛选培养基(NB+100 mg L–1G418+100 mg L–1阿莫西林克拉维酸钾, pH 5.8)中, 筛选获得的抗性苗转移至生根培养基(Murashige and Skoog, MS+100 mg L–1阿莫西林克拉维酸钾, pH 5.8)中, 得到T0代植株。繁种后收获T1代种子, 本研究使用的是T3代纯合转基因水稻种子。
1.4 转基因植株检测
使用Axygen公司的AxyPrep Genomic DNA purification试剂盒, 提取水稻叶片基因组DNA。以基因组DNA为模板进行PCR检测, 所用基因特异引物为GsERF6- PCR-F/R (表1)。反应体系15 μL: 2×EasyPCR Super Mix 7.5 μL, 上下游引物(10 μmol L–1)各0.3 μL, ddH2O 4.9 μL以及模板DNA 2 μL。PCR反应条件: 95℃预变性5min, 95℃变性30 s, 60℃退火30 s, 72℃延伸60 s, 30个循环后, 72℃终延伸10 min。
使用Thermo公司的TRIzol试剂法提取PCR阳性植株总RNA, 经诺唯赞生物科技股份(南京)有限公司gDNA wiper清除基因组DNA, 再利用此公司试剂盒HiScript III RT SuperMix反转录合成cDNA。以此为模板进行半定量PCR检测, 所用基因特异引物为GsERF6-RT-F/R, 内参基因引物为OsElf1-α-F/R (表1)。RT-PCR反应体系20 μL: 2× EasyPCR Super Mix 10 μL, 上下游引物(10 μmol L–1)各0.4 μL, ddH2O 7.2 μL以及cDNA 2 μL。PCR反应条件: 95℃预变性5 min, 95℃变性30 s, 60℃退火30 s, 72℃延伸15 s, 28个循环(内参25个循环); 72℃终延伸10 min, 4℃终止反应。
1.5 水稻盐碱胁迫处理
将纯合转基因株系和野生型水稻种子浸泡发芽, 待破胸露白, 移植到田园土、君子兰土、蛭石以体积比5∶3∶2均匀混合在营养钵中, 于人工气候室(25℃、相对湿度70%、光照14 h/黑暗10 h)培养至三叶期。选取长势一致的水稻幼苗, 分别用含有0 mmol L–1和200 mmol L–1NaHCO3(pH 8.4)溶液浇灌进行盐碱胁迫处理12 d, 每4 d浇灌1次处理液, 每次浇灌2 L[21]。观察植株生长状态, 并测定存活率和地上部分相对含水量, 存活率(%)=存活株数/总株数×100, 地上部分相对含水量(%)=(鲜重-干重)/鲜重×100。每个处理设置3个独立的生物学重复, 每个生物学重复中每个株系不少于60株。
1.6 水稻盐碱胁迫生理指标检测
分别在处理前和处理7 d后剪取水稻幼苗的地上部分,以单株水稻为1个样本, 每个指标测定20个样本。按照李合生等[22]方法测定超氧化物歧化酶(SOD, EC: 1.15.1.1)、过氧化物酶(POD, EC: 1.11.1.7)和过氧化氢酶(CAT, EC: 1.11.1.6)活性; 采用氮蓝四唑(nitrotetrazolium blue chloride, NBT)和3,3’-二氨基联苯胺(3,3’-diaminobenzi-dine, DAB)染色液进行染色[23]; 采用硫酸蒽酮比色法[24]测定可溶性糖含量; 采用茚三酮比色法[24]测定脯氨酸含量。
1.7 实时荧光定量PCR表达水平检测
在盐碱胁迫基因表达检测时, 选择水培法[20]对水稻进行培养和盐碱处理, 便于保证盐碱处理强度的一致性, 减少误差。将野生型和纯合转基因株系水稻种子浸泡发芽,待芽长约2 mm时, 移至含有Youshida营养液(pH 5.5~5.8)的96孔水培盒里, 于人工气候室培养, 其间每2 d换1次营养液。挑选长势一致的三叶期幼苗, 用0 mmol L–1和40 mmol L–1NaHCO3溶液处理6 h, 取相同部位相同长度水稻叶片, 迅速置于液氮速冻, 于-80℃保存备用。
提取总RNA并反转录合成cDNA。采用全式金生物技术股份有限(北京)公司试剂盒TransStart Top Green qPCR SuperMix于实时荧光定量PCR仪CFX96 Touch (BioRad公司)上进行实时荧光定量PCR (quantitative real-time PCR)检测。反应体系15 μL: 2×Top qMix 7.5 μL, 上游和下游引物(10 μmol L–1)各0.3 μL, ddH2O 4.9 μL以及cDNA 2 μL。PCR反应条件: 94℃预变性30 s, 94℃变性5 s, 60℃退火15 s, 72℃延伸20 s, 40个循环。所用和基因特异引物见表1。以未处理时野生型水稻中基因表达量作为1, 采用2–DDCt法计算基因相对表达量。
1.8 数据处理及统计分析
采用Microsoft Excel 2016进行数据处理及绘图, 用Student’s检验进行显著性分析。
2 结果与分析
2.1 GsERF6基因生物信息学分析
通过文献检索结合国家水稻数据中心数据库, 查找已报道的水稻耐逆ERF转录因子, 结合Blastp比对从水稻中鉴定出5个相似性较高的同源蛋白(值4e-26), 分别为OsERF105 (LOC_Os04g46240)、OsSta2 (LOC_Os02g43820)、OsBIERF3 (LOC_Os02g43790)、OsAP23 (LOC_Os03g05590)和OsERF15 (LOC_Os03g64260) (图1)。多重序列比对结果显示, GsERF6与水稻的ERF家族蛋白序列相似性很高, 且均包含1个蛋白序列高度保守的AP2结构域(图2)。
2.2 GsERF6过表达转基因水稻的获得
2.2.1过表达载体的构建 将含有基因CDS区的片段插入植物表达载体pCAMBIA230035SU, 该载体由组成型启动子35S驱动基因表达, 且含有新霉素植物选择标记基因(图3-A)。采用基因特异引物PCR扩增获得长度为840 bp的目的条带(图3-B), PCR产物经USER酶切后, 与经I及I线性化的pCAMBIA230035SU连接。经菌落PCR鉴定获得阳性克隆pC35SU-GsERF6 (图3-C), 用于下一步农杆菌介导的水稻遗传转化。
2.2.2 水稻遗传转化及转基因植株的鉴定 在获得的多个抗性的水稻株系中, 随机选择8株(#1~#8)采用基因特异引物进行PCR检测。图4-A显示, 抗性植株均能扩增出400 bp的特异性条带, 而野生型(对照)中未检测到条带, 表明基因已整合到水稻基因组中。进一步提取2个转基因株系(#1和#2)总RNA, 进行反转录获得cDNA后, 以水稻作为内参基因,进行半定量RT-PCR检测。图4-B显示, 经过25个循环, 内参基因均扩增出条带; 28个循环, 对照未能扩增出条带,而转基因水稻中可检测到基因的转录。
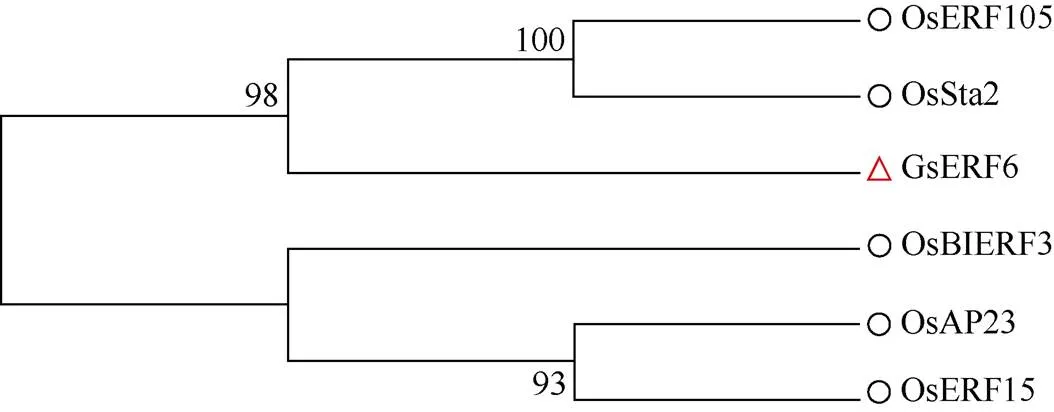
图1 GsERF6与水稻中同源ERF蛋白进化关系分析
“”代表水稻ERF基因; “D”代表野生大豆ERF基因。
“” stands for ERF gene of rice; “D” stands for ERF gene of wild soybean.
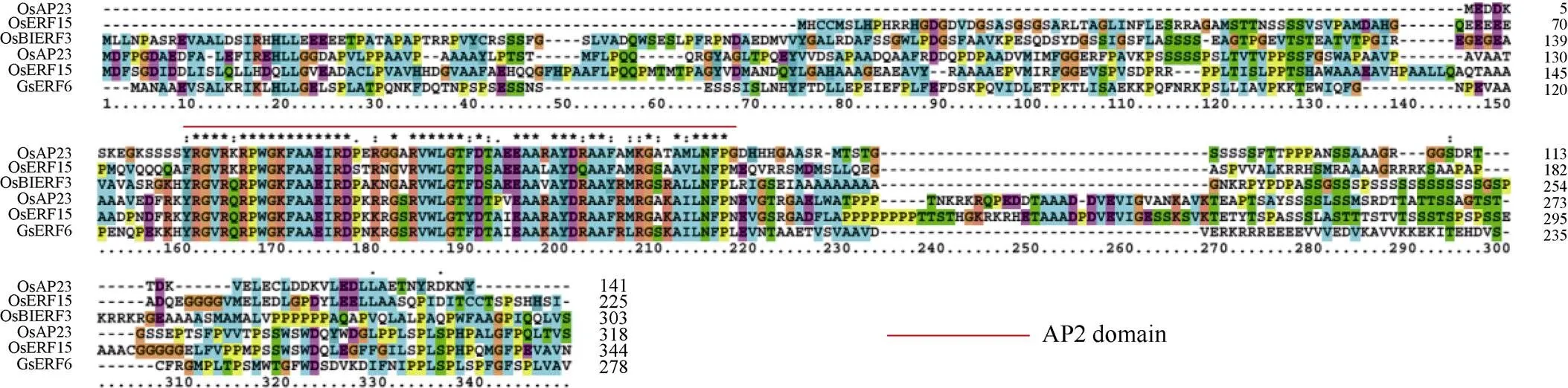
图2 GsERF6与水稻中同源ERF蛋白序列比对

图3 GsERF6植物超量表达载体的构建
A:过表达载体示意图; B:基因的克隆; M: DNA marker; 1~3: GsERF6 PCR产物; C: pC35SU-GsERF6菌落PCR鉴定; M: DNA marker; −: 阴性H2O对照; +: 阳性质粒对照; 1: pC35SU-GsERF6。
A: theoverexpression vector; B: gene clone of; M:DNA marker; 1–3: PCR product of GsERF6; C: PCR identificationof recombinantclones harboring the pC35SU-GsERF6 con-struct; M: DNA marker; −: negative H2O control; +: positive plas-mid control; 1: PCR products of the pC35SU-GsERF6 clones.
2.3 GsERF6转基因水稻耐盐碱性分析
选取2个pC35SU-GsERF6 T3代纯合转基因水稻株系(#1和#2), 以野生型水稻作为对照, 进行耐盐碱性分析。如图5-A所示, 正常生长条件下(0 mmol L–1)时转基因水稻长势与对照不存在显著差异; 而200 mmol L–1NaHCO3处理12 d后,转基因水稻各株系生长状况明显优于对照, 具体表现为对照植株地上部分严重萎蔫、失绿, 而转基因水稻生长状态较好。统计学分析显示, 200 mmol L–1NaHCO3处理下,转基因水稻#1、#2存活率分别为75%和61%, 显著高于对照31% (图5-B); 同时转基因水稻地上部分相对含水量(85%、82%)也显著高于对照(72%), 说明转基因水稻耐盐碱性明显提高(图5-C)。
2.4 GsERF6转基因水稻ROS积累及抗氧化物酶活性分析
DAB和NBT染色结果显示, 未进行盐碱处理时转基因水稻与对照植株染色颜色较浅, 盐碱处理后对照染色程度明显深于过表达植株(图6-A, B), 说明O2܋和H2O2积累量高于过表达植株。进一步对盐碱处理前后的转基因水稻和对照进行SOD、POD和CAT活性测定。结果显示, 正常情况下转基因水稻与对照植株体内SOD、POD和CAT活性无显著差异, 盐碱处理后上述抗氧化酶活性均有不同程度提高(图6-C~E), 但转基因水稻中酶活显著高于对照, 表明可能通过提高抗氧化物酶活性, 减少ROS积累, 从而提高水稻耐盐碱性。
2.5 GsERF6转基因水稻渗透调节物质含量分析
对转基因水稻渗透调节物质含量分析发现, 盐碱胁迫前,转基因水稻与对照植株体内可溶性糖和脯氨酸含量均无显著差异; 盐碱胁迫条件下, 上述2个物质的含量均不同程度地提高, 但转基因水稻中的可溶性糖和脯氨酸含量显著高于对照(图7-A, B)。
2.6 GsERF6转基因水稻盐碱应答基因表达分析
为进一步探究基因调控水稻耐盐碱性的作用机制, 我们分析了对照和转基因水稻胁迫下2个盐碱应答标记基因([25]和[26])的表达水平。如图8所示, 正常生长条件下,和的表达在对照和转基因水稻中无显著差异; 经40 mmol L–1NaHCO3处理6 h后二者表达水平在对照和转基因水稻中均上调。其中, 盐碱胁迫处理后转基因水稻中(图8-A)和(图8-B)的表达水平均极显著高于对照。综上表明,过表达可通过提高水稻中盐碱应答基因的表达, 来调控植株对盐碱胁迫的响应。
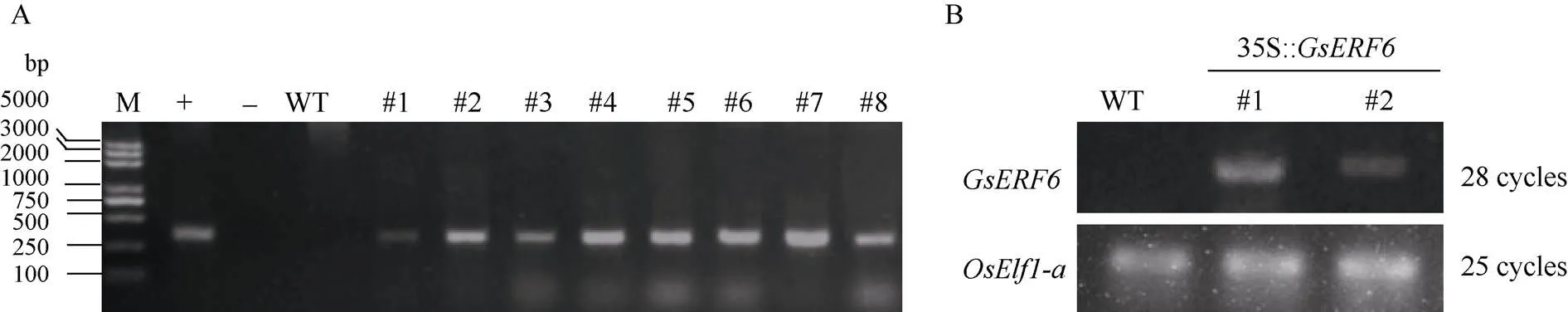
图4 GsERF6转基因株系的分子检测
A: 转基因抗性植株PCR检测; B: 转基因植株RT-PCR检测; M: DNA marker; −: 阴性H2O对照; +: 阳性质粒对照; WT: 野生型对照; #1~#8: 独立株系。
A: PCR detection of resistant plants transformed with; B: RT-PCR detection oftransgenic rice; M: DNA marker; −: negative H2O control; +: positive plasmid control; WT: wild type control; #1–#8:resistant seedlings.
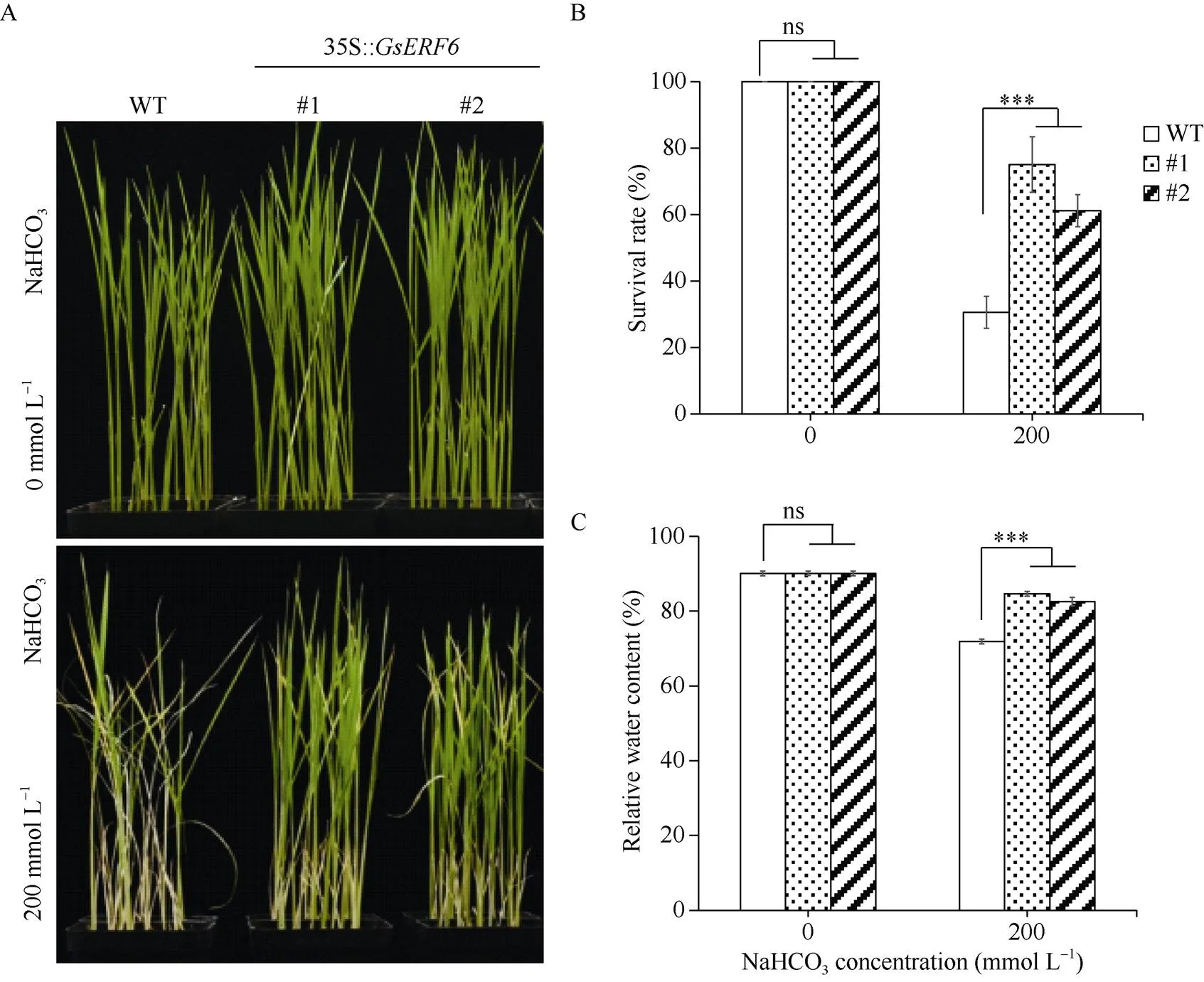
图5 GsERF6转基因水稻幼苗期耐盐碱性分析
A: NaHCO3处理前后各株系生长状态; B: 水稻幼苗存活率; C: 水稻幼苗相对含水量。差异显著性分析采用-test方法。
A: phenotype of WT and transgenic rice seedlings treated with NaHCO3; B: the survival rates of WT and transgenic rice seedlings; C: the relative water content of WT and transgenic rice seedlings. The significant difference was evaluated by the Student’s-test. *:< 0.05; **:< 0.01; ***:< 0.001.
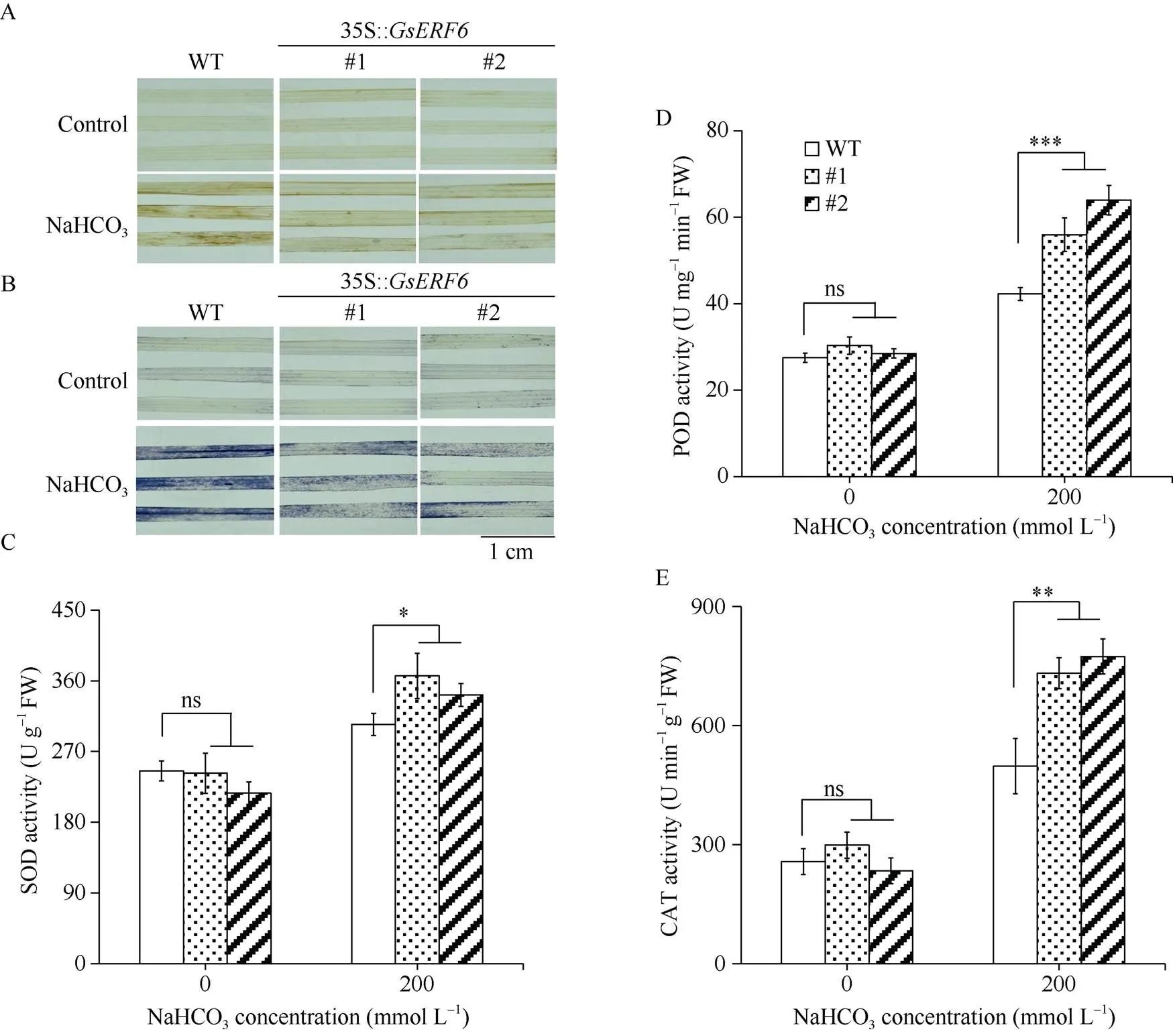
图6 GsERF6转基因水稻盐碱胁迫下ROS积累及抗氧化物酶活分析
A: DAB染色; B: NBT染色; C: SOD活性; D: POD活性; E: CAT活性。差异显著性分析采用-test方法。
A: DAB staining; B: NBT staining; C: SOD activity; D: POD activity; E: CAT activity. The significant difference was evaluated by the Student’s-test. *:< 0.05; **:< 0.01; ***:< 0.001.
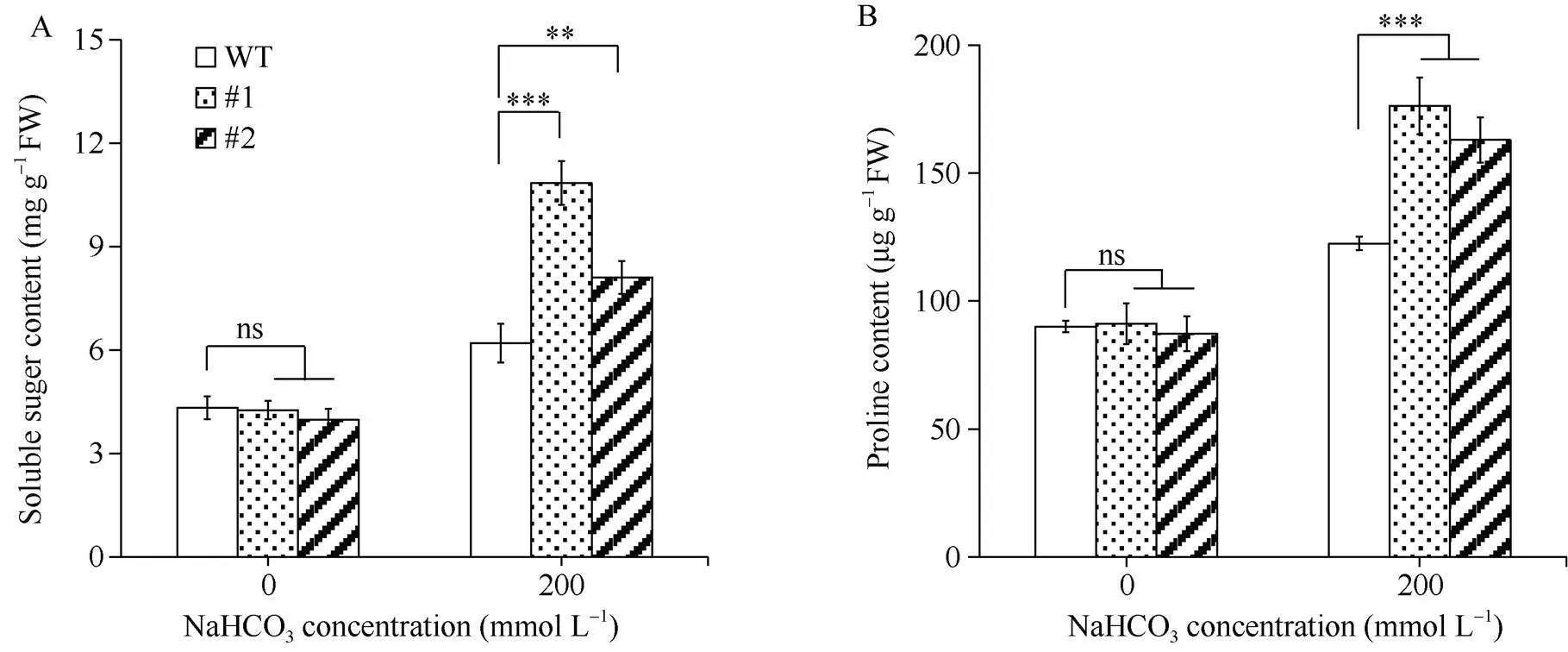
图7 GsERF6转基因水稻盐碱胁迫后渗透调节物质含量分析
A: 可溶性糖含量; B: 脯氨酸含量。差异显著性分析采用-test方法。
A: soluble sugar content; B: proline content. The significant difference was evaluated by the Student’s-test. *:< 0.05; **:< 0.01; ***:< 0.001.
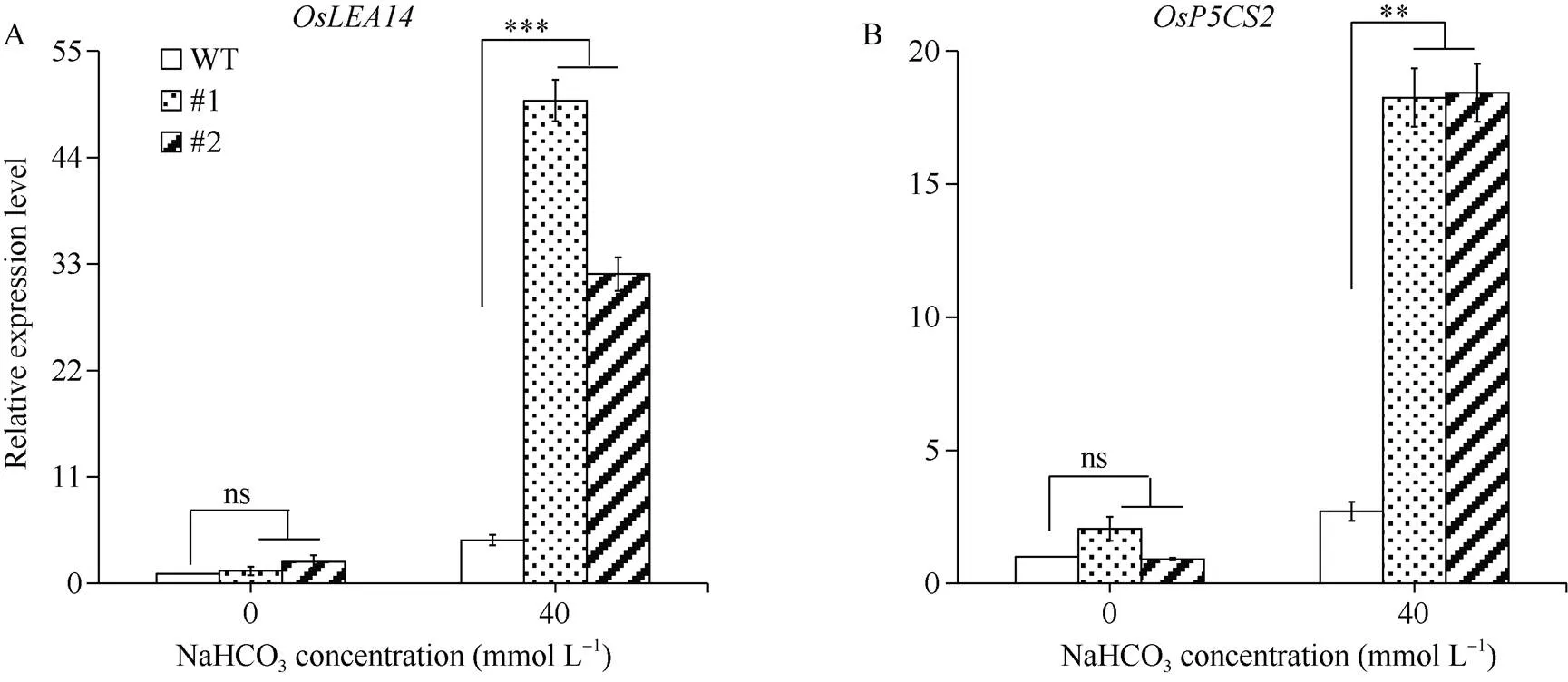
图8 GsERF6转基因水稻盐碱应答基因表达分析
A:基因表达量变化; B:基因表达量变化。差异显著性分析采用-test方法。
A: the relative expression level ofgenes; B: the relative expression level ofgenesThe significant difference was evaluated by the Student’s-test. *:< 0.05; **:< 0.01; ***:< 0.001.
3 讨论
GsERF6是课题组前期从野生大豆中鉴定的1个调控苏打盐碱耐性的ERF转录因子。Yu等[18]发现在拟南芥中过表达提高了转基因株系在HCO3−(NaHCO3和KHCO3)处理下的种子萌发和幼苗生长。本研究首先在水稻中鉴定到5个GsERF6同源蛋白(图1), 都包含1个高度保守的AP2结构域, 保守的氨基酸序列为丙氨酸-14 (Ala-14)和天冬氨酸-19 (Asp-19) (图2)。有研究表明水稻、和基因均参与盐胁迫应答[27-29]。本研究证实NaHCO3处理后,转基因水稻的存活率和相对含水量显著高于野生型(图5), 说明基因在水稻中过表达同样提高了苏打盐碱胁迫的耐性。
ROS是参与调节植物生长发育和响应逆境胁迫的重要指标[30]。植物在受到盐碱胁迫时, 会产生过量的O2܋和H2O2等有害物质, 打破体内ROS动态平衡状态, 造成细胞氧化损伤, 最终导致细胞死亡[31]。Wang等[6]研究表明, 拟南芥中ERF6可以被MPK6 (mitogen-activated protein kinase 6)磷酸化, 从而与ROSE7/GCC box (ROS- responsive cis-acting element 7)结合调节ROS响应基因转录参与氧化胁迫应答。Zhang等[13]发现,的过表达减少了盐胁迫下拟南芥叶片中H2O2的积累, 且ROS清除基因(、和)的表达在转基因植株中显著上调。本研究与上述得出相似结论,在水稻中的表达, 降低了盐碱胁迫下O2܋和H2O2含量的积累, 增强了抗氧化酶SOD、POD和CAT的活性(图6), 通过提高ROS的清除效率, 减轻转基因水稻氧化损伤。
可溶性糖、脯氨酸等渗透调节物质积累有助于植物应对外界逆境胁迫[32]。研究发现黄花苜蓿()中响应高盐、高温、干旱等非生物胁迫, 在盐胁迫下过表达植株体内脯氨酸含量显著提高[33]。此外,等多个ERF转录因子均被报道参与植物体内氧化应激和渗透调节过程[34-35]。本研究发现转基因水稻在盐碱胁迫下的可溶性糖和脯氨酸含量显著高于野生型(图7), 表明基因参与盐碱胁迫下植物体内氧化还原和渗透调节过程。
本研究中, 我们发现盐碱处理下,转基因水稻中和基因的表达显著高于对照(图8)。有研究表明烟草中过表达, 通过增加脯氨酸含量减轻高盐、高渗透胁迫对烟草造成的氧化损伤[26]。与此结果一致,在拟南芥中过表达同样促进了、、等多个非生物胁迫应答基因的表达[17]。PtrERF108可特异性结合棉子糖合酶基因启动子的GCC-box, 并激活其表达, 进而调控植株体内RafS活性和棉子糖含量, 增强枳的抗寒性[11]。我们前期通过酵母转录激活实验证实GsERF6蛋白具有转录激活活性, 且亚细胞定位实验表明GsERF6蛋白定位于细胞核[17]。我们推测可能直接或间接调控和基因的表达, 从而参与水稻盐碱胁迫应答过程。鉴于其与其他ERF转录因子高度保守的AP2 DNA结合结构域, 我们推测GsERF6也能够识别和结合DRE/CRT和GCC-box元件。
[1] 李健, 王逸茹, 张凌霄, 孙明昊, 秦阳, 郑军. 玉米基因在盐胁迫应答中的功能研究. 作物学报, 2020, 46: 1351–1358.
Li J, Wang Y R, Zhang L X, Sun M H, Qin Y, Zheng J. Functional analysis ofgene from maize in response to salt stress., 2020, 46: 1351–1358 (in Chinese with English abstract).
[2] 刘奕媺, 于洋, 方军. 盐碱胁迫及植物耐盐碱分子机制研究. 土壤与作物, 2018, (7): 201–211.
Liu Y M, Yu Y, Fang J. Saline-alkali stress and molecular mechanism of saline-alkali tolerance in plants., 2018, (7): 201–211 (in Chinese with English abstract).
[3] 邵玺文, 冉成, 金峰, 郭丽颖, 耿艳秋. 松嫩平原苏打盐碱地水稻栽培技术研究进展与展望. 吉林农业大学学报, 2018, 40: 379–382.
Shao X W, Ran C, Jin F, Guo L Y, Geng Y Q. Advances and prospects in research of rice cultivation technology in saline- sodic soil of Songnen plain., 2018, 40: 379–382 (in Chinese with English abstract).
[4] Deokar A A, Kondawar V, Kohli D, Aslam M, Jain P K, Karuppayil S M, Varshney R K, Srinivasan R. The CarERF genes in chickpea (L.) and the identification of CarERF116 as abiotic stress responsive transcription factor., 2015, 15: 27–46.
[5] Bui L T, Giuntoli B, Kosmacz M, Parlanti S, Licausi F. Constitutively expressed ERF-VII transcription factors redundantly activate the core anaerobic response in., 2015, 236: 37–43.
[6] Wang P, Du Y, Zhao X, Miao Y, Song C P. The MPK6-ERF6-ROS-responsive-acting element7/GCC box complex modulates oxidative gene transcription and the oxidative response in., 2013, 161: 1392–1408.
[7] 陈悦, 孙明哲, 贾博为, 冷月, 孙晓丽. 水稻AP2/ERF转录因子参与逆境胁迫应答的分子机制研究进展. 作物学报, 2022, 48: 781–790.
Chen Y, Sun M Z, Jia B W, Leng Y, Sun X L. Research progress regarding the function and mechanism of rice AP2/ERF transcription factor in stress response., 2022, 48: 781–790 (in Chinese with English abstract).
[8] Yao W, Wang L, Zhou B, Wang S, Li R, Jiang T. Over-expression of poplar transcription factorgene confers salt tolerance in transgenic tobacco., 2016, 198: 23–31.
[9] Zhang G, Chen M, Chen X, Xu Z, Li L, Guo J, Ma Y. Isolation and characterization of a novel EAR-motif-containing genefrom soybean (L.)., 2010, 37: 809–818.
[10] Zhang Y, Ming R, Khan M, Wang Y, Dahro B, Xiao W, Li C, Liu J H. ERF9 of(L.) Raf. undergoes feedback regulation by ethylene and modulates cold tolerance via regulating agene., 2022, 20: 183–200.
[11] Khan M, Hu J, Dahro B, Ming R, Zhang Y, Wang Y, Alhag A, Li C, Liu J H. ERF108 from(L.) Raf. functions in cold tolerance by modulating raffinose synthesis through transcriptional regulation of., 2021, 108: 705–724.
[12] Wang M, Dai W, Du J, Ming R, Dahro B, Liu J H. ERF109 of trifoliate orange ((L.) Raf.) contributes to cold tolerance by directly regulating expression ofinvolved in antioxidative process., 2019, 17: 1316–1332.
[13] Zhang Z, Wang J, Zhang R, Huang R. The ethylene response factor AtERF98 enhances tolerance to salt through the transcriptional activation of ascorbic acid synthesis in., 2012, 71: 273–287.
[14] Serra T S, Figueiredo D D, Cordeiro A M, Almeida D M, Tiago L, Isabel A A, Alvaro S, Lisete F, Bruno C M, Oliveira M M, Nelson J M., a negative regulator of salt stress response in rice, is regulated by two AP2/ERF transcription factors., 2013, 82: 439–455.
[15] Liu D, Chen X, Liu J, Ye J, Guo Z. The rice ERF transcription factornegatively regulates resistance toand salt tolerance., 2012, 63: 3899–3911.
[16] 葛瑛, 朱延明, 吕德康, 董婷婷, 王维世, 谭上进, 刘彩虹, 邹平. 野生大豆碱胁迫反应的研究. 草业科学, 2009, 26(2): 47–52.
Ge Y, Zhu Y M, Lyu D K, Dong T T, Wang W J, Tan S J, Liu C H, Zou P. Research on responses of wild soybean to alkaline stress., 2009, 26(2): 47–52 (in Chinese with English abstract).
[17] Yu Y, Liu A L, Duan X B, Wang S T, Sun X L, Duanmu H Z, Zhu D, Chen C, Cao L, Xiao J L, Li Q, Nisa Z U, Zhu Y M, Ding X D., an ethylene-responsive factor from, mediates the regulation of plant bicarbonate tolerance in., 2016, 244: 681–698.
[18] Yu Y, Duan X B, Ding X D, Chen C, Zhu D, Yin K D, Cao L, Song X W, Zhu P H, Li Q, Nisa Z U, Yu J Y, Du J Y, Song Y, Li H Q, Liu B D, Zhu Y M. A novel AP2/ERF family transcription factor from, GsERF71, is a DNA binding protein that positively regulates alkaline stress tolerance in., 2017, 94: 509–530.
[19] Jensen J K, Hansen B G, Halkier B A, Norholm M H H, Nour E H H. Advancing uracil-excision based cloning towards an ideal technique for cloning PCR fragments., 2006, 34: e122.
[20] 李晶岚, 陈鑫欣, 石翠翠, 刘方惠, 孙静, 葛荣朝.基因过表达和RNA干涉对水稻苗期耐盐性的影响. 作物学报, 2020, 46: 1217–1224.
Li J L, Chen G X, Shi C C, Liu F H, Sun J, Ge R C. Effects ofgene overexpression and RNAi on the salt-tolerance at seedling stage in rice., 2020, 46: 1217–1224 (in Chinese with English abstract).
[21] 潘欣. 转、基因耐盐碱水稻的筛选与抗性分析. 东北农业大学硕士学位论文, 黑龙江哈尔滨, 2010.
Pan X. Analysis of Tolerance and Screening of Rice withandGene on Salt Stress and Alkali Stress. MS Thesis of Northeast Agricultural University, Harbin, Heilongjiang, China, 2010 (in Chinese with English abstract).
[22] 李合生. 植物生理生化实验原理和技术. 北京: 高等教育出版社, 2000. pp 167–169, 184–185.
Li H S. Principles and Techniques of Plant Physiological Biochemical Experimental. Beijing: Higher Education Press, 2000. pp 167–169, 184–185 (in Chinese).
[23] Kaur N, Sharma I, Kirat K, Pati P K. Detection of reactive oxygen species inL. (rice)., 2016, 24: e2061.
[24] 张宪政. 作物生理研究法. 北京: 中国农业出版社, 1992. pp 201–202.
Zhang X Z. Crop Physiology Research Method. Beijing: China Agriculture Press, 1992. pp 201–202 (in Chinese).
[25] Jung S E, Bang S W, Kim S H, Seo J S, Yoon H B, Kim Y S, Kim J K. Overexpression of, a vascular tissue-specific transcription factor gene, confers drought tolerance in rice., 2021, 22: 7656.
[26] 张霞, 唐维, 刘嘉, 刘永胜. 过量表达水稻和基因提高烟草脯氨酸的生物合成及其非生物胁迫抗性. 应用与环境生物学报, 2014, 20: 717–722.
Zhang X, Tang W, Liu J, Liu Y S. Co-expression of riceandgenes in transgenic tobacco resulted in elevated proline biosynthesis and enhanced abiotic stress tolerance., 2014, 20: 717–722 (in Chinese with English abstract).
[27] Kumar M, Choi J, An G, Kim S R. Ectopic expression ofenhances salt stress tolerance in rice., 2017, 8: 316.
[28] Cao Y F, Wu Y F, Zheng Z, Song F M. Overexpression of the rice EREBP-like geneenhances disease resistance and salt tolerance in transgenic tobacco., 2005, 67: 202–211.
[29] Zhuang J, Jiang H H, Wang F, Peng R H, Yao Q H, Xiong A S. A rice OsAP23, functioning as an AP2/ERF transcription factor, reduces salt tolerance in transgenic., 2013, 31: 1336–1345.
[30] Angelos E, Brandizzi F. NADPH oxidase activity is required for ER stress survival in plants., 2018, 96: 1106–1120.
[31] Sun X L, Sun M Z, Jia B W, Qin Z W, Yang K J, Chen C, Yu Q Y, Zhu Y M. Amethionine sulfoxide reductase B5a interacts with the Ca2+/CAM-binding kinase GsCBRLK and activates ROS signaling under carbonate alkaline stress., 2016, 86: 514–529.
[32] Zhang H M, Zhu J H, Gong Z Z, Zhu J K. Abiotic stress responses in plants., 2022, 23: 104–119.
[33] 张春霄. AP2/EREBP家族基因对提高豆科植物生物胁迫和非生物胁迫抗性的功能分析. 内蒙古大学硕士学位论文, 内蒙古呼和浩特, 2019.
Zhang C X. The Functional Analysis ofGene in AP2/EREBP Family with Improve Biological and Abiotic Stress Resistance about Leguminous Plant. MS Thesis of Inner Mongolia University, Hohhot, Inner Mongolia, China, 2019 (in Chinese with English abstract).
[34] Jin Y, Pan W, Zheng X, Cheng X, Liu M, Ma H, Ge X., an ERF family transcription factor, regulates drought stress response in reproductive tissues., 2018, 98: 51–65.
[35] Lu L, Qanmber G, Li J, Pu M, Chen G, Li S, Liu L, Qin W, Ma S, Wang Y, Chen Q, Liu Z. Identification and characterization of the ERF subfamily B3 group revealedimproves salt tolerance in upland cotton., 2021, 12: 705883.
Effects ofoverexpression on salt-alkaline tolerance in rice
CAI Xiao-Xi**, HU Bing-Shuang**, SHEN Yang, WANG Yan, CHEN Yue, SUN Ming-Zhe, JIA Bo-Wei, and SUN Xiao-Li*
Crop Stress Molecular Biology Laboratory, College of Agriculture, Heilongjiang Bayi Agriculture University, Daqing 163319, Heilongjiang, China
Ethylene response factors (ERFs) are a family of plant specific transcription factors that play important roles in abiotic stress. Bioinformatic analysis revealed that GsERF6, aERF transcription factor that positively regulated salt-alkaline tolerance, shared high sequence identity with homologous ERF proteins with one highly conserved AP2 domain in rice. To explore the effect ofoverexpression on the salt-alkaline tolerance in rice, we transformedinto rice and obtained two homozygous transgenic lines via PCR and RT-PCR. Phenotypical and physiological assays indicated that, compared with the wild type rice under 200 mmol L–1NaHCO3treatment, the survival rate, relative water content, the activities of SOD, POD, and CAT, the soluble sugar and proline contents, were significantly increased, while ROS accumulation was significantly decreased inoverexpression lines. The qRT-PCR showed that transcript levels ofandwere significantly up-regulated intransgenic line after 40 mmol L–1NaHCO3treatment for 6 hours. In summary,overexpression in rice contributed to ROS scavenging, osmotic regulation, and activation of stress responsive genes, thus improving the salt-alkaline tolerance of transgenic rice.
rice; salt-alkaline tolerance; ethylene response factors;;
10.3724/SP.J.1006.2023.22008
本研究由国家自然科学基金项目(32101672, 31971826, U20A2025)和中央支持地方高校改革发展资金人才培养支持计划项目(202201005)资助。
This study was supported by the National Natural Science Foundation of China (32101672, 31971826, U20A2025) and the Special Funds from the Central Finance to Support the Development of Local Universities (202201005).
孙晓丽, E-mail: sunxiaoli2016@byau.edu.cn
**同等贡献(Contributed equally to this work)
才晓溪, E-mail: 18746616279@163.com; 胡冰霜, E-mail: alisa961102@gmail.com
2022-01-28;
2022-06-07;
2022-06-21.
URL: https://kns.cnki.net/kcms/detail/11.1809.S.20220620.1740.004.html
This is an open access article under the CC BY-NC-ND license (http://creativecommons.org/licenses/by-nc-nd/4.0/).
