甘蔗PIN-LIKES基因家族的鉴定与表达分析
2023-02-10潘洁明田绍锐梁艳兰朱宇林周定港阙友雄
潘洁明 田绍锐 梁艳兰 朱宇林 周定港 阙友雄 凌 辉,* 黄 宁,*
甘蔗PIN-LIKES基因家族的鉴定与表达分析
潘洁明1田绍锐3梁艳兰2朱宇林1周定港4阙友雄2凌 辉1,*黄 宁1,*
1玉林师范学院农学院, 广西玉林 537000;2福建农林大学 / 农业农村部福建甘蔗生物学与遗传育种重点实验室, 福建福州 350002;3西南大学植物保护学院, 重庆 400700;4湖南科技大学生命科学学院/ 经济作物遗传改良与综合利用湖南省重点实验室, 湖南湘潭 411201
PILS (PIN-LIKES)是一类新发现的生长素输出载体, 协助生长素的极性运输。本研究立足甘蔗割手密基因组和栽培品种转录组数据, 利用生物信息学分析技术, 分别在割手密和栽培品种中鉴定到11个PILS (PIN-LIKES,)基因和4个PILS(spp. hybridPIN-LIKES,)基因。结果表明, 11个SsPILS基因家族成员分布于6条染色体上, 基因内含子数量介于5~11个。系统进化树分析发现, 割手密PILS与水稻PILS有较高同源性, 归属于3个不同的系统发育分支。转录组数据分析显示,的同源基因在受高粱花叶病毒和黑穗病菌胁迫甘蔗栽培种中差异表达。通过RT-PCR扩增技术, 克隆获得基因序列(NCBI accession number: OM258732)。该基因全长cDNA为1332 bp, 包含一个1233 bp的完整开放阅读框, 编码410个氨基酸, 理论等电点(pI)为6.17, 不稳定系数为39.31, 平均疏水性值为0.689, 预测ScPILS1c为稳定酸性疏水蛋白。其编码蛋白的二级结构主要包括α-螺旋(48.54%)和无规则卷曲(35.37%), 这与三级结构预测结果相符。qRT-PCR表达分析揭示,基因在甘蔗中的表达具有组织特异性, 在蔗髓中表达量最低、皮中的表达量最高, 同时该基因的表达受H2O2及黑穗病菌的显著性诱导。亚细胞定位表明,基因的编码蛋白主要定位于细胞膜。以上结果为深入研究甘蔗PIN-LIKES基因的结构和功能积累了基础资料。
甘蔗; PILS; 生物信息学; 实时荧光定量PCR; 基因家族
生长素(auxin)是最早被科学家达尔文(Darwin)父子发现的一种植物激素[1-2], 该激素参与调控植物生长发育[3-6]、植物逆境响应[7-9]等。研究表明生长素突出的特征是极性运输, 并通过极性运输产生浓度梯度, 实现在植物体内的差异分布[10-12]。生长素极性运输的调控包括输入载体(Influx carrier)和输出载体(Efflux carrier)[13]。PIN (PIN-FORMED)是典型的生长素输出载体, 在细胞膜上的极性分布, 是决定生长素极性运输方向的关键[14-18]。
在拟南芥()中, 存在8个PIN蛋白[19-21]和7个PILS (PIN-LIKES)蛋白[13]。PILS蛋白家族在空间结构上与PIN家族相似, 但序列相似度仅有10%~18%[22]。Barbez等[13]构建的系统进化树表明, 7个拟南芥PILS蛋白包含3个亚组: AtPILS1/AtPILS3/AtPILS4、AtPILS5/AtPILS7和AtPILS2/ AtPILS6。水稻中包括7个PILS蛋白, 其中OsPILS7a/ OsPILS7b/OsPILS1与AtPILS1/AtPILS3/AtPILS4为一个亚组, OsPILS5与AtPILS5/AtPILS7为一个亚组, OsPILS2/OsPILS6a/OsPILS6b 与 AtPILS2/AtPILS6为一个亚组[23]。前人研究显示, PILS具有生长素载体结构域Mem_trans (PF03547)[24], 参与调节细胞内生长素的稳态, 并能引起生长素信号转导速率的变化, 进而调控细胞的差异生长[25]。Feraru等[25]和Beziat等[26]研究发现, PILS2、PILS3、PILS5和PILS6蛋白可以调控细胞内生长素的结合速率, 进而影响生长素核信号的传导。Barbez等[13]和Feraru等[22]报道, 在烟草细胞中诱导PILS2的表达能够增加生长素的积累, 而PILS2、PILS5双突变的原生质体则呈现明显的生长素输出量[13,22]。
甘蔗在全球多地区广泛种植, 是我国最重要的糖料作物和生物能源作物[27-31]。据报道, 我国约90%的食糖来自甘蔗[32-33]。甘蔗可作为新能源的原料应用前景广阔[34-36]。Lalman等[37]报道全球40%的燃料乙醇来源于甘蔗。甘蔗是保证全球食糖安全和新能源材料的重要作物, 对甘蔗基因的深入研究可以更好地了解生长素在甘蔗生长发育及逆境响应过程中的调控机制, 有助于满足甘蔗的种植需求。迄今, 在甘蔗中尚未见关于PILS基因的相关报道。本研究立足甘蔗割手密()基因组数据[38]和不同胁迫下甘蔗栽培品种()转录组数据, 基于已报道的拟南芥PILS基因家族的序列及结构特征, 对甘蔗割手密及栽培品种PILS基因家族进行鉴定, 克隆和生物信息学分析, 检测甘蔗栽培品种PILS基因家族成员在不同生物胁迫下的表达模式, 以期为深入探讨甘蔗PIN-LIKES基因家族成员的结构和功能积累基础资料, 并为甘蔗抗逆分子育种提供潜在的基因资源。
1 材料与方法
1.1 甘蔗割手密PILS基因家族鉴定及分析
以拟南芥PILS基因家族序列为原始序列, 在甘蔗割手密基因组的蛋白序列中进行blast比对, 选择相似性大于65%的序列通过Pfam (http://pfam.xfam. org/)进行结构域分析, 筛选其中具有Mem_trans结构域的序列为甘蔗割手密PILS基因家族成员。根据甘蔗割手密基因组注释信息, 通过TBtools软件[39]中的Quick run MCScanX wrapper工具对SsPILS基因家族成员的染色体分布及基因家族成员间的共线性进行分析。
1.2 甘蔗栽培品种PILS基因家族的鉴定及在生物胁迫下的表达分析
以拟南芥和甘蔗割手密PILS基因家族成员的氨基酸序列为原始序列, 分别使用blastp和Pfam工具, 在受高粱花叶病毒[40]和黑穗病菌胁迫下的甘蔗栽培品种转录组数据库[41]中进行同源比对和结构域分析,筛选其中具有Mem_trans结构域的序列为甘蔗栽培种PILS基因家族成员, 并对筛选到的序列进行表达模式分析。通过DNAMAN对甘蔗割手密及甘蔗栽培品种PILS基因家族成员进行多序列比对。
1.3 生物信息学分析
1.3.1 甘蔗割手密PILS基因家族的生物信息学分析 使用ProtParam (https://web.expasy.org/protparam/)对SsPILS基因家族成员进行氨基酸个数、分子量、等电点和不稳定系数分析。使用CELLO v.2.5 (http:// cello.life.nctu.edu.tw/)对SsPILS基因家族成员进行亚细胞定位预测。使用MEGA7.0对甘蔗割手密、拟南芥[13]、水稻 ()[23]中的PILS的氨基酸序列进行比对(MAFFT), 并通过PhyloSuite v1.2.1[42]进行系统进化树构建, 建树模型为LG+G4, 步长为1000。根据SsPILS氨基酸序列信息, 分别通过MEME (http://meme-suite.org/tools/meme)和SEA (https://meme-suite.org/meme/tools/sea), 进行保守基序查找及功能分析。基于甘蔗割手密基因组序列及注释信息, 通过TBtools[39]中的Gene Structure View对SsPILS的基因结构进行分析和展示。
1.3.2基因的生物信息学分析 利用ORF finder (http://www.ncbi.nlm.nih.gov/gorf/gorf.html)在线工具对的开放阅读框(open reading frame, ORF)和蛋白保守结构域进行预测; 分别使用在线工具SMART (http://smart.embl-heidelberg.de/ smart/set_mode.cgi?NORMAL=1)、TMHMM (https:// services.healthtech.dtu.dk/service.php?TMHMM-2.0)、ProtParam (https://web.expasy.org/protparam/)、PRABI- GERLAND (https://npsa-prabi.ibcp.fr/cgi-bin/npsa_ automat.pl?page=/NPSA/npsa_hnn.html)、SWISS- MODEL (https://swissmodel.expasy.org/)和ProtScale (https://web.expasy.org/protscale/)对编码蛋白质的保守结构域、跨膜结构、一级、二级、三级结构和疏水性/亲水性进行预测。
1.4 ScPILS1c基因的克隆与表达分析
1.4.1 植物材料及处理 本研究中基因克隆及定量分析所用的植物材料均为甘蔗栽培品种ROC22。参考Ling等[43]的报道, 选择5株长势一致健康的ROC22植株进行不同组织的取样, 取样组织分别为皮、叶片、芽和蔗髓。同时选择ROC22的健康植株, 用水将其冲洗后砍成单芽茎段。分别对其进行以下处理: (1) 将单芽茎段种植在经过灭菌处理的基质中待甘蔗苗长出4~6片叶子时, 选取长势相近的甘蔗苗用于组培苗的培养。组培苗生根后, 将其移出温室并水培7 d, 挑选长势一致的组培苗分别在其叶片喷施无菌水及500 mmol L-1过氧化氢(H2O2), 每个处理设置3个生物学重复, 6 h取样。(2) 参考Que等[41]报道的甘蔗接种黑穗病菌的方法, 对单芽茎段进行水培, 待蔗芽长至1.0~2.0 cm时, 分别接种黑穗病菌和无菌水, 每个处理设置3个生物学重复, 7 d取样。用锡箔纸包裹取样样品, 放入液氮处理, 然后在-80℃条件下保存。
1.4.2 总RNA提取和cDNA合成 采用TRIzol法[44]提取甘蔗总RNA后, 使用DNaseI对RNA样品进行处理, 同时使用PrimeScript RT-PCR Kit (Perfect for Real Time)合成cDNA。
1.4.3基因的克隆 根据同时在受高粱花叶病毒[40]和黑穗病菌胁迫下的[41]甘蔗栽培品种转录组中均差异表达的基因序列, 设计克隆引物PILS1c-F/R (表1), 参考黄宁等[44]报道的方法进行PCR扩增, PCR产物经纯化后连接至pMD19-T载体, 并转化至大肠杆菌DH5α后送生工生物工程(上海)股份有限公司测序。

表1 本研究所用引物
1.4.4基因的组织特异性表达分析及其受H2O2和黑穗病菌胁迫后的表达模式 采用qRT-PCR技术, 以q-PILS1c-F/R (表1)为引物, 检测基因在甘蔗不同组织中的表达情况, 并检测基因在甘蔗受过氧化氢(H2O2)胁迫和黑穗病菌胁迫后的表达情况。本试验采用内参基因和组合对基因表达量数据进行校正[45],和引物见表1。PCR反应体系参考黄宁等[44]报道的方法。每种样品进行3次技术重复。利用在ABI PRISM7500 real-time PCR system软件对获得的数据进行初步分析后, 导出数据文件, 按照2-ΔΔCt算法进行结果分析[46]。
1.4.5基因编码蛋白的亚细胞定位分析 以pMD19-T-ScPILS1c质粒DNA为模板, 借助亚细胞定位引物YFP-PILS1c-F/R (表1)进行PCR扩增。利用同源重组试剂盒ClonExpress II One Step Cloning Kit (诺唯赞生物科技有限公司, 中国南京), 将基因ORF (不含终止密码子)连接到亚细胞定位载体pCAMBIA2300-YFP上, 获得重组质粒35S::ScPILS1c::YFP。将重组阳性质粒35S::ScPILS1c:: YFP和空载质粒35S::YFP分别转化根癌农杆菌GV3101, 液体培养(含50 μg mL-1卡那霉素和50 μg mL-1利福平) 28℃ 下250转 min-1振荡培养48 h, 调节菌液浓度(含200 μmol L-1乙酰丁香酮)至0.8 (OD600), 分别将含有35S::ScPILS1c::YFP和35S:: YFP质粒的农杆菌注射侵染烟草叶片, 48 h后, 于激光共聚焦显微镜(Leica TCS SP8, 德国)下观察亚细胞定位情况[44]。
2 结果与分析
2.1 甘蔗割手密PILS基因家族的鉴定及共线性分析
在甘蔗割手密基因组中, 共鉴定获得11个与拟南芥PILS基因家族成员相似性大于65%且具有Mem_trans结构域的序列。结果显示, 11个SsPILS共定位到6条染色体上, 分别为染色体1C、1D、2A、2B、2C、3C (图1-A)。基因组内共线性分析结果显示, SsPILS基因家族成员间存在2个共线性基因对, 其中和互为复制基因、和互为复制基因(图1-B)。
2.2 甘蔗割手密PILS基因家族生物信息学分析
2.2.1 理化性质分析和亚细胞定位预测 本研究对获得的11个甘蔗割手密PILS基因推导的编码蛋白的理化性质进行分析(表2)发现, 除SsPILS1a氨基酸数目较大为847外, SsPILS1b~SsPILS7b的氨基酸数目差别不大, 介于301~436之间, 相对分子量为32.59~89.11 kD。除SsPILS7b蛋白等电点为6.12外, 其余10个SsPILS、蛋白等电点均大于7。Plant-mPloc软件预测结果显示SsPILS蛋白均定位于质膜。
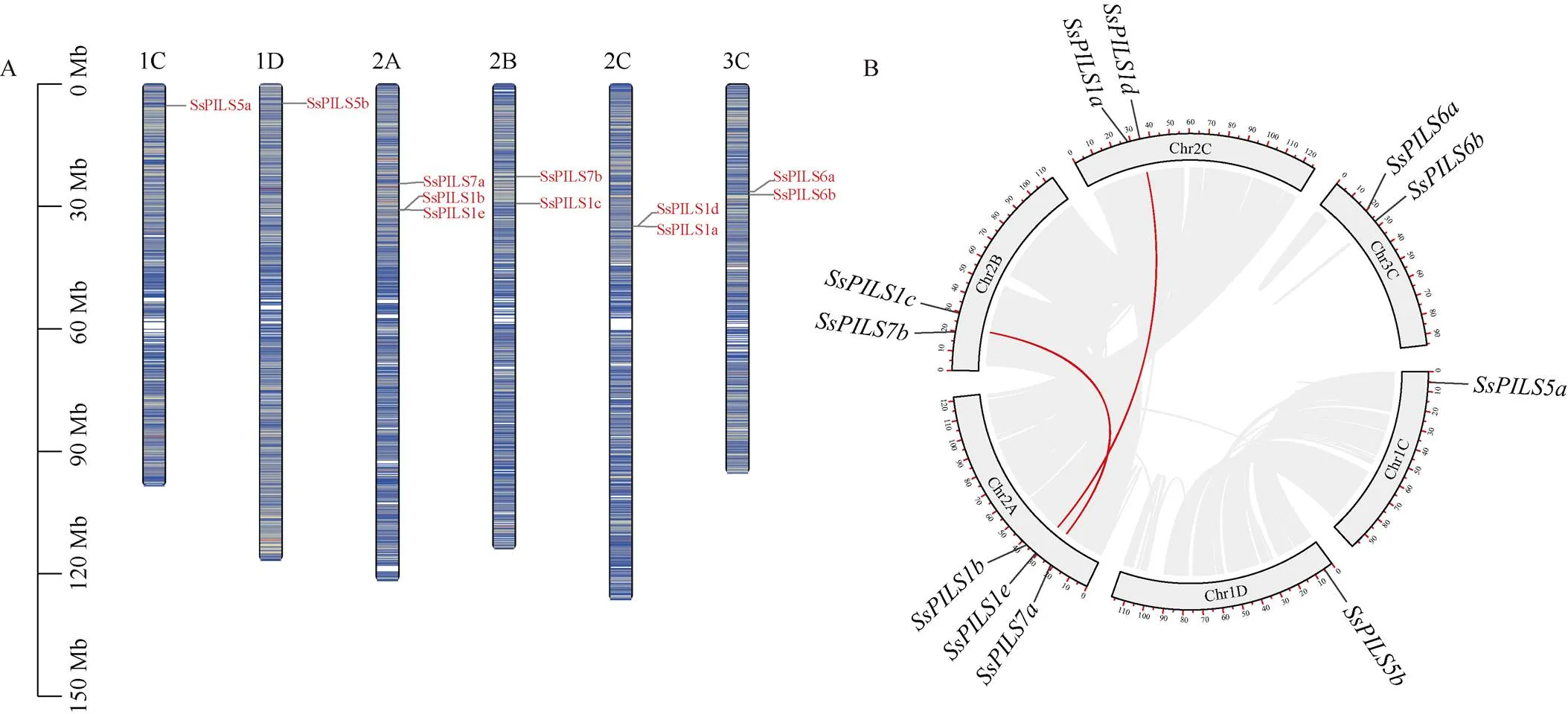
图1 甘蔗割手密PILS基因家族的染色体分布(A)及共线性分析(B)
红色连线表示甘蔗割手密PILS基因家族间的共线性, 灰色连线表示割手密所有基因间的共线性。
The red line indicates synteny within PILS and the grey line indicates synteny within all genes ingenome.
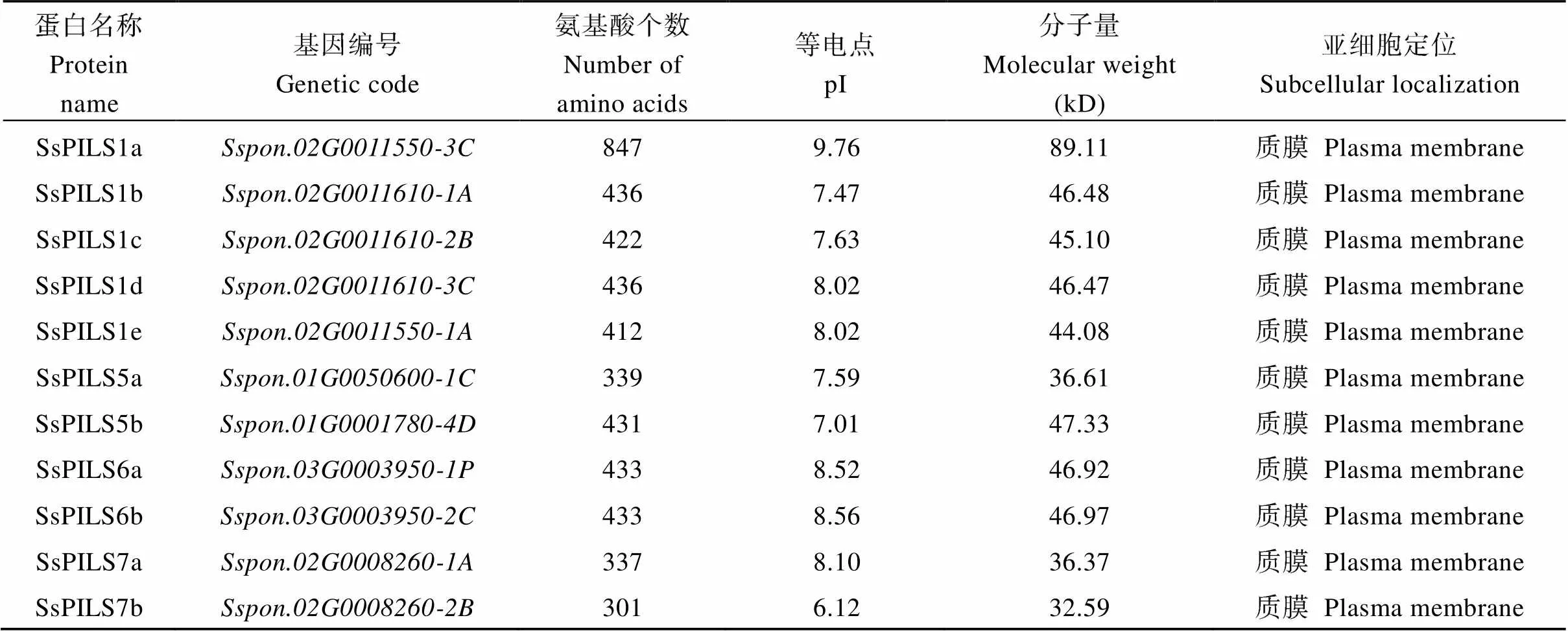
表2 甘蔗割手密PILS蛋白的理化性质分析及亚细胞定位预测
2.2.2 系统进化分析 甘蔗割手密、水稻和拟南芥中的PILS聚类成3个系统发育分支, 其中, SsPILS1a~SsPILS1e分布于分支I, SsPILS5a、SsPILS5b、SsPILS7a和SsPILS7b分布于分支II, SsPILS6a和SsPILS6b分布于分支III。并且甘蔗割手密PILS与水稻OsPILS的进化距离相较于拟南芥AtPILS更近(图2)。
2.2.3 保守基序及基因结构分析 通过MEME工具在SsPILSs中进行保守基序查找发现, SsPILSs包含3~5个保守基序, 且N端均具有保守基序motif 3 (图3-A), 说明SsPILS基因家族基序具有一定保守性。经SEA在motif数据库PROSITE fixed-length motifs (PROSITE 2021_04)中比对发现, 除motif 2和motif 5在数据库中未找到比对结果外, motif 1、motif 3和motif 4分别为N-肉豆蔻酰化位点、酰胺化位点和N-糖基化位点。基因结构分析表明,包含5至11个内含子, 其中具有5个内含子,、具有11个内含子,、具有8个内含子。、、具有9个内含子,、、具有10个内含子。除、、、和具有UTR外, 其他均无UTR (图3-B)。
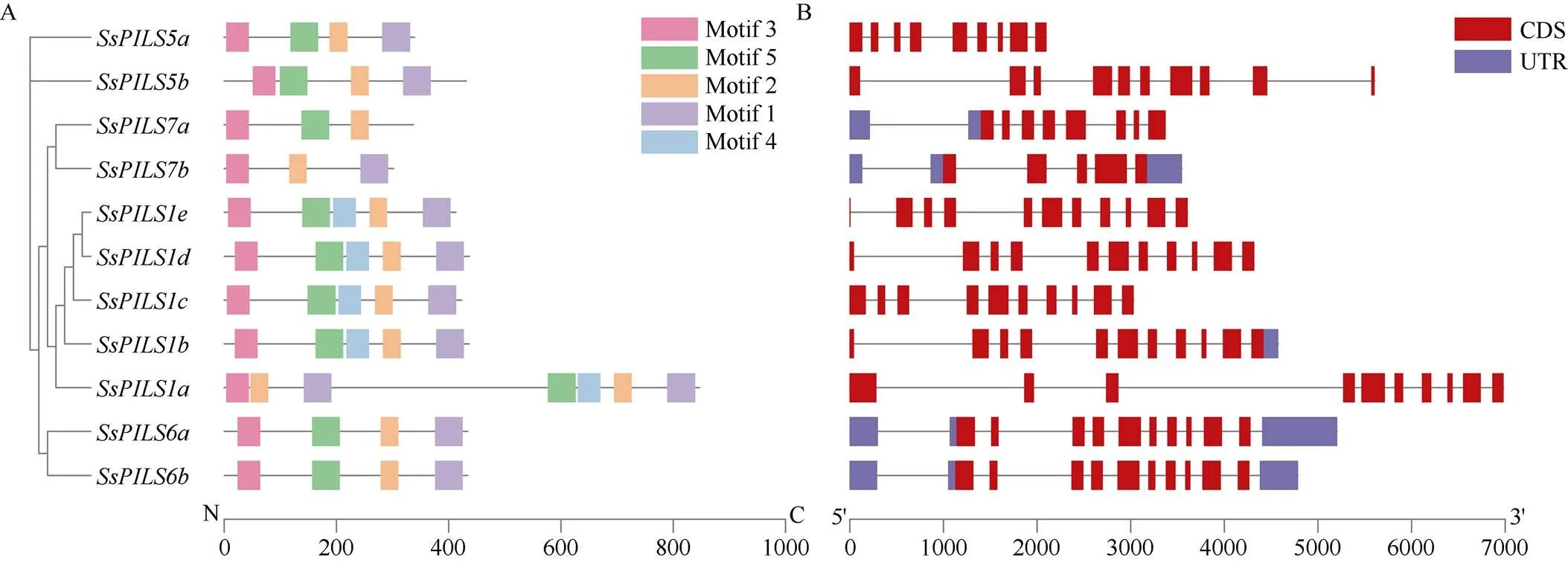
图3 甘蔗割手密PILS基因家族蛋白保守基序(A)和基因结构分析(B)
A图中, 不同颜色方框表示不同保守基序, 黑线表示氨基酸序列。B图中, 不同颜色方框表示不同基因结构, 黑线表示内含子。
Different colored boxes indicate different conserved motifs, and black lines indicate amino acid sequences in Fig. 3-A. Different colored boxes indicate different gene structures, and black lines indicate introns in Fig. 3-B.
2.3 甘蔗栽培品种PILS基因家族的鉴定及在生物胁迫下的表达分析
在受高粱花叶病毒和黑穗病菌胁迫下的甘蔗栽培品种转录组数据中, 一共筛选获得4条基因, 经多序列比对后发现, 4个基因序列之间的同源性大于95%, 因此将它们分别命名为、、和。利用转录组数据对筛选到的甘蔗栽培种PILS基因家族成员进行表达模式分析发现, 与对照相比, 受高粱花叶病毒胁迫后在甘蔗栽培品种中的表达量变化不大(图4-A), 但在黑穗病菌胁迫下的甘蔗栽培品种中没有检测到表达(图4-B)。此外, 受高粱花叶病毒胁迫后, 除被诱导表达、被抑制表达外, 其他基因表达量变化不大(图4-A)。受黑穗病菌胁迫后的24 h、48 h和120 h 3个时间点, 易感黑穗病品种ROC22和抗黑穗病品种YC71-374中,和基因的表达量和表达模式相差不大。同时, 在ROC22中,在受黑穗病菌胁迫后的24 h、48 h和120 h, 表达量基本不变, 但在YC05- 179受侵染120 h时, 该基因被诱导表达(图4-B)。综上所述,在受高粱花叶病毒和黑穗病菌胁迫后的甘蔗中均差异表达, 且均被诱导表达。
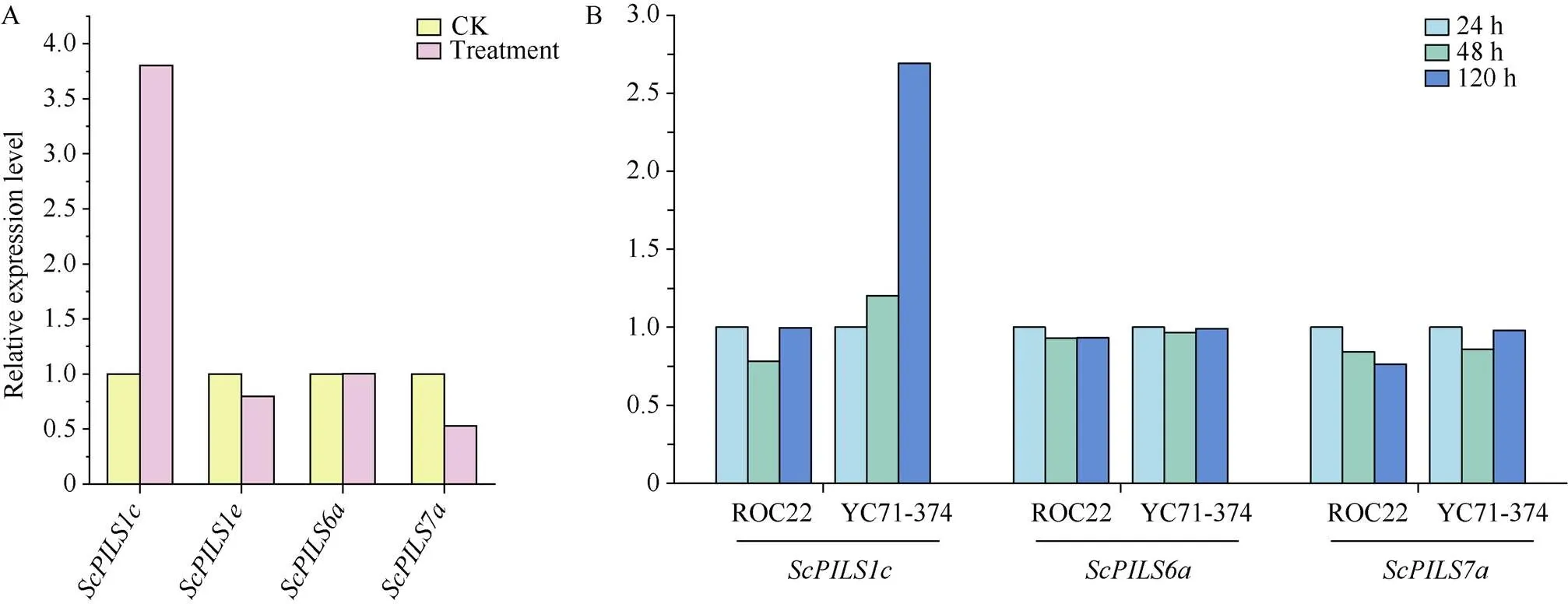
图4 ScPILS基因在不同胁迫下的表达
A: 高粱花叶病毒胁迫; B: 黑穗病菌胁迫。
A:infection; B:infection.
2.4 ScPILS1c基因的克隆与序列分析
本研究克隆获得的基因与基因的相似性为96.45%, 其cDNA全长为1332 bp, 应用ORF Finder对其序列进行分析, 结果显示其包含一条1233 bp的完整开放阅读框, 编码410个氨基酸(图5-A)。SMART和TMHMM分析结果显示, ScPILS1c蛋白具有Mem_trans保守结构域(图5-B), 并包含10个跨膜结构(图5-C)。应用在线工具ProtParam对编码的蛋白的一级结构预测结果显示ScPILS1c分子量为43.86 kD, 分子式为C2011H3213N497O558S18。根据预测结果计算得出的不稳定性指数(II) 为39.31, 等电点为6.17。由此推测ScPILS1c蛋白质为一个酸性稳定的蛋白质。蛋白二级结构预测显示, ScPILS1c主要由α螺旋(48.51%)、无规则卷曲(35.37%)和延伸链(16.10%) 三部分组成。三级结构预测表明, ScPILS1c的建模模板为5aymA, 其与模板叠合后, 覆盖度为0.94, RMSD值为1.36, 建模结果可信度高(图5-D)。三维结构结果显示, ScPILS1c蛋白以α螺旋为主, 其次是无规则卷曲与二级结构预测结果相同(图5-E)。
应用ProtScale在线工具对PILS进行疏水性/亲水性预测(图6)发现, 在基因的编码氨基酸序列中, 大部分残基为疏水性, 第257位具有最高分值(3300), 疏水性最强; 第226位具有最低分值(-2589), 亲水性最强。根据蛋白的平均疏水性(grand average of hydropathicity, GRAVY)值进行预测, 该蛋白的GRAVY值为0.689, 为疏水性蛋白。
2.5 甘蔗栽培品种PILS1c基因的表达分析和亚细胞定位
2.5.1基因的组织特异性表达分析 组织特异性的qRT-PCR分析显示, 甘蔗基因在皮、叶片、芽和蔗髓中组成型表达, 且在芽中表达量最低, 其次为蔗髓, 在叶片和皮中表达量较高(图7)。
2.5.2基因受H2O2和黑穗病菌胁迫后的表达模式分析 qRT-PCR表达分析中, 受H2O2胁迫后,基因表达量相对于对照组上调表达, 且显著高于对照组(图8-A); 受黑穗病菌胁迫7 d后,基因的表达量相对于对照组上调表达, 并且在受黑穗病菌胁迫后显著高于对照组(图8-B)。据此推测,基因受H2O2及黑穗病菌显著诱导表达。
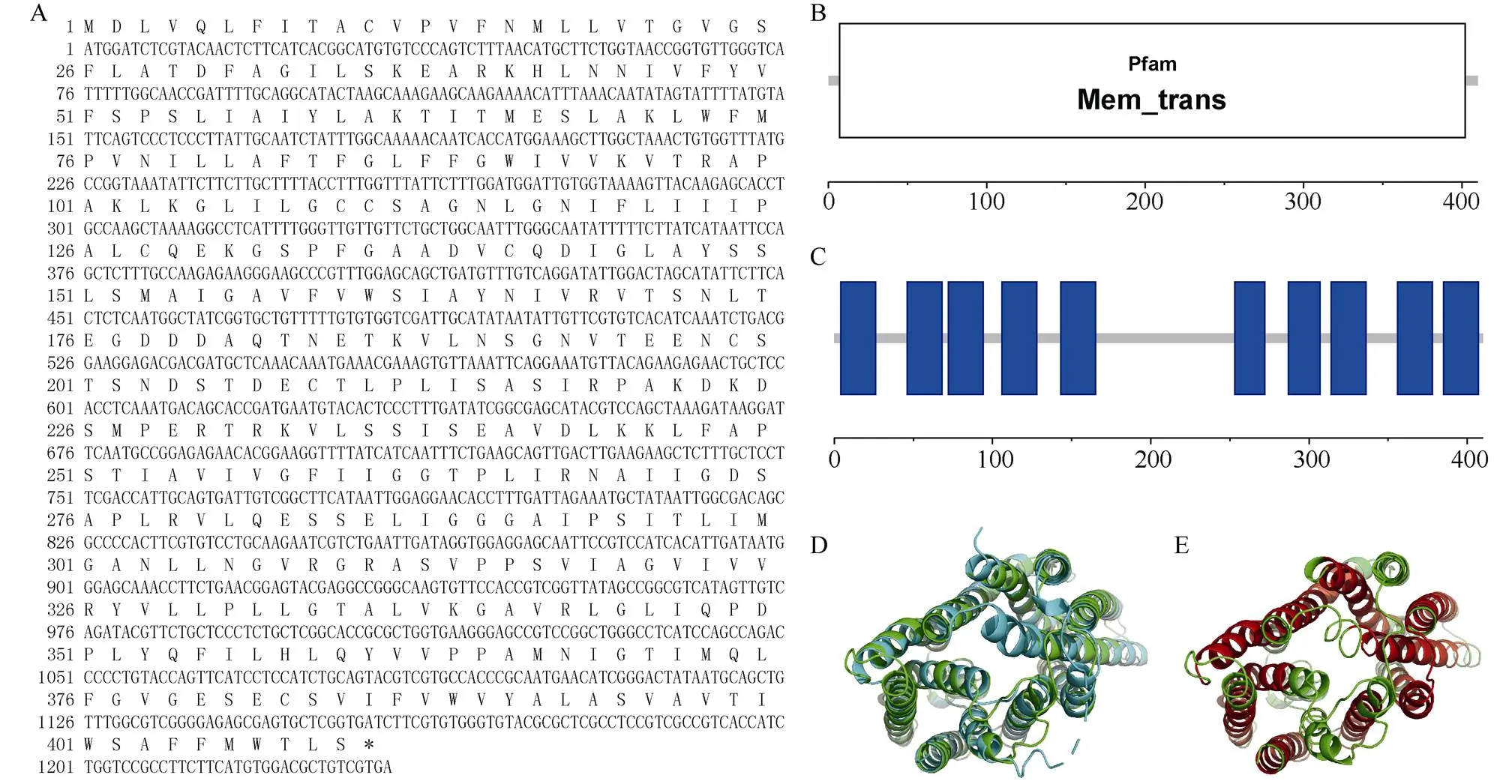
图5 ScPILS1c蛋白生物信息学分析
A:基因的开放读码框及其所编码蛋白; B: ScPILS1c蛋白的保守结构域; C: ScPILS1c蛋白的跨膜结构; D: ScPILS1c三级结构模型与5aymA比对; E: ScPILS1c三级结构模型, 红色为跨膜结构域。
A: the open reading frame ofgene and its encoded protein; B: the conserved domain of ScPILS1c protein; C: the transmembrane helices of ScPILS1c protein; D: the comparison of third structure model of ScPILS1c with 5aymA; E: the third structure model of ScPILS1c, and red color indicates transmembrane domain.
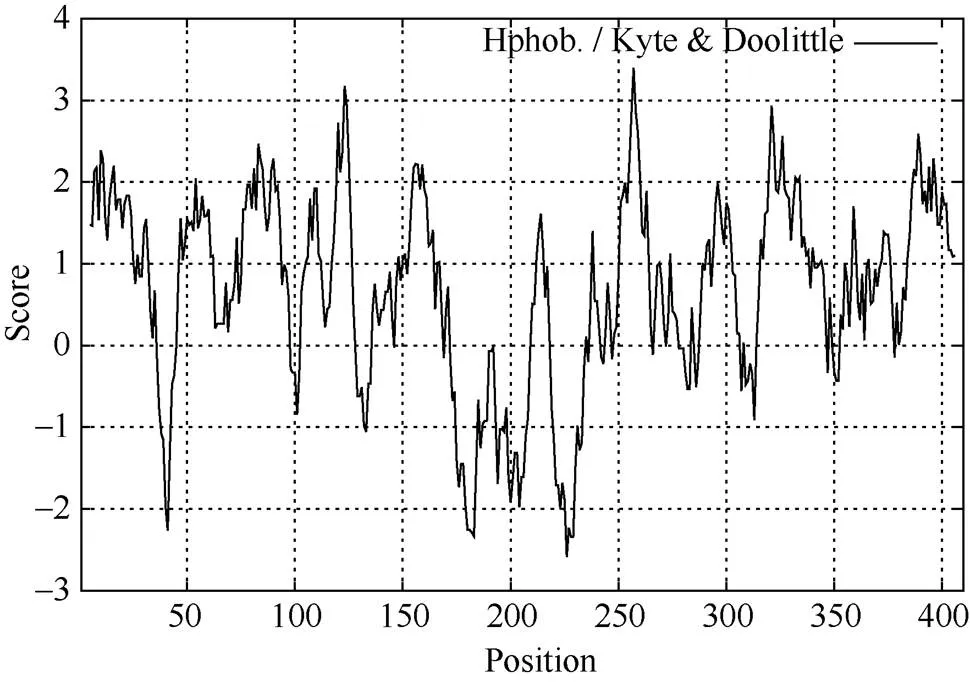
图6 ScPILS1c蛋白氨基酸疏水性/亲水性预测
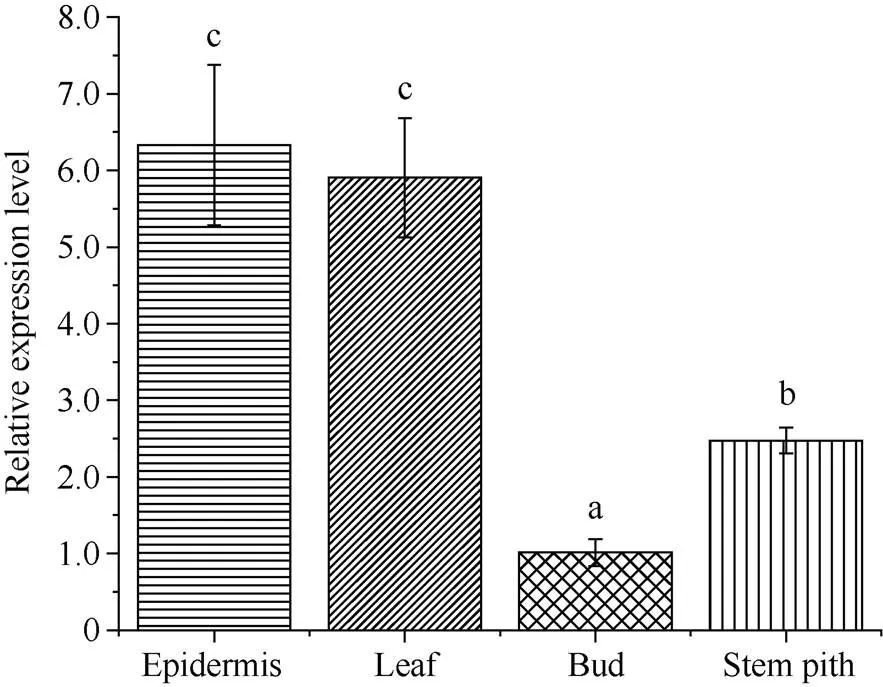
图7 ScPILS1c基因在不同组织中的表达
柱上不同小写字母表示在0.05水平差异显著。
Different lowercase letters above the bars indicate significant difference at the 0.05 probability level.
2.5.3基因编码蛋白的亚细胞定位分析 通过农杆菌介导将35S:ScPILS1c:YFP导入本氏烟叶片下表皮内细胞内, 进行瞬时表达分析, 并借助激光共聚焦显微镜观察融合蛋白的亚细胞定位情况。由图9可知, 在注射空载35S: YFP的本氏烟叶片中, YFP黄色荧光比较均匀的分布在细胞边缘, 细胞内也有明亮的荧光点。而35S:ScPILS1c:YFP的本氏烟叶片, 在细胞膜上有不连续的黄色荧光点, 细胞内有空泡状的荧光点, 细胞质中有网格状荧光, 说明定位可能和细胞膜系统相关。该结果与亚细胞定位预测的结果一致。
3 讨论
生长素是植物体内的重要激素, 受到研究者的高度关注, 生长素的极性运输参与调控植物的形态建成、向性反应、导管结构和器官结构等多个方面[47-48]。与此同时, 生长素在植物逆境响应中的作用也有不少报道[7-9]。生长素输出载体蛋白PIN家族和PILS家族[2, 49]参与和调控植物发育的各个阶段, 是决定生长素极性运输方向的关键。越来越多的研究表明,基因能够通过调节生长素的稳态, 影响生长素信号传导[22], 但是其研究多集中于植物生长发育, 响应生物及非生物胁迫的报道较少。如: BR(brassinosteroids)信号通过影响的转录以及翻译, 使基因的表达受到抑制[50], PILS蛋白的数量减少, 从而诱导生长素信号传导[25]。、和共同控制内侧生长素信号的消耗, 从而诱导顶端弯钩的伸展[51]。植物色素B依赖光信号通路直接调节基因的活性, 抑制核生长素信号传导并控制植物生长[26]。以上研究结果表明,基因可以通过调节生长素的消耗、积累和极性运输调控植物器官的形成和发育, 在植物生长发育中具有关键的作用[23]。
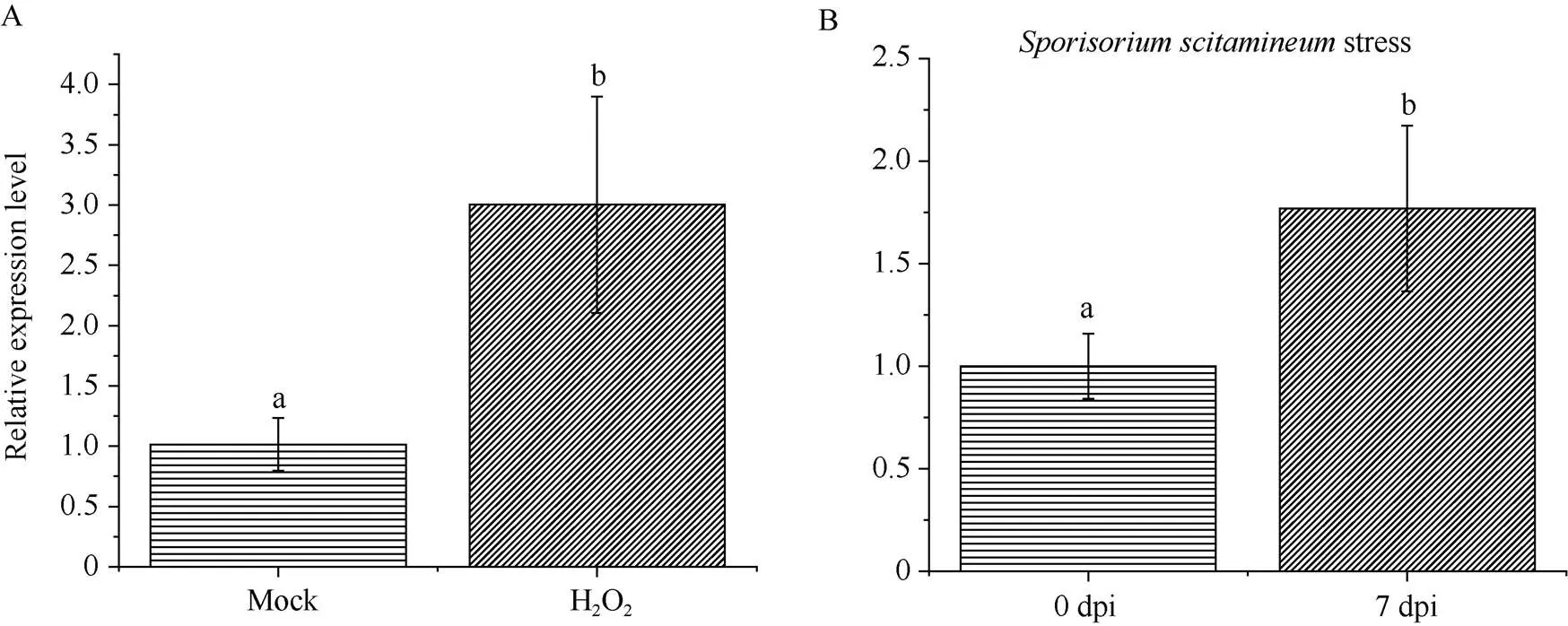
图8 ScPILS1c基因在不同胁迫下的表达
A: H2O2胁迫; B: 黑穗病菌胁迫。柱上不同小写字母表示在0.05水平差异显著。
A: H2O2stress; B:stress. Different lowercase letters above the bars indicate significant difference at the 0.05 probability level.
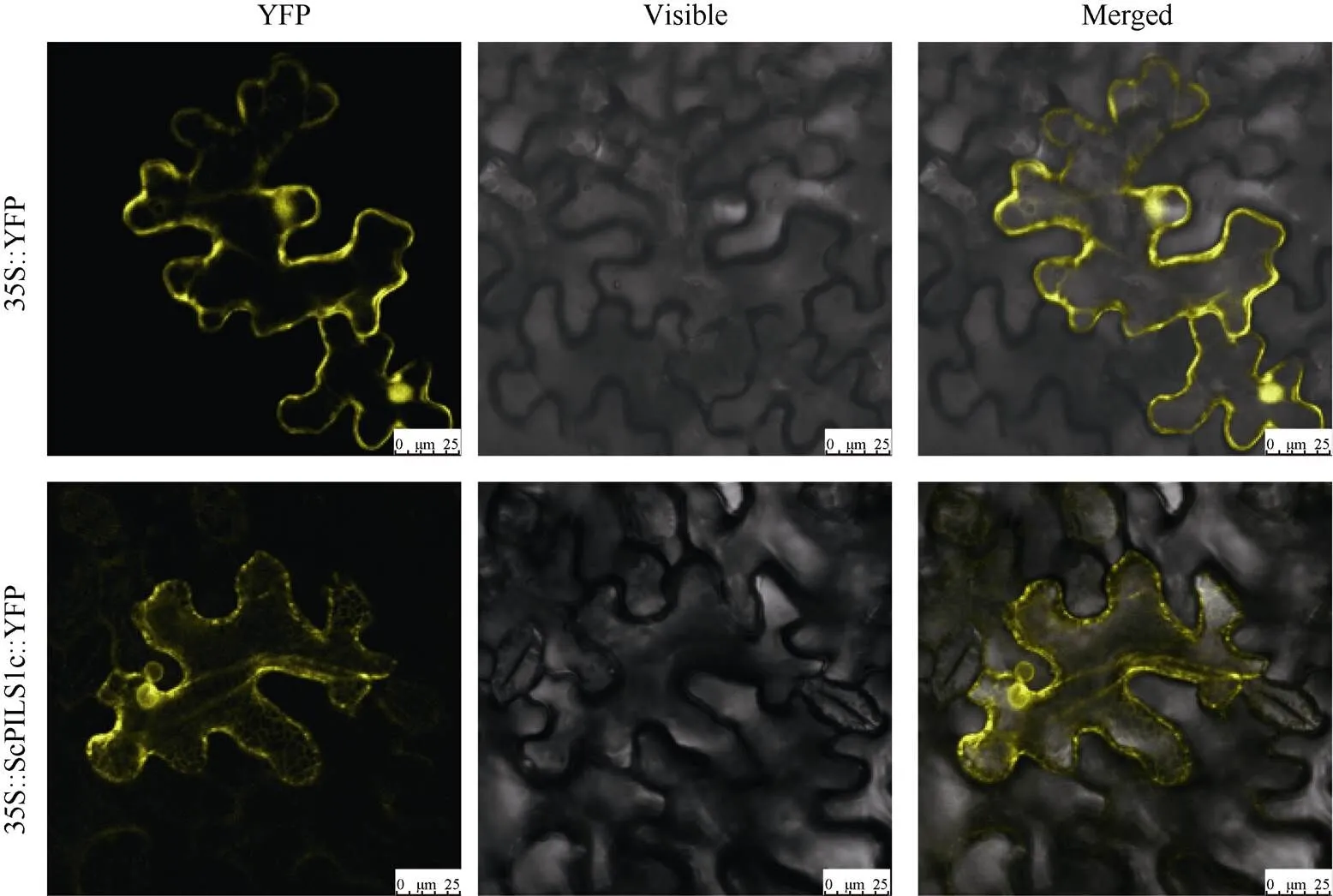
图9 甘蔗ScPILS1c在本氏烟下表皮细胞中的亚细胞定位
目前, PILS的研究主要集中在拟南芥和水稻中[23,52],在甘蔗中未见PILS研究的相关报道。本研究在甘蔗割手密基因组中共鉴定到11个基因, 对比前人研究发现, 不同作物中基因所编码氨基酸的理化性质相差不大, 比如本研究中甘蔗割手密PILS氨基酸长度除SsPILS1a氨基酸数目为847外, 其他多数在301~436之间, 内含子在5~11个之间; 而大豆的19个PILS氨基酸长度在259~445之间, 内含子在1~11个之间[53]; 水稻的7个PILS氨基酸长度在414~1280之间, 内含子在1~10个之间[23]。我们推测,基因所编码蛋白质在植物中保守性高。根据前人研究发现, 拟南芥的AtPILS蛋白、大豆的GmPILS蛋白均定位在内质网上[53], 而本研究中的ScPILS1c蛋白定位于细胞膜及细胞内膜系统这与前人的研究结果不同, 推测ScPILS1c与拟南芥中AtPILS蛋白和大豆中GmPILS蛋白具有不同的细胞定位, 可能行使不同的分子功能, 参与不同的生物学进程。甘蔗基因属于PILS基因家族第I亚组, 在各组织中均表达, 但在蔗髓中表达量最低, 皮中的表达量最高。而拟南芥主要在茎中表达, 在叶和花部的表达量很低[22]。由此可见, 不同植物中基因的组织特异表达模式是不同的。
前人研究表明, 生长素信号传导通路相关基因[54]和[55]能够积极响应生物胁迫。过表达柑橘转基因植株中游离生长素含量减少, 受柑橘黄单胞菌侵染后, 游离生长素含量减少速度加快, 对柑橘黄单胞抗性增强[54]。木薯基因沉默植株对细菌性萎蔫病菌更加感病[55]。基因作为生长素信号转导通路相关基因, 也可能参与植物对生物胁迫的响应过程。本研究通过qRT- PCR技术分析基因受黑穗病菌胁迫后的表达情况发现, 受黑穗病菌胁迫后,基因显著上调表达, 结合[54]和[55]的相关报道可以推测作为生长素输出载体可能通过调节生长素的运输, 使甘蔗体内或某些组织中生长素含量减少, 以此增强甘蔗对生物胁迫的抗性。植物在受到各种胁迫后, 体内H2O2含量增加, 并作为信号分子参与植物抗逆反应, 外源施加H2O2可模拟受胁迫后的植物内环境, 这被广泛用于植物基因功能抗逆方面的研究[44,56-60]。烟草基因[56]、甘蔗栽培种基因[44]、玉米基因[57]、小麦基因[58]、棉花和基因[59]、甘薯基因[60]在H2O2胁迫下显著诱导表达; 甘蓝型油菜基因[61]、甘蔗基因[62]、小麦基因[63]受H2O2胁迫后下调表达。在本研究中, 受H2O2胁迫后,基因显著上调表达。综上所述,基因的表达受H2O2、高粱花叶病毒以及黑穗病菌胁迫等逆境条件的调控, 推测该基因通过调控生长素信号传导通路在甘蔗抗逆境方面发挥一定的作用。
4 结论
本研究在甘蔗割手密和栽培品种中分别鉴定获得11个和4个基因。11个甘蔗割手密基因, 分布于6条染色体, 各自含有5~11个内含子。系统进化树分析发现, 相对于拟南芥, 割手密与水稻亲缘关系更近, 归属于3个不同系统发育分支。同时,的同源基因基因在高粱花叶病毒及甘蔗黑穗病菌胁迫下的转录组中均差异表达。从甘蔗栽培品种中克隆获得了基因的cDNA序列, 其全长为1332 bp, 包含一个1233 bp的完整开放阅读框, 编码410个氨基酸, 为稳定酸性疏水蛋白。二级结构主要包括α-螺旋和无规则卷曲。qRT-PCR表达分析揭示,基因的表达具有组织特异性, 且在蔗髓中表达量最低, 皮中的表达量最高。此外, 该基因的表达受H2O2及黑穗病菌的显著诱导。亚细胞定位分析表明,基因主要定位于细胞膜。以上结果可为甘蔗基因在逆境胁迫响应机制中的功能和作用机制解析提供参考。
[1] Gao Y B, Zhang Y, Zhang D, Dai X H, Estelle M, Zhao Y D. Auxin binding protein 1 (ABP1) is not required for either auxin signaling ordevelopment., 2015, 112: 2275–2280.
[2] Enders T A, Strader L C. Auxin activity: past, present, and future., 2015, 102: 180–196.
[3] Chandler J W. Auxin response factors., 2016, 39: 1014–1028.
[4] Miao Z Q, Zhao P X, Mao J L, Yu L H, Yang Y, Hui T, Liu Z B, Xiang C B. HOMEOBOX PROTEIN52 mediates the crosstalk between ethylene and auxin signaling during primary root elongation by modulating auxin transport-related gene expression., 2018, 30: 2761–2778.
[5] Ishida J K, Wakatake T, Yoshida S, Takebayashi Y, Kasahara H, Wafula E, Depamphilis C W, Namba S, Shirasu K. Local auxin biosynthesis mediated by a YUCCA flavin monooxygenase regulates haustorium development in the parasitic plant., 2016, 28: 1795–1814.
[6] Di Mambro R, De Ruvo M, Pacifici E, Salvi E, Sozzani R, Benfey P N, Busch W, Novak O, Ljung K, Di Paola L, Mare A F, Costantino P, Grieneisen V A, Sabatini S. Auxin minimum triggers the developmental switch from cell division to cell differentiation in theroot., 2017, 114: E7641–E7649.
[7] 颜爽爽, 邱正坤, 余炳伟, 明方艳, 陈长明, 雷建军, 曹必好. 植物生长素响应高温胁迫研究进展. 园艺学报, 2020, 47: 2238–2246.
Yan S S, Qiu Z K, Yu B W, Ming F Y, Chen C M, Lei J J, Cao B H. Advances in phytohormone auxin response to high temperature., 2020, 47: 2238–2246 (in Chinese with English abstract).
[8] Schaker P D, Palhares A C, Taniguti L M, Peters L P, Creste S, Aitken K S, Van Sluys M A, Kitajima J P, Vieira M L, Monteiro-Vitorello C B. RNAseq transcriptional profiling following whip development in sugarcane smut disease., 2016, 11: e0162237.
[9] Yang Y Y, Gao S W, Su Y C, Lin Z L, Guo J L, Li M J, Wang Z J, Que Y X, Xu L P. Transcripts and low nitrogen tolerance: regulatory and metabolic pathways in sugarcane under low nitrogen stress., 2019, 163: 97–111.
[10] Strader L C, Zhao Y. Auxin perception and downstream events., 2016, 33: 8–14.
[11] 邹纯雪, 门淑珍. 生长素的外输载体PIN蛋白家族研究进展. 中国细胞生物学学报, 2013, 35: 574–582.
Zou C X, Men S Z. Research advances in auxin efflux carrier PIN proteins.,2013, 35: 574–582 (in Chinese with English abstract).
[12] 潘建伟, 张晨燕, 周哉材. 生长素极性输出载体PIN的研究进展. 浙江师范大学学报(自然科学版), 2018, 41: 436–443.
Pan J W, Zhang C Y, Zhou J C. Research progress of auxin efflux transporter PIN proteins.(Nat Sci Edn),2018, 41: 436–443 (in Chinese with English abstract).
[13] Barbez E, Kubeš M, Rolčík J, Béziat C, Pěnčík A, Wang B, Rosquete M R, Zhu J, Dobrev P I, Lee Y, Zažímalovà E, Petrášek J, Geisler M, Friml J, Kleine-Vehn J. A novel putative auxin carrier family regulates intracellular auxin homeostasis in plants., 2012, 485: 119–122.
[14] Santos F, Teale W, Fleck C, Volpers M K, Ruperti B, Palme K. Modelling polar auxin transport in developmental patterning., 2010, 12: 1438–8677.
[15] Wang J, Jin Z, Yin H, Yan B, Ren Z Z, Xu J, Mu C J, Zhang Y, Wang M Q, Liu H. Auxin redistribution and shifts in PIN gene expression during., 2014, 61: 688–696.
[16] Friml J. Subcellular trafficking of PIN auxin efflux carriers in auxin transport., 2010, 89: 231–235.
[17] Murphy A S . Seven things we think we know about auxin transport., 2011, 4: 487–504.
[18] Zwiewka M, Bilanoviová V, Seifu Y W, Nodzyński T. The buts and bolts of PIN auxin efflux carriers., 2019, 10: 985.
[19] Luschnig C, Vert G. The dynamics of plant plasma membrane proteins: PINs and beyond., 2014, 141: 2924–2938.
[20] Adamowski M, Friml J. PIN-dependent auxin transport: action, regulation, and evolution., 2015, 27: 20.
[21] Habets M E, Offringa R. PIN-driven polar auxin transport in plant developmental plasticity: a key target for environmental and endogenous signal., 2014, 203: 362–377.
[22] Feraru E, Vosolsobe S, Feraru MI, Petrasek J, Kleine-Vehn J. Evolution and structural diversification of PILS putative auxin carriers in plants., 2012, 3: 227.
[23] Mohanta T K, Mohanta N, Bae H. Identification and expression analysis of PIN-Like (PILS) gene family of rice treated with auxin and cytokinin.(Basel), 2015, 6: 622–640.
[24] Song S L, Wang Z P, Ren Y M, Sun H M. Full-length transcriptome analysis of the,S, andfamilies involved in somatic embryogenesis ofDC. Fisch., 2020, 21: 453.
[25] Feraru E, Feraru M I, Barbez E, Waidmann S, Sun L, Gaidora A, Kleine-Vehn J. PILS6 is a temperature-sensitive regulator of nuclear auxin input and organ growth in., 2019, 116: 3893–3898.
[26] Beziat C, Barbez E, Feraru M I, Lucyshyn D, Kleine-Vehn J. Light triggers PILS-dependent reduction in nuclear auxin signalling for growth transition., 2017, 3: 17105.
[27] Guo J L, Xu L P, Su Y C, Wang H B, Gao S W, Xu J S, Que Y X., a metallothionein gene of sugarcane, plays an important role in the regulation of heavy metal tolerance/accumulation., 2013, 2013: 904769.
[28] Verma A K, Agarwal A K, Dubey R S, Solomon S, Singh S B. Sugar partitioning in sprouting lateral bud and shoot development of sugarcane., 2013, 62: 111–115.
[29] 刘燕群, 李玉萍, 梁伟红, 宋启道, 秦小立, 叶露. 国外甘蔗产业发展现状. 世界农业, 2015, (8): 147–152.
Liu Y Q, Li Y P, Liang H W, Song Q D, Qin X L, Ye L. Current status and development of the abroad sugarcane industry., 2015, (8): 147–152 (in Chinese with English abstract).
[30] 王明强, 李文凤, 黄应昆, 王晓燕, 卢文洁, 罗志明. 我国大陆蔗区发生的甘蔗病毒病及防控对策. 中国糖料, 2010, (4): 55–58.
Wang M Q, Li Y P, Huang Y K, Wang X Y, Lu W J, Luo Z M. Occurrence and controlling strategies on sugarcane viral diseases in Chinese mainland., 2010, (4): 55–58 (in Chinese with English abstract).
[31] 周晚秋, 娄春, 何子林, 傅峻涛. 巴西生物燃料技术现状与发展. 中外能源, 2017, 22: 24–31.
Zhou W Q, Lou C, He Z L, Fu J T. Status quo and development of Brazilian biofuel technologies., 2017, 22: 24–31 (in Chinese with English abstract).
[32] 翁卓, 黄寒. 中国制糖产业竞争力对比与政策建议—基于对巴西、印度、泰国考察的比较. 甘蔗糖业, 2015, (4): 65–72.
Weng Z, Huang H. Comparative analysis on China’s sugar industry competitiveness: based on the comparison of Brazil, India and Thailand sugar industry.,2015, (4): 65–72 (in Chinese with English abstract).
[33] 刘晓雪, 王新超. 2017/18榨季中国食糖生产形势分析与2018/19榨季展望. 农业展望, 2018, 14(11): 40–46.
Liu X X, Wang X C. Domestic sugar production situation in 2017/18 crushing season and its prospect for 2018/19 crushing season., 2018, 14(11): 40–46 (in Chinese with English abstract).
[34] 李谭面.来宾市甘蔗高产稳产高糖栽培技术. 乡村科技,2017, (15): 63–64.
Li T M. Cultivation techniques of sugarcane with high and stable yield and sugar in Laibin city., 2017, (15):63–64 (in Chinese with English abstract).
[35] 李瑞美,潘世明,王水琦,林一心.糖能兼用甘蔗新品种闽糖92-142的选育研究. 福建农业学报,2006, 21:98–100.
Li R M, Pan S M, Wang S Q, Lin Y X. Selection and breeding of new sugarcane variety mintang 92-142., 2006, 21:98–100 (in Chinese with English abstract).
[36] 蔡文燕,吴水金.能源甘蔗—甘蔗糖业发展的新亮点. 甘蔗糖业,2006, (3): 22–25.
Cai W Y, Wu S J. Energy sugarcane-a new bright spot in the development of cane sugar industry.,2006, (3):22–25 (in Chinese with English abstract).
[37] LalmanJA, ShewaWA, Gallagher J, Ravella S. Biofuels production from renewable feedstocks. Springer, 2016, pp. 193–220.
[38] Zhang J, Zhang X, Tang H, Zhang Q, Hua X, Ma X, Zhu F, Jones T, Zhu X, Bowers J, Wai C M, Zheng C, Shi Y, Chen S, Xu X, Yue J, Nelson D R, Huang L, Li Z, Xu H, Zhou D, Wang Y, Hu W, Lin J, Deng Y, Pandey N, Mancini M, Zerpa D, Nguyen J K, Wang L, Yu L, Xin Y, Ge L, Arro J, Han J O, Chakrabarty S, Pushko M, Zhang W, Ma Y, Ma P, Lv M, Chen F, Zheng G, Xu J, Yang Z, Deng F, Chen X, Liao Z, Zhang X, Lin Z, Lin H, Yan H, Kuang Z, Zhong W, Liang P, Wang G, Yuan Y, Shi J, Hou J, Lin J, Jin J, Cao P, Shen Q, Jiang Q, Zhou P, Ma Y, Zhang X, Xu R, Liu J, Zhou Y, Jia H, Ma Q, Qi R, Zhang Z, Fang J, Fang H, Song J, Wang M, Dong G, Wang G, Chen Z, Ma T, Liu H, Dhungana S R, Huss S E, Yang X, Sharma A, Trujillo J H, Martinez M C, Hudson M, Riascos J J, Schuler M, Chen L Q, Braun D M, Li L, Yu Q, Wang J, Wang K, Schatz M C, Heckerman D, Van Sluys M A, Souza G M, Moore P H, Sankoff D, Van Buren R, Paterson A H, Nagai C, Ming R. Allele-defined genome of the autopolyploid sugarcaneL, 2018, 50: 1565–1573.
[39] Chen C, Chen H, Zhang Y, Thomas HR, Frank MH, He Y, Xia R. TBtools: an integrative toolkit developed for interactive analyses of big biological data., 2020,13: 1194–1202.
[40] Ling H, Huang N, Wu Q, Su Y, Peng Q, Ahmed W, Gao S, Su W, Que Y, Xu L. Transcriptional insights into the sugarcane-sorghum mosaic virus interaction., 2018,11: 163–176.
[41] Que Y, Su Y, Guo J, Wu Q, Xu L. A global view of transcriptome dynamics during sporisorium scitamineum challenge in sugarcane by RNA-Seq., 2014 , 9:e106476.
[42] Zhang D, Gao F L, Jakovlić I, Zou H, Zhang J, Li WX, Wang GT.PhyloSuite: an integrated and scalable desktop platform for streamlined molecular sequence data management and evolutionary phylogenetics studies., 2020, 20: 348–355.
[43] Ling H, Wu Q B, Guo J L, Xu L P, Que Y X. Comprehensive selection of reference genes for gene expression normalization in sugarcane by real time quantitative RT-PCR., 2014, 9:e97469.
[44] 黄宁,惠乾龙,方振名,李姗姗,凌辉,阙友雄,袁照年.甘蔗β-胡萝卜素异构酶基因家族的鉴定、定位和表达分析. 作物学报,2021,47:882–893.
Huang N, Hui Q L, Fang Z M, Li S S, Ling H, Que Y X, Yuan Z N. Identification, localization and expression analysis of beta-carotene isomerase gene family in sugarcane., 2021,47:882–893 (in Chinese with English abstract).
[45] Staff P O.Correction: comprehensive selection of reference genes for gene expression normalization in sugarcane by real time quantitative RT-PCR.,2015,10: e0118444.
[46] Livak K J, Schmittgen T D. Analysis of relative gene expression data using real-time quantitative PCR and the 2−∆∆CTmethod., 2001, 25: 402–408.
[47] Heisler M G, Ohno C, Das P, Sieber P, Reddy G V, Long J A, Meyerowitz E M. Patterns of auxin transport and gene expression during primordium development revealed by live imaging of theinflorescence meristem., 2005, 15: 1899–1911.
[48] Geldner N. Auxin transport inhibitors block PIN1 cycling and vesicle trafficking., 2001, 413: 425–428.
[49] Zhou J J, Luo J. The PIN-FORMED auxin efflux carriers in plants., 2018, 19: 2759.
[50] Sun L, Feraru E, Feraru MI, WangZY, CorrespondenceKV. PIN-likes coordinate brassinosteroid signaling with nuclear auxin input in., 2020, 30: 1579–1588.
[51] Béziat C, Kleine-Vehn J. The road to auxin-dependent growth repression and promotion in apical hooks., 2018, 28:519–525.
[52] Sauer M, Kleine-Vehn J. PIN-FORMED and PIN-LIKES auxin transport facilitators., 2019, 146: dev168088.
[53] 董衍坤, 黄定全, 高震, 陈栩. 大豆PIN-like (PILS)基因家族的鉴定、表达分析及在根瘤共生固氮过程中的功能初探. 作物学报, 2021, 47: 1–20.
Dong Y K, Huang D Q, Gao Z, Chen Y. Preliminary study of identification, expression profile of soybeanPIN-like (PILS) gene family and its function in symbiotic nitrogen fixation in root nodules., 2021,47: 1–20 (in Chinese with English abstract).
[54] Zou X, Long J, Zhao K, Peng A, Chen M, Long Q, He Y, Chen S. Overexpressingandreduces susceptibility tosubsp.by repressing auxin signaling in citrus (Osbeck).,2019, 14: e0220017.
[55] Fan S, Chang Y, Liu G, Shang S, Tian L, Shi H. Molecular functional analysis of auxin/indole-3-acetic acid proteins (Aux/IAAs) in plant disease resistance in cassava., 2020,168: 88–97.
[56] 陈倩, 卓维, 李佳皓, 鲁黎明, 李立芹. 烟草基因的克隆及生物信息学和表达模式分析. 烟草科技, 2018, 51(1): 1–7.
Chen Q, Zhuo W, Li J H, Lu L M, Li L Q. Cloning, bioinformatics and expression pattern analysis of., 2018,51(1):1–7 (in Chinese with English abstract).
[57] 郭玉敏,张云华.玉米基因克隆及诱导表达分析.分子植物育种,2020,18:719–728.
Guo Y M, Zhang Y H. Cloning and induced expression analysis ofin.,2020,18:719–728 (in Chinese with English abstract).
[58] 于永昂,睢晓湉,张蕾,张夏冰.小麦转录因子基因克隆与抗旱性分析.西北农林科技大学学报(自然科学版),2022, 50(4):32–41.
Yu Y A, Sui X T, Zhang L, Zhang X B. Cloning and function against drought stress oftranscription factor gene.(Nat Sci Edn),2022, 50(4):32–41 (in Chinese with English abstract).
[59] 秦朋飞,尚小光,宋健,郭旺珍.棉花酰基辅酶A结合蛋白(ACBP)家族基因的发掘及在非生物胁迫抗性中的功能鉴定.作物学报,2016,42:1577–1591.
Qin P F, Shang X G, Song J, Guo W Z. Genome-wide identification of acyl-CoA-binding protein (ACBP) gene family and their functional analysis in abiotic stress tolerance in cotton., 2016,42:1577–1591 (in Chinese with English abstract).
[60] 靳容,蒋薇,刘明,赵鹏,张强强,李铁鑫,王丹凤,范文静,张爱君,唐忠厚.甘薯Dof基因家族挖掘及表达分析.作物学报, 2022, 48: 608–623.
Ji R, Jiang W, Liu M, Zhao P, Zhang Q Q, Li T X, Wang D F, Fan W J, Zhang A J, Tang Z H. Genome-wide characterization and expression analysis of Dof family genes in sweetpotato., 2022, 48: 608–623 (in Chinese with English abstract).
[61] 袁大双, 邓琬玉, 王珍, 彭茜, 张晓莉, 姚梦楠, 缪文杰, 朱冬鸣, 李加纳, 梁颖. 甘蓝型油菜基因的克隆及功能分析. 作物学报, 2022, 48: 840–850.
Yuan D S, Deng W Y, Wang Z, Peng X, Zhang X L, Yao M N, Miu W J, Zhu D M, Li J N, Liang Y. Cloning and functional analysis ofgene in., 2022, 48: 840–850 (in Chinese with English abstract).
[62] 黄宁, 范金燕, 赖洁玲, 刘秦, 阙友雄, 朱宇林, 凌辉. 甘蔗类质子梯度调节蛋白基因的生物信息学及表达分析. 分子植物育种, 2021.
Huang N, Fan J Y, Lai J L, Liu Q, Que Y X, Zhu Y L, Ling H. The bioinformatics and expression analysis of a sugarcane proton gradient regulation like 1 gene., 2021 (in Chinese with English abstract).
[63] 孟钰玉, 魏春茹, 范润侨, 于秀梅, 王逍冬, 赵伟全, 魏新燕, 康振生, 刘大群. 小麦基因的表达响应逆境胁迫并与SCF复合体接头蛋白TaSKP1相互作用. 作物学报, 2021, 47: 224–236.
Meng Y Y, Wei C R, Fan R Q, Yu X M, Wang X D, Zhao W Q, Wei X Y, Kang Z S, Liu D Q.gene shows induced expression pattern in wheat responses to stresses and interacts with adaptor protein SKP1 from SCF complex., 2021, 47: 224–236 (in Chinese with English abstract).
Identification and expression analysis of PIN-LIKES gene family in sugarcane
PAN Jie-Ming1, TIAN Shao-Rui3, LIANG Yan-Lan2, ZHU Yu-Lin1, ZHOU Ding-Gang4, QUE You-Xiong2, LING Hui1,*, and HUANG Ning1,*
1College of Agriculture, Yulin Normal University, Yulin 537000, Guangxi, China;2Key Laboratory of Sugarcane Biology and Genetic Breeding (Fujian), Ministry of Agriculture and Rural Affairs / Fujian Agriculture and Forestry University, Fuzhou 350002, Fujian, China;3College of Plant Protection, Southwest University, Chongqing 400700, China;4College of Life Science, Hunan University of Science and Technology/ Key Laboratory of Economic Crops Genetic Improvement and Integrated Utilization, Xiangtan 411201, Hunan, China
PILS (PIN-LIKES) is a newly reported auxin efflux carrier, which is involved in the polar transport of auxin. In this study, bioinformatics analysis indicated that 11 SsPILS genes and 4 ScPILS genes were identified from thegenomic and the sugarcane cultivars transcriptomic data. 11 SsPILS gene family members were located at 6 chromosomes, and had 5–11 introns, respectively. Phylogenetic tree analysis showed that these PILS fromwas clustered into 3 different branches and had highly homologous with PILS from. The transcriptomic data revealed that,, the ortholog of, was differentially expressed in the sugarcane cultivars underandstress.(NCBI accession number: OM258732), cloned by RT-PCR, had 1332 bp in length, containing a 1233 bp complete open reading frame and encoding 410 amino acids residues. The isoelectric point, instability coefficient, and average hydrophobicity value of ScPILS1c were 6.17, 39.31, and 0.689, respectively. The ScPILS1cwas predicted as a stable acid hydrophobicity protein. The secondary structure of ScPILS1c protein was mainly composed of α-helix (48.54%) and random coils (35.37%) and was highly consistent with the prediction of tertiary structure. The qRT-PCR demonstrated that the relative expression ofwas tissue-specific and the highest expression was observed in epidermis while the lowest in pith.was significantly induced by H2O2treatment andinfection. Transient expression onleaves suggested thatwas localized on cell membrane. This study provides a reference for further research on the structure and function ofgene in sugarcane.
sugarcane;gene; bioinformatics; qRT-PCR; gene family
10.3724/SP.J.1006.2023.24022
本研究由国家自然科学基金项目(31801424, 32160435)和玉林师范学院高层次人才启动项目(G2020ZK03)资助。
This study was supported by the National Natural Science Foundation of China (31801424, 32160435) and the Scientific Research Foundation of Yulin Normal University (GZ2020ZK03).
黄宁, E-mail: hning2012@126.com; 凌辉, E-mail: linghuich@163.com
E-mail: jiemingpan@163.com
2022-01-17;
2022-05-05;
2022-05-12.
URL: https://kns.cnki.net/kcms/detail/11.1809.S.20220511.1914.004.html
This is an open access article under the CC BY-NC-ND license (http://creativecommons.org/licenses/by-nc-nd/4.0/)
