基于MPNet 与BiLSTM 的COVID-19 临床文本命名实体识别方法
2023-02-08蔡晓琼郑增亮苏前敏郭晶磊
蔡晓琼,郑增亮,苏前敏,郭晶磊
(1 上海工程技术大学 电子电气工程学院,上海 201620;2 上海中医药大学 基础医学院,上海 201203)
0 引言
生物医学命名实体识别,是生物医学信息提取领域最基本的任务之一,其准确性对后续研究工作至关重要。随着生物医学研究与信息化技术的迅速发展,临床医学文献数量呈指数级增长,这些资源中存在大量非结构化数据,迫切需要开发一些自动化技术,来解决生物医学领域的单词分割、实体识别、关系抽取、主题分类等问题。由于生物医学实体的多样性和变异性,识别生物医学实体是一项非常具有挑战性的任务。目前,生物医学命名实体识别方法主要分为基于字典和规则的方法,以及基于深度学习的方法。
基于规则的命名实体识别方法主要依赖于人工定义的词典和规则,可以根据特定领域的词典和句法-词汇模式来进行设计。一些著名的基于规则的命名实体识别模型包括LaSIE-II、Sra、FASTUS 和LTG 等[1-4]。与传统方法相比,引入深度学习技术的命名实体识别方法,通过在大规模语料库中进行训练与学习,自动提取语义特征,使模型具有较强的泛化能力,同时降低了人力与时间成本。目前常用的词嵌入方法是由Mikolov 等[5]提出的Word2Vec模型。但该方法训练获得的词向量是静态的,无法解决一词多义问题。随着自然语言处理技术的发展,ELMo[6]、BERT[7]和XLNet[8]等预训练语言模型的推出,为下游任务提供了极大的帮助,对模型进行微调后,在命名实体识别任务上取得了较好的效果。但ELMo 输出的语义表征是单向的,无法获得更全面的双向信息;BERT 和XLNet 采用的MLM 和PLM 方法各自存在局限性,在用于下游任务微调时,会造成预训练和微调不匹配。为了解决以上问题,南京大学和微软共同提出了基于MLM 和PLM各自优点的预训练模型MPNet[9],弥补了MLM 无法学习tokens 之间依赖关系的不足,同时克服了PLM 无法获得下游任务中可见完整信息的问题。
针对目前临床试验文本研究匮乏、语料不足与标注质量不高等问题,本文结合UMLS 语义网络和专家定义方法建立了COVID-19 临床实体识别语料库,并将预训练语言模型MPNet 引入COVID-19 临床实体识别任务中,提出了一种基于MPNet 与BiLSTM 的医学实体识别模型。
1 MPNet-BiLSTM-CRF 命名实体识别模型
本文提出的MPNet-BiLSTM-CRF 命名实体识别模型主要结构如图1 所示。采用MPNet 预训练模型作为嵌入层对输入进行语义提取生成动态词向量;采用BiLSTM 捕捉长距离依赖关系;最后由CRF推理层进行最佳标注序列解码,预测出全局最优标签。该方法在经典BiLSTM-CRF 的基础上进行了改进,引入了MPNet 语言模型,在预测掩码标记的同时以更多的信息为条件,从而获得更好的学习表征,并减少了微调阶段的差异。
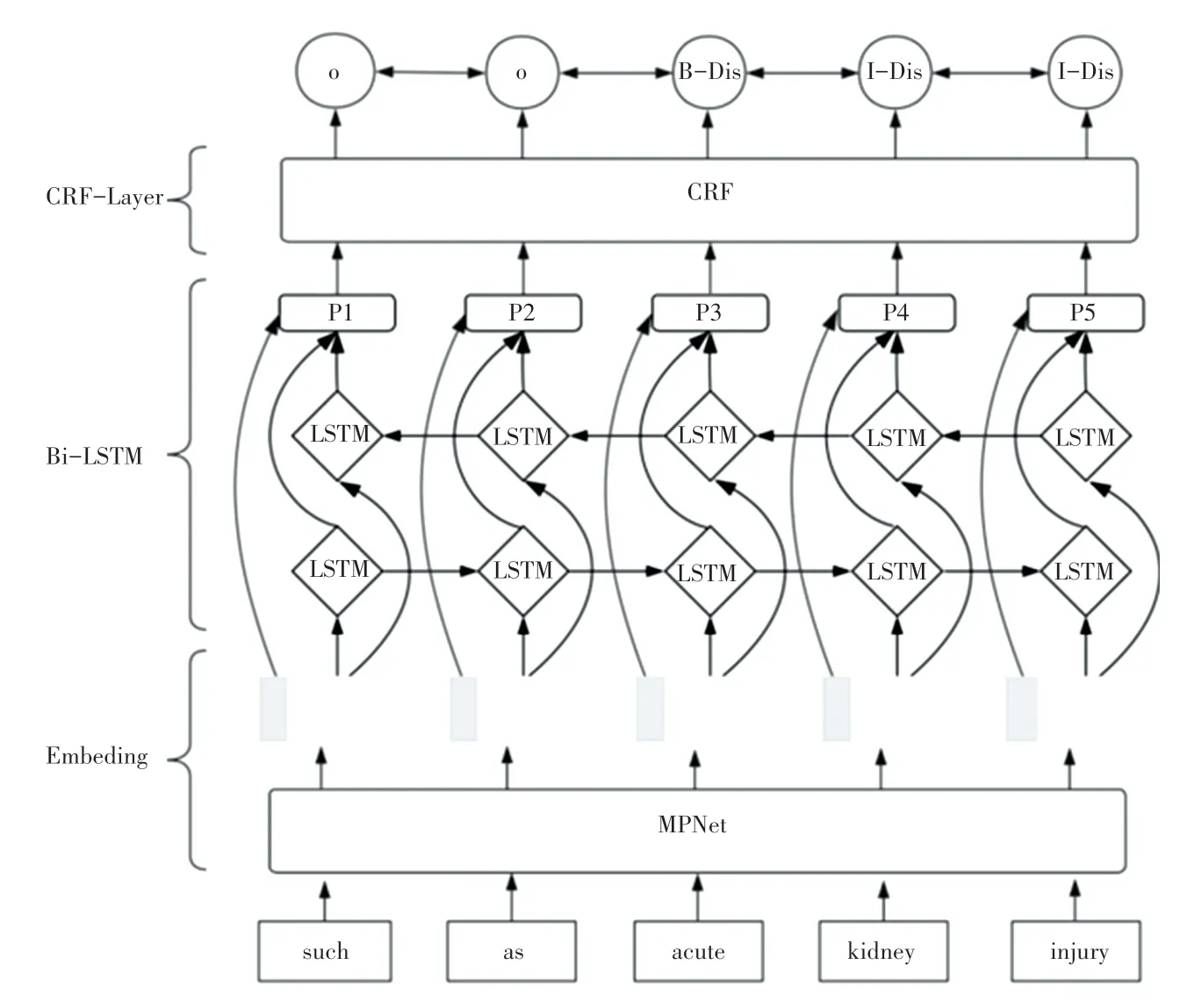
图1 MPNet-BiLSTM-CRF 模型结构图Fig.1 The structure of MPNet-BiLSTM-CRF model
1.1 MPNet 预训练模型
MPNet 模型的注意力掩码机制:首先设长度n =6 的输入序列为(x1,x2,x3,x4,x5,x6),若随机生成的序列为(x5,x4,x2,x6,x3,x1),预测值分别是x6、x3和x1,则非预测序列表示为(x5,x4,x2,[MASK],[MASK],[MASK] ),对应位置序列为(P5,P4,P2,P6,P3,P1)。其次,为 了能让预测部分 的[MASK]看到之前预测的tokens,MPNet 采用了PLM 双流自注意力机制完成自回归生成,并为内容流和查询流设置了不同的掩蔽机制。例如,MPNet在预测上述序列中的x3时,能在非预测部分看到(x5+P5,x4+P4,x2+P2),同时在预测部分看到之前预测的(x6+P6),从而避免MLM 中依赖关系遗漏的问题。此外,为了确保预训练中输入信息与下游任务中输入信息的一致性,MPNet 在非预测部分增加了掩码符号和位置信息([MASK]+P6,[MASK]+P3,[MASK]+P1),使模型能看到完整的句子。当预测x3时,能在非预测部分看到原始的(x5+P5,x4+P4,x2+P2)以及引入了额外tokens和位置信息的([MASK]+P3,[MASK]+P1),同时在预测部分看到之前预测的(x6+P6)。通过上述办法对查询流和内容流进行位置补偿后的模型,能够大幅减少预训练与微调之间输入不一致的问题。
假定句子为“to take up seasonal flu vaccination”,模 型 输 入 序 列 为[to,take up,seasonal,flu,vaccination],需 要 预 测 的token 是[seasonal,flu,vaccination],MPNet 的因子化如下:

1.2 Transformer
自Vaswani 等[10]提出Transformer 这一基于自注意力机制的深度学习模型以来,已被广泛应用到NLP 领域中解决各种复杂问题,几款主流的预训练语言模型(如BERT、XLNet 和ALBERT 等)都以Transformer 作为其骨干网络。传统语言模型通常基于CNN 或RNN 编码器进行训练,在长周期语境建模中能力较为欠缺,并且在学习单词表征时存在位置偏差,尤其RNN 是按顺序处理输入的,即一个字一个字地处理,对计算机硬件的并行能力要求较高。为了克服现有深度学习模型的缺点,Transformer 在每个关键模块中引入了Attention 机制,大幅提升了模型对文本的特征提取能力。此外,Transformer 还引入了多头注意力机制(Multi-Head Attention),使模型能够使用各个序列位置的各个子空间的表征信息来进行序列数据处理,其相当于多个不同的自注意力模块的集成,从而构成蕴含完整语料信息的多粒度特征。标准的Transformer 结构如图2 所示。
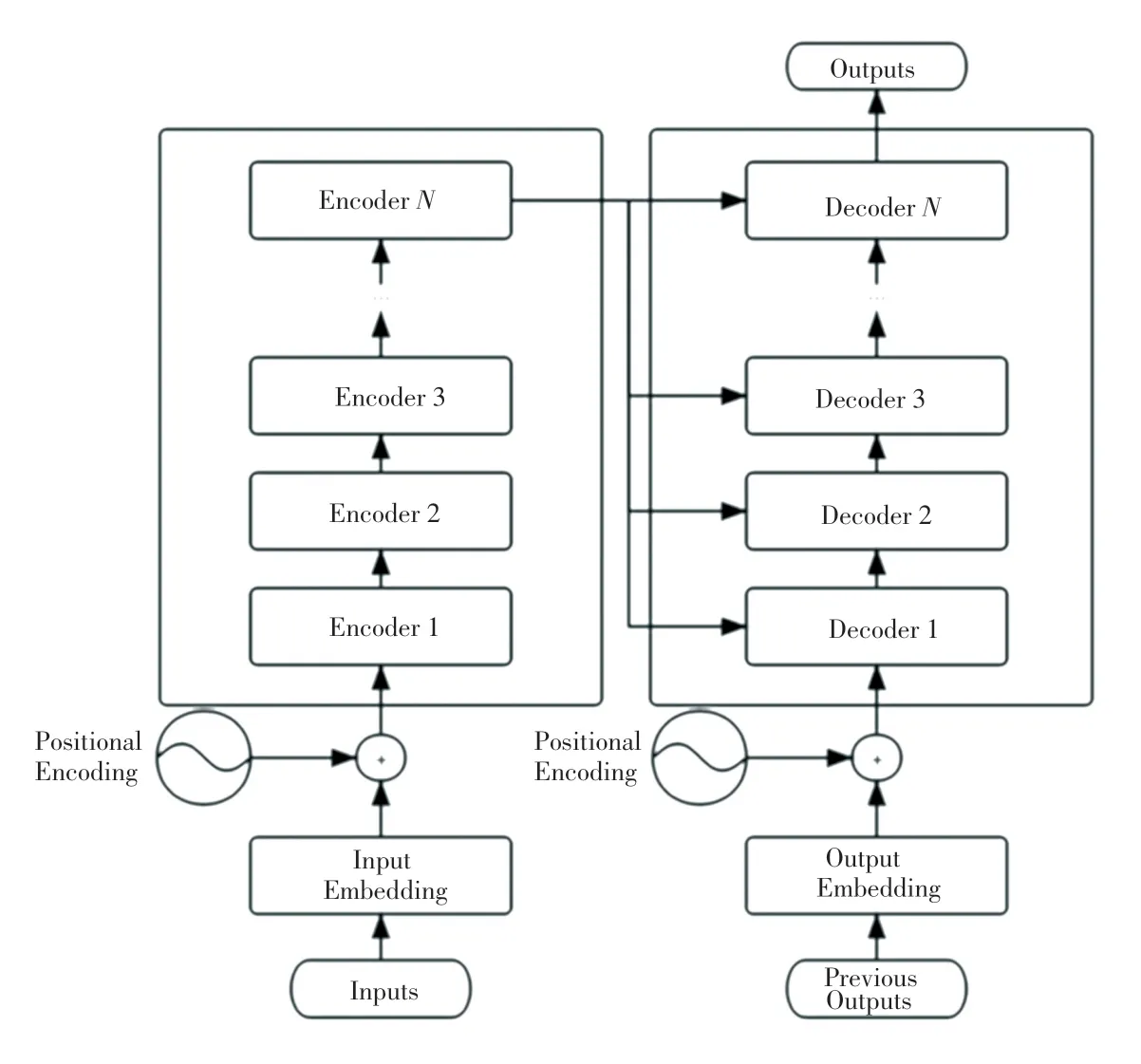
图2 标准Transformer 结构图Fig.2 Standard Transformer structure diagram
通 过 图2 可 知,Transformer 由encoder 和decoder 两部分组合而成。其中,encoder 通过6 个编码器叠加构成,每个encoder 中都包含一个自注意力层和前馈神经网络层,两个子层之间通过残差网络结构进行连接,后接一个正则化层。为了方便各层之间的残差连接,模型中所有子层的输出维度均为512。Decoder 也由6 个完全相同的解码器叠加组成,除了与编码器完全一致的两个子层外,Decoder 还设置了一个多头注意力层。多头注意力层是由多个自注意力层拼接而成的,可以捕捉到单词之间各维度上的相关系数。
1.3 双向长短期记忆网络(BiLSTM)模型
为了解决传统循环神经网络(Recurrent Neural Network,RNN)在训练较长语句时可能导致梯度爆炸和梯度消失问题,Hochreiter 等人[11]提出了长短期记忆网络(Long Short-Term Memory,LSTM),该模型能够在长文本训练中捕捉长距离依赖特征。与标准RNN 模型相比,LSTM 在其结构基础上增加了门控机制和记忆单元两个模块。其中,记忆单元用于存储文本特征,门控机制则对记忆单元中的存储信息进行筛选。LSTM 模型分别设置了输入门、遗忘门和输出门保护和控制细胞状态,通过累加更新传递信息的方式,消除了RNN 模型在处理长文本任务时可能出现的问题,其单元结构如图3 所示。
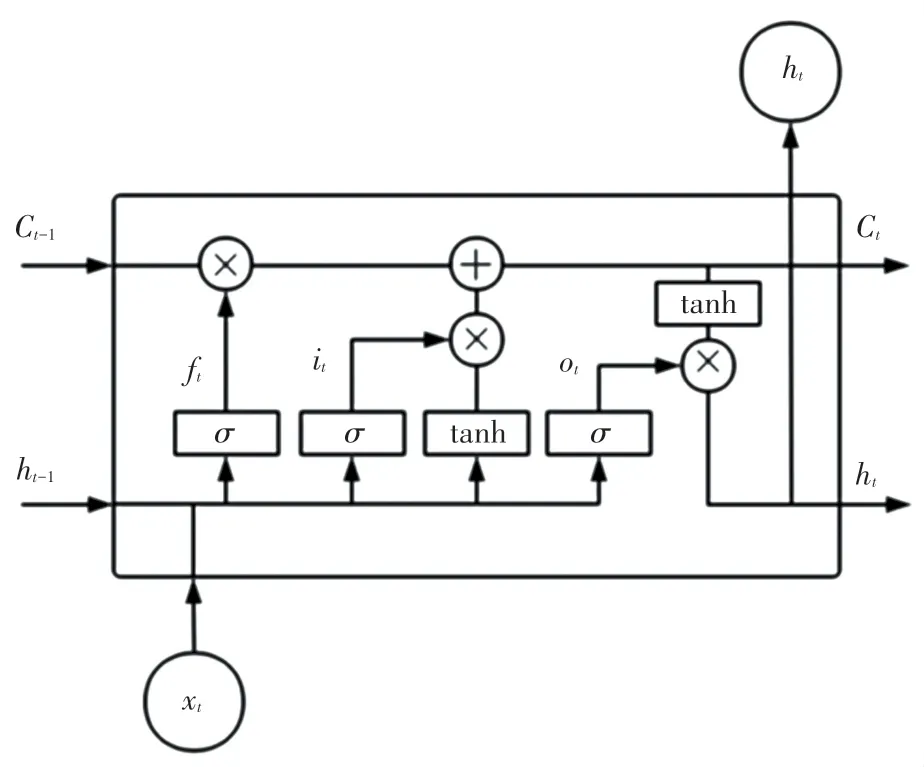
图3 LSTM 单元结构图Fig.3 LSTM unit structure
LSTM 模型是由t时刻的输入词Xt、细胞状态Ct、临时细胞状态隐含状态ht、遗忘门ft、记忆门it及输出门ot组成,通过对细胞状态中信息遗忘和记忆新的信息,使得对后续时刻计算有用的信息得以传递,而无用的信息被摒弃,并在每个时间步都会输出隐含状态ht。其中遗忘、记忆与输出通过上一时刻的隐含状态ht-1和当前输入Xt计算出来的遗忘门ft、记忆门it和输出门ot来控制,遗忘门计算如公式(2):
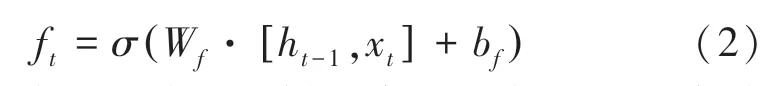
其中,输入为前一时刻的隐含状态ht -1和当前时刻的输入词Xt。
记忆门的值it和临时细胞状态分别由公式(3)和公式(4)计算得出:
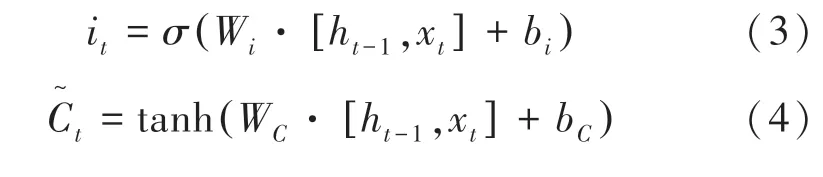
根据输入为记忆门的值it、遗忘门的值ft、临时细胞状态和上一时刻细胞状态Ct -1计算t时刻细胞状态Ct为
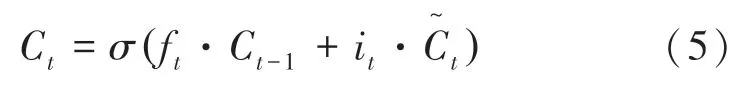
前一时刻的隐含状态ht -1、当前时刻的输入词Xt和当前时刻细胞状态ht,计算t时刻输出门的值ot和隐含状态ht为
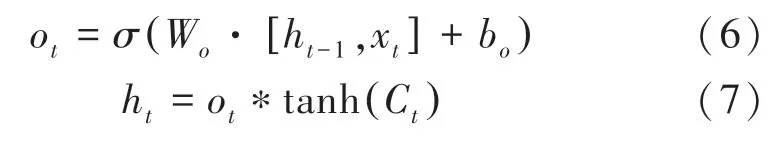
其中,σ为sigmoid函数;tanh 为双曲正切激活函数;W和b分别表示链接两层的权重矩阵和偏置向量。经过LSTM 模型计算后,最终即可得到与句子长度相同的隐含状态序列{h0,h1,…,hn-1}。
对于处理NLP 任务(尤其对序列标注任务),上下文内容无论对单词、词组还是字符,在整个研究过程中都尤为重要。通常情况下,LSTM 常用单元为前向传播,然而在研究序列问题时,前向LSTM 无法处理下文的内容信息,从而导致模型无法学习到下文的知识,影响最终模型效果。而双向长短期记忆网络(Bi -directional Long Short -Term Memory,BiLSTM)既能获取上文信息,又能捕获下文内容,对双向信息都能进行记忆,通过同时得到前后两个方向的输出,来提高整个NLP 模型的性能。BiLSTM模型结构如图4 所示。
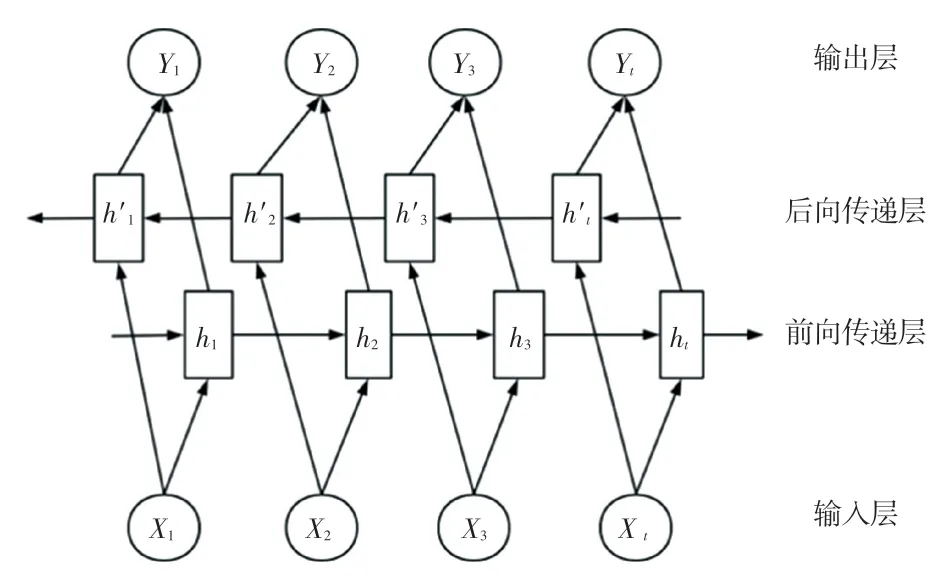
图4 BiLSTM 模型结构图Fig.4 BiLSTM model structure
1.4 条件随机场(CRF)
条件随机场(Conditional Random Field,CRF)是Lafferty 等[12]基于最大熵模型和隐马尔可夫模型所提出的一种判别式概率无向图学习模型,常用于标注和切分有序数据的条件概率模型。在序列任务标注任务中,令X =(X1,X2,…,Xn)表示模型的观察序列,Y =(Y1,Y2,…,Yn)表示状态序列,P(Y |X)为线性链条件随机场,则在随机变量X取值为x的条件下,计算状态序列Y取值为y的条件概率分布P(Y |X),计算公式为
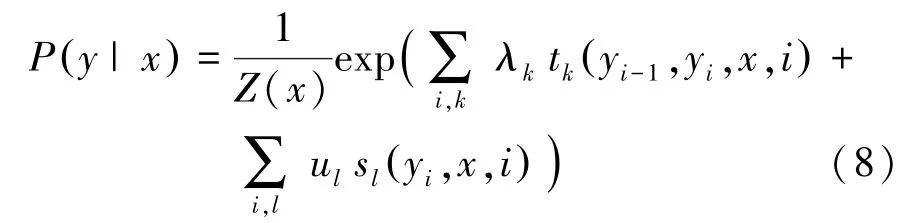
其中,tk、sl为特征函数,其函数取值为0 或1(当满足特征条件时,函数取值为1,反之为0);λk、ul为对应权重;Z(x)为归一化因子,其计算公式为
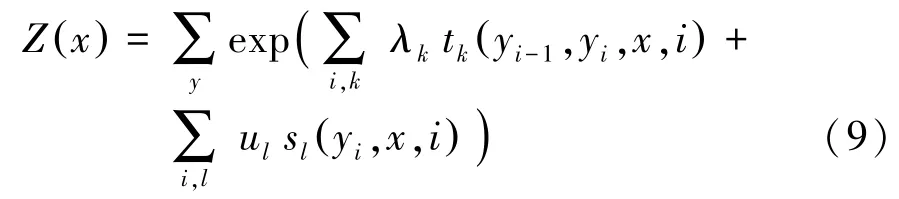
为了对上述公式进行简化,可将转移特征、状态特征及其权重用统一的符号进行表示,简化后的公式为
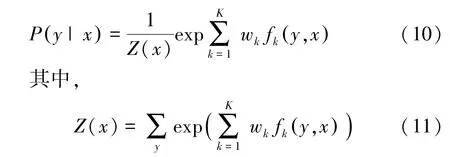
CRF 中最重要的就是训练模型的权重。通常情况下,使用最大化对数似然函数进行CRF 模型的训练,通过式(12)计算在给定条件下,标签序列Y的条件概率:
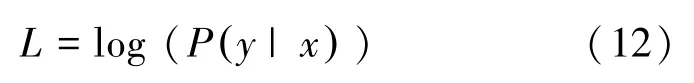
最后,在CRF 模型预测过程中采用维特比(Viterbi)算法来求解全局最优序列,通过该算法可以计算出与预测对象对应的最大概率标签,计算公式为
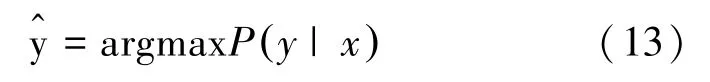
2 实验结果与分析
2.1 实验数据
本文使用的是美国临床试验注册中心(ClinicalTrials,CT)中的COVID-19 相关临床试验注册数据,CT 官方网址为https://clinicaltrials.gov。CT 中收录了临床研究者在世界各地进行的私人或公共资助的临床研究项目,其中包含有关人类志愿者医学研究的信息。随着COVID-19 的爆发,越来越多相关的临床试验在此平台进行了注册,临床记录中富含COVID-19 相关的临床医学知识。考虑到试验结束前可能存在信息更新不完整的问题,本文基于已完成的697 项干预性试验数据进行COVID-19 临床试验的命名实体识别实验。
2.2 实验配置
本研究采用的NER 模型基于PyTorch 深度学习框架,实验环境配置见表1。

表1 实验环境配置Tab.1 Experimental environment configuration
本研究采用微软发布的MPNet 模型,由12 个Transformer 层组合而成,隐藏层维度设为768,12 个注意头模式;使用GELU 作为其激活函数,BiLSTM隐藏单元为128。在训练阶段,MPNet-BiLSTM-CRF的最大序列长度为256,batch_size 为128,MPNet 学习率设为3e-5,Dropout 为0.1,其他模块Dropout 为0.3,并通过Adam 优化算法对模型进行训练。
2.3 概念定义与标注策略
临床试验注册中的非结构化文本主要涉及了具有特定意义的相关医学实体。例如:药物名称“Remdesivir”、医疗程序“bronchoalveolar lavage”、疾病名称“COVID-19”等等。不同的研究对医疗实体的标注规则和定义都有一定差异,统一医学语言系统(Unified Medical Language System,UMLS)收录了超过500 万条生物医学术语,至少200 万种医学概念,目前已广泛应用于文献分类、临床研究和中英文电子病历等领域中。本研究参考了UMLS 定义的实体类别以及文献[13]中提出的医学实体标注规范,对临床试验注册内容规定了实体标准和含义,结合文本内容定义了8 种类别的临床实体,COVID-19临床文本命名实体识别示例见表2。
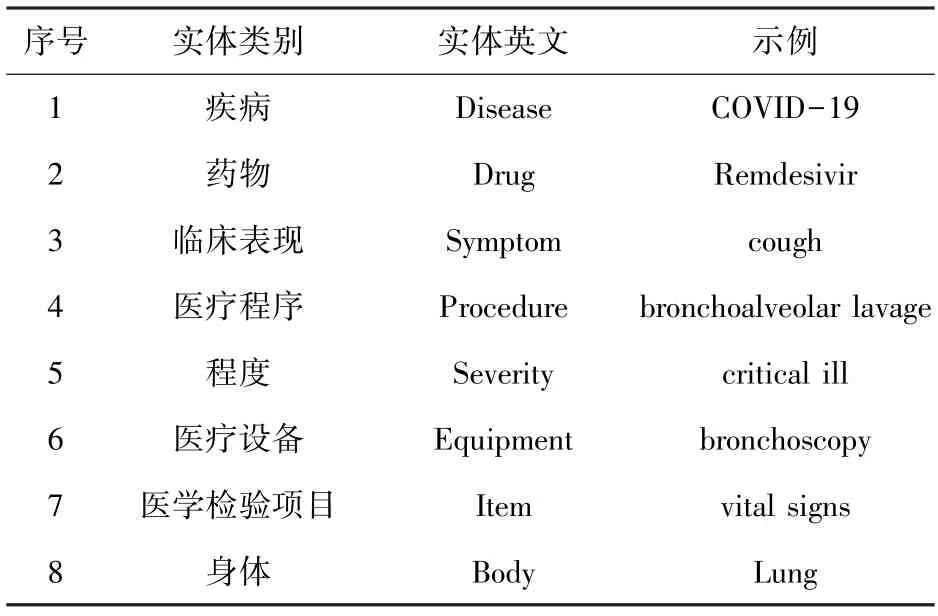
表2 实体类型定义与示例Tab.2 Entity type definitions and examples
NER 任务旨在提取文本中的命名实体,如名称或带有适当NER 类的标签,本文使用BIO 标注方式对序列进行标注。在这种格式中,不属于实体的标记被标记为“O”,“B”标记对应实体的第一个单词,“I”标记对应同一实体的其余单词。“B”和“I”标签后跟连字符(或下划线),后跟实体类别缩写(如Dru、Dis、Syp 等),表3 是对COVID-19 临床实体预测标签的示例。
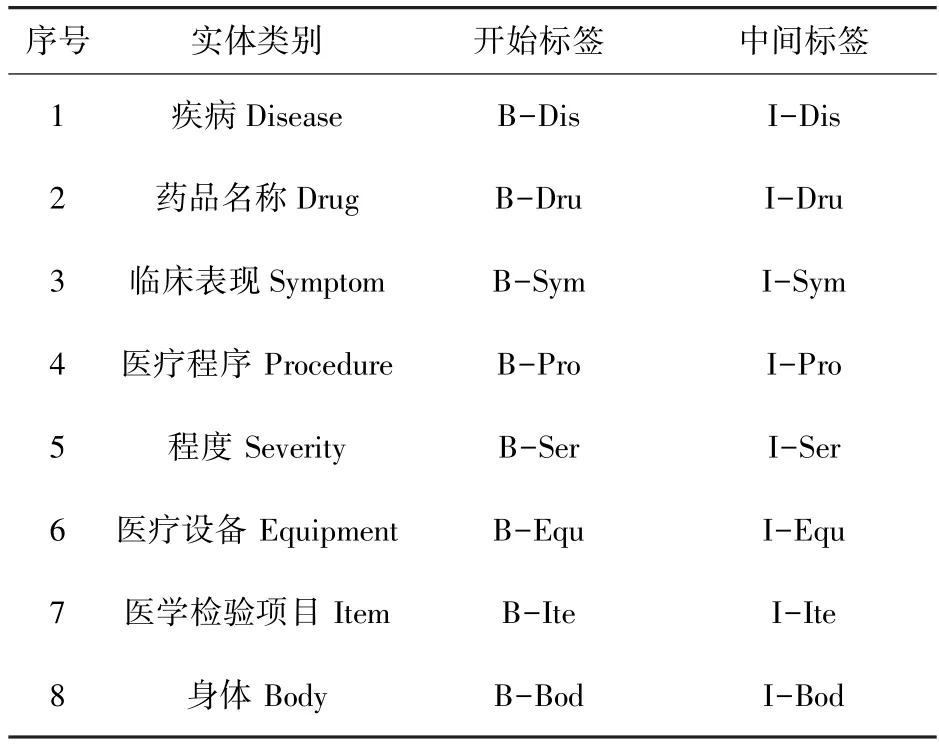
表3 实体预测标签定义Tab.3 Entity prediction label definitions
2.4 评价指标
本研究采用精确率(P)、召回率(R)和F1 值(F1)作为模型的评价指标。P是指正确识别的实体占全部预测实体的比重,R是正确识别的实体占语料库中所有实体的比重。各指标对应的计算公式如下:

其中,TP指正确地将正例预测为正的数量;FP指错误地将负例预测为正的数量;FN指错误地将正例预测为负的数量。
2.5 对比实验
为了验证本文提出的MPNet-BiLSTM-CRF 融合模型对COVID-19 临床试验注册实体具有较好的识别效果,与以下几种方法进行对比实验:
(1)经典的BiLSTM-CRF 模型:该模型采用word2vec 训练得到的词,嵌入向量作为输入,然后通过双向LSTM 层和CRF 完成编码与识别任务。目前该模型已广泛应用于中英文生物医学NER 任务中,并取得了良好的效果。
(2)Att-BiLSTM-CRF 融合模型[14]:该模型通过引入Attention 机制,确保模型能够专注于标记本文中同一token 的多个实例之间的一致性。
(3)XLNet-BiLSTM-CRF 模型[15]:该模型使用XLNet 预训练语言模型提取句子特征,然后将经典神经网络模型与获取的特征相结合,在公共医疗数据集上识别效果较好。
(4)BERT-BiLSTM-CRF 模型:通过使用BERT替换上个模型中的XLNet 方法。与现有方法相比,该模型在英文生物医学NER 任务中具有更好的表现[16]。
2.6 结果分析
对于不同的实体类型,各模型的实验结果见表4。由表4 可知,本文提出的MPNet-BiLSTM-CRF模型在大部分实体上表现较好,少数几类实体上表现略逊于基于XLNet 的融合模型,但F1 值的差别不明显。在所有模型的实验结果中,“Disease”、“Symptom”和“Severity”的F1 值较高,这是由于在摘要中这3 类实体结构简单且包含的信息种类较少,模型能够充分学习这些文本的特征。通过分析另外几种实体发现,存在训练数据集较少导致过拟合现象,另外部分实体结构复杂且出现次数少(例如为特定情况下COVID-19 防治选用的药物、检查或治疗措施),导致模型难以充分提取其特征。在后续COVID-19 临床试验摘要的NER 任务中,可以通过适量加入专业的英语语料库来增加语义特征,从而优化模型的识别能力。由表5 中展示的实验结果可知,在COVID-19临床试验注册数据集中,MPNet-BiLSTM-CRF 模型与其它4 种模型相比,整体的精确率、召回率和F1值都有所提高。经典BiLSTM-CRF 模型实体识别的F1 值为69.42%,引入Attention 机制后的模型F1值提升了2.49%。注意力矩阵能够计算当前的目标单词与序列中所有单词的相似性,通过权重矩阵为不同重要程度的单词分配相应的权重值,计算出文本的全局向量作为BiLSTM 输出的加权和。对比ERT-BiLSTM-CRF 和Att-BiLSTM-CRF 模型,前者实验结果的3 项指标均有提升,F1 值比后者提升了1.26%,说明BERT 预训练语言模型能更好地捕捉语义关系和单词特征。BERT-BiLSTM-CRF 与XLNet-BiLSTM-CRF 模型相比,精确率分别是72.60%和74.57%,F1 值分别为73.17%和74.01%。相比之下,基于XLNet 的NER 模型效果略优于BERT。而本文提出的MPNet-BiLSTM-CRF 融合模型与XLNet-BiLSTM-CRF 模型相比,F1 值提高了1.06%,通过使用融合了BERT 和XLNet 优点的MPNet 预训练语言模型,增强了序列的特征表示,同时弥补了两者的缺陷,从而提高了模型整体的识别能力。此外,MPNet 模型的识别能力提高后,速度并未有明显下降,因此MPNet 作为词嵌入层将具有更优秀的表现。
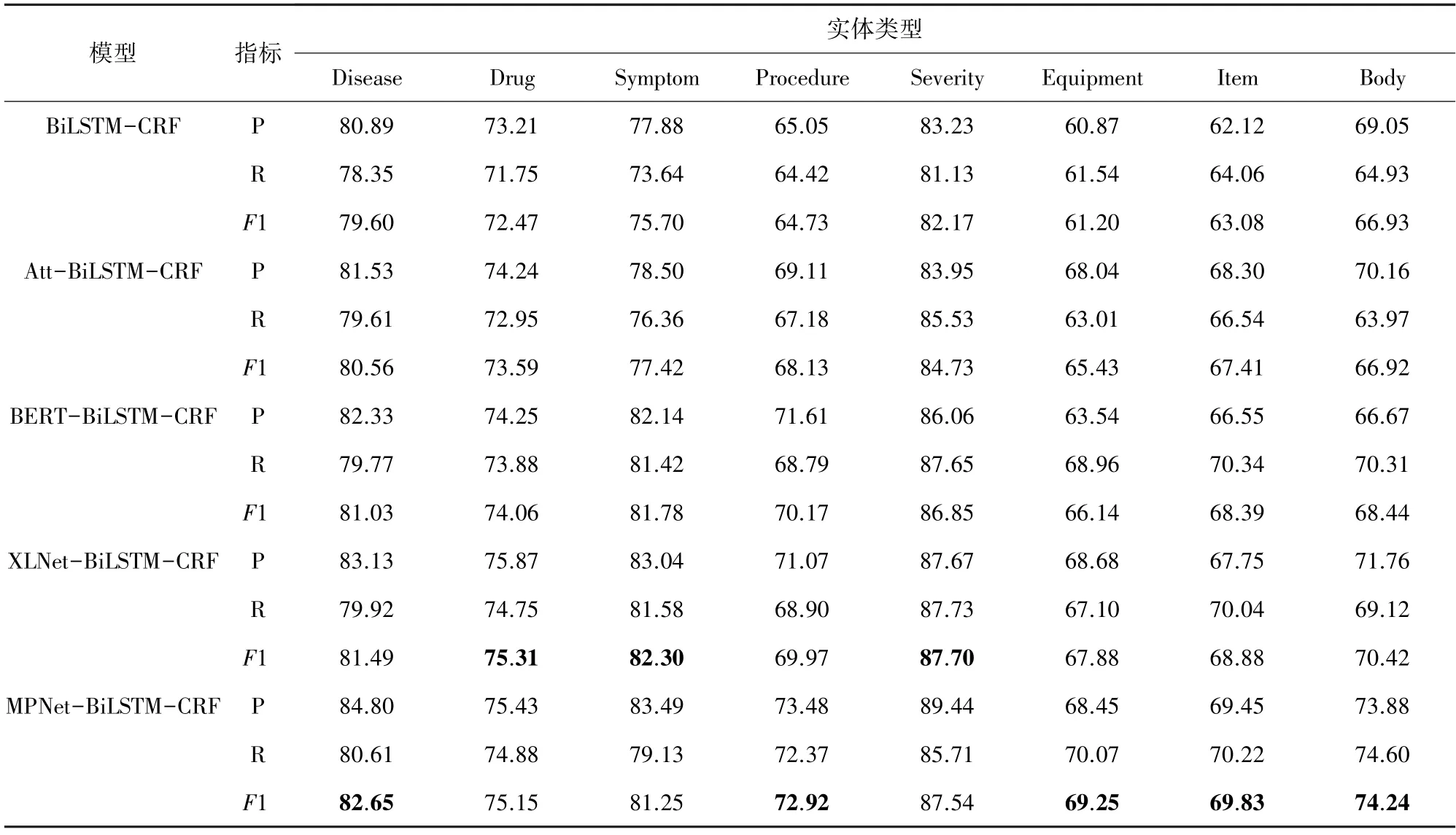
表4 不同实体类型的识别结果Tab.4 Identification results of different entity types
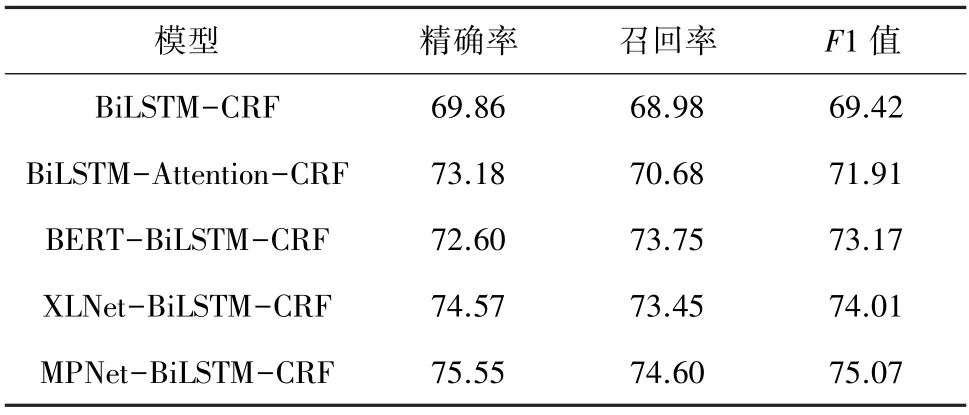
表5 各模型整体对比结果Tab.5 Overall comparison results for each model %
3 结束语
本文提出了一种基于MPNet 和BiLSTM 的医学实体识别模型,联合BiLSTM 网络适应长文本特征提取和CRF 序列标注方法,能适应于COVID-19 临床试验注册文本中新兴医学实体的识别任务。实验设置了多组对比模型以验证本文方法的有效性,结果表明其识别性能优于基准模型以及近年来被广泛研究的基于主流预训练模型的实体识别方法,并且能够较好地实现COVID-19 相关临床医学实体的识别任务,对医学领域相关研究具有一定参考价值。本实验数据仅包含697 份临床试验注册记录中的摘要文本,存在实体种类多但数量不均衡的问题,因此在接下来的工作中,将纳入更多临床文本来丰富语料库,为挖掘COVID-19 临床文本中隐含的医学知识与临床价值做准备。
