甘薯、番茄和拟南芥中丝氨酸羟甲基转移酶基因家族功能研究
2023-01-05赵玉琪周志林姚改芳胡康棣
赵玉琪, 周志林, 唐 君, 姚改芳, 胡康棣, 张 华
(1.合肥工业大学 食品与生物工程学院,安徽 合肥 230601; 2.江苏徐淮地区徐州农业科学研究所,江苏 徐州 221131)
0 引 言
丝氨酸羟甲基转移酶(serine hydroxymethyltransferase,SHMT)是一种保守的磷酸吡哆醛依赖性酶[1],可以催化丝氨酸与甘氨酸之间的可逆转换(丝氨酸+亚甲基四氢叶酸→甘氨酸+N5,N10-亚甲基四氢叶酸+水)[2],这一反应过程提供了一碳单位,而一碳单位是甲基生物合成、核苷酸生物合成以及叶酸和氨基酸代谢所必需的[3-4]。研究表明,SHMT参与植物生长发育以及胁迫的抗性响应,对高等植物的一碳代谢、光呼吸和氧化还原动态平衡也很重要[5-6]。例如,拟南芥中的一个SHMT基因突变导致植株在CO2环境下生长时表现出致死的光呼吸表型[7]。通过对水稻进行转录组分析,发现盐胁迫条件下的SHMT3基因表达增强,同时,过量表达水稻SHMT3(OsSHMT3)的拟南芥转基因植株表现出对盐胁迫的耐受性,表明OsSHMT3在提高植物对盐胁迫的耐受性中的关键作用[1]。此外,在高温胁迫条件下,2个坛紫菜SHMT(PhSHMT)基因均表现为先上调后下调再上调的趋势,表明SHMT基因可能在坛紫菜应答高温胁迫过程中发挥作用[8]。同时,大豆SHMT基因的一个隐性突变可引起细胞死亡,在多种胁迫条件下导致萎黄和坏死组织的形成[9]。强光、盐、干旱等非生物胁迫加重拟南芥atshm1、水稻osshm1突变体缺陷表型,深入研究显示SHMT可以降低非生物胁迫导致的植物细胞内活性氧的水平,减轻了细胞内氧化损伤[10]。水稻中过表达OsSHMT1的植株表现出更高的光合效率和更高的生产力[11]。另外,SHMT可作为除草剂作用的新靶点,当编码含有SHMT肽段的核苷酸序列缺失的植物会表现出生长迟缓或叶片萎黄[12]。此外,植物中SHMT活性已在不同的细胞器中检测到,表明其在不同代谢途径中发挥着广泛的作用[13]。研究表明,豌豆和大麦的叶绿体中检测到SHMT活性,开花植物中都存在质体定位的SHMT。拟南芥有7个编码SHMT蛋白的基因,它们编码的蛋白具有广泛的亚细胞定位,AtSHM1~5的定位已确定,而AtSHM6和AtSHM7基因则需要进一步的证实[14]。研究发现,小麦幼叶中的所有SHMT活性都位于线粒体基质[15],OsSHM1蛋白定位于线粒体[5]。鉴于SHMT蛋白家族保守的生物学功能,系统研究植物中SHMT基因家族功能,对产酶相关化合物的代谢功能和对该基因的基因工程应用具有重要意义。
甘薯是一种具有复杂遗传基础的异源六倍体植物,由于缺乏系统的基因组信息以及遗传资源的匮乏,导致甘薯复杂性状的分子机制研究较为滞后。随着栽培甘薯的2个二倍体野生近缘种基因组序列的完成,甘薯的分子生物学研究大大加快[16]。拟南芥是一种研究植物生理与分子生物学的模式植物,而番茄是研究果实发育的重要植物。SHMT作为一类非常重要而且保守的蛋白,该家族在甘薯潜在的功能尚不清楚。因此,本文通过蛋白同源检索,对甘薯、番茄、拟南芥中的SHMT蛋白家族进行进化树分析、motif分析、蛋白结构域分析、蛋白高级结构分析等,并对蛋白的理化性质及潜在的亚细胞定位进行分析,进一步验证了这一蛋白家族在进化上的保守性,随后通过数据挖掘,分析了番茄中SHMT基因家族在不同组织及果实发育不同阶段的表达水平变化,此外,对模式植物拟南芥SHMT家族在不同胁迫下的应答进行分析,发现AtSHMT在响应胁迫中可能发挥作用。因此,本文通过多种生物信息学分析以及数据挖掘,为甘薯中SHMT基因家族功能的研究奠定基础。
1 材料与方法
1.1 SHMT家族系统进化树分析
利用拟南芥中已经报道SHMT家族成员AtMSA1(At1g36370)的蛋白序列,通过BLAST工具从phytozome在线网站下载拟南芥和番茄中的14条SHMT蛋白序列,其中拟南芥、番茄中各7条,利用Sweetpotato Genomics Resource网站检索甘薯蛋白数据库获得7个潜在的SHMT蛋白序列。利用MEGA7.0软件使用Neighbor-Joining法构建SHMT基因系统发育树。
1.2 SHMT家族motif、结构及理化性质分析
利用MEME网站对甘薯、番茄、拟南芥SHMT蛋白序列进行motif分析。利用SMART网站对甘薯、番茄、拟南芥SHMT蛋白进行结构域分析。利用GSDS 2.0网站,将番茄、拟南芥、甘薯SHMT基因家族的cDNA序列与基因组DNA序列导入,分析SHMT基因家族的外显子和内含子结构。利用EXPASY网站获取甘薯、番茄、拟南芥21个SHMT蛋白的序列长度、等电点、分子量等理化参数。
1.3 SHMT蛋白家族定位和结构预测
利用NLS Mapper网站预测蛋白的核定位信号,随后通过WoLF PSORT网站进一步预测21个SHMT蛋白的亚细胞定位。
利用甘薯、番茄、拟南芥SHMT蛋白家族序列,通过SOPMA和SWISS在线工具,对蛋白的二级结构和三级结构进行预测分析。
1.4 番茄中SHMT基因表达量
利用Solanaceae Genomics Network网站对番茄中的SHMT基因在番茄Micro Tom品种中不同组织和果实发育不同阶段的基因表达进行数据挖掘,并利用基迪奥网站进行热图绘制。
1.5 AtSHMT基因在胁迫条件下的表达分析
利用网站Eplant对拟南芥中SHMT基因在胁迫条件下表达水平进行分析,胁迫条件包括冷害胁迫(4 ℃)、盐胁迫(150 mmol/L NaCl)、热胁迫(38 ℃下处理3 h,随后在25 ℃恢复),并预测基因的潜在功能。
2 结果与分析
2.1 SHMT家族系统进化树分析
将甘薯SHMT(itfSHMT)、番茄SHMT(SlSHMT)、拟南芥SHMT共21条SHMT蛋白序列利用MEGA7.0 软件构建系统发育树,结果如图1所示,从图1可以看出,21个SHMT蛋白在进化树上形成4个分支,其中itfSHMT1、itfSHMT2、SlSHMT1、AtSHMT1、AtSHMT2、itfSHMT3、SlSHMT2在同一个分支上,说明这7个SHMT基因亲缘性较高;AtSHMT3、itfSHMT4、SlSHMT3、SlSHMT4在同一个分支上;AtSHMT4、AtSHMT5、SlSHMT5、itfSHMT5在同一个分支上;AtSHMT6、AtSHMT7、SlSHMT6、SlSHMT7、itfSHMT6、itfSHMT7处于同一个分支。每一个分支上均具有来自甘薯、番茄、拟南芥的SHMT蛋白,表明来自不同物种中SHMT基因可能具有相似的基因功能。

图1 甘薯、番茄和拟南芥中SHMT家族进化树分析
2.2 SHMT家族motif分析
利用MEME网站分析甘薯、番茄、拟南芥中21个SHMT蛋白的motif结构。21个SHMT蛋白的保守motif分布如图2所示,图2中:不同颜色的色条代表不同的保守基序;下方数字表示蛋白的氨基酸数目。
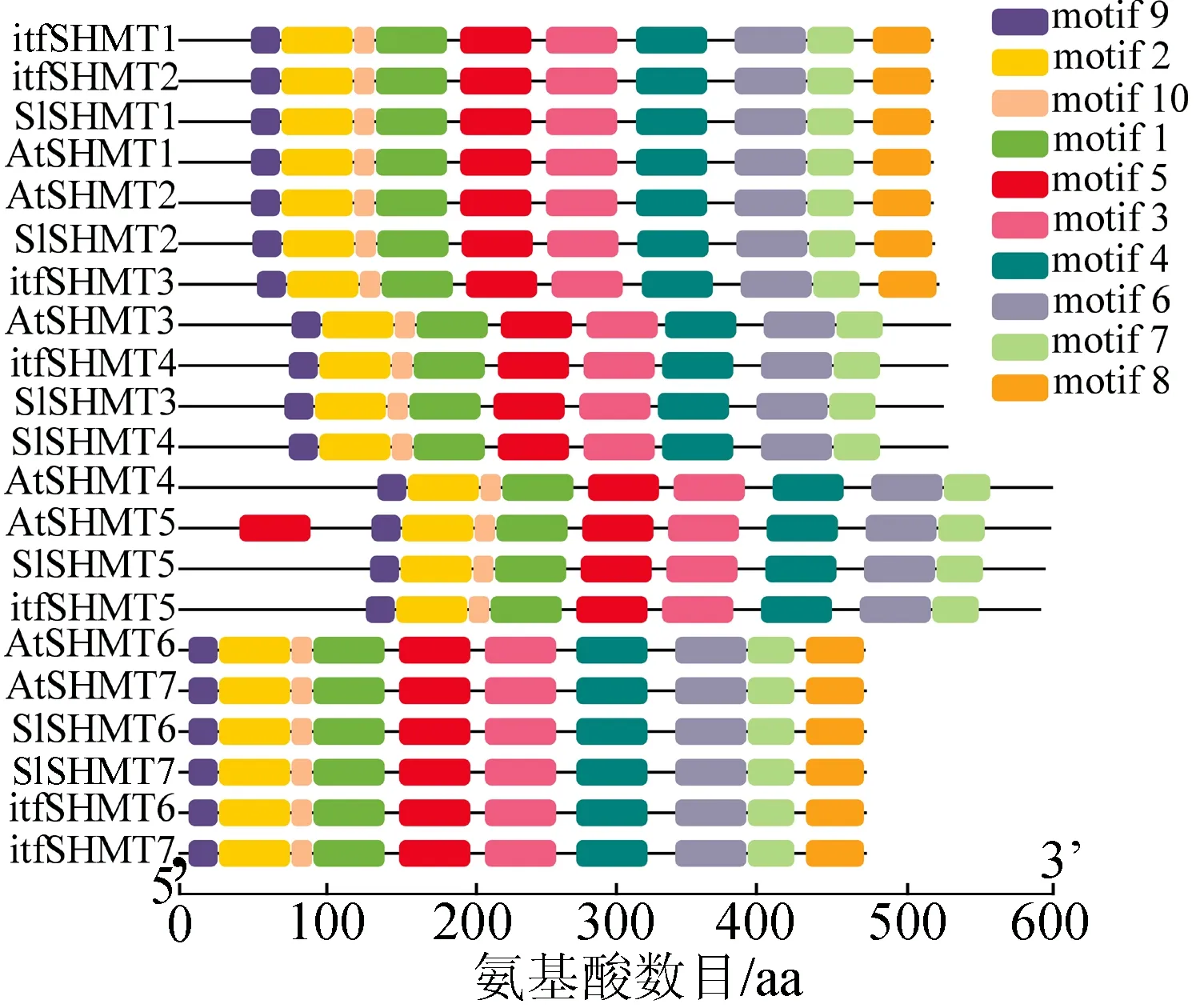
图2 甘薯、番茄、拟南芥中SHMT蛋白motif分析
从图2可以看出,进化树中分支1,即itfSHMT1、itfSHMT2、SlSHMT1、AtSHMT1、AtSHMT2、itfSHMT3、SlSHMT2具有相同的motif组成和排列顺序,这与基因家族进化树的结果相吻合;分支2中的SlSHMT3、SlSHMT4、AtSHMT3与itfSHMT4的motif结构也很相似,与进化树结果一致;分支3中的AtSHMT5虽然相比SlSHMT5、itfSHMT5、AtSHMT4多了一个motif5,但其他motif位置都保持一致;分支4中的AtSHMT6、AtSHMT7,SlSHMT6、SlSHMT7以及itfSHMT6、itfSHMT7的motif数量及排列均比较一致,表明它们可能具有保守的基因功能。蛋白motif结果分析表明,进化树中的4个分支分别具有相对保守的蛋白motif结构,说明motif分析结果与进化树的一致性。
2.3 SHMT蛋白家族结构域分析
利用SMART网站对甘薯、番茄、拟南芥中21个SHMT蛋白的结构域进行分析,结果如图3所示。
从图3可以看出,21个蛋白都有典型的SHMT结构域。
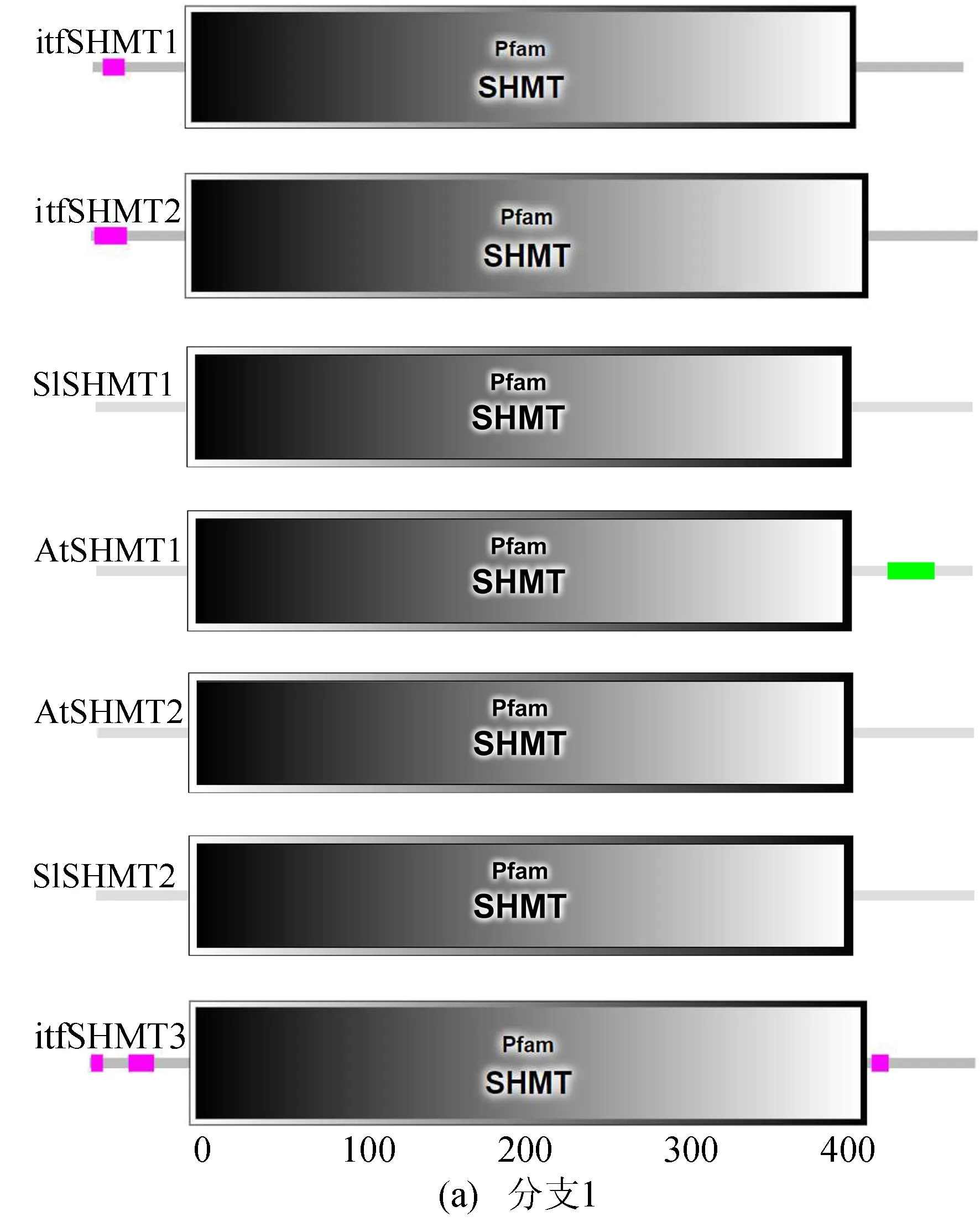
图3 甘薯、番茄、拟南芥中SHMT蛋白结构域分析
从图3a可以看出,与进化树相对应的分支1中itfSHMT1、itfSHMT2、SlSHMT1、AtSHMT1、AtSHMT2、SlSHMT2、itfSHMT3的SHMT结构域共有398个氨基酸,其中SlSHMT2的SHMT结构域位于第56~454个氨基酸之间,从图3b可以看出,itfSHMT3的SHMT结构域位于第59~457个氨基酸之间,该分支其余5个蛋白结构域位于第55~453个氨基酸之间;进化树分支2中AtSHMT3、itfSHMT4、SlSHMT3、SlSHMT4的SHMT结构域共有390个氨基酸,其中AtSHMT3的SHMT结构域位于第83~473个氨基酸之间,itfSHMT4、SlSHMT4的SHMT结构域位于第81~471个氨基酸之间,SlSHMT3结构域位于第78~468个氨基酸之间;从图3c可以看出,进化树分支3中AtSHMT4、AtSHMT5、SlSHMT5、itfSHMT5的SHMT结构域共有405个氨基酸,它们的结构域分别位于第142~547个、第138~543个、第137~542个、第134~539个氨基酸之间;从图3d可以看出,进化树分支4中AtSHMT6,AtSHMT7、SlSHMT6、SlSHMT7、itfSHMT6、itfSHMT7的结构域共有400个氨基酸,结构域皆位于第12~412个氨基酸之间。由上述分析可以看出,甘薯、拟南芥、番茄当中的SHMT蛋白结构域约由400个氨基酸残基构成,且同源性越高的蛋白结构域位置及氨基酸数目越相似。
2.4 SHMT基因家族结构分析
利用GSDS 2.0在线软件分析甘薯、番茄、拟南芥中21个SHMT基因结构中内含子和外显子的分布情况,如图4所示。

图4 甘薯、番茄、拟南芥中SHMT基因结构分析
图4中:蓝色代表基因上游或下游的非编码序列;黄色代表基因的外显子区域;实线代表基因的内含子区域。
从图4可以看出,进化树分支1中相对应的itfSHMT1、itfSHMT2、SlSHMT1、AtSHMT1、AtSHMT2、itfSHMT3、SISHMT2有15个外显子和14个内含子;分支2中AtSHMT3、itfSHMT4、SlSHMT3、SlSHMT4有11个外显子和10个内含子;分支3中AtSHMT4、AtSHMT5、SlSHMT5、itfSHMT5有4个外显子和3个内含子;分支4除了AtSHMT6只有3个外显子2个内含子外,AtSHMT7、SlSHMT6、SlSHMT7、itfSHMT6、itfSHMT7均具有4个外显子3个内含子。由上述分析可知,进化树上处于同一分支的SHMT基因亚家族,具有相似数目的外显子和内含子,进一步说明SHMT基因在不同物种间具有保守性。
2.5 SHMT蛋白家族理化性质分析
对甘薯、番茄、拟南芥中的21个SHMT氨基酸序列进行理化分析结果,见表1所列。
由表1可知,进化树分支1中相对应的itfSHMT1、itfSHMT2、SlSHMT1、AtSHMT1、AtSHMT2、itfSHMT3、SISHMT2蛋白理化性质相近,氨基酸数目最多相差4个氨基酸残基,蛋白分子量相差不到1 000 g/mol,pI均为碱性;分支2中AtSHMT3、itfSHMT4、SlSHMT3、SlSHMT4、氨基酸数目、蛋白分子量差异均不大,pI值均为碱性,但AtSHMT3的pI明显高于其他3个蛋白。
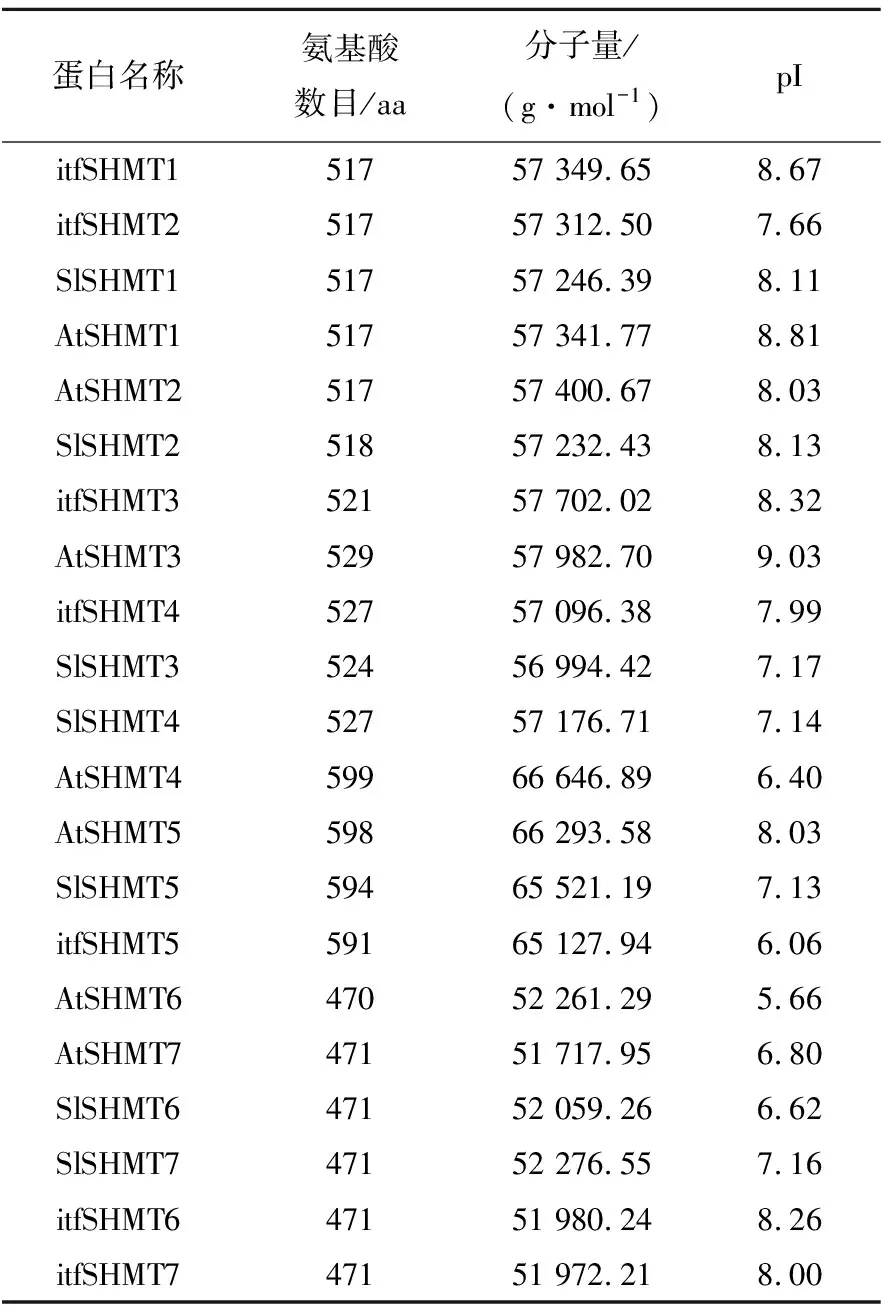
表1 番茄、甘薯、拟南芥中SHMT蛋白的理化性质
由表1还可知,分支3中AtSHMT4、AtSHMT5、SlSHMT5、itfSHMT5、氨基酸数目、蛋白分子量皆高于另外3个分支的基因或蛋白,pI值也各有不同,位于6.40~8.03之间;分支4中,AtSHMT6比AtSHMT7、SlSHMT6、SlSHMT7、itfSHMT6、itfSHMT7氨基酸数目仅少1个氨基酸残基,蛋白分子量和pI值也有略微差异。由上述分析可知,进化树上处于同一分支的SHMT蛋白具有相似的理化性质,进一步说明SHMT蛋白理化性质在不同物种间具有保守性。
2.6 SHMT蛋白家族亚细胞定位预测
SHMT基因的核定位序列(nuclear localization sequence,NLS)及亚细胞定位分析,结果见表2所列。通过NLS Mapper网站预测,其中拟南芥SHMT基因中有3个含有潜在的NLS,番茄SHMT基因只有SlSHMT5含有潜在的NLS,甘薯SHMT基因中只有itfSHMT1和itfSHMT5含有潜在的NLS,说明小部分SHMT蛋白可能在细胞核中发挥作用。通过亚细胞定位预测网站发现绝大多数SHMT可能在线粒体和叶绿体发挥主要作用,有的SHMT蛋白还可以在细胞核、内质网、质体、过氧化物酶体、细胞质、细胞骨架发挥作用;甚至AtSHMT4、AtSHMT2、itfSHMT4蛋白可能同时具有不同的亚细胞定位,进而行使自身的功能。

表2 番茄、甘薯、拟南芥中SHMT蛋白的核定位序列及亚细胞定位预测
2.7 SHMT蛋白高级结构预测
利用SOPMA网站对甘薯、番茄、拟南芥SHMT蛋白序列的二级结构进行预测分析,如图5所示。图5中:蓝色代表α-螺旋;紫色代表无规则卷曲;红色代表延伸链;绿色代表β-转角。SHMT蛋白主要由α-螺旋和无规则卷曲构成。其中,拟南芥、甘薯、番茄中SHMT蛋白构成α-螺旋占比分别为38.63%~47.78%、40.10%~46.81%、40.07%~46.91%;β-转角分别占比5.10%~6.00%、5.73%~6.72%、5.73%~6.72%;延伸链分别占比11.70%~16.19%、11.91%~13.85%、11.89%~15.32%;最后无规则卷曲占比分别为35.03%~41.47%、33.97%~42.64%、33.01%~38.72%。由图5可知,处于同一分支的蛋白具有较为相似的二级结构排布,表明在蛋白二级结构上的保守性。
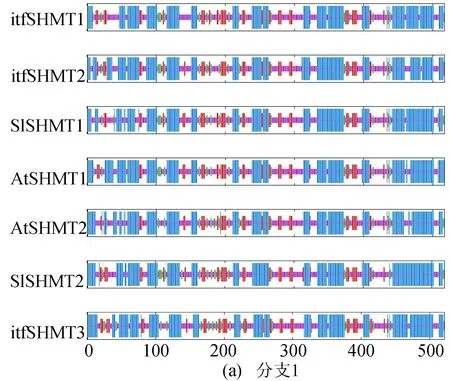
图5 甘薯、番茄、拟南芥中SHMT蛋白二级结构分析
为研究甘薯、番茄、拟南芥中的21个SHMT蛋白在三维结构上的保守性,利用SWISS网站在线构建SHMT的甘薯、番茄、拟南芥三维结构模型,如图6所示。
从图6可以看出,3个物种的蛋白三维结构基本分为4种构象,其中进化树分支1中itfSHMT1、itfSHMT2、SlSHMT1、AtSHMT1、AtSHMT2;itfSHMT3、SlSHMT2蛋白三维结构相似;分支2中AtSHMT3、itfSHMT4、SlSHMT4、SlSHMT3构象相近;分支3中AtSHMT4、AtSHMT5、SlSHMT5、itfSHMT5构象相近;分支4中itfSHMT6、itfSHMT7、SlSHMT6、SlSHMT7、AtSHMT6、AtSHMT7具有相似的构象。这与进化树分析结果比较吻合,结果表明,甘薯、拟南芥、番茄中SHMT蛋白具有高度保守的三级结构。
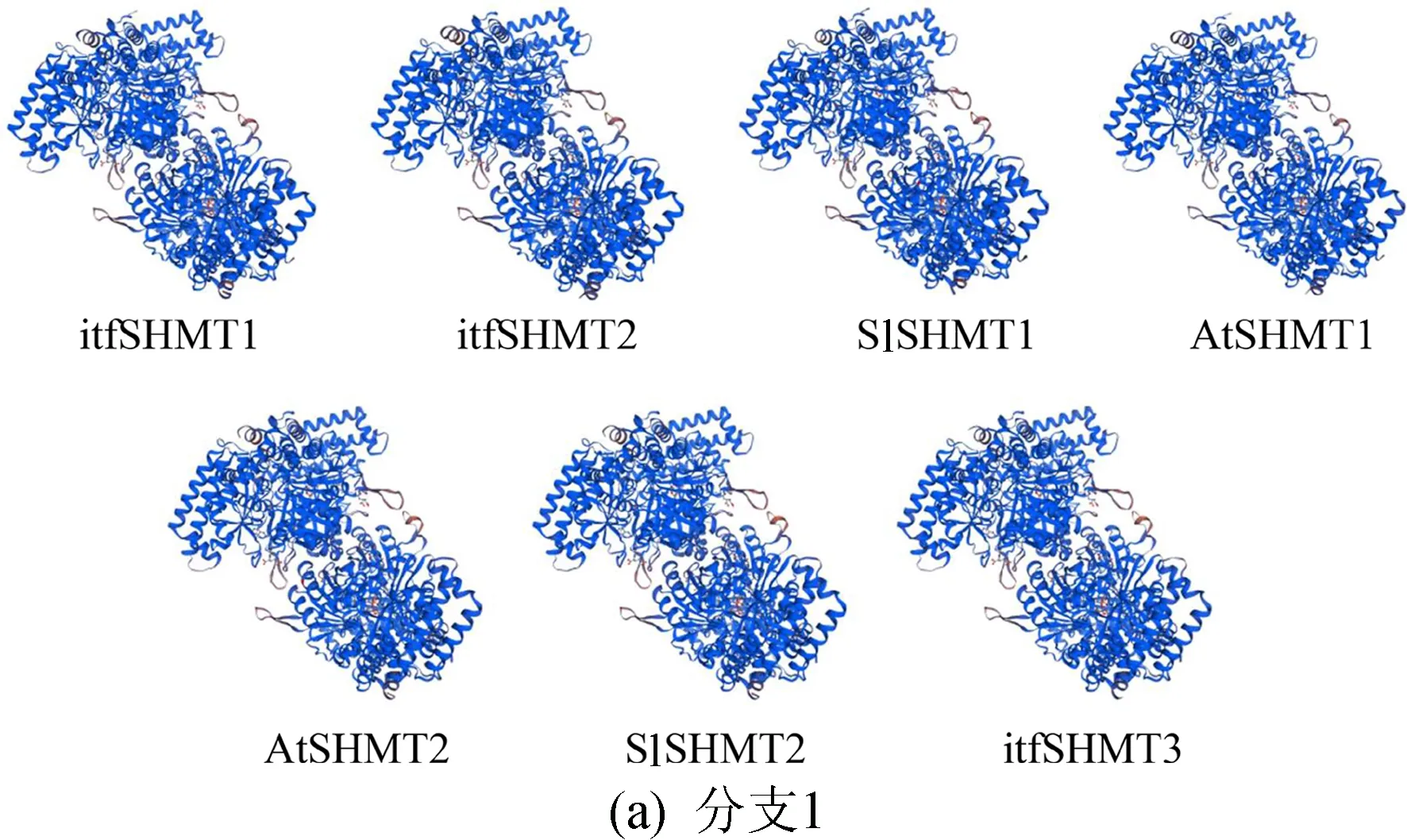
图6 甘薯、番茄、拟南芥中SHMT蛋白三维结构分析
2.8 番茄中SHMT基因的组织表达水平分析
利用Solanaceae Genomics Network网站和基迪奥网站分析番茄中SHMT基因的组织表达水平,为甘薯中该基因家族的功能研究提供参考。番茄中7个SHMT基因在番茄不同组织及果实不同发育阶段的表达量,如图7所示。
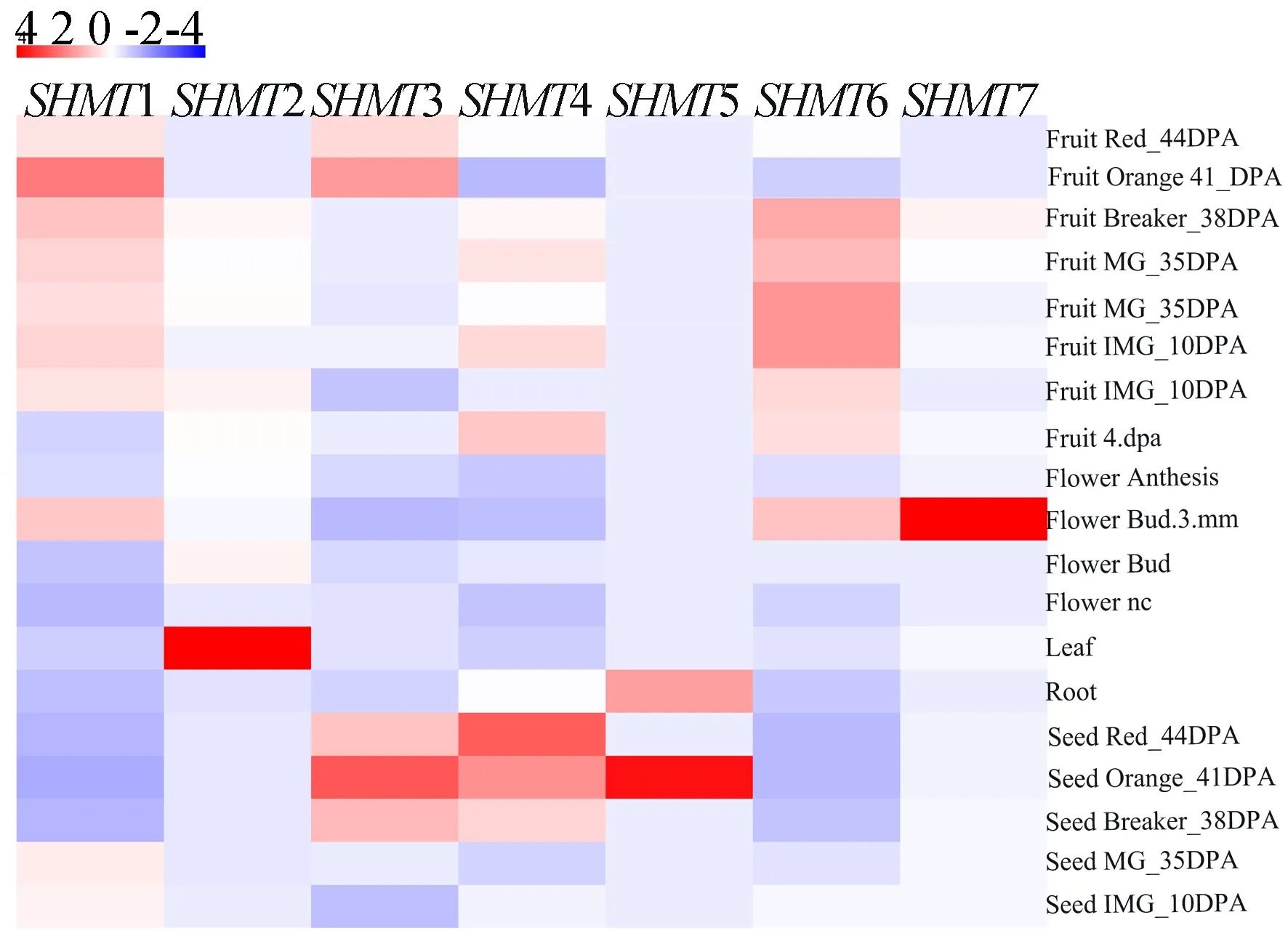
图7 番茄中SHMT基因在不同组织中的表达量热图分析
图7中,DPA代表开花后天数。由图7可知,SlSHMT1和SlSHMT6在番茄种子、叶、根中表达量较低,但在花及不同时期的果实中表达量显著上升,说明这2个基因可能在番茄开花、果实成熟过程中发挥了重要作用;SlSHMT2、SlSHMT5、SlSHMT7分别在叶片、种子、花中表达量偏高,而在其他组织中几乎检测不到,体现了这3个基因表达的组织特异性,表明这3个基因可能参与番茄光合作用、种子萌发及开花过程的调控;SlSHMT3、SlSHMT4的表达量与种子发育过程呈现明显的正相关,说明它们可能参与番茄种子中胚的发育,同时两者与果实成熟也具有一定的关联。
2.9 AtSHMT在胁迫条件下的基因表达量
鉴于SHMT基因家族在植物中的保守性,本文分析了拟南芥中7个SHMT基因在冷、热及盐胁迫下基因表达变化,如图8所示,从图8可以看出,在冷胁迫中,只有AtSHMT5在胁迫6 h后表达量呈现上升趋势,对冷胁迫响应明显;盐胁迫中AtSHMT1在根中表达量下降,AtSHMT5表达量在3~24 h明显高于对照组,表明AtSHMT1可能参与盐胁迫的负调控,而AtSHMT5则可能参与正调控;热胁迫条件下,AtSHMT1在根和幼苗中表达量急剧上升又急剧下降到正常水平,AtSHMT3在6 h时表达量急剧上升,24 h又恢复到正常水平,AtHMT6在热胁迫过程中表达量一直低于对照组,说明AtHMT1、AtHMT3、AtHMT6可能参与了拟南芥对热胁迫的反应。综合上述可知,在热胁迫中AtSHMT3、AtSHMT6表达量明显上调,表明AtSHMT3、AtSHMT6在拟南芥抵御高温时的潜在作用;而AtSHMT5在冷胁迫中表达量上调明显,表明该基因可能参与拟南芥冷害胁迫的调控。
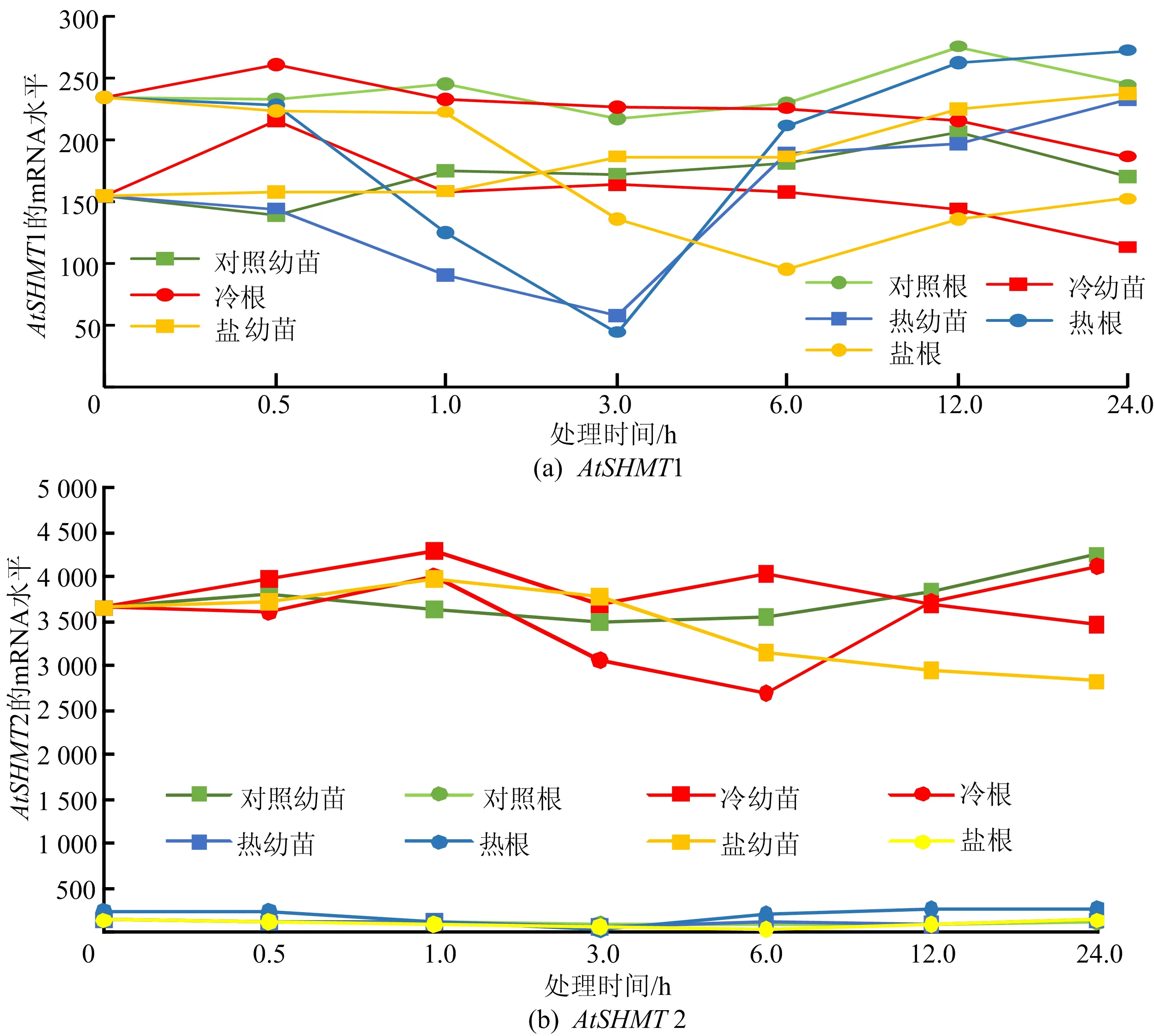
图8 拟南芥中不同胁迫条件下各SHMT基因的表达量分析
3 结 论
SHMT在植物生长中的重要作用不言而喻,迄今为止对SHMT的研究依旧是生命科学领域重要的课题,不少相关领域的学者已经对SHMT的功能做了深入的探索和研究。然而,SHMT在甘薯和番茄中的功能尚不可知,因此,本文以甘薯、番茄、拟南芥SHMT基因家族为研究对象,通过进化树分析、基因结构域分析、motif分析、蛋白的二级结构及三级结构预测,发现SHMT蛋白家族具有较高的保守性,并且发现蛋白结构信息和进化树的分支情况,具有显著的一致性。随后通过理化性质分析、核定位序列预测、亚细胞定位预测等,发现部分SHMT具有广泛的亚细胞定位。通过对番茄中7个SHMT基因进行表达量分析,研究SHMT基因家族在番茄生长发育中的功能和作用。例如,SlSHMT1和SlSHMT6可能在番茄开花、果实成熟过程中发挥了重要作用;SlSHMT2、SlSHMT5、SlSHMT7可能参与番茄光合作用、种子萌发及开花过程的调控;SlSHMT3、SlSHMT4可能参与番茄种子中胚的发育,同时两者与果实成熟也具有一定的关联。通过获取不同胁迫(冷害、高温及盐胁迫)下拟南芥中7个SHMT的基因表达量,分析SHMT在拟南芥中可能参与多种胁迫。例如,AtSHMT3、AtSHMT6在拟南芥抵御高温时可能有潜在作用;而AtSHMT5可能参与拟南芥冷害胁迫的调控。因此,通过对甘薯、番茄、拟南芥中的21个SHMT基因的生物信息学分析及表达数据挖掘,归纳出特定SHMT基因可能具有的功能,对深入研究SHMT基因在甘薯、番茄、拟南芥中的生物功能具有重要作用。
