基于环境DNA的日本河流生物多样性文献计量分析及其进展
2022-11-25刘泽军胡茂川刘丙军郝爱民井芹宁
贺 凯,刘泽军,胡茂川,刘丙军,郝爱民,井芹宁
(1. 中山大学 土木工程学院,广东 珠海 519082;2. 广东省环境资源利用与保护重点实验室,广州 510640;3. 温州大学 生命与环境科学学院,浙江 温州 325035;4. 亚热带水环境生态保护浙江省国际科技合作基地,浙江 温州 325035)
0 前 言
传统水生生物监测主要依靠观测者对物种进行形态学鉴定,因而对从事物种鉴定人员的专业知识要求较高,同时传统的水生生物监测方法可能会给水生生物或者其所在的水生生态系统带来一定程度的危害导致其具有一定的局限性[1]。而环境DNA技术作为新兴的水生生物监测方法,基于当前基因测序技术,可以通过检测游离在水环境中的DNA分子检测出对应生物种类的存在。其相较于传统水生生物形态学监测方法而言具有经济、高效、准确、灵敏等优势[2]。因此,近年来环境DNA检测技术发展迅速,被广泛应用于流域入侵物种、濒危物种监测、生物量评估和生物多样性调查等[3-5]。
环境DNA指在自然环境中广泛分布的游离DNA分子,主要通过生物脱落的皮肤、唾液、粪便、配子、分泌物等方式获得[6]。环境DNA分子大小不一,存在部分大于180 μm或小于0.2 μm的分子,DNA分子的大小可能与生物种类相关。运用环境DNA的水生生物调查研究始于1987年,由美国田纳西大学研究团队开发从环境(湖泊沉积物)中提取和纯化细胞外DNA的方法[7],随后法国Ficetola等[8]通过针对栖息地池塘中提取牛蛙(Ranacatesbeiana)DNA信息,以此研究牛蛙栖息习惯。Minamoto等首次运用环境DNA宏条形码技术于鱼类、两栖动物、鸟类和哺乳动物等水环境生物监测研究[9]。Doi等也通过对鲤鱼环境DNA分析检测来证明环境DNA宏条形码技术在鱼类乃至水生生物的定量评估中的潜力[10-11]。 Miya开发具有代表性的通用引物“MiFish”用于环境DNA宏条形码技术,目前该引物可检测到200多个鱼种[12-14]。日本通过出版环境DNA研究和分析手册等[15-16]指导环境DNA技术应用研究,并建立基于大规模、高分辨率数据的生态系统解释、预测和保护系统。
日本开展环境DNA研究较早,有着广泛的应用案例。近年环境DNA研究也被引导强化同其他研究领域的融合与合作,如生态系统的可持续利用和环境保护。本文在系统介绍环境DNA技术及其影响因素的基础上,梳理日本运用环境DNA技术在河流生态监测与多样性评价方面的研究成果,旨在为中国未来生态监测和生物多样性研究方面提供素材和借鉴。
1 日本环境DNA技术的发展历程
日本在环境DNA技术的研究起步较早,Minamoto等[9]在对水族箱水样进行DNA提取和分析后,确认所有水族箱鱼种都被检出。根据水族箱分析结果对3个自然河道现场采集的水样开展环境DNA分析,并检出自然河道中4个鱼种,检测结果同以往常规方法(观察和捕捉方法)确定的鱼种一致。环境DNA方法可以提供简化的鱼类等水生生物调查和监测,日本科学家在之后的研究中成功利用环境DNA技术检出日本淡水湖中鱼类的物种组成,证明该方法在日本水环境中鱼类调查研究中的适用性。Takahara等[10]利用环境DNA技术在室内水箱和室外实验池中发现鲤鱼(CyprinuscarpioL.)丰度与水中环境DNA浓度之间存在正相关关系,并利用环境DNA对天然潟湖中的鲤鱼生物量进行评估。从2008年至今,环境DNA技术在水生生态系统的研究经历了定性到定量,由检测单一物种到同时检测多种物种,从调查淡水水域扩展到海洋,调查物种从小型底栖动物扩展到两栖动物、哺乳动物等的发展历程[17-20]。
为研究日本环境DNA技术发展历程运用CiteSpace[21]软件对日本相关文献进行关键词聚类分析,数据库为Web of Science (WOS)核心数据库(2022年6月28日),搜索策略为TS=((“environmental DNA”OR “eDNA”) AND (Japan OR Japanese)),共搜索文献141篇。历年文献发表数量及年被引次数见图1,从图中可以看出环境DNA发文数量及年被引用次数均呈逐年上涨的趋势,而发展突变点出现于2013—2016年,表明在该时间段环境DNA开始步入大众科研关注领域。
2 环境DNA技术流程介绍及其影响因素
环境DNA技术分析主要包括样品采集、样品处理、DNA提取、PCR扩增、基因测序、生物信息学以及统计学分析。具体技术处理流程如图2所示。

图1 日本环境DNA发文数量及年被引次数

图2 环境DNA技术处理流程
Jo等以环境DNA的生物放大作用,以马鲛鱼(Trachurusjaponicus)为标本,对不同水温和鱼类生物量条件下的DNA脱落和降解过程进行实验,发现降解过程随环境DNA大小发生变化,同时环境DNA浓度及其降解率也随着水温的升高和生物量增加而增加[22]。溝口等利用环境DNA技术对海上风电设施附近开展鱼类监测,用来识别其周围水域的鱼种,对渔业影响评估方法的改善有很大的促进作用[23]。Kabamoto等按照灌溉季节和非灌溉季节在全国10个农业改良项目区的122个地点开展采样,通过环境DNA技术在农业水道中检出15~40种鱼类,通过比较采样调查和以往调查数据结果,评估环境DNA技术在快速流动水环境(如农业水道等)中水生态调查的适用性并讨论检测假阳性等技术问题[24]。需要注意的是,其研究强调人类活动影响下环境DNA污染问题,如检测鱼DNA检测分析中存在实验室污染等。
聚合酶链式反应(PCR)是一种体外核酸扩增技术,用于放大与扩增特定的DNA片段。因此,确定要扩增的DNA片段对后续的eDNA测序分析具有至关重要的作用。在理想情况为了成功地进行eDNA测序分析,PCR引物对和扩增出的中间eDNA片段应满足以下要求:① 稳定性,设计的通用PCR引物需要无偏差扩增各目标类群的基因片段,且不出现基因片段缺失;② 一致性,扩增的片段应包含足够的变异以允许明确的分类分配;③ 特异性,引物应是目标类群的特定引物(如MiFish)。否则,来自非目标分类群的DNA(原核生物和非目标真核生物的DNA)将在扩增产物中占主导地位。根据文献调研对通用PCR引物进行统计发现(见图3),MiFish引物占比最多(51.2%),其次是Cyt b引物(34.9%),日本常见鱼类环境DNA引物(部分)基因序列表如表1所示。这些通用引物,不仅在日本,在我国生物多样性研究中也开展了相关应用研究[25-26]。从日本环境DNA研究地理分布图上看,MiFish引物在日本全国的应用案例较多,最近的日本政府调查项目也主要采用MiFish引物开展eDNA分析进行生物多样性监测[27]。例如:日本国土交通省开展全国河流环境普查试点研究[28];日本环境省生物多样性中心推动环境DNA标准化项目(https://www.biodic.go.jp/edna/edna_top.html)。而Cyt b引物则是在以濑户内海为主的西日本地区应用较为广泛(见图4)。

图3 日本环境DNA发表论文引物频率组成
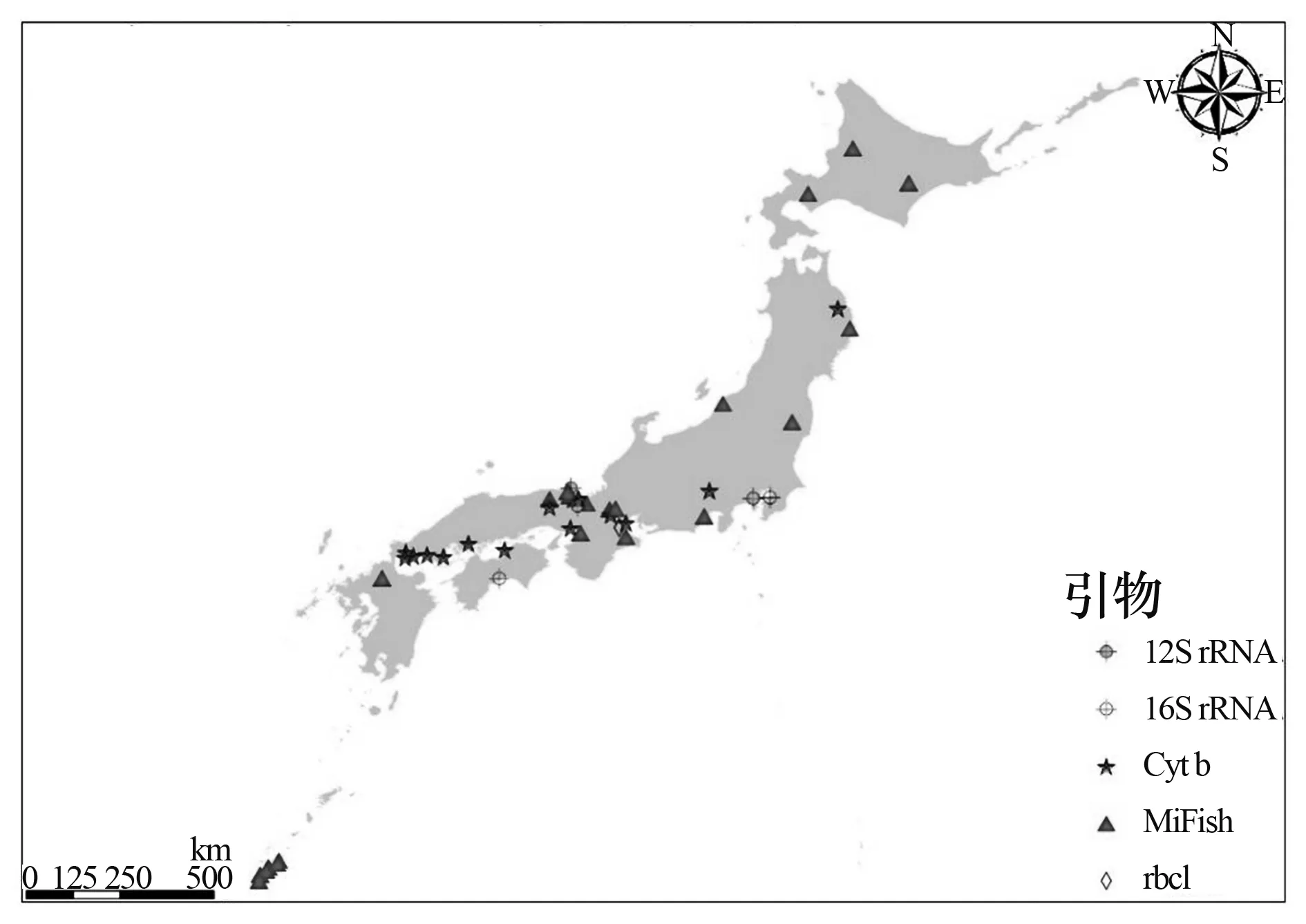
图4 日本环境DNA研究地理分布

表1 日本常见鱼类环境DNA引物(部分)基因序列表
3 环境DNA技术在日本河流生物多样性研究中的应用
随着环境DNA技术的不断发展和基因库的不断完善,越来越多研究人员运用环境DNA技术开展水生生物的物种鉴定。同时由于外来入侵物种、濒危物种和稀有物种的监测调查一直是传统生物监测研究的难题,环境DNA技术为上述问题的解决提供了一种新的思路。本部分针对生物多样性调查、外来入侵物种的监测、濒危物种和稀有物种的监测以及生物量评估方面的环境DNA研究及其技术应用进行介绍梳理。
通过Web of Science搜索文献导入CiteSpace软件,去重后剩余140篇有效文献,文献研究时间段为2005—2022年,本次研究以3 a为时间间隔,选取每年引用频率前25的关键词进行聚类分析,共得到15类聚类(单一聚类关键词小于5类时不显示),有效聚类9类(0号 影响因素、1号 浮游植物、2号 日本鳗鱼、3 号日本竹荚鱼、5号 日本鳗鲡、7号栖息地相关、8号 海洋、9号 大肠杆菌 14号、鉴别),由于当前研究处于发展阶段,文献较少,无法进行关键词突现性研究,本次研究以不同年间关键词共现作为趋势研究,相关聚类见图5(a)(2008—2010年)、图5(b)(2020—2022年),其中圆形大小表示关键词出现次数,同一圆形不同颜色环形为不同年份出现次数,连线表示关键词存在共同出现,粗连线表示a、b特定年间关键词存在共同出现。从图中可以看出,除环境DNA,qPCR等纲领性关键词外,以生物质、丰度、河流、鱼类、数量、濒危物种、洄游、温度、降解、琵琶湖等词出现频率较高,表明当前环境DNA研究更多关注于物种(多指鱼类)丰度、濒危物种以及季节性物种的监测领域,对于实验条件的关注则集中在温度以及引物、环境DNA的降解速度等领域。图5(a)(2008—2010年)显示日本早期环境DNA技术主要以基因宏条形码技术、qPCR扩增为主,并开始应用于琵琶湖流域生物监测。从图5(b)(2020—2022年)可以看出当前日本环境DNA技术应用开始全面扩宽。在研究区域上,由传统河湖研究迈向海洋;在技术研究上开始深入引物设计,引物种类得到扩充,对实验最优条件也进行了一定的探索;在技术应用上从种群监测步向多元研究,在物种多样性保护、濒危物种、入侵物种监测、季节性洄游鱼类监测等领域均进行了较多研究。关键词聚类表明当前日本对环境DNA技术的研究已进入较深入阶段。

图5 日本环境DNA关键词聚类分析
3.1 生物多样性调查
生物多样性的有效保护对于人类的生存和生态系统的维持至关重要。生物多样性评估基本上依赖于物种监测,但传统的物种形态学监测难以满足生物多样性研究要求。因此,通过从环境样本中提取DNA来获取物种、种群和群落信息的方法能够弥补传统物种鉴定的不足。在鱼类多样性调查中,环境DNA宏条形码方法作为一种全面检测鱼类等水生生物鉴定技术被提出。Miya等[12]开发的通用PCR 引物组(MiFsh-U/E)目前已在全球范围内广泛使用。该引物是根据880个物种的线粒体完整基因组序列和160个板鳃亚类(鲨鱼和鳐鱼)物种的部分线粒体序列,针对12S rRNA基因的长可变区(163~185 bp)设计,检出实验室180种海洋鱼类中的168种(93.3%),并在附近的珊瑚礁调查中也检测到93种。
在其他水生生物多样性调查中,Kayano等通过检测附着板中的环境DNA,表明淡水珍珠牡蛎在河流中的大量存在对水生环境中的附着微动物种群产生影响,并影响河流中氮的迁移转化[31]。Narumi等人使用线粒体12s rRNA基因靶点的定量PCR技术对沉积物中的水蚤(Daphniagaleata)的DNA浓度进行检测,并量化上述物种在水中的生物量[32]。Tateishi等通过研究日本绒螯蟹蜕皮对环境DNA释放的影响,揭示了日本绒螯蟹(Eriocheirjaponica)的分布和季节性变化,发现其幼体在海水中生长,成熟体栖息在淡水区的栖息规律。Aratani等通过开发龙虱科(Dytiscidae)和蝎蝽科(Nepidae)的环境DNA引物集,用于保护列入世界自然遗产的奄美大岛等地的水生昆虫,并评估当地的生物多样性种群特征[33]。Hirai对太平洋中的海洋浮游生物进行了全面的宏条形码分析,而这些浮游生物未成熟的个体很难通过形态学来识别,研究重点是占主导地位和种群高度多样化的尾鳍动物等浮游生物,运用环境DNA宏条形码技术评估尾鳍动物的多样性和群落结构[34]。此外,环境DNA技术也被应用于重要渔业物种的饮食分析和浮游生物监测评估食物网结构和生态系统。菅原建立一种专门检测贻贝的环境DNA方法,并将其应用于湖泊环境的监测调查[35]。池田专注于mtDNA、16s rRNA和12s rRNA区域,并开发相关引物集用于检测水生昆虫,并提出黄嘴蜉蝣(Epeorusaesculus)存在未知亚种的可能。Sakata开发并应用了两栖动物的通用引物[36]。Akatsuka等调查高悬浮物条件下的水样采集方法,以确定海草床的变化,并在入海口开展海带鳗鲡(Zosterajaponicaeelgrass)群落及其生物量的数值计算。同时结果表明,受目标习性和繁殖期影响,水样采集的适应时间因地而异,对生物多样性实地调查具有重要作用[37]。Yonezawa等通过水中的环境DNA揭示了栖息在水边并以水中的鱼和水生昆虫为食的亚洲水鼩(Chimarrogaleplatycephalus)的栖息地分布[38]。 此外,Ishige等分析马来西亚婆罗洲盐池水样的环境DNA,发现猩猩(Pongopygmaeus)、爪哇野牛(Bosjavanicuslowi)、亚洲象(Elephasmaximus)等多种动物,均显示了环境DNA同传统形态学观察监测相比的优越性[39]。Ushio等使用新开发的MiBird引物在从动物园鸟笼的饮水区采集的样本检测出鸟类的环境DNA[13]。
3.2 外来入侵物种的监测
外来物种入侵是指外来种由原生地经自然或人为途径进入另一个生态环境,并在该生态环境系统中定居、自行繁殖建群和扩散而逐渐占领新栖息地的一种生态现象[40-42]。环境DNA技术可以促进对水生生物入侵物种的检测和监测,促进入侵物种的监测和预防工作开展。Takahara等利用环境DNA技术对入侵的蓝鳃太阳鱼(Lepomismacrochirus)环境DNA进行了检测,发现蓝鳃太阳鱼入侵情况在人类活动的影响较少的岛屿池塘中较轻,具有比传统观察方法更好的生物入侵检测率[43]。Maruyama等发现入侵物种蓝鳃太阳鱼中较小的个体每单位体重释放更多的DNA到水中,从而影响环境DNA浓度估算生物量和种群密度的结果[44]。此外,Uchii等研究结果表明,根据线粒体DNA的单核苷酸差异可以区分同一物种内非本地基因转移,并量化鲤鱼中本地和非本地DNA的相对比例,进而开发量化基于单核苷酸差异的本地和非本地DNA的相对比例的检测方法[44]。Mizumoto等人通过环境DNA手段发现日本蟾蜍(Bufojaponicusformosusk)的繁殖区广泛分布在北海道的12条河流的32个地点,由于其毒性而影响本地物种的生存,但其夜间的性质和隐藏习惯导致其很难被发现和消除[45]。同时Jo等人对多个池塘样本采用多重实时PCR方法,发现不同季节的3种非本地鱼种(Lepomismacrochirus;蓝鳃鱼,Micropterussalmoides;大嘴鲈和Micropterusdolomieu:大口黑鲈)和3种濒临灭绝的本地鱼类(Hemigrammocyprisrasborella:间纹鲤;Oryziaslatipes:日本青鳉;Misgurnusanguillicaudatus:泥鳅)在不同季节检出率不同。对其分布情况进行评估,总体结果显示夏季的环境DNA检测率较高,并发现非本地物种在人类活动影响大、池塘表面积大、pH值高的池塘中更容易被检出,而本地物种在非本地物种检测率高的池塘中检出率较低[47]。Fkumoto等使用环境DNA来确定大鲵(Andriasjaponicus)和中国大鲵(Andriasdavidianus)的分布,这是一个通过入侵并与本地物种杂交影响本地物种栖息地的入侵物种,根据其活动周期等确定相应的调查时期[48]。Yamakawa和Miya修改MiFish 引物的部分序列,用于检测沼泽中的非本地木龟(Chelydraserpentina),并正在开发MiTurtle引物集,其序列对淡水龟类具有高度特异性。在研究区域除了非本地木龟(C.serpentina),还发现了日本石龟(Pelodiscussinensisver.japonica)、棱皮龟(Mauremysreevesii)、日本石龟(Mauremysjaponica)、非本地物种包括红耳龟(Trachemysscripta)、箱龟(CuoragalbinifronsMoegi,居住在中国南部)、鳄龟(Macrochelystemminckii)、马蹄龟(Sternotheruscarinatus)等[49]。针对入侵物种柳杉(Limnopernafortunei)在许多地方造成供水设施的阻塞问题,Ito等建立其环境DNA调查方法,并且正在Kasumigaura(霞浦)水系统中进行入侵监测。同时在5个水库对入侵物种柳杉进行常规调查方法(目测、采样和幼虫观察)和环境DNA调查。在所有用常规方法检测到柳杉的水库以及用常规方法未检测到的水库中,都检测到柳杉DNA。基于种群密度和环境DNA浓度之间的关系,在实验水库中也揭示了定量趋势[50]。池田对日本特有物种日本小龙虾(Cambaroidesjaponicus)和可能影响其生境的入侵物种内田小龙虾(Pacifastacusleniusculus)在北海道的分布进行了调查[36],环境DNA调查在检测这两个物种方面有优势,因为它们隐藏在砾石下,所以很难观察和捕获。这项研究的结果显示,内田小龙虾可能入侵上游地区,影响本地物种。
3.3 濒危物种和稀有物种的监测
由于濒危物种和稀有物种生物量较少,传统的调查方法存在精确度偏低与破坏栖息生存环境等问题,仅靠传统的捕捉和观察调查很难进行评估,而环境DNA分析技术能有效规避这些问题。Sakata等[51]在Omonogawa(大野川)河的99个水样中的2个检测到细鳞鱊(Acheilognathustypus),并在检出水区开展捕捉调查,确认其中一个地点存在成熟的细鳞鱊雄性和雌性以及作为产卵宿主的双壳类。这一发现是该河流11 a来的首次发现,有效证明环境DNA调查是确定大河中稀有物种分布和繁殖区域的有用工具。在这些地方Akamatsu等利用线粒体的DNA区开发一套引物探针,并在野外应用,发现检测到的DNA片段与潜水目测观察到的个体数量呈正相关,表明环境DNA可用于以符合国家规定的方式进行调查[52]。
3.4 生物量及生态影响评估
环境DNA技术有助于鱼类生物量或丰度的非侵入性量化,但目前DNA采集方法和环境条件对水样中环境DNA浓度的影响并没有系统的标准和了解,这样对运用环境DNA技术量化评估鱼类生物量造成限制。Doi等人在2016年利用潜水观察和摄食痕迹等方法调查河流中的常驻香鱼,对香鱼(Plecoglossusaltivelis)进行定量分布调查和环境DNA研究,两者之间存在明显的关系,证明环境DNA是一个来估计河水中香鱼的丰度与生物量、分布的有效方法[10]。日本山口大学的研究小组利用环境DNA来评估大坝建设对流域生境的影响,并验证改进项目的效果。研究表明,随着支流流量的增加,香鱼栖息地的密度在夏季也会增加,并建议在支流中建设适合香鱼栖息的主要区域[53-54]。Hanaoka等使用环境DNA来验证为恢复江之川滨原大坝下游的河流环境而实施的沉积物供应项目的效果,发现由于土壤径流的改善作用,沙质底部鱼类生物量有所增加[55]。此外,利用环境DNA技术揭示繁殖季节环境DNA浓度的昼夜变化以及繁殖行为中环境DNA颗粒大小特征并通过核DNA与线粒体DNA的比率评估产卵行为,根据河水温度和环境DNA浓度关系的实地研究数据,利用气候变化导致的河水温度上升模型,预测未来香鱼栖息地的减少。根据香鱼幼崽释放的DNA数量及河水环境DNA浓度调查幼崽的实际数量,并评估河口堰塞湖流速降低对其的影响。洪水事件中,香鱼生境密度和生境使用的变化研究中发现,香鱼在洪水来临之前与护岸比率呈负相关,与流速呈正相关;而在洪水来临之后,只有护岸比率呈负相关,表明香鱼生境受到洪水干扰导致的支流规模的影响[56]。Imashiro等同时检查Shimanto(四万十市)河中的香鱼和Flavobacteriumpsychrophilum(一种引起香鱼冷水病的革兰氏阴性长杆菌)的环境DNA,发现冷水病不是发生在5月(即原来的高峰期),而是在11月和12月发生在河口附近的产卵地[57],同时研究显示,产卵和携带真菌的香鱼的存在是感染扩散的驱动力。Ushio等通过使用illumina MiSeq并添加内部DNA标准,成功获得了沿海地区70多种鱼类的定量环境DNA时间序列数据。基于这些数据,Kondo对鱼类之间以及鱼类与水母之间的生态关系进行了时间序列分析,并与物理观测数据相结合,建立了预测海湾中竹荚鱼(Trachurusjaponicus)表层、中层和底部丰度的模型[58]。
3.5 环境DNA评估河道设施对鱼类影响
当前使用环境DNA技术评估河道设施对鱼类影响的研究较少。Aranishi等对关东河干流Shidzumi水库附近60 km长度的香鱼洄游河段进行了其纵向分布的环境DNA调查,发现在水库上游13 km及水库内广泛检测到香鱼DNA,而在下游未检测到其DNA,验证了大坝对季节性洄游鱼类的阻碍作用[59]。Yamanaka等通过对3个目标洄游鱼种(温带鲈鱼、平头鲻鱼、鲻鱼)的DNA片段的物种特异性检测,证明了目标物种的eDNA的存在与否与已知的物种季节性迁移模式一致,且在配置鱼梯的大坝上游最靠近河口的地方检测到温带鲈鱼和平头鲻鱼的DNA,而在另外两个未配置鱼梯的大坝上游未检测到上述两者的DNA,表明鱼梯能帮助这些物种通过绕过大坝的鱼梯成功向上游迁移[60]。Morita等利用电捕鱼与环境DNA结合的方法研究了日本北海道岛西南部大岛渚半岛30条有水坝的溪流中白斑红点鲑鱼(Salvelinusleucomaenis)种群灭绝的脆弱性,比较了1999年至2014年间溪流中白点鲑种群的灭绝概率,发现其局部灭绝的概率与流域面积成反比,而安装鱼梯重新连接隔断的生境后,能有效缓解白斑红点鲑鱼的局部灭绝危机[61]。
4 总结与展望
利用环境DNA了解生物多样性需要对多物种进行宏条形码的全面检测并结合特定物种的检测来确定物种的存在与否和它们的生物量。日本环境DNA协会已经出版了一本全面总结环境DNA作为一种调查和分析技术的手册《环境DNA调查和实验手册》,同时日本环境省自然保护局生物多样性中心也出版了《使用环境DNA分析技术的淡水鱼类调查方法手册》等技术资料。
环境DNA技术最严重的缺陷是存在污染的风险,即可能出现假阳性的结果。环境DNA在样品的采集、运输、保存等过程中都容易发生交叉污染,在实验室分析过程中亦会受到试剂的污染。污染问题是造成环境DNA实验结果误差的一个重要影响因素。可能导致分析错误的因素包括假阴性的问题,环境DNA技术需要依靠数据库,但是现如今的数据库生物信息并不完善,特别是针对感兴趣的生物体的生态环境(如低活性或者有限的时间内活动),缺乏参考物种名称的序列信息,以及良好的检测系统的发展不足影响环境 DNA 的最终检测结果。此外,环境DNA分析方法需要进一步规范完善,很多研究人员在环境DNA样品采集、保存、提取等过程采取的方法各不相同,并没有形成系统的分析方法,今后的研究中可以对环境DNA技术操作流程进行进一步的优化和完善。环境中的DNA会被微生物和紫外线辐射所降解,所以目前很难用具体的数值来预测和评估其降解率、平流和扩散。环境DNA技术的检测结果在时间和空间这两个维度的精度较低,检测精度有待提升。环境DNA技术可以通过特定物种DNA的有无直观的表征河道建筑对鱼类活动的影响,可以通过检测洄游性鱼类的DNA来评估河道设施对河流生态健康的影响,但由于当前环境DNA技术仍处于发展阶段,在定量评价上需要未来的进一步研究。
每一种调研方法都有其优点和缺点,因此了解和使用环境DNA作为生物多样性研究的工具的优缺点是非常重要的。环境DNA可以大大增加在时间和空间上获得生物多样性数据的可能性,它可以用来了解生物体与环境中各种因素之间的相互作用。通过进一步优化环境DNA样品采集、保存方法、DNA提取等分析方法,预计环境 DNA技术未来将进一步发展跨学科的综合研究,以解读生物体与其环境中各种因素之间的相互作用,例如其将食物链的能量流动、动物摄食等研究领域的作用展示出来。
