融合多维特征的医学知识图谱分步实体对齐方法
2022-11-18胡佳慧赵琬清陈凌云
娄 培,胡佳慧,赵琬清,陈凌云,方 安
知识图谱作为一个结构化的图模型,可以很好地描述现实中各种实体及相互之间的关系,在信息检索等领域有着非常重要的作用。知识图谱也被广泛用于医疗领域,利用知识图谱描述病历中相关的医疗知识,可以提高数据的利用率,推动智能医疗的发展,为医生的决策支持起到重要的辅助作用[1]。
知识图谱的构建流程包括本体构建、数据抽取、数据融合、数据存储、可视化展示。由于构建医学知识图谱的数据来源广泛,从异构数据源中抽取的医学名词差异化严重,导致图谱中存在实体重复、冗余等问题,进而无法很好地完成检索、知识推理、图谱问答等一系列任务,因此需要进行数据融合,使其成为一个有机整体,有利于进行更全面的知识共享[2]。本文以医学知识图谱为切入点,将电子病历数据与网络资源进行融合,以期构建一个内容更全面的知识图谱,在此过程中,提出了一种基于多维特征的实体融合方法,可以更好地适用于医学知识图谱知识融合。
1 相关研究
知识图谱实例层的融合主要以实体对齐任务为主,即将知识库中含义相同但标识符不同的实体进行知识发现和合并。实体对齐方法主要分为基于实体属性的方法和基于表示学习的方法两大类[3]。有学者设计了融合属性信息的双向对齐图卷积网络模型,通过融合更多的图谱信息提高了实体对齐准确率[4]。有学者利用度量空间的数学特征过滤掉大量不满足映射条件的实例对,使用三角形不等式计算实体间相似性[5]。有学者开发了Silk Linking Framework 工具包,用于发现Web 数据源之间的数据链接,允许用户定义规则并基于字符、数字、日期相似度进行实体对齐[6]。还有学者使用机器学习方法进行实体对齐,如有学者使用词频-逆文档频度(term frequency-inverse document frequency,TF-IDF)、隐含狄利克雷分布(latent Dirichlet allocation,LDA)计算文本型实体的相似度,使用支持向量机(support vector machine,SVM)、Logistic 回归等方法进行建模,得到了较好的结果[7];有学者使用主动学习方法训练一个分类器对记录进行匹配[8];有学者提出了一种简单贪婪匹配算法(SiGMa),适用于大规模知识库的实体对齐,利用图的结构信息和局部实体属性进行相似性度量[9]。
近年来,基于表示学习的实体对齐算法受到了广泛的关注。该算法将知识图谱中的实体映射到低维的向量空间,在低维空间中可以高效地计算实体、关系及其之间的联系,解决知识图谱数据稀疏性问题[10]。通过将实体映射成向量以便使用公式计算各实体间的相似度,在知识融合时具有很好的效果。有学者使用语义和结构特征训练基于联合表示学习的实体对齐模型,以提升实体对齐的效果[11]。有学者使用有监督实体对齐方法,通过TransE 算法把实体和关系表示成向量,计算向量之间的语义距离判断两个实体是否对齐[12]。有学者提出了一种基于表示学习的知识图嵌入和实体对齐算法,基于预先对齐的实体对,将头、尾实体与相应的上下文向量建模,将实体嵌入到公共空间中,在统一框架下解决了实体的嵌入和对齐问题[13]。有学者提出了一种利用联合知识嵌入实现实体对齐的方法,根据一个小的对齐实体种子集,将不同图谱的实体和关系联合编码到一个统一的低维语义空间中,根据实体的语义距离在这个联合语义空间中进行对齐[14]。有学者提出一种融入实体描述的知识图谱表示学习模型(description-embodied knowledge representation learning,DKRL),利用连续词袋模型(continuous bag of words,CBOW)和卷积神经网络(convolutional neural networks,CNN)将实体的描述信息转换成向量,然后利用TransE 进行训练[15]。
通用领域的实体对齐任务已受到广泛关注,医学领域也在逐渐开展相关研究。有学者提出了一种通过挖掘Web 数据建立糖尿病知识库的方法,从垂直门户的半结构化内容中提取知识,然后进一步将它们映射到统一的知识图中,使用基于距离的期望最大化(expectation maximization,EM)算法进行知识融合[16]。有学者构建了中文症状知识库,以从医疗网站中提取的数据和中文百科网站中提取的症状为补充,利用实体类型对齐、实体映射和属性映射融合抽取到的数据,以解决不同数据源之间的数据重复问题[17]。有学者构建了一个生物医学知识图谱,从出版物、百科全书、医疗门户网站和在线社区中提取和融合了数据,并使用逻辑推理进行了一致性检查[18]。
医学领域数据专业性强,多以临床术语记述,且包含医学习惯用语、缩略语等。其中,疾病名称和症状名称的不规范表述现象尤为严重,在图谱构建过程中容易造成歧义和冗余。因此,本文针对医学知识图谱结构和医学术语表述的特点,提出了一种基于多维特征融合的实体对齐方法。
2 融合多维特征的医学知识图谱分步实体对齐方法
2.1 模型框架
针对医学数据的特点,在构建医学知识图谱过程中习惯将图谱本体层划分为疾病、症状、治疗、病因等实体类型,图谱呈现为以疾病为中心的一阶发散结构,根据这类图结构特点,本文提出一种融合多维特征的医学知识图谱分步实体对齐方法。在待对齐的数据三元组中,将疾病类型定义为头实体,其他类型归类为尾实体,首先对齐尾实体节点,对齐后得到语义一致的尾实体表达,然后进行头实体对齐。
广义上的实体对齐方法分为成对对齐和集体对齐两种,成对对齐只根据一个实体对中两个实体本身的信息进行匹配,集体对齐会考虑整个知识图谱的信息进行实体匹配[19]。本文综合考虑头、尾实体本身的相似度和邻居节点的相似度,从实体的语义、结构、字符3 方面提取特征,利用监督学习方法训练实体对齐模型,模型框架见图1。
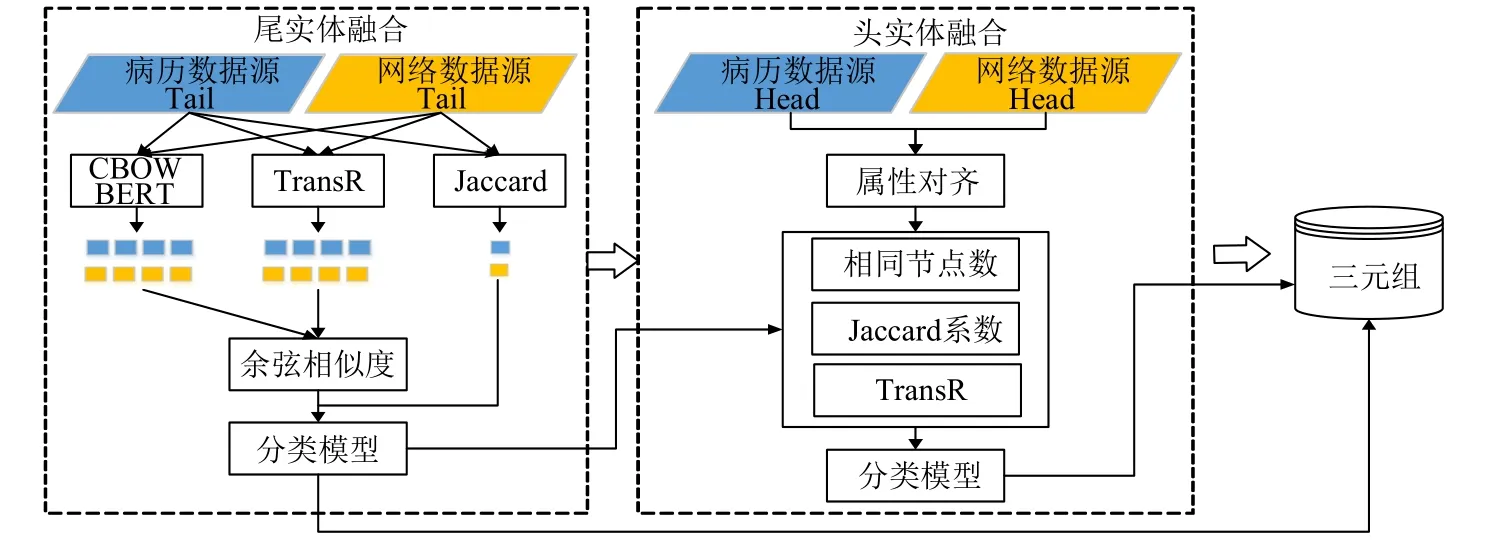
图1 多维特征融合的头尾实体对齐模型框架
2.2 尾实体对齐
在尾实体对齐中,通过寻找异构数据源中相同类型实体之间的最佳对齐方式来融合数据。选取实体的3 个特征(即语义、结构、字符特征)构建模型。通过基于Transformer 的双向编码模型(bidirectional encoder representations from transformer,BERT)、Word2Vec 模型得到实体语义信息的向量表示,利用TransR 模型得到实体结构信息的向量表示,进行向量拼接并计算余弦距离得到相似度,通过Jaccard 系数获得实体的字符相似度特征。将多个特征送入分类模型训练得到尾实体对齐结果(图2)。
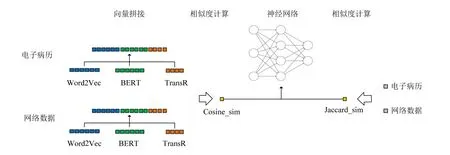
图2 尾实体对齐模型
2.2.1 语义特征
文本型数据对齐中广泛使用到语义相似度计算,通过词向量中携带的语义信息进行特征提取。本文使用两种语义模型对实体构造嵌入,将异构数据源实体映射到同一向量空间,通过度量空间距离衡量语义上的相似度。
Word2Vec 模型是一种分布式的向量表示方法,通过从大数据集中学习高质量的词嵌入计算单词的矢量表示[20]。由于Word2Vec 训练的准确率与训练数据有直接关系,且医学知识图谱有很强的领域专业性,因此结合通用语料和医学领域专业语料作为训练语料。本文使用CBOW 模型将词映射到固定维度空间,对实体词xi使用随机梯度下降的优化方法,最小化目标函数,更新并输出向量,得到实体的向量表示w2v_vec(xi)。
基于BERT 模型的自然语言处理任务通过预训练过程和微调过程两个过程实现[21]。将训练语料输入BERT 模型中,通过训练Next Sentence Prediction和Masked 语言模型两个任务得到词语的表达。模型的输入是位置信息、词、句子3 种向量的叠加。通过微调预训练模型将语料中的知识迁移至向量BERT_vec(xi)中(图3)。
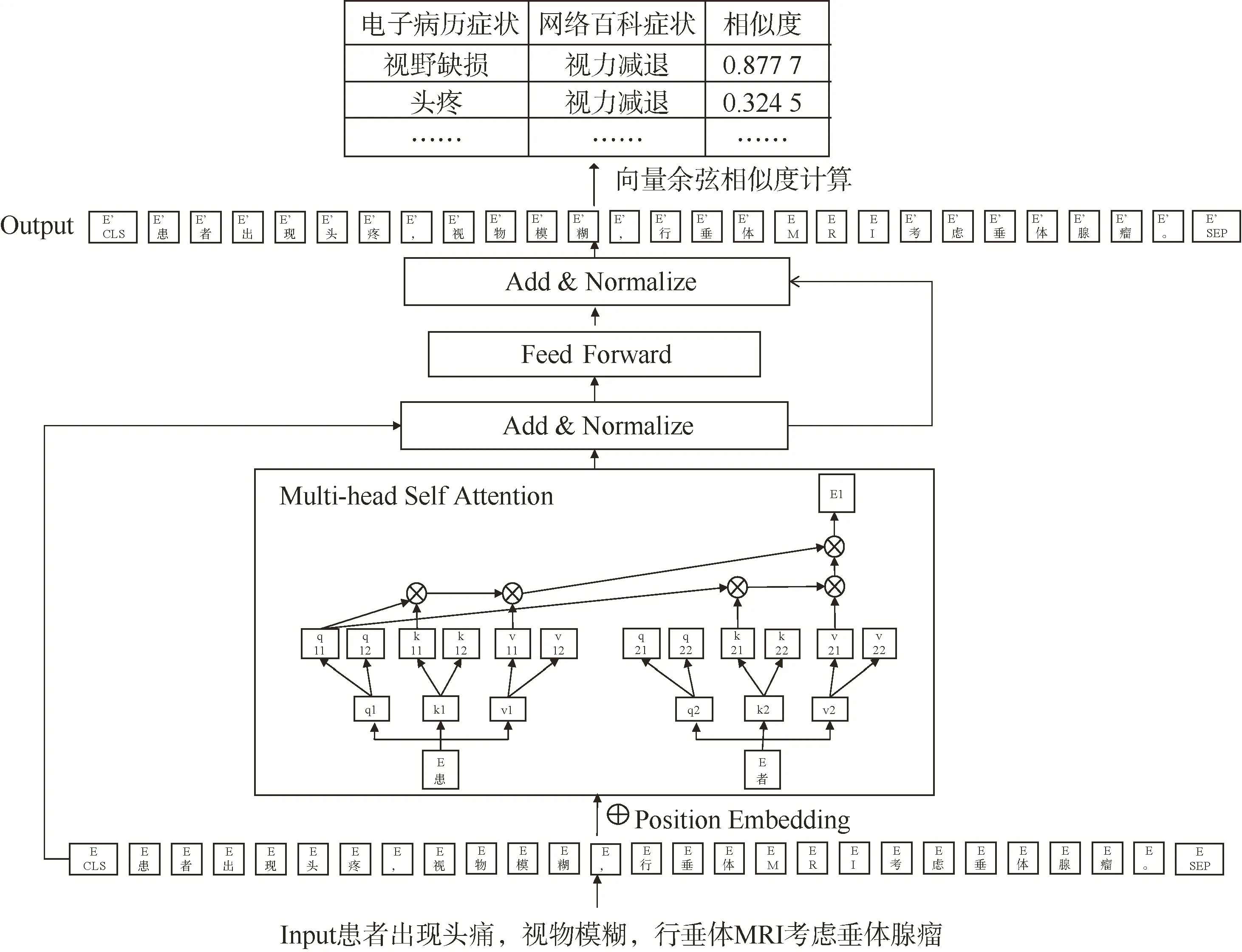
图3 BERT 模型语义相似度计算
2.2.2 图结构特征
自然语言处理模型通常需要对大规模语料进行训练,学习隐含的语义信息,知识表示学习不依赖于文本信息,而是通过将实体映射到低维空间来获得数据的深度特征。TransE 模型借鉴了Word2Vec 的平移不变性思想,对实体和关系进行分布式表示,但由于模型设计简单只能学习“一对一”关系,而医学知识图谱中包含众多“一对n”关系。因此,本文使用了将不同关系分别映射至不同空间的TransR 模型[22]。
将抽取的三元组数据作为正例(h,r,t),对每个正例三元组,随机替换头实体或尾实体生成负例{(h′,r,t) ∪ (h,r,t′)}。通过映射矩阵将关系r映射到不同空间,对每个三元组,将其损失函数定义为公式(1)。利用梯度下降法对参数进行更新,得到含有实体图结构特征的向量表示Trans_vec(xi)。
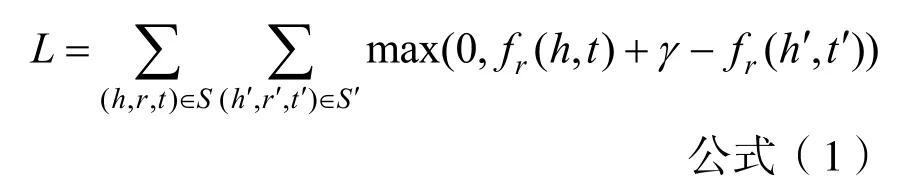
2.2.3 字符集合特征
Jaccard 系数可以比较样本集之间的相似性和差异性。将异构数据源中的每个实体分别映射到集合M、W,Jaccard 系数定义为集合M与W交集的大小与并集大小的比值,Jaccard 值越大说明相似度越高。使用Jaccard 系数计算一个实体对中相同字符数与总字符数之比,得到实体对的字符集合特征Sim_symjac。
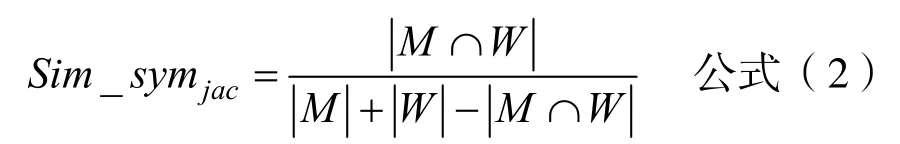
2.2.4 相似度计算
通过对异构数据源中的实体构造嵌入,得到了实体的语义特征和结构特征向量。利用向量的运算特性将上述w2v_vec、BERT_vec、Trans_vec向量横向拼接。通过计算拼接后向量在二维空间的夹角余弦值评估它们的相似度,夹角θ越小,余弦值越大,得到两个数据源中实体对的相似度Sim_symcos,计算公式为公式(3)和公式(4)。在实体对齐任务中,因为数据的类别注释只有相同或不同两种类型,因此将其转换为二分类问题,将实体对的余弦相似度值和Jaccard 系数值作为分类模型的输入进行训练,得到融合结果,计算公式为公式(5)。
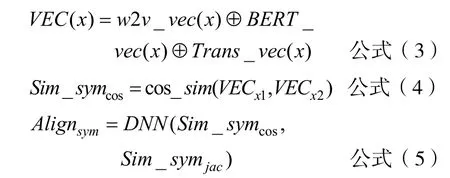
2.3 头实体对齐
尾实体融合完成后,对头实体(疾病)进行融合。当三元组中尾实体类型和关系类型相同而对应的头实体名称不同时,考虑头实体的属性相似性和结构相似性进行实体对齐(图4)。
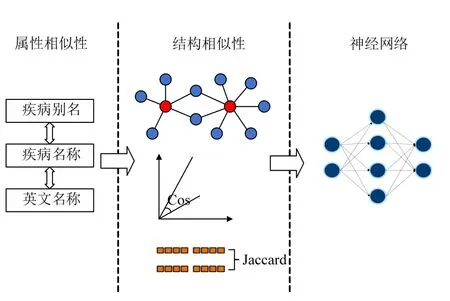
图4 头实体对齐模型
2.3.1 属性相似性
利用疾病的别名属性和英文名称属性可以进行实体对齐,如果两个数据源中头实体具有相同的疾病名称、别称、英文名称,即可认为两个实体相同,如垂体生长激素腺瘤的英文别名为Marie,中文别名包括生长激素腺瘤、GH 腺瘤等。
2.3.2 结构相似性
当实体不能通过别名进行对齐时,根据结构相似度判断两实体的相似性。以异构数据源中的两个疾病集合为例,对头实体x,y的相似度需考虑以下3 个因素:两头实体包含的相同尾节点个数Num_tail,计算公式为公式(6);两个数据源中头实体对应的所有尾实体集合中相同字符数与总字符数的比值,计算公式为公式(7);通过TransR 模型计算头实体所得的向量余弦相似度Sim_TransR。在相似度特征值计算完成后,仍将结果输入到分类模型进行头实体分类,计算公式为公式(8)。

3 实验与分析
3.1 数据来源
由于医学知识图谱实体对齐研究中没有先验数据集,因此本文以垂体瘤专病知识图谱构建为例验证所提出的分步实体对齐方法的有效性(图5)。数据来源包括两个部分。一部分数据来源于我国某三甲医院神经外科治疗中心,包含300 名垂体瘤患者的临床诊疗信息。在临床医生的指导下对疾病、症状实体进行标注,使用基于单字特征的条件随机场(conditional random field,CRF)进行实体识别[23],最终得到80 种疾病和762 个相关症状实体。另一部分数据从医学网站和质量较高的百科类网站中选取。将电子病历中抽取的疾病实体作为检索词,在医学网站进行检索。由于单一的门户网站包含的疾病种类有限,信息检索不全面,因此选取了“寻医问药网”[24]、UpToDate 临床顾问[25]、百度百科[26]、“春雨医生”[27]4 个数据质量较高、结构化程度较好的网站。通过设计正则等规则构建数据抽取器,实现对网站中疾病和症状数据的自动化抽取,得到58 种疾病和435 个相关症状实体。
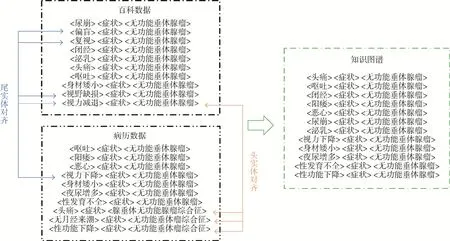
图5 无功能垂体腺瘤数据对齐实例
最终,得到垂体瘤疾病相关的3 154 个“疾病-症状”三元组进行模型实验,训练集与测试集按7∶3 进行划分。本文使用了监督学习模型,在专业医生的帮助下对实体对的相似性进行人工标注,如果该实体对可以融合,标记为1,否则标记为0。
3.2 模型设置
实验在Windows 10 环境下进行,模型基于TensorFlow 框架构建。在训练基于Word2Vec 的语义相似度模型时,使用Python 的gensim 库提供的方法进行词向量训练。使用CBOW 模型,参数定义如下:min_count=1,hs=1,window=10,dim=150维。对谷歌预训练好的BERT 模型进行微调,参数定义如下:epoch=100,batchsize=100,λ=0.001,dim=768 维。将构建好的正负例“疾病-症状”三元组输入TransR 模型,定义向量维度dim=50 维。
分类模型训练调用Sklearn.linear_model 子类下的回归模型、Sklearn.tree.DecisionTreeClassifier子类下的决策树模型和深度神经网络(deep neural networks,DNN)模型。使用DNN 模型时设置输入层3 个神经元,隐藏层4 个神经元,输出层2 个神经元,设置参数为n_epoch=30,batch_size=32,show_metric=True。
3.3 实验结果
在尾实体对齐模型中,采用logistic 回归、决策树和神经网络3 种不同的分类模型进行训练,结果如表1 所示。训练结果显示,神经网络表现最好,准确率为99.58%,F1 值为99.58%。模型可以很好地将具有相似特征的实体进行合并。
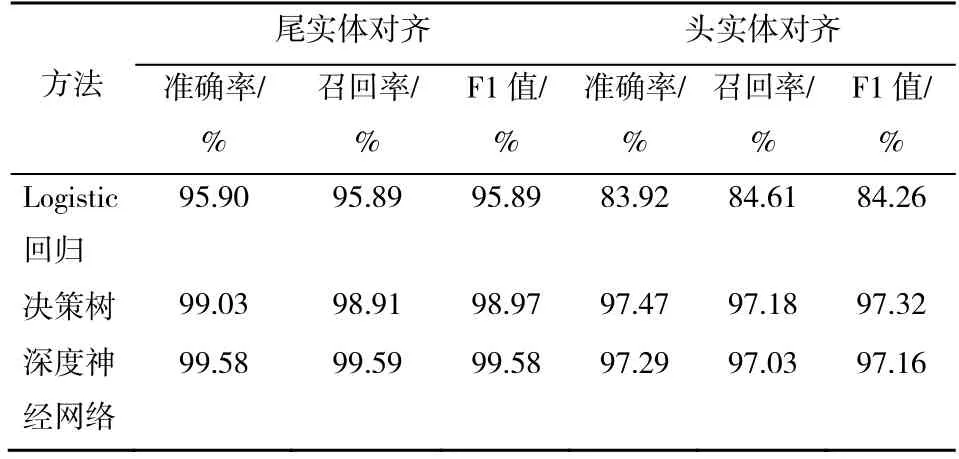
表1 融合多维特征的医学知识图谱分步实体对齐模型结果
将尾实体融合后的三元组用于头实体融合实验。其中17 个实体可直接映射,6 个实体可通过别名属性进行融合,包括垂体生长激素腺瘤、垂体微腺瘤、库欣综合征、甲状腺功能减退等。仍利用上述3 种分类模型进行头实体对齐训练,模型对齐了42 个头实体。实验结果显示,头实体对齐实验中决策树的分类效果更好,其准确率达到97.47%,F1值达到97.32%。
使用Logistic 回归作为基线模型,在头实体、尾实体融合中,基线模型的准确率达到了83.92%,说明本文选择的特征可以很好地表达实体信息。相较于尾实体,头实体数据量小,神经网络在训练过程中出现过拟合,决策树在头实体对齐实验中表现更好;而随着数据量的增加,在尾实体对齐时,神经网络的优势则体现出来。
4 结语
本文介绍了一种融合多维特征的医学知识图谱分步实体对齐方法,利用抽取到的电子病历和网络资源中的疾病和症状信息,对实体对齐模型进行了研究。根据医学数据特征,本文提出了分步的头实体、尾实体对齐方法,并全面考虑到了数据的语义、结构、字符相似度特征。实验结果显示,头实体、尾实体融合的准确率都很高,均达到了97%以上,说明该方法可以有效将异构数据源融合在一起,适用于多源异构医学知识图谱的构建。
本文也存在一定的局限,由于数据量较小,在实体对相似度计算时没有考虑计算复杂度,今后将考虑引入分块的思想,将知识图谱划分成n 个小规模的图谱进行匹配以降低计算复杂度,同时还将尝试把知识图谱用在医疗决策支持等场景中,帮助医生和患者解决实际问题。
