结合语义和依存关系的药物相互作用关系抽取
2022-11-14罗熹曾智颖王建新安莹
罗熹 曾智颖 王建新 安莹




摘要:从生物医学文本中抽取药物相互作用对可以快速更新药物数据库,具有非常重要的意义与医学应用价值.现有的神经网络模型往往仅从句子序列或其他外部信息中学习到单一片面的特征,难以充分挖掘句中潜在的长距离依赖特征获得全面的特征表示.本文提出一种结合语义和依存关系的药物相互作用关系抽取方法,该方法在利用Bi-GRU网络分别从句子序列和目标药物实体的最短依存路径序列中学习语义特征表示的同时,进一步结合多头自注意力机制挖掘单词之间潜在的依存关系,通过充分融合多源特征来有效提升生物医学文本中药物相互作用对的识别和抽取性能.在DDIExtraction-2013数据集上的实验结果表明,该方法超过现有的药物相互关系抽取方法获得了75.82%的F1值.
关键词:药物相互作用;关系抽取;循环神经网络;多头自注意力机制;最短依存路径
中圖分类号:TP391文献标志码:A
Drug-drug Interaction Extraction Combining Semantics and Dependency
LUO Xi1,2,ZENG Zhiying1,WANG Jianxin1,AN Ying3
(1. School of Computer Science and Engineering,Central South University,Changsha 410075,China;2. Key Laboratory of Network Crime Investigation of Hunan Provincial Colleges,Hunan Police Academy,Changsha 410138,China;3. Big Data Institute,Central South University,Changsha 410083,China)
Abstract:Automatically extracting unknown drug-drug interactions from biomedical literature can update the drug database quickly,which is of great importance and medical value in application. Existing neural network models often can only learn a single one-sided feature in a certain aspect from sentence sequences or other external information,but it is difficult to fully mine the potential long-distance dependency features from sentences to obtain a comprehensive feature representation. This paper proposes a novel drug-drug interaction extraction method combin - ing semantics and dependency. In this method,we not only use the Bi-GRU network to learn the semantic feature representation from the sentence sequence and the shortest dependency path of the target drug entities,but also combine the multi-head self-attention mechanism to further capture the potential dependencies between words. Finally,these multi-source features are fully fused to effectively improve the performance of drug-drug interaction extraction. The experimental results on the DDIExtraction-2013 dataset show that our method outperforms other existing methods and obtains an F1 value of 75.82%.
Key words:drug-drug interaction;relation extraction;recurrent neural networks;multi-head self-attention mechanism;the shortest dependency path
药物相互作用(Drug-Drug Interaction,DDI)是指同时或相继使用两种或两种以上药物时,某一药物作用大小、持续时间甚至作用性质受到其他药物或化学物质的影响而发生明显改变或产生药物不良反应的现象[1].不良的药物相互作用可以减缓或者延迟药物的吸收,导致病人的治疗周期增长,治疗效果减弱,严重的更会危及生命甚至导致死亡[2-3].在临床治疗中,患者在很多时候不可避免地会需要服用多种药物,DDI的存在可能导致患者面临严重的用药风险,影响临床治疗的效果.随着联合用药的日益普遍,如何避免不良药物相互作用的发生已经成为临床安全性的一道难题.目前来看,尽可能多地了解药物作用的相关信息,是解决不良药物相互作用发生的有效途径,对药物研发和临床治疗有着重要的意义.
随着研究人员对于DDI研究关注度的不断提升,近年来涌现了大量的相关研究成果.这些研究成果大多以自由文本的形式存在于医学文献中,这使得医学文献成为了获取最新DDI信息的最有效来源之一.目前虽然存在一些结构化的药物数据库可供用户查询药物的相关信息,但是现有的数据库大多通过人工采集的方式从文本中挖掘药物相互作用关系来建立相关数据库.这种过分依赖人工干预的方式导致现有药物数据库的构建和维护极为耗时费力,知识的更新缓慢低效,覆盖范围也十分有限,难以满足数据规模爆炸式增长、数据复杂度不断提高的大数据环境下药物相关研究和临床应用的实际需求.因此,实现非结构化生物医学文本中的DDI自动提取具有极为重要的研究意义和应用价值.
传统的基于统计的机器学习的方法虽然能够从文本中自动地抽取DDIs,但是这类方法在特征提取的过程中通常依赖于人工制定的特征工程,抽取效果也不太理想.基于深度学习的方法避免了复杂的特征工程并且取得了不错的效果,但是现有的方法往往仅从句子序列或其他外部信息中学习到片面的特征,难以充分地利用文本中多方面的相关信息获得全面的特征表示.
本文提出了一种结合语义和依存关系的药物相互作用关系抽取方法.在通过Bi-GRU网络从句子序列中充分学习语义特征表示后,利用多头自注意力机制从中挖掘句子内部单词之间潜在的依存关系,获得长距离依赖特征.同时,还通过引入目标药物实体间的最短依存路径,保留能表明候选药物对关系的重要单词,过滤掉其他无关的词,并利用Bi- GRU网络从最短依存路径序列中学习特征.最后充分地融合句子序列和候选药物对之间最短依存路径之中的特征来实现文本中药物相互作用对的识别和抽取.
1相关工作
生物医学和医学信息化的发展为药物相关研究积累了各种形式的大量生物医学数据,越来越多的研究成果都以非结构化文本的形式展示在互联网上,如何及时从这些开放领域的文本中获取有价值的信息,成为一个亟待解决的问题.实体关系抽取的主要目的是从文本中识别实体并抽取实体之间的语义关系,从而解决原始文本中目标实体之间的关系分类问题,它也是构建复杂知识库系统的重要步骤. 药物相互作用关系抽取(Drug-Drug Interaction Extraction,DDIE)作为实体关系抽取中一个具体领域的子任务,也得到了广泛的关注.
近年来,研究人员在DDIE任务上做出了许多努力.现有方法可以分为以下三类:基于规则的方法、基于机器学习的方法和基于深度学习的方法.早期的研究大多采用基于规则的方法,通过制定一系列规则从文本中抽取存在相互作用的药物对[4-5].但是在生物医学文献中,由于描述药物相互作用的语句结构复杂多变,基于人工规则的方法在药物相互作用关系抽取任务中表现不佳.DDIExtraction-2011评测[6]和DDIExtraction-2013评测[7]的成功开展,为研究人员提供了一个已标注的药物相互作用关系语料库,它在区分药物对是否存在相互作用的同时,还将药物相互作用的类别进行了划分.这个语料库的发布给研究人员提供了标准有效的数据支撑,极大地促进了药物相互作用抽取相关研究的发展.近年来,许多基于机器学习的方法開始被用于药物相互作用关系抽取任务并取得了较好的效果.
基于机器学习的方法大体可分为两类:基于特征的方法和基于核的方法.基于特征的方法利用人工提取的各种特征将候选实例表示为相应的特征向量以实现DDI的分类.常用的特征有单词特征、上下文特征、句法特征等.例如Kim等人[8]提出了一种基于丰富特征的抽取DDI的方法,包括单词特征,依赖图特征,解析树特征等.然而基于特征的机器学习方法存在着明显的局限性:特征的人工提取十分耗时且依赖于研究人员的专业知识技能水平的高低.基于核的方法通过构建不同的核函数来量化两个对象之间的相似性,Chowdhury等人[9]提出了一种用于DDI提取的混合核方法,包括基于特征的内核,浅语言内核等.但是核函数的有效设计是一个极富挑战性的问题.因此,传统的基于统计的机器学习方法在药物相互作用关系抽取方面的性能仍然难以令人满意.
随着深度学习技术的发展,深度神经网络模型不依赖传统特征工程的自动特征学习能力,使其在自然语言处理的多个子领域得到了广泛的应用并获得了一定的成功.近年来已经提出了一些基于深度学习的方法用来从文本中抽取DDI.基于深度学习的方法可以利用深度神经网络的学习能力从数据中自动捕获相关特征,并在一定程度上有效地提高DDIE的性能,因此它们吸引了广泛的注意并且逐渐成为主流方法.
起初,基于深度学习的方法往往只考虑了文本中句子序列的语义特征.Liu等人[10]在2016年首次使用CNN模型来进行DDI的抽取,该方法将文本中的单词转化为词向量并结合位置信息作为特征输入,避免了传统方法在提取特征过程中对于自然语言处理工具的过度依赖.Quan等人[11]则利用多通道融合不同版本的词向量来获得包含更丰富语义信息的词向量表示,再利用卷积神经网络从句子序列中抽取DDI,获得了优于传统方法的性能.
然而,仅仅通过句子序列分析虽然能获得相关的语义线索,却难以学习到句中包含的句法信息.因此,许多研究人员尝试利用自然语言处理工具进行句子解析以获取更多的句法特征来提高关系抽取的准确性.最常用的就是依存句法分析[12](Dependency Parsing).依存句法分析可以通过分析句内成分之间的依存关系揭示其句法结构,目前在自然语言处理领域取得了广泛的应用.在药物相互作用关系抽取任务中,依存分析的有效性也得到了有效验证.比如Wang等人[13]通过依存句法分析得到句子的依存分析树,通过对其使用深度优先搜索和广度优先搜索得到相应的序列数据(DFS和BFS),再通过Bi-LSTM网络从DFS、BFS和原句子序列中分别学习得到特征.Zhao等人[14]提出了一种结合了GRU和GCN(Graph Convolutional Network)的深度神经网络,分别用于从句子序列和句法图中学习相关特征来对DDI进行分类.这些方法虽然在DDI抽取性能上取得了一定的提高,但是它们通常依赖于句子解析的准确性,而目前的自然语言处理工具并不完善,不可避免的存在一定的解析错误,尤其是对复杂长句的解析上效果较差,因此大大影响了它们的有效性.
除此之外,随着注意力机制在各类自然语言处理任务中取得了成功的应用,相关研究人员也开始将其引入到药物相互作用关系抽取任务中来.Wang等人[15]提出的Input Attention机制通过计算每个单词与药物实体的点积来衡量它们之间的相似度,从而给句中单词分配不同的权重.Zhou等人[16]提出了position-aware Attention机制,把全句各单词的位置信息加以考虑来捕捉特定单词对目标实体关系的影响.这些方法通过Attention机制选择性地关注来自输入的重要信息,在DDIExtraction-2013数据集上取得了不错的效果.但是这些方法忽略了句法特征的重要性,尤其是长距离依赖特征对于DDI识别和抽取的关键作用.
自注意力机制[17]是一种特殊的注意力机制,它可以根据当前单词与同一序列中其他单词的关联度来评估其重要性,已经被广泛应用到各种自然语言处理任务中,比如Paulse等人[18]在抽象式摘要任务中通过自注意力机制来捕捉长距离依赖关系,Tan等人[19]利用多头自注意力机制完成语义标注任务等,都取得了优秀的表现.本文提出的方法将自注意力机制引入到DDIE任务中来挖掘单词间的潜在关联以及句子中的长距离依赖.同时,为了获得更丰富更全面的特征表示,采用多个并行头来捕获单词间的相互依赖性.
综上所述,尽管基于深度学习的方法取得了优于基于机器学习的方法的表现.但是,现有的神经网络模型学习到的特征往往是单一片面的,难以充分挖掘句中潜在的长距离依赖特征获得全面的特征表示.因此,本文结合多头自注意力机制提出了一种能融合文本更深层的语义特征和其他多层面多角度特征的方法来实现DDI的抽取.
2结合语义和依存关系的DDIE方法
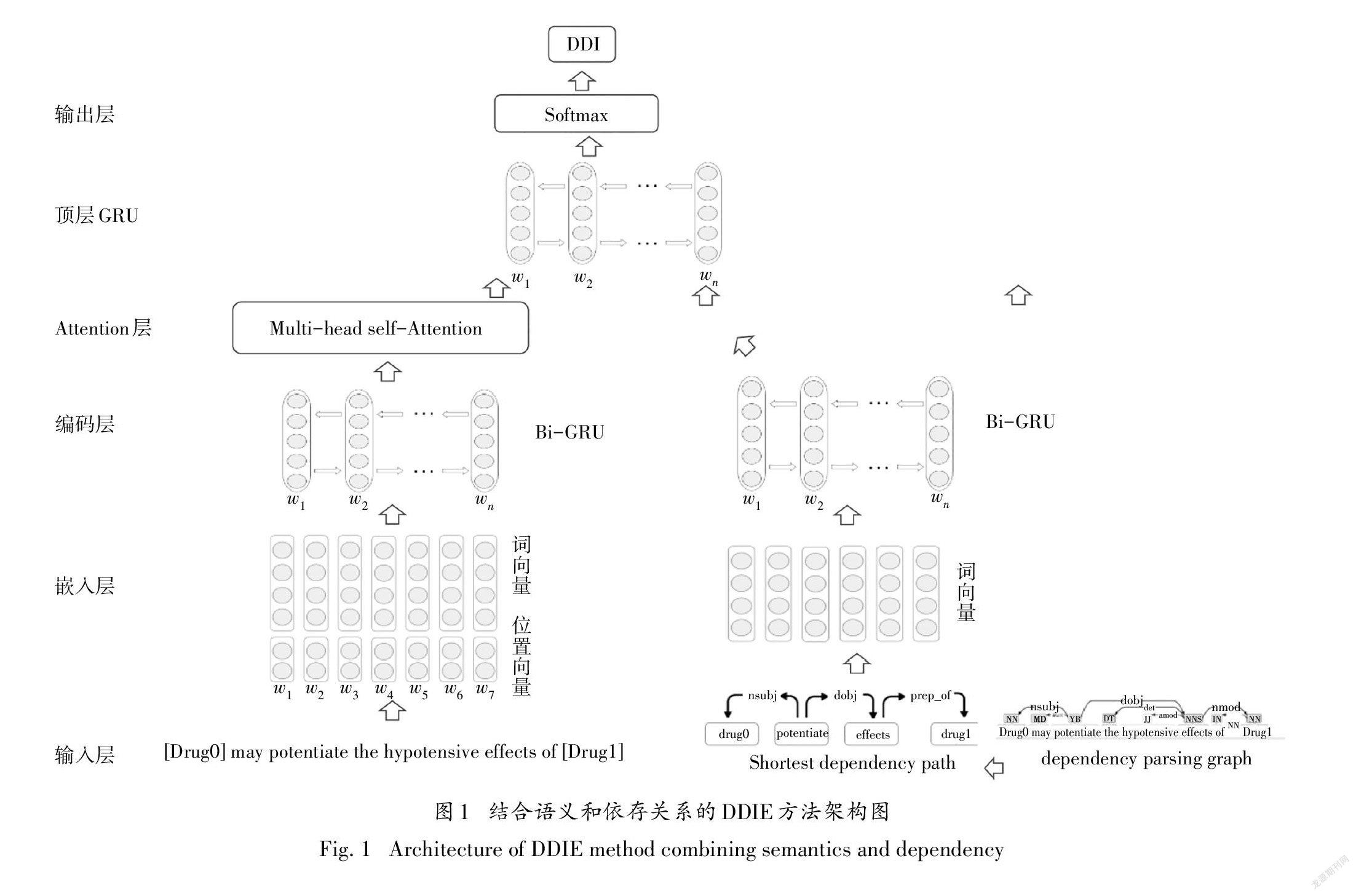
本节将对本文方法进行详细描述,其模型结构如图1所示.将包含候选药物对的句子作为输入,本文模型可以自动从该句子中提取特征,然后确定该药物对是否存在相互作用.其主要流程如下:
1)预处理:对句子进行相关预处理并利用Stanford parser解析句子得到目标药物实体间的最短依存路径.
2)嵌入层:通过嵌入层把句子序列和最短依存路径序列中的单词映射为预先训练好词向量,对于句子序列中的单词还根据其与目标药物的相对距离生成位置向量.
3)编码层:使用Bi-GRU神经网络分别从句子序列和最短依存路径序列中学习语义和句法特征.
4)Attention层:使用多头自注意力机制从融合了上下文信息的句子序列中挖掘单词之间的长距离依赖关系得到最终特征表示.
5)输出层:通过顶层Bi-GRU融合多源特征实现DDI的识别和抽取.
2.1预处理
我们首先对数据集中的句子进行相应的预处理操作.为了增强模型的泛化能力,按照在句中出现的顺序将候选实例中的两个药物名分别替换成“DRUG1”和“DRUG2”,句中其他药物名均替换成“DRUG0”.同时,语料库中有大量表示药物剂量、百分比等的数值表达式,它们是导致假阴性的重要原因之一.因此,我们将句中表示药物剂量、百分比等的整数和小数数值型实体分别用“num”和“float”标记代替.此外,为了简化句子,在预处理步骤中还进一步删除了句中冗余内容,如出现在括号中不包含目标药物实体的补充说明内容.
另外,由于候选药物相互作用对的选取方式是考虑句中所有药物实体可能的组合,导致数据集中负样本的数量远多于正样本的数量.例如对于句子“In patients receiving mercaptopurine (Purinethol)or azathioprine (Imuran),the concomitant administration of 300-600 mg of allopurinol per day will require a reduction in dose to approximately one-third to one-fourth of the usual dose of mercaptopurine or aza- thioprine.”,共包含7个药物实体,将它们两两组合生成候选实例,则该句中一共存在62个候选的药物相互作用对.但是其中只有两个正样例,其余均为负样例.在DDIExtraction-2013训练集中,正样例数为4 020,而负样例数为23 772,正负样例的比例达到了1:5.9,数据存在明显的不平衡现象.为了减轻样本不平衡对模型训练效果的影响,根据先前的工作[20-21],我们采用了下面的样本过滤规则,将满足其中任一条件的候选实体识别为负样例并予以滤除.过滤规则具体描述如下:
规则1:候选关系实例中的两个药物实体名相同或者一个药物名是另一个药物名的别名或缩写名.
规则2:候选关系实例中两个药物名出现在同一个并列结构中且该并列结构包含两个以上药物名.
在经过预处理步骤后,得到包含有两个目标药物实体的句子序列:
S={w,w,…,w,…,w,…,w)(1)
其中w(i∈[1,n])為句中的单词,w=“DRUG1”和w=“DRUG2”(u,v∈[1,n],u≠v)分别为候选药物对中的两个药物实体.
2.2生成最短依存路径
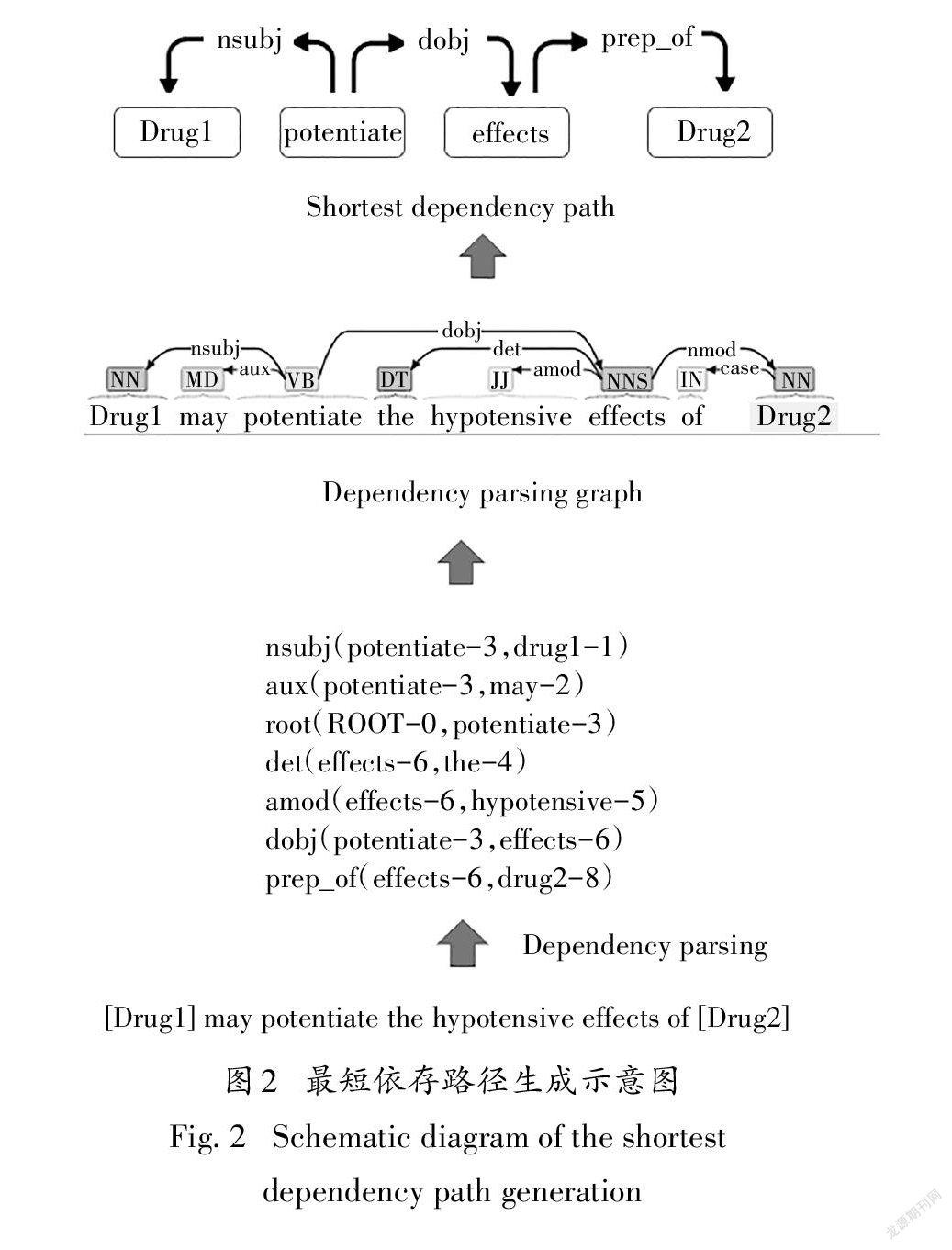
为了获得更多的特征来提升药物相互作用关系抽取模型的性能,本文使用Stanford parser126]来对句子进行依存句法分析,并根据依存分析的结果得到句子的依存关系图.以依存关系图中的“DRUG1”为起始节点,“DRUG2”为结束结点,找到它们之间的最短路径X,即为两目标药物实体之间的最短依存路径:
X={s,s,…,s}(2)
其中s(i∈[1,m])为最短依存路径序列中的单词.两个药物实体在依存关系图中可能存在多条路径,但两个节点之间的最短路径最可能携带有关它们相互关系的最有价值的信息.因此,在关系抽取任务中,可以通过最短依存路径显著缩小目标实体之间的线性顺序距离来捕获他们之间的关系.最短依存路径生成的具体流程如图2所示.
原句子经过Stanford parser依存句法分析后得到若干个表示句中两个单词之间关系的三元组.比如对于三元组“dobj(potentiate-3,effects-6)”,表示“effects”是“potentiate”的直接宾语,“dobj”表示直接宾语关系,3和6分别表示“potentiate”和“effects”在句中的位置.根据依存分析结果得到句子的依存关系图,依存关系图使用节点来表示句子中的单词,使用边来描述单词之间的依存关系.最后从依存关系图中找到目标药物实体间的最短依存路径.可以看到,最短依存路径将重要的词保留在两个实体之间的语法路径上,同时过滤掉次要的辅助词,并给出了相邻词之间的依存关系.因为表征单词之间关系的依赖关系词没有一致的向量表示方法,通过随机初始化再训练的方式很难学习其中复杂的关系,所以我们只保留路径中的单词作为最短依存路径.另外,句中除了两个目标药物实体外,往往还存在多个其他药物实体.为了避免这些无关实体对识别目标药物实体相互关系带来的负面影响,本文对每条最短依存路径中“DRUG0”的出现次数进行统计,并将“DRUG0”出现次数大于或等于路径长度一半的最短依存路径进行置空处理.
2.3嵌入层
完成预处理后,包含有两个目标药物实体的句子S及其对应的最短依存路径X输入到模型的嵌入层以生成句子序列和最短依存路径序列的嵌入向量表示.我们直接利用了Pyysalo等人[22]基于大量PubMed和English Wikipedia文献训练得到的词向量.同时,为了进一步获得句中其他单词与目标药物实体的关联关系,我们还将句中任意单词w到两个目标药物实体w和w的相对距离标记为p和o,用来表示句中单词的位置信息.
pi1=i-u,p=i-v(3)
然后,通过嵌入层将句中单词和其与目标药物之间的相对距离映射为对应的向量:
最后,句子序列S和其对应的最短依存路径X可分别表示为如下的嵌入矩阵E和E:
2.4编码层
循环神经网络(RNN)在处理文本序列信息时具有独特的优势,很适合序列建模,但是易于遭遇梯度消失和梯度爆炸问题,为了解决这一问题,Long Short Term Memory(LSTM)[23]网络和Gated RecurrentUnit(GRU)[24]网络相继被提出.本文中,我们采用了结构更为简洁的GRU网络.
z为更新门,用来表示第(t-1)个节点的状态有多少需要传递到下一节点:
z=σ(W-[h,e])(9)
g=σ(W·[h,e])(11)
上式中的σ表示sigmoid激活函数,tanh表示双曲正切激活函数,W,W,W都是学习到的参数矩阵.但是单个GRU网络在计算当前状态时只考虑了过去的状态信息,而忽略后续的状态信息.因此,本模型采用双向GRU(Bi-GRU)网络,充分地利用过去和未来的信息来获得更全面的上下文特征,并得到e的向量表示y.
y=[y,y,…,y,…,y](15)
对于最短依存路径的嵌入矩阵E亦经过Bi- GRU网络得到相应的特征表示y:
y=y=Bi-GRU(E)(16)
2.5Attention层的相关性,并将其输入到softmax函数中,得到与值相对应的最终权值,最后与值进行加权求和得到该查询经过键值对映射后的Attention值,具体计算过程如下.
最后,来自h个并行头的结果被拼接起来并映射为句子序列的最终特征表示r:
M=contact(head,head,…,head)(19)
r=M·W(20)
2.6輸出层
最后将从句子序列和最短依存路径中学习到的特征表示r和y拼接起来输入顶层Bi-GRU神经网络中获得最终的融合特征表示:
g=[r,y](21)
y=Bi-GRU(g)(22)
其中g的维度为2d,y为融合各特征后得到的最终特征表示,维度为d.最后将y放入全连接层并通过softmax函数得到该候选药物对属于第,类的概率为:
y(p∈i)=softmax(W·r+b)(23)
其中W和b为权重参数.
3实验结果与分析
3.1数据集
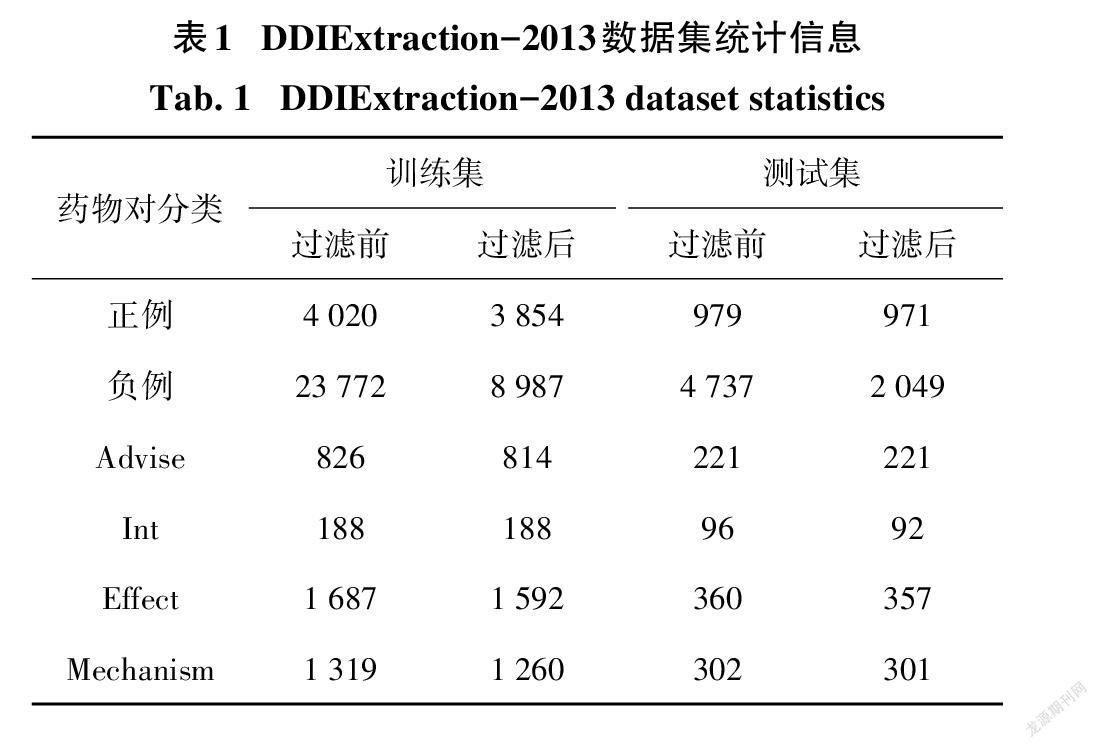
本文使用的实验数据均来自DDIExtraction- 2013评测数据集.该数据集在DDIExtraction-2011 测评数据集包含的来自DrugBank数据库DDI文本的基础上,还进一步增加了MedLine摘要文本,共计包含905份文本(其中730份DrugBank文本,175份Medline文本).通过随机选择的方式,语料库中77% 的文本被用作训练集,而剩余的部分则作为测试集. 数据集中的所有文本都以XML格式进行存储,文本中的药物名和药物相互作用候选对均由相关领域专家完成人工标注,不仅标明了各候选实例是否存在相互作用,还对存在相互作用的药物对细分为以下四类:
Int:句中说明了两个目标药物之间会发生相互作用,但未做出进一步说明.
Advise:句中说明了两个目标药物之间会发生相互作用,同时给出了建议.
Effect:句中说明了两个目标药物之间会发生相互作用,同时描述了相互作用产生的影响.
Mechanism:句中说明了两个目标药物之间会发生相互作用,并介绍了该DDI的药代动力学机制.
根据预处理步骤中所述的过滤规则,我们对数据集中的实例进行了相应的预处理.过滤前后的数据集统计信息如表1所示.从表中可以看出,训练集中总共过滤掉了166个正样例和14 785个负样例. 需要说明的是,对于测试集,将满足过滤规则的总共8个正样例判定为负样例,并计入假阴性统计数据,其余满足过滤规则的所有负样例直接计入真阴性统计数据中,参与最终的评估.
3.2实验设置和评估指标
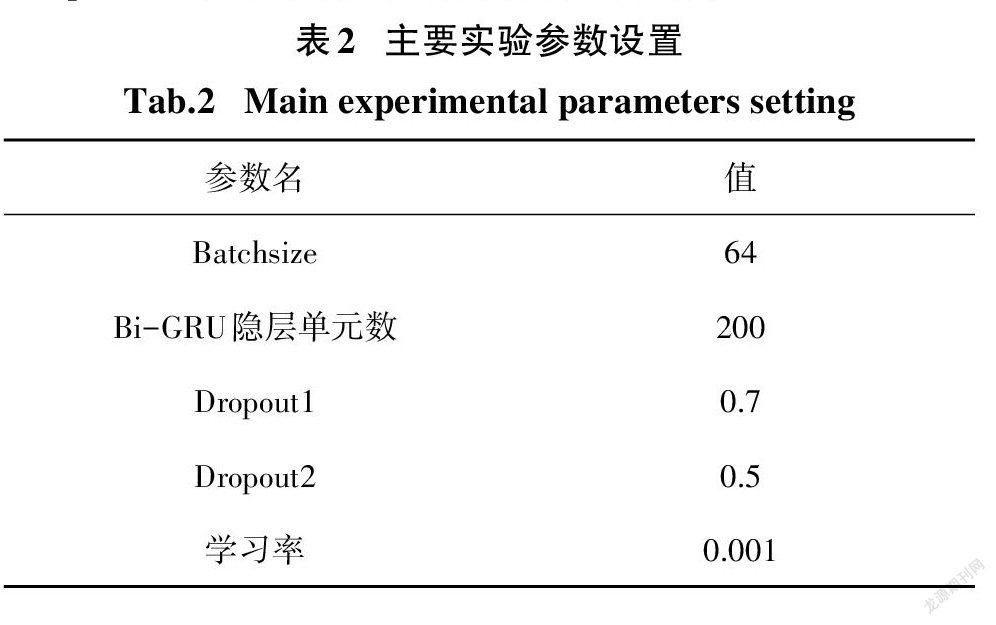
本文使用以TensorFlow为后端的Keras框架来实现相关的对比模型.词嵌入和位置嵌入的维度分别设为200和15.为了防止过拟合,我们在模型的嵌入层和输出层都采用了Dropout策略(Dropout1和Dropout2).主要实验参数设置如表2所示.
本文采用了DDIExtraction-2013评测的评估标准,当且仅当某一药物对被识别出存在相互作用并被正确分类到具体某个关系类型时,方认为该药物对被正确地识别.具体的评价指标包括:准确率P(precision)、召回率R(Recall)和F值(F-score).相应的计算方法如下.
1)微准确率:以抽取的关系对的准确程度来对模型进行评估,公式如下:
2)微召回率:从查全率的角度来对模型进行评估,公式如下:
3)微F值:对模型从查全率和查准率进行综合考量,公式如下:
其中TP(True Positive)是指被正确分类为正样本的
样本个数,FP(False Positive)指负样本中被错误分类为正样本的样本个数,FN(False Negative)指正样本中错误分类为负样本的样本个数.
3.3Baseline方法
为了评价本文提出方法的有效性,我们与以下几种baseline方法进行了比较.
FBK-irst[9]:该方法是一种基于核的方法,通过使用混合内核(包括浅层内核,基于特征的内核等)实现DDI的提取.
Feature-based kernel[25]:该方法是一种基于特征的方法,通过集成了多个相关特征例如单词特征,短语特征,词汇特征等来抽取DDI.
Basic-CNN[10]:该方法利用卷积神经网络从词向量和位置向量中学习特征来实现从生物医学文本中抽取DDI.
MCCNN[11]:该方法通过不同的通道融合多种词向量获得包含更丰富语义信息的词向量表示,再利用卷积神经网络从中抽取DDI.
GRU+GCN[14]:该方法是一种结合了GRU和GCN(Graph Convolutional Network)的深度神经网络,分别从句子序列和句法图中学习特征来对DDI进行分类.
UGC-DDI[21]:该方法通过融合用户生成信息和句子信息,再利用LSTM从中提取特征来抽取DDI.
Dep-LSTM[13]:该方法是一个基于Bi-LSTM的模型,结合了句子序列特征和基于依存关系的特征.
LSTM+Att[16]:该方法提出一种关注位置信息的注意力机制,并利用Bi-LSTM从句中学习特征完成DDI的抽取.
BERE[27]:該方法使用混合编码网络来更好地结合语义特征和句法特征表示每个句子,并通过考虑相关语句后使用特征聚合网络进行DDI的抽取.
3.4结果分析
3.4.1特征融合的有效性验证
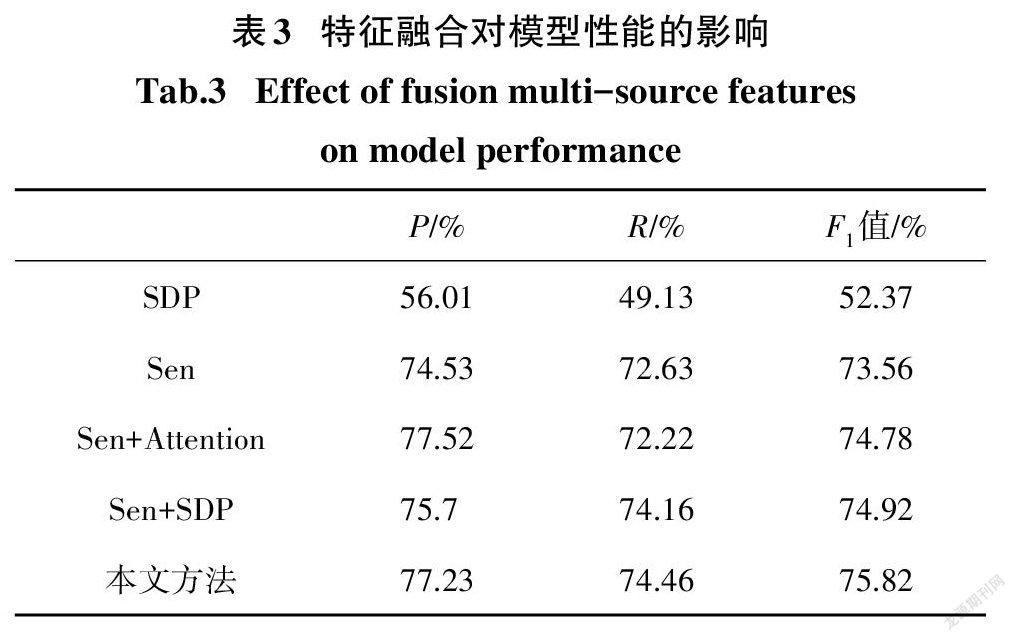
为了验证融合语义和依存关系特征的有效性,我们在本文模型的基础上实现了4种单纯使用句子序列特征或最短依存路径特征的简化模型,并与我们提出的结合语义和依存关系的DDIE方法进行了性能比较.这些对比模型包括:1)单纯利用最短依存路径特征的Bi-GRU模型(SDP);2)单纯利用句子序列特征的BI-GRU模型(Sen);3)单纯利用句子序列特征并结合了多头自注意力机制的Bi-GRU模型(Sen+Attention);4)SDP和Sen进行并行结合后版本(Sen+SDP).
由表3所示的实验结果可以看到,SDP模型的准确率、召回率和F值是所有对比模型中最低的.这是因为最短依存路径只保留了目标药物实体之间部分最重要的单词,容易丢失许多其他有用的信息,因而导致其识别精度受到较大影响.相比最短依存路径,句子序列包含了更为完整和丰富的信息,因此,直接利用句子序列进行特征学习的Sen模型获得了远高于SDP模型的性能.通过融合句子序列和最短依存路径两方面的特征,Sen+SDP模型获得了明显优于其他基于单一特征模型的性能,其准确率、召回率和F值分别达到了75.7%、74.16%和74.92%.此外,Sen+Attention虽然也仅仅使用了句子序列特征,但是,由于多头自注意力机制的引入,增强了模型获取长距离依赖特征的能力,所以,其超过其他两种基于单一特征的方法并获得了接近Sen+SDP的性能.本文提出的模型在融合语义和依存关系的基础上,利用多头自注意力机制进一步提升了模型的特征表示学习能力,因此取得了所有对比方法中最好的DDI分类效果,充分证明特征融合以及多头自注意力机制对于提升DDI的识别能力起到了有益的促进作用.
3.4.2与baseline方法的性能比较
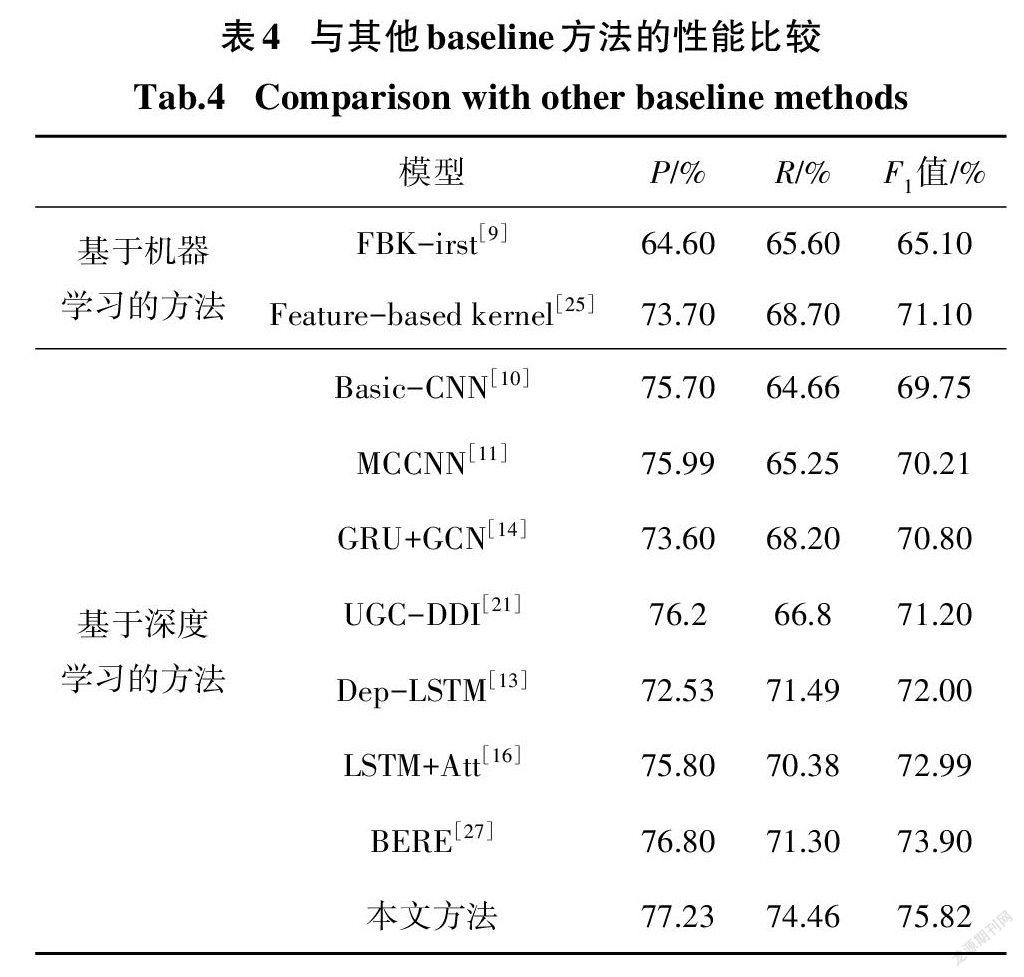
表4展示了几种代表性的药物相互作用关系抽取方法与本文方法的性能对比结果.从表中可以看出,基于机器学习的方法与基于深度学习的方法相比具有一定的差距.如,FBK-irst[9]的F值只有65.10%,是所有比较方法中最低的.尽管Feature- based kernel[25]通过使用多个人工制定的规则和特征有效地提高了DDI的识别性能,但其F值也仅为71.1%.这主要是因为这些方法通常利用依赖人工干预的传统特征工程来实现特征的提取,特征选择的主观性和不全面性往往会极大地影响该类方法的实际性能.
相比之下,基于深度学习的方法可以自动地、更广泛全面地捕获数据中的相关特征,具有更强的特征学习和表示能力.其中,MCCNN[11]通过不同的通道结合五个版本的词向量得到最终的特征表示,但是,由于忽略了句中单词的位置信息,仅获得了略高于Basic-CNN[10]的70.21%的F值.而GRU+GCN[14]和Dep-LSTM[13]融合了原始句子序列中的单词和语义特征以及从依存路径或句法图中得到的其他句法结构特征,所以,二者的性能均高于MCCNN. UGC- DDI[21]方法通过使用UGC(user generated content)资源为DDI的抽取提供更多有用的外部特征信息,因而也取得了较高的F值.LSTM+Att[16]除了从句子序列中学习特征外,还通过注意力机制来捕获全句各单词的位置信息,从而将模型的F值进一步提高到了72.99%.BERE[27]则是通过Tree-GRU等获得句子的向量表示后,进一步将实体的上下文特征嵌入到句子向量中,最后基于注意力机制加权求和得到最终特征表示进行分类,取得了73.9%的F值.值得注意的是,得益于对句子序列的语义特征、最短依存路径中的句法结构特征以及由多头自注意力机制提取的单词间依赖关系,本文方法的F值达到了75.82%,获得了最佳的性能.
3.4.3差错分析
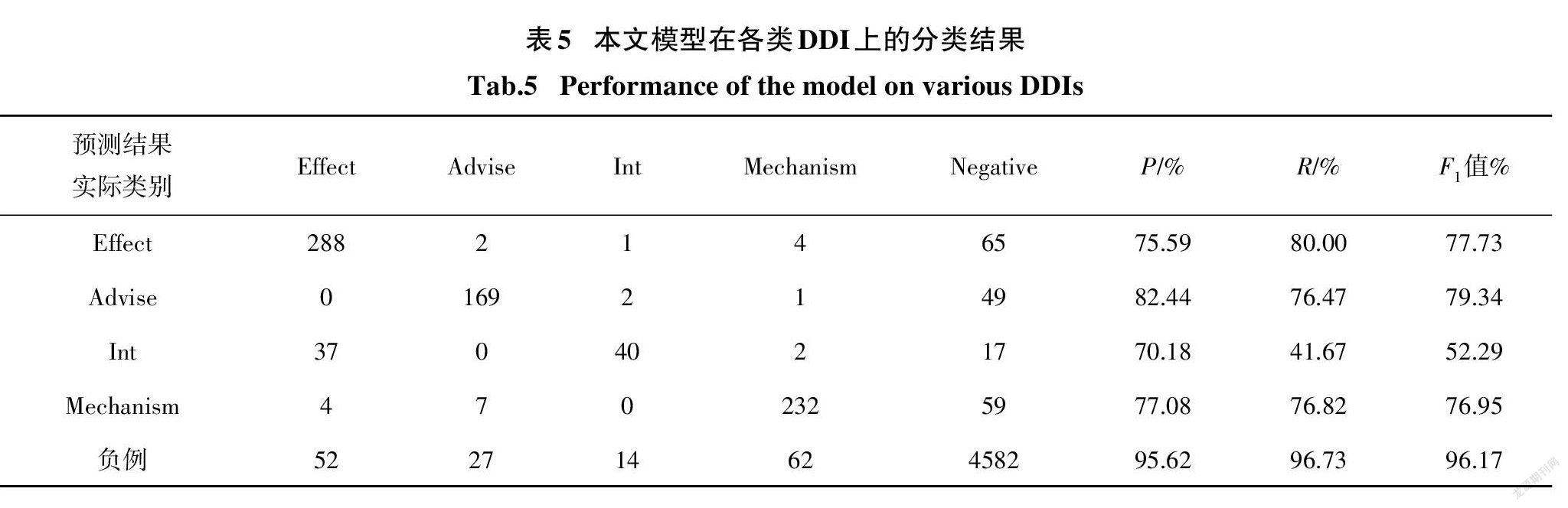
表5展示了本文方法对不同类别DDI的分类结果.从表中可以看出,一共有405个样本被错误分类.在4737个负样本中,有155个被误分为正样本,约占错误分类样本的38%;在979个正样本中有190个被错误分类为负样本,约占错误分类样本的47%. 这主要是因为描述药物相互作用的句子往往结构多变,对于在训练集中出现较少的句式难以学习到其中的特征.另外,通过观察正负样本被错误分类的句子,我们发现,大多数句中除了包含两个目标药物实体外还存在多个其他的非目标药物实体,这给目标药物实体对的关系识别带来了一定的干扰.从正样本中的四类DDI的识别效果来看,我们的模型在“Effect”“Advise”和“Mechanism”三类DDI上取得了较好的表现.其中,“Advise”类型DDI的抽取获得了最高的79.34%的F值.这是因为关于用药建议的描述形式通常较为标准和清晰,使得它们相对更易于区分. 然而,对于“Int”类型的DDI,由于训练样本较少(仅188个,不到其他类型训练样本量的25%),其分类性能最差,仅获得了52.29%的F值.同时,从表中我们还可以看到,在总共96个“Int”类型的测试样本中,56个被错误分类.其中,有37个(约66%)是因“Int”类型被误分类为“Effect”类型所致.这是由于部分句子仅模糊地描述了两种药物联合使用后的效果,从而导致模型在“Effect”类型和“Int”类型之间容易发生混淆.
4结论
药物相互作用关系抽取是生物医学关系抽取中的重要任务,现有的基于深度学习的方法往往仅从句子序列或其他外部信息中学习单一片面的特征,无法充分地利用文本中多方面的相关信息获得全面的特征表示,针对这一问题,本文提出了一种结合语义和依存关系的药物相互作用关系抽取方法.该方法在从句子序列中学习语义特征的基础上,利用Bi- GRU网络从目标药物实体的最短依存路径中获取相关句法特征,同时进一步结合多头自注意力机制来挖掘句子内部单词之间潜在的依存关系,获得长距离依赖特征,最终通过充分地融合多源特征有效地提升了生物医学文本中药物相互作用關系识别和抽取的整体性能.本文的不足之处在于,我们的方法只关注了同一句子内药物相互作用关系的提取,而没有考虑不同句子中药物实体之间的关系.在未来的工作中,我们将扩展模型句子间药物相互作用关系的识别和抽取能力,并在其他相关数据集上进一步验证模型的有效性.
参考文献
[1] CHO K,VAN MERRIENBOER B,GULCEHRE C,et al. Learning phrase representations using RNN encoder-decoder for statistical machine translation [C]// Proceedings of the 2014 Conference on Empirical Methods in Natural Language Processing (EMNLP). Doha,Qatar:Association for Computational Linguistics,2014:1724-1734.
[2] CHOWDHURY M F M,LAVELLI A. FBK-irst:A multi-phase kernel based approach for drug-drug Interaction detection and classification that exploits linguistic information [C]//Proceedings of the 7th International Workshop on Semantic Evaluation. Atlanta,Georgia,USA:Association for Computational Linguistics,2013:351-355.
[3] HINES LE,MURPHY J E. Potentially harmful drug - drug interactions in the elderly:a review[J]. The American Journal of (Geriatric Pharmacotherapy,2011,9(6):364-377.
[4] HOCHREITER S,SCHMIDHUBER J. Long short-term memory [J].Neural Computation,1997,9(8):1735-1780.
[5] HONG L,LIN J,LI S,et al. A novel machine learning framework for automated biomedical relation extraction from large-scale literature repositories[J]. Nature Machine Intelligence,2020,2:347-355.
[6] HONIG P K,WORTHAM D C,ZAMANI K,et al. Terfenadine- ketoconazole interaction:Pharmacokinetic and electrocardiographic consequences [J]. JAMA The Journal of the American MedicalAssociation,1993,269(12):1513-1518.
[7] KIM S,LIU H,YEGANOVA L,et al. Extracting drug - drug interactions from literature using a rich feature-based linear kernel approach[J]. Journal of Biomedical Informatics,2015,55:23-30.
[8] KLEIN D,MANNING C. Accurate Unlexicalized Parsing[C]// Proceedings of the 41st Annual Meeting of the Association for Computational Linguistics. Sapporo,Japan:Association for Computational Linguistics,2003:423-430.
[9] LIU S,TANG B,CHEN Q,et al. Drug-drug interaction extraction via convolutional neural networks[J]. Computational and Mathematical Methods in Medicine,2016,2016:1-8.
[10] MIRANDA V,FEDE A,Nobuo,et al. Adverse drug reactions and drug interactions as causes of hospital admission in oncology[J]. Journal of Pain & Symptom Management,2011,42(3):342-353.
[11] NIVRE J. Dependency parsing[J]. Language & Linguistics Compass,2010,4(3):138-152.
[12] PAULUS R,XIONG C,SOCHER R. A deep reinforced model for abstractive summarization[C]// Proceedings of the 6th International Conference on Learning Representations. Vancouver,BC,Canada:ICLR,2018. DOI:10.48550/arXiv.1705.04304.
[13] PYYSALO S,GINTER F,MOEN H,et al. Distributional semantics resources for biomedical text processing[C]// Proceedings of the 5th International Symposium on Languages in Biology and Medicine. 2013:39-44.
[14] QUAN C,HUA L,SUN X,et al. Multichannel convolutional neural network for biological relation extraction[J]. BioMed Research International,2016,2016:1-10.
[15] RAIHANI A,LAACHFOUBI N. Extracting drug-drug interactions from biomedical text using a feature-based kernel approach [J]. Journal of Theoretical & Applied Information Technology,2016,92 (1):109-120.
[16] SEGURA-BEDMAR I,MARTINEZ P,HERRERO-ZAZO M. SemEval-2013 task 9:Extraction of drug-drug interactions from biomedical texts (DDIExtraction 2013)[C]//Proceedings of the 7th International Workshop on Semantic Evaluation. Atlanta,Georgia,USA:Association for Computational Linguistics,2013:341-350.
[17] SEGURA-BEDMAR I,MARTINEZ P,PABLO-SANCHEZ C D. A linguistic rule-based approach to extract drug-drug interactions from pharmacological documents[J]. BMC Bioinformatics,2011,12(Suppl2):S1.DOI:10.1186/1471-2105-12-S1-S1.
[18] SEGURA-BEDMAR I,MARTINEZ P,PABLO-SANCHEZ C D. The 1st DDIextraction-2011 challenge task:Extraction of drugdrug interactions from biomedical texts[C]// Proceedings of the 1st Challenge Task on Drug-Drug Interaction Extraction. Huelva,Spain:CEUR,2011:1-9.
[19] TAN Z,WANG M,XIE J,et al. Deep semantic role labeling with self-attention[C]//Proceedings of the 32nd AAAI Conference on Artificial Intelligence. New Orleans,LA,USA:AAAI press,2018:4929-4936.
[20] TARI L,ANWAR S,LIANG S,et al. Discovering drug-drug inter- actions:a text-mining and reasoning approach based on properties of drug metabolism[J]. Bioinformatics,2010,26(18):i547 -i553.
[21] VASWANI A,SHAZEER N,PARMAR N,et al. Attention is all you need [C]// Proceedings of the 31st Annual Conference on Neural Information Processing Systems. Long Beach,CA,USA:Neural information processing systems foundation,2017:5999 - 6009.
[22] WANG Linlin,ZHU Cao,Melo G D,et al. Relation classification via multi-level attention cnns[C]//Proceedings of the 54th Annual Meeting of the Association for Computational Linguistics. Berlin,Germany:Association for Computational Linguistics,2016:1298-1307.
[23] WANG W,YANG X,YANG C,et al. Dependency-based long short term memory network for drug-drug interaction extraction [J]. BMC Bioinformatics,2017,18(S16):578. DOI:10.1186/ s12859-017-1962-8.
[24] XU B,SHI X,YIN Y,et al. Incorporating user generated content for drug drug interaction extraction based on full attention mechanism[J]. IEEE Transactions on Nanobioscience,2019,18(3):360-367.
[25] ZHAO D,WANG J,LIN H,et al. Extracting drug-drug interactions with hybrid bidirectional gated recurrent unit and graph convolutional network[J]. Journal of Biomedical Informatics,2019,99:103295.DOI:10.1016/j.jbi.2019.103295.
[26] ZHAO Z,YANG Z,LUO L,et al. Drug-drug interaction extraction from biomedical literature using syntax convolutional neural net- work[J]. Bioinformatics,2016,32(22):3444-3453.
[27] ZHOU D,MIAO L,HE Y L. Position-aware deep multi-task learning for drug - drug interaction extraction[J]. Artificial Intelligence in Medicine,2018,87:1-8.
