作物茬口对黑土农田土壤反硝化细菌数量及群落结构的影响
2022-11-10刘俊杰刘株秀顾海东张兴义王光华刘晓冰
王 璐,刘俊杰,刘株秀,顾海东,张兴义,王光华,刘晓冰
(1 东北农业大学资源与环境学院,黑龙江哈尔滨 150030;2 中国科学院东北地理与农业生态研究所 /黑土区农业生态重点实验室,黑龙江哈尔滨 150081)
反硝化作用是硝酸盐 (NO3-)逐步被还原成亚硝酸盐 (NO2-)、一氧化氮 (NO)、一氧化二氮 (N2O)和终产物氮 (N2)的四步还原过程[1-2]。NO2-还原为NO是反硝化细菌区别于其他硝酸盐呼吸细菌的关键过程[3],此步还原过程是由亚硝酸盐还原酶 (NIR)催化的,该酶具有两种结构不同但功能相同的形式,分别为nirS基因编码的cd1细胞色素还原酶和nirK型基因编码的含铜还原酶[4-5]。N2O是重要的温室气体,其全球变暖潜力比等摩尔量的CO2高出300倍[6]。农田是温室气体的主要来源,农业土壤排放的N2O约占全球人为排放N2O的60%,而土壤微生物介导的反硝化作用被认为是人为N2O排放的关键因素[7-8]。目前如何通过调控土壤反硝化作用减少N2O的排放备受关注[9]。
土壤微生物在土壤生态系统中发挥重要作用,是评价土壤生态系统功能的重要指标[10]。土壤微生物介导了包括固氮作用、硝化作用和反硝化作用等重要的土壤氮素循环过程[11]。大量研究证实,连作导致土壤理化性质恶化、土壤生物群落失衡,不仅影响农田生态系统的稳定性,而且显著降低作物产量,而轮作被认为是保持土壤肥力和土壤健康的一种合理耕作方式。周丽等[12]对四川紫色土反硝化细菌nirS型基因丰度的研究发现,玉米-大豆轮作体系的nirS型基因丰度显著高于连作处理。Behnke等[13]研究发现,与玉米-大豆轮作比,玉米连作的nirS型基因丰度无显著变化,但显著增加了nirK型基因丰度。Jude等[14]研究发现,轮作处理大豆茬口下,nirK型基因丰度显著高于轮作体系下的小麦茬口,而不同作物茬口的nirS型基因丰度没有显著变化。此外,研究结果表明,土壤类型[15]、作物茬口[16]和轮作年限[17]均对反硝化细菌数量和群落组成具有显著影响。因此,以反硝化nirS和nirK型基因为标记,开展不同作物茬口土壤反硝化微生物群落变化特征研究,有助于进一步认知土壤微生物参与的农田生态系统的生物地球化学循环过程。
黑龙江省农作物种植面积达10.99万hm2,粮食产量占全国的1/4,调出量占1/3,是我国粮食安全的“压舱石”和“稳压器”。黑龙江省是我国大豆的主产区,随着国家作物种植结构的调整和大豆产业振兴计划的实施,加之耕地面积和经济效益的制约,东北地区大豆连作现象有所加重,必将制约中国大豆生产力的提高,威胁中国大豆产业的可持续发展,是目前亟待解决的现实问题。连作则会导致土壤肥力不均衡,降低土壤肥力,引发连作障碍,从而导致土传病害加重,而轮作是改善土壤肥力、减少养分流失的有效技术措施[18]。目前,基于黑土农田土壤不同作物茬口下反硝化细菌群落的变化特征的研究相对较少。因此,开展该地区不同作物茬口下土壤反硝化细菌数量、多样性指数、群落组成及其与反硝化速率间的作用关系研究,对选择适宜的种植方式提高土壤氮素利用率和改善土壤生态功能具有重要意义。鉴于此,本研究采用qPCR和高通量测序技术对黑土区玉米连作、大豆连作和玉米-大豆轮作体系下土壤反硝化细菌数量和群落特征进行综合分析,旨在从微生物生态学角度,明确不同作物茬口下反硝化细菌群落的变化特征,阐明不同作物茬口影响N2O排放的微生物学机制,为提出减少温室气体排放的种植方式提供理论依据。
1 材料与方法
1.1 试验设计与土壤样品采集
利用2013年建立于中国科学院东北地理与农业生态所海伦水土保持监测研究站的长期定位试验开展研究。试验站位于黑龙江省海伦市前进乡光荣村(47°23′N,126°51′E),海拔高度为 210 m;是温带大陆性季风气候,冬季干燥寒冷,夏季炎热多雨,平均年降水量为530 mm,年均气温1.5℃,极端最高温度为37℃,极端最低温度为-39.5℃,土壤类型为典型黑土,该地区是雨养农业区。
该定位试验2013年开始种植玉米,2014年种植大豆,由此循环。该样地采用人工播种、人工除草、机械中耕的方法。选用定位试验中的4个处理,分别为:大豆连作 (CC)、玉米连作 (SS)、玉米-大豆轮作玉米茬口 (CSC)和玉米-大豆轮作大豆茬口 (SSC)处理。随机区组设计,4次重复,每小区12条垄,垄距为0.7 m,垄长为9 m,每个小区面积为75.6 m2。种植的大豆和玉米品种分别为‘东生1号’和‘兴垦5号’,种植大豆小区施用磷酸二铵150 kg/hm2、尿素50 kg/hm2和硫酸钾50 kg/hm2;玉米小区施用磷酸二胺150 kg/hm2、尿素70 kg/hm2和硫酸钾50 kg/hm2,并在玉米生长的V6期追施225 kg/hm2尿素。
于2018年大豆成熟期和玉米蜡熟期,采集16个土壤样品,土样过2 mm筛后分为3份,一份于-80℃保存,用于土壤微生物DNA提取;一份风干,用于土壤理化性质测定;一份于4℃保存,用于反硝化速率测定。
1.2 土壤理化性质测定
土壤pH采用pH计 (PHS-3C,中国)测定 (w土:v水为 1 : 5 )。利用元素分析仪 (VarioELIII,德国)测定土壤全碳和全氮含量。土壤全磷、速效磷、铵态氮 (NH4+-N)和硝态氮 (NO3--N)含量用连续流动分析仪 (SKALAR SAN++,荷兰)测定。利用电感耦合等离子发射光谱 (ICPS-7500,日本)测定土壤全钾和有效钾含量。
1.3 土壤反硝化速率
采用乙炔抑制法测定土壤的反硝化速率[19]。取4℃保存的土壤样品于28℃活化7天,取10 g活化后的土样放入集气瓶中,加入10 mL无菌蒸馏水,提供厌氧环境。集气瓶瓶盖插入两个针头,一个充氮气,一个排空气,待排除瓶内空气后,抽取15 mL氮气,加乙炔15 mL,震荡1 h,25℃培养12 h。由于乙炔气体的存在抑制了N2O向N2的还原,所以N2O成为了反硝化作用的最终产物,最后通过气相色谱 (GC-2010,日本)测定集气瓶中N2O的浓度,用以表征土壤反硝化作用的强弱。
1.4 土壤总DNA提取、qPCR和Illumina MiSeq测序
称取土壤样品0.5 g,采用Fast DNA®Spin Kit for Soil (MP Biomedicals,美国)试剂盒,按照说明书提取土壤总DNA,并使用NanoDrop®2000 (Thermo Scientific, USA)检测DNA浓度和纯度,将提取的DNA放入-20℃冰箱中保存。
利用引物 Cd3aF (5′-GTSAACGTSAAGGARACSGG-3′)/R3cdR (5′-GASTTCGGRTGSGTCTTGA-3′)[20]和 F1aCu (5′-ATCATGGTSCTGCCGCG-3′)/R3Cu (5′-GCCTCGATCAGRTTGTGGTT-3′)[21],分别对反硝化细菌nirS型基因和nirK型基因进行扩增。利用 LightCycler®480 thermocycler (Roche Applied Science)进行qPCR反应。将拷贝数已知的质粒DNA,按10倍梯度稀释做标准曲线,每个标准样品做4次重复。qPCR反应体系包括10 μL SYBR®Premix Ex Taq (Takara,大连,中国)、1 μL 土壤样品 DNA、1 μL 10 μmol/L 正向引物、1 μL 10 μmol/L反向引物和7.0 μL H2O。反应条件为:95℃初变性30 s,然后95℃变性5 s,60℃退火和延伸30 s,共25个循环,最后50℃保持30 s。
采用上述相同的引物,对nirS和nirK型基因进行高通量测序分析。区别于qPCR分析,高通量测序分析每个引物5′端被样本特异的barcode所修饰,以区分土壤样本的处理来源。PCR体系共25 μL,包含 2.5 μL 10× PCR buffer、2 μL 2.5 mmol/L dNTPs、0.25 μL Taq 酶 (5 U/μL) (TaKaRa,大连,中国)、0.5 μL正向和反向引物,2 μL样品DNA为模板,用灭菌超纯水补足至25 μL。PCR反应条件:95℃初变性5 min,然后95℃变性1 min,63℃复性1 min和72℃延伸1 min,共30个循环,最后72℃延伸5 min。每个样品做3次重复,PCR产物经凝胶电泳检测,并利用胶纯化试剂盒 (TaKaRa,大连,中国)进行纯化。利用Illumina MiSeq平台 (上海美吉生物公司,中国)对纯化后的PCR产物进行测序分析。
1.5 测序数据分析
基于Illumina PE 300测序平台,测序结果通过QIIME Pipeline Version 1.9.0软件进行分析 (http://qiime.org/tutorials/tutorial.html)。首先,将获得的反硝化细菌FASTQ原始序列文件,进行序列拆分和质量控制[22]。利用FLASH (fast length adjustment of short reads)软件,对质控序列进行双端拼接,去除序列短于200 bp以及平均质量得分小于20的序列信息[23]。将得到的优质序列,以97%相似度水平进行OTU(operational taxonomic units)聚类。将每个OTU的代表性序列翻译成氨基酸序列,然后在NCBI上比对。使用Mega软件将代表性序列与参考序列对比,构建系统发育树。将每个样品的序列,按最小序列数进行抽平处理,用于后续生物信息学分析。
1.6 统计分析
基于SPSS (Version 18.0)软件,利用单因素方差分析 (one-way ANOVA)对不同处理的土壤理化性状、反硝化速率和反硝化细菌相对丰度进行差异显著性分析。利用R (Version 3.3.1)软件 (R Development CoreTeam, 2006)的“vegan”包,进行基于Bray-Curtis距离的主坐标分析 (principal co-ordinates analysis,PCoA)和冗余分析 (redundancy analysis,RDA),以分析不同处理间反硝化细菌群落的差异及其主要驱动因素。用R软件的“pheatmap”包进行反硝化细菌相对丰度与反硝化速率间相关性的Heatmap分析。此外,利用SPSS AMOS软件 (Version 24.0,Chicago,IL: Amos Development Corporation)构建结构方程模型 (structural equation models,SEM),以解析土壤理化性质、nirS型和nirK型反硝化细菌基因丰度和群落结构对反硝化速率的影响。采用方差膨胀系数(variance inflation factor,VIF)分析,构建以土壤全氮、C/N和NO3--N为最佳土壤理化因子的SEM结构方程模型。
2 结果与分析
2.1 土壤理化性质变化的差异
表1显示,4个处理的土壤pH、全碳、全钾以及速效钾含量无显著差异 (P>0.05)。与连作处理CC和SS相比,轮作处理的CSC和SSC显著提高了土壤全氮含量,而降低了土壤C/N (P<0.05)。此外,CSC显著增加了铵态氮 (NH4+-N)和硝态氮 (NO3--N)含量,但显著降低了土壤全磷含量,SSC显著增加了土壤速效磷含量 (P<0.05)。

表1 不同作物茬口对土壤理化性状的影响Table 1 The effects of crop stubble in different cropping systems on soil physical and chemical properties
2.2 土壤反硝化速率及反硝化细菌丰度变化差异
4个处理间土壤反硝化速率差异显著 (P<0.05)(图1)。其中,CC处理的土壤反硝化速率最高,达到21.15 μg/(g·h);其次是轮作处理CSC和SSC,反硝化速率分别为16.93和16.48 μg/(g·h)。SS处理的土壤反硝化速率最低,为7.00 μg/(g·h),相比于CC、CSC和SSC处理,分别降低66.9%、58.7%和57.5% (P<0.05)。
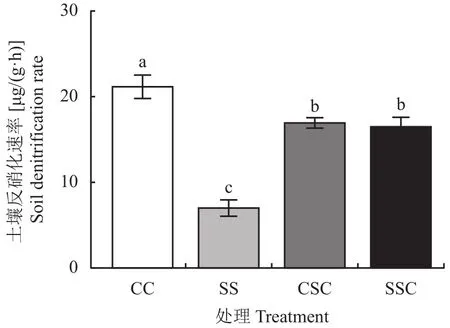
图1 不同作物茬口对土壤的反硝化速率的影响Fig.1 Effects of crop stubble in different cropping systems on soil denitrification rate
不同处理条件下,反硝化细菌nirK型基因拷贝数为 6.83×106~10.31×106/ (g, soil),nirS型基因拷贝数为 2.59×107~3.48×107/ (g, soil) (图2)。轮作处理SSC和CSC中nirS和nirK型基因拷贝数均显著高于SS处理 (P<0.05),SSC处理的nirK型基因拷贝数显著高于CC处理 (P<0.05),而SSC和CSC的nirS型基因拷贝数与CC处理无显著差异 (P>0.05)。
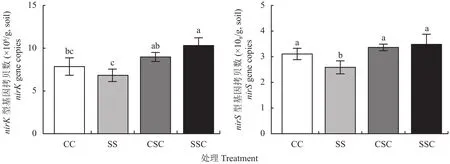
图2 不同作物茬口对反硝化基因丰度的影响Fig.2 The effects of crop stubble in different cropping systems on denitrification gene abundance
2.3 土壤反硝化速率与反硝化细菌的相关性
本研究分别获得了471和1055个nirS和nirK型反硝化细菌OTUs。土壤nirS和nirK型反硝化细菌OTUs与反硝化速率的相关性分析结果 (图3)表明,nirS型基因中,OTU225 (罗河杆菌属Rhodanobacter)、OTU357 (草螺菌属Herbaspirillum)等30个OTUs与土壤反硝化速率呈显著负相关关系;OTU63 (噬氢菌属Hydrogeaophaga)、OTU472 (慢生根瘤菌属Bradyrhizobium)等9个OTUs与土壤反硝化速率呈显著正相关关系,其余OTUs与土壤反硝化速率则无显著相关性。在nirK基因中,OTU346 (慢生根瘤菌属Bradyrhizobium)、OTU159 (中慢生根瘤菌属Mesorhizobium)等24个OTUs与土壤反硝化速率呈显著正相关;而OTU281 (根瘤菌属Rhizobium)、OTU258 (中慢生根瘤菌属Mesorhizobium)等65个OTUs与反硝化速率呈负相关,其余966个OTUs与土壤反硝化速率无显著相关性。
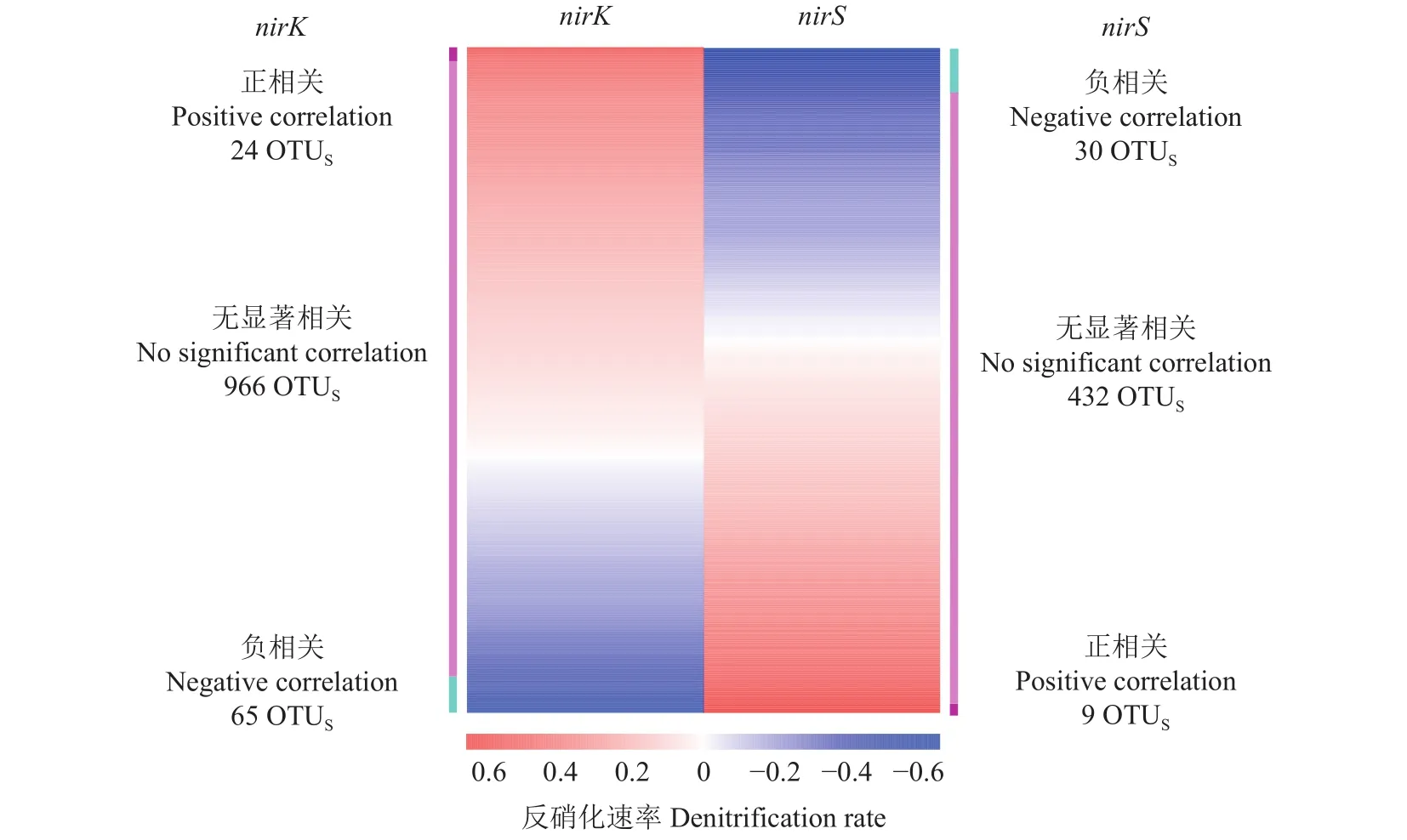
图3 土壤反硝化速率与反硝化细菌的相关性分析Fig.3 Correlation between soil denitrification rate and denitrifying bacterial communities
2.4 nirS型和nirK型反硝化细菌群落的变化
图4列出了土壤nirS和nirK型反硝化细菌中与反硝化速率显著正相关的8个属 (OTUs)在不同茬口土壤总的相对丰度。CC和CSC处理的nirS和nirK型反硝化细菌相对丰度均高于甚至显著高于SS处理。除OTU261 (慢生根瘤菌属Bradyrhizobium)和OTU307 (中慢生根瘤菌属Mesorhizobium)的相对丰度显著高于CSC处理外,CC处理其余的OTUs基因丰度与CSC处理的多无显著差异。nirK型反硝化细菌中,CC处理OTU346 (慢生根瘤菌属Bradyrhizobium)、OTU376 (假单胞菌属Pseudomonas)和OTU307 (中慢生根瘤菌属Mesorhizobium)在CSC和SSC处理中的相对丰度差异不显著,而OTU261 (慢生根瘤菌属Bradyrhizobium)在轮作体系SSC处理中相对丰度显著高于CSC处理,且均明显高于SS处理 (P<0.05)。nirS型反硝化细菌中,轮作体系CSC处理的OTU472(慢生根瘤菌属Bradyrhizobium)、OTU129 (新草螺菌属Noviherbaspirillum)和OTU100 (草螺菌属Herbaspirillum)相对丰度均显著高于SSC处理,且OTU129和OTU100的相对丰度显著高于SS处理(P<0.05)。
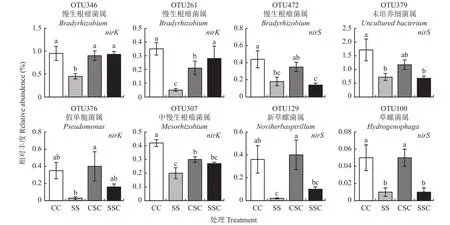
图4 不同作物茬口土壤下nirK和nirS型细菌中与反硝化速率正相关的8个属(OTUs)的基因相对丰度Fig.4 The relative gene abundance of the eight OTUs is positively related to the denitrifying rate of nirK or nirS in soils with different crop stubbles
2.5 不同茬口反硝化细菌群落结构及其驱动因素
基于bray-curtis距离的PCoA分析结果(图5)表明,PCoA1和PCoA2分别解释了nirS型反硝化细菌群落结构变化的47.72%和7.83%。根据Adonis分析,nirS型反硝化细菌群落中SSC处理和CSC处理各自单独聚类 (Adonis test,P=0.028),而CC和SS处理群落相似度较大 (Adonis test,P=0.212)。对于nirK型反硝化细菌而言,PCoA1和PCoA2分别解释了群落结构变异的34.36%和14.46%。Adonis分析表明,CC处理和SS处理的nirK型反硝化细菌群落存在显著差异 (Adonis test,P=0.036),而SSC处理和CSC处理在PCoA分析图谱中聚集较为紧密,其群落相似度较高 (Adonis test,P=0.134)。此外,RDA分析结果表明,NO3--N和C/N分别是nirS和nirK型反硝化细菌群落分异的最主要驱动因子。
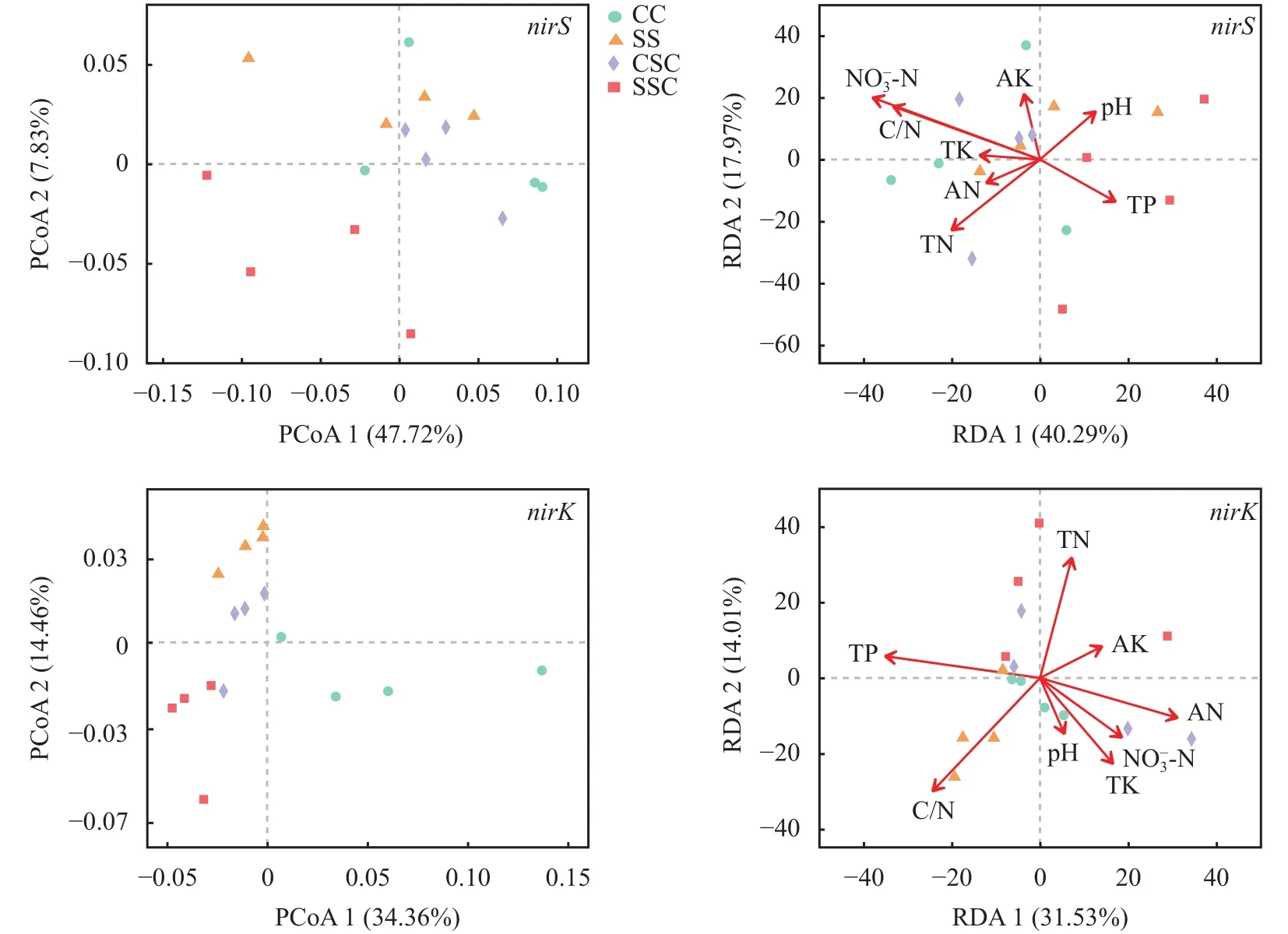
图5 不同作物茬口土壤中反硝化细菌群落结构及其驱动因素Fig.5 Denitrifying bacterial community structures and their driving factors in soils with different crop stubbles
2.6 土壤理化性质、nirS和nirK型反硝化细菌群落结构和基因丰度对反硝化速率的影响
本研究利用结构方程模型 (SEM)探究了土壤理化性质、反硝化基因丰度及反硝化细菌群落结构对土壤反硝化速率的影响 (图6)。SEM分析结果表明,土壤全氮与nirS型 (R2=-0.62)和nirK型 (R2=-0.66)反硝化细菌的基因丰度呈显著负相关,与土壤反硝化速率呈显著正相关 (R2=0.78)。土壤C/N和NO3--N均通过影响nirS型 (R2=0.65)和nirK型 (R2=0.59)反硝化细菌群落多样性,间接影响土壤反硝化速率。此外,nirS型反硝化细菌群落多样性与反硝化速率呈显著正相关 (R2=0.92),nirK型反硝化细菌群落多样性与反硝化速率呈显著负相关 (R2=-0.81),而nirS和nirK型基因丰度与土壤反硝化速率无显著相关关系。
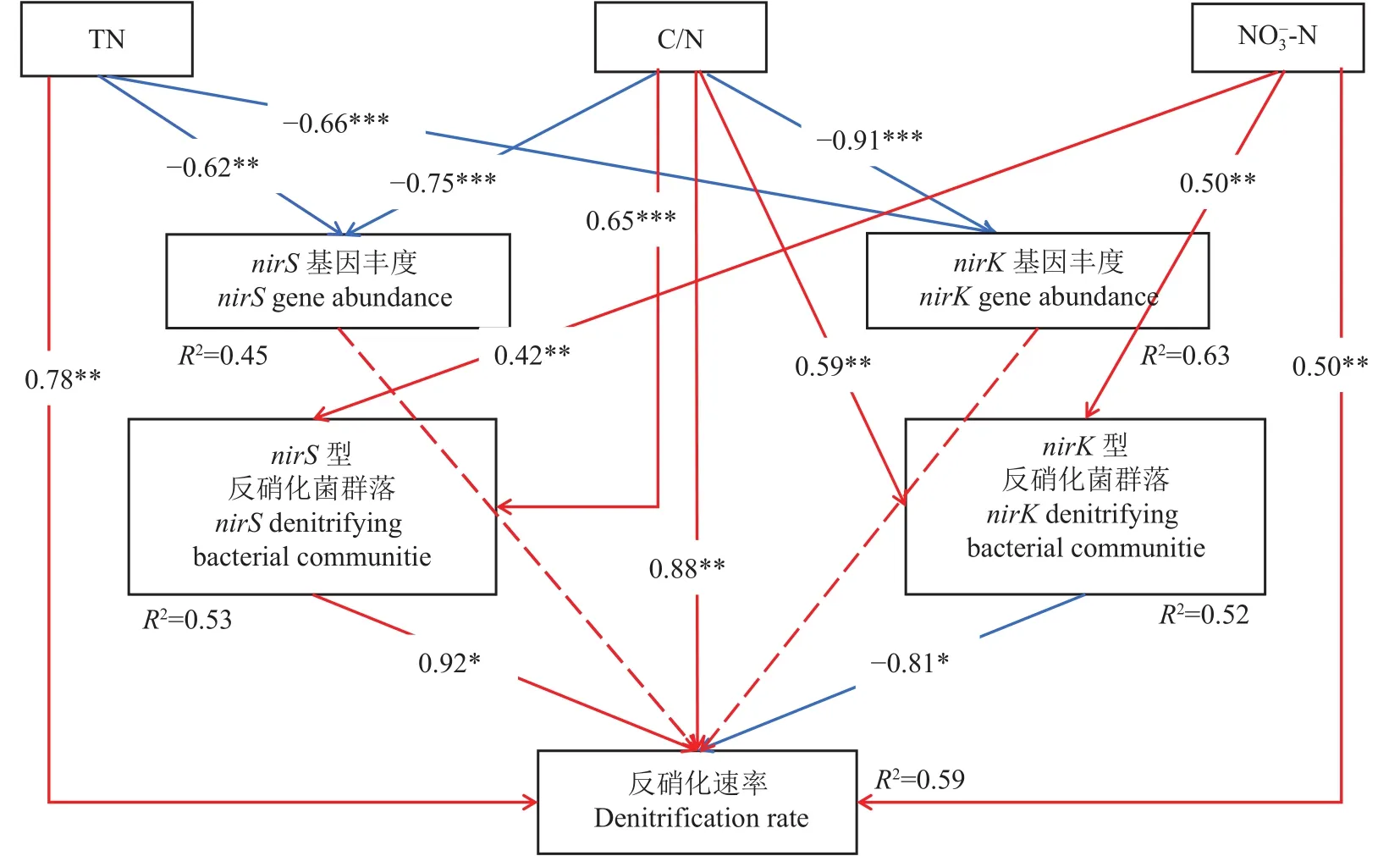
图6 土壤理化性质与反硝化细菌群落对反硝化速率的直接与间接影响Fig.6 Direct and indirect effects of soil physical and chemical properties and denitrifying bacterial communities on soil denitrification rate
3 讨论
3.1 作物茬口对土壤理化性质、反硝化细菌丰度和反硝化速率的影响
作物茬口显著影响着土壤的理化性质。与大豆连作相比,玉米-大豆轮作显著增加了土壤pH、全氮、全碳和速效养分含量[24-25]。本研究中,不同作物茬口的土壤pH和全碳含量无显著差异,可能与本研究的试验持续时间较短 (第5年)、轮作倒茬仅两次有关。然而,与玉米连作和大豆连作处理相比,玉米-大豆轮作体系处理显著增加了土壤全氮含量(表1)。利用相同的土壤样本,Zou等[26]研究发现,玉米-大豆轮作导致大豆根瘤菌数量增加,大豆从空气中固定更多的氮素,进而增加了土壤全氮含量。
在本试验中,nirS型反硝化细菌的基因丰度远远高于nirK型细菌,表明短期内nirS型反硝化细菌可能比nirK型细菌在土壤反硝化中发挥更重要的作用[27-28]。Wang等[29]研究发现,nirK型反硝化细菌主要在深层土壤中富集,而本研究采集的土壤样品为表层 (0—15 cm)土壤,这可能是导致nirK型反硝化细菌丰度低于nirS型反硝化细菌的原因。本研究同时发现,玉米连作处理的土壤反硝化速率显著高于其他处理,这很可能与玉米种植时高的氮肥投入量有关。已有研究均发现,氮肥能显著增加土壤反硝化细菌数量,进而促进土壤反硝化作用[30-31]。黄国宏等[32]研究表明,土壤N2O排放随着种植作物的不同而发生改变,种植玉米的土壤N2O排放量比种植大豆的土壤增加了103.7%。
3.2 作物茬口对nirS和nirK型反硝化细菌群落组成的影响
本研究中,通过分析nirS和nirK型反硝化细菌相对丰度与反硝化速率呈正相关的OTUs,发现玉米连作处理的反硝化细菌相对丰度显著高于大豆连作处理,说明种植玉米施用的氮量可导致反硝化微生物数量的增加,其在加速作物对养分吸收利用的同时也加速了土壤反硝化速率[33-34]。有趣的是,Zou等[26]利用相同土壤样本对不同作物茬口下土壤固氮微生物群落的研究发现,nifH型基因丰度在种植相同茬口的作物中,轮作处理下的基因丰度均显著高于连作处理。然而,本研究发现,nirS和nirK基因丰度未呈现与nifH基因丰度变化一致性规律,尽管玉米-大豆轮作玉米茬的相对丰度多显著高于连作大豆,仅玉米-大豆轮作大豆茬的OTU346 (Bradyrhizobiumsp.)和OTU261 (Bradyrhizobiumsp.)丰度高于大豆连作,说明反硝化细菌群落组成受到氮肥施用量和作物茬口的双重制约,且氮肥施用是反硝化细菌群落分异的主导因素[35-37]。此外,nirS型反硝化细菌草螺菌属 (Hydrogenophaga)在玉米茬口下丰度显著高于大豆茬口,Gabriela等[38]研究证明氮肥施用量相比于作物茬口对草螺菌属的影响更大。与nirK型反硝化细菌变化特征相比,nirS型反硝化细菌群落中玉米-大豆轮作大豆茬口和大豆连作处理相对丰度及大豆-玉米轮作玉米茬口和玉米连作处理相对丰度均无显著差异。已有研究发现,nirS型反硝化细菌对于土壤中可利用资源的竞争能力显著高于nirK型反硝化细菌,致使nirS型反硝化细菌的环境适应能力更强且不易受到作物茬口的影响[39-40]。
3.3 土壤理化性质和反硝化细菌群落对土壤反硝化速率的影响
基于PCoA结果显示,玉米-大豆轮作体系处理中nirS型反硝化细菌群落差异显著,大豆连作和玉米连作处理群落相似度较高,而nirK型反硝化细菌群落则呈现完全相反的趋势,说明不同作物茬口对nirS和nirK型反硝化细菌群落结构的影响存在分异。Teele等[41]研究表明,nirS型反硝化细菌群落偏好氧浓度较高的环境,而nirK型反硝化细菌群落对土壤氧气水平不敏感,为此轮作导致的土壤氧气含量升高是导致nirS型反硝化细菌在轮作处理中差异显著,而连作处理下群落相似度高的主要原因。结合RDA分析和SEM分析结果发现,土壤全氮、土壤C/N和土壤硝态氮对nirS和nirK型反硝化群落结构存在显著影响,这与以往研究结果一致,即土壤氮素作为反硝化作用的底物其强烈影响反硝化微生物群落的结构[42-43]。
已有研究发现,nirK型反硝化细菌群落对调节反硝化速率有显著影响[44-45]。本研究SEM结构方程分析同样发现,nirS型反硝化细菌群落与反硝化速率显著正相关,说明东北黑土中nirS型反硝化细菌群落可能对土壤反硝化作用起到更重要的作用。这一结论与Yin等[46]研究结果一致,即黑土区农田土壤中携带nirK型基因的反硝化细菌与反硝化速率无显著相关关系,而nirS型反硝化细菌是土壤反硝化作用的主要贡献者。本研究发现,反硝化基因丰度与反硝化速率间不存在显著相关性 (nirS:P=0.395;nirK:P=0.597),此外,SEM分析也发现,nirS和nirK型基因丰度对反硝化速率变化没有显著影响,而反硝化细菌群落组成与反硝化速率显著相关 (图6)。采用qPCR技术对土壤反硝化基因丰度变化研究均发现,土壤反硝化基因丰度与土壤反硝化速率之间不存在显著相关性[47-49]。本研究和上述报道均证明,反硝化基因丰度仅表征了反硝化微生物的潜在生理功能,而反硝化细菌的多样性特征和群落组成是土壤反硝化作用发生的决定因素。
4 结论
连作玉米茬口土壤中nirS和nirK型反硝化细菌的相对丰度多显著高于连作大豆茬口,轮作玉米和大豆茬口与连作大豆茬口也存在显著性差异,但未表现出一致的规律性变化特征。轮作体系中大豆茬口和玉米茬口nirS型反硝化细菌群落构成差异显著,而连作玉米和连作大豆在nirK型反硝化细菌群落构成间存在显著差异。反硝化速率与nirS型反硝化细菌群落构成显著正相关,与nirS和nirK型基因丰度无显著相关性。土壤NO3--N和C/N含量是驱动黑土中nirS型反硝化细菌群落构成的主要因素。
