不同施肥处理农田土壤中噬菌体与细菌携带抗生素抗性基因的比较
2022-11-05胡雪莹张越郭雅杰仇天雷高敏孙兴滨王旭明
胡雪莹 张越 郭雅杰 仇天雷 高敏 孙兴滨 王旭明
(1. 东北林业大学林学院, 哈尔滨150040;2. 北京市农林科学院生物技术研究所 农业基因资源与生物技术北京市重点实验室,北京 100097)
抗生素广泛使用导致的细菌耐药问题已对全球公共卫生构成了重大威胁。抗生素耐药菌携带的抗生素抗性基因(antibiotic resistance genes,ARGs)被认为是一种新型的环境污染物[1],引起国内外的广泛关注。土壤是自然环境中ARGs的最大储存库[2]。很多土壤微生物具有内在的抗生素抗性[3],而人类活动如粪肥施用、再生水灌溉等,使土壤ARGs的多样性和丰度显著增加[4-5]。土壤ARGs可通过接合、转化和转导作用进行水平转移[6],使抗生素抗性在土壤微生物间扩散传播。其中,质粒等可移动遗传元件(mobile genetic elements,MGEs)介导的接合作用被认为是细菌ARGs水平转移的主要方式[7-8]。尽管噬菌体介导的转导作用可以转移供体细胞内的任何DNA片段(包括质粒DNA)给受体细胞,但是噬菌体与抗生素抗性之间的关系一直未得到重视和深入研究[2]。这在一定程度上限制了人们对土壤微生物抗生素抗性的全面认知,以及对耐药细菌快速进化机制的详细了解。
噬菌体是地球上丰度最高的生物体,其数量约为1031-1033,是细菌的10倍[9]。最近的研究表明,河水、土壤、污水、畜禽粪便等环境噬菌体基因组中都存在ARGs[10-13]。农田土壤中种类丰富、数量巨大的细菌和噬菌体为它们的相互作用以及基因转移提供了物质基础。由于噬菌体能通过转导作用转移ARGs,因此噬菌体DNA中ARGs的风险可能比细菌中的更大[14]。尽管土壤噬菌体中ARGs的存在已被证实,但对其多样性和丰度的认识还不够全面,不同肥料长期施用对土壤噬菌体中ARGs的影响也鲜有研究报道。本文以北京某蔬菜基地长期施用化肥和有机肥的土壤为研究对象,采用微滴数字PCR(droplet digital PCR,ddPCR)绝对定量检测方法,初步探讨土壤噬菌体携带ARGs的多样性与丰度,揭示化肥和有机肥施用对土壤噬菌体中ARGs的影响差异,并与土壤细菌中的ARGs进行对比分析,旨在为评估噬菌体对土壤抗生素抗性的贡献奠定基础。
1 材料与方法
1.1 材料
选择位于北京大兴区某蔬菜基地(39°66'N,116°57'E)的温室,于2020年10月采集土壤样品。该温室面积488 m2(61 m×8 m),于2009年开始采用3种不同的施肥处理方式:不施肥(CK)、单施化肥(IF)、单施有机肥(OF)。每个处理设置3个小区,各处理之间设50 cm隔断,以避免干扰。每年种植2茬蔬菜,上半年种植茄子,下半年种植甜椒,在每茬蔬菜种植前进行1次施肥,每次施肥量如表1所示。采样时地表种植作物为甜椒,选用五点采样法采集每种处理中3个小区的表层土壤(0-20 cm)作为3个重复。所有样品用冰袋覆盖运输,当日到达实验室后过2 mm筛,均匀分成两份,一份用于理化性质测定,另一份保存于-80℃冰箱中,用于后续DNA提取。

表1 不同施肥处理的施肥量Table 1 Fertilizer amount of different treatments
1.2 方法
1.2.1 土壤理化性质的测定 对3种不同施肥处理农田土壤中的重金属元素(As、Hg、Cu、Cr、Cd、Pb、Zn)、酸碱度(pH)、有机质(OM)、有效磷(AP)、有效钾(AK)、总氮(TN)进行测定。重金属As、Hg用硝酸-盐酸消解后用原子荧光光谱法测定,Cu、Cr、Cd、Pb、Zn用硝酸-盐酸消解后用火焰原子吸收光谱法和石墨炉原子吸收光谱法测定。在土水比为1∶2.5的条件下,用pH计测定土壤pH。采用重铬酸盐氧化法测定土壤OM,碳酸氢钠法测定AP,醋酸铵萃取-火焰光度法测定AK,凯氏定氮法测定TN。
1.2.2 土壤细菌DNA和噬菌体DNA的提取 将20 g土壤样品和20 mL PBS缓冲液1∶1混合,加入50 mL无菌离心管中,加入丝裂霉素C至终浓度1 μg/mL[15]。混合液于 30-50 r/min 下 28℃过夜培养,涡旋1 h后于4℃ 3 000×g离心10 min,保留上清液,将土样重新悬浮,重复3次。收集好的上清液依次经 5 μm、0.45 μm 及 0.22 μm 孔径 PES滤膜过滤以去除土样中的细菌[16]。将滤膜截留的细菌用试剂盒(FastDNA SPIN Kit For Soil,美国)提取DNA,按照说明书要求进行操作;获得的含噬菌体的滤液,用100 kD超滤管(Millipore,美国)超滤浓缩至1 mL,加入DNase I和RNase A至终浓度为100 U/mL[12],37℃ 孵 育 2 h, 加 入 EDTA(0.5 mol/L,pH=8.0)至终浓度20 mmol/L,65℃加热5 min,用试剂盒(TIANGEN,TIANamp Virus DNA/RNA Kit,中国)提取噬菌体DNA。用16S rRNA通用引物对每个噬菌体DNA提取物进行PCR检测,超纯水作为阴性对照,保留结果为阴性(即不含细菌DNA)的噬菌体DNA样本,再用Qubit 3.0检测DNA浓度,以便后续分析。
1.2.3 ARGs及MGEs的ddPCR定量检测 采用ddPCR对细菌DNA和噬菌体DNA中ARGs和MGEs进行定量检测。本研究中,选择了氨基糖苷、β-内酰胺、大环内酯-林肯酰胺-链阳性菌素B(MLSB)类等9大类共25种ARGs亚型和1种常见的MGE(I类整合子,intl1)进行分析(表2)。ddPCR是在水-油系统中将含有核酸分子的反应体系形成数万个独立的纳升级微滴,经PCR扩增后,对微滴中核酸靶分子进行检测来实现绝对定量。ddPCR反应体系为 20 μL:QX200 ddPCR EvaGreen Supermix(美国,Bio-Rad)10 μL,上、下游引物(10 μmol/L)各 0.2 μL,ddH2O 8.6 μL,模板 1 μL。反应程序为 95℃预变性10 min,95℃变性30 s,退火30 s(各引物序列和退火温度见表2),72℃延伸30 s,40 个循环后于4℃保存,升温速率2.5℃/s。每个样品测定3个技术重复。ARGs和intl1拷贝数(copies/g)=检测得到的拷贝数(copies/μL)×体系(20 μL)×DNA稀释倍数×DNA洗脱体积(μL)/DNA提取的样品质量(g)。
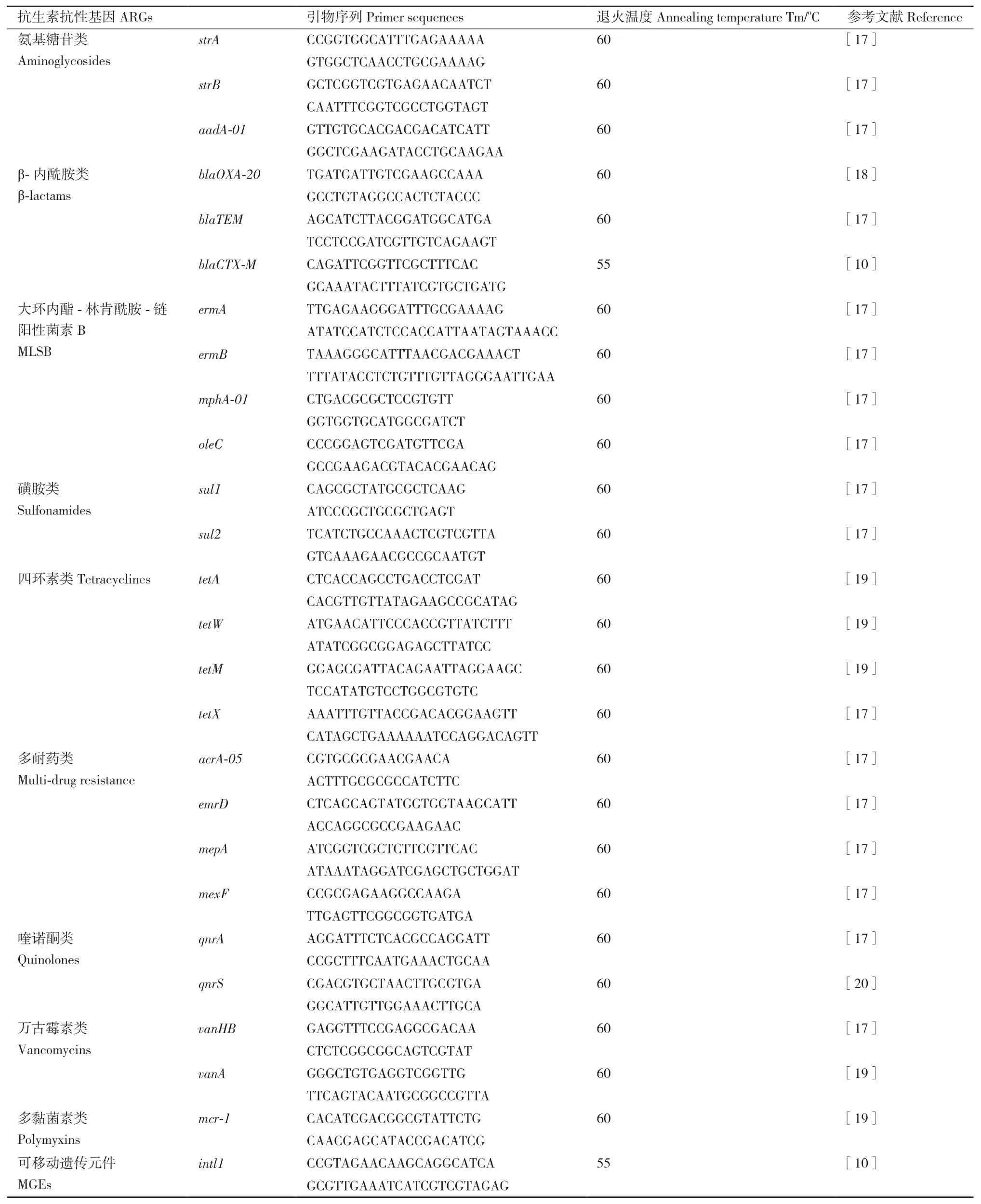
表2 ARGs及I类整合子intl1 PCR引物信息Table 2 PCR primer information of ARGs and class I integrase gene(intl1)
1.2.4 数据分析 检测数据经Excel进行初步计算整理,采用IBM SPSS 24.0软件进行单因素方差分析和相关性分析,在P<0.05时确定统计显著性。利用Origin 2021软件进行直方图与拟合曲线的绘制。采用R 3.6.1进行环境因子与ARGs关系的冗余分析(redundancy analysis,RDA)。
2 结果
2.1 不同施肥处理农田土壤噬菌体与细菌中ARGs的多样性
对3种施肥处理的土壤样品中噬菌体和细菌携带ARGs和intl1进行检测,结果如表3所示。总体上看,土壤细菌DNA中共检出23种ARGs亚型,噬菌体DNA中共检出20种。所有土壤细菌和噬菌体样品均未检出的ARGs有2种:β-内酰胺类抗性基因blaOXA-20和喹诺酮类抗性基因qnrA。同时发现,所有在噬菌体中检测到的ARGs都能在细菌中检出,二者均检出了intl1。细菌携带的ARGs在IF和OF中检出率最高,均为92%;在CK中,还有β-内酰胺类抗性基因blaCTX-M未被检测出,ARGs检出率为88%。对于所有处理的土壤噬菌体,除了未检出以上目标基因之外,也没有发现MLSB类抗性基因ermA、ermB和磺胺类抗性基因sul1。此外,CK处理未检测出strA、blaCTX-M、qnrS,ARGs检出率为68%;IF处理未检测出blaCTX-M和mcr-1,ARGs检出率为72%;OF处理还未检测出qnrS,ARGs检出率为76%。以上结果表明,土壤细菌DNA中ARGs的多样性普遍高于噬菌体DNA;有机肥施用不但能增加土壤细菌中ARGs的多样性,也能增加土壤噬菌体中ARGs的多样性。
2.2 不同施肥处理农田土壤噬菌体与细菌中ARGs的丰度水平
对不同施肥处理农田土壤(CK、IF、OF)中噬菌体和细菌携带ARGs的丰度水平进行分析。结果表明,土壤噬菌体中ARGs的总丰度在(5.92×102)-(1.55×103)copies/g之间(图1-B),占据显著优势的是多耐药类抗性基因,其丰度在(2.52×102)-(1.15×103)copies/g之间,MLSB类和β-内酰胺类抗性基因的丰度相对其它类型抗性基因也处于比较高的水平。方差分析(ANOVA)表明,噬菌体携带ARGs的总丰度在OF处理中要显著高于CK和IF(P<0.05),MLSB类和多耐药类抗性基因丰度也有相似规律,但万古霉素类抗性基因丰度在IF处理(1.40×102copies/g)中要显著高于CK和OF(P<0.05)。由图1-D可看出,不同施肥处理农田土壤中噬菌体携带intl1的丰度有显著差异(P<0.05),在IF处理中显著低于CK和OF,丰度范围为(1.89×102)-(7.09×102)copies/g。此外,不同施肥处理土壤细菌中ARGs的总丰度在(1.35×105)-(1.30×106)copies/g之间(图1-A),比土壤噬菌体中ARGs的总丰度高2-3个数量级,且在不同施肥处理中具有显著性差异(P<0.05)。与土壤细菌中ARGs相似,土壤噬菌体中也是多耐药类抗性基因的丰度占显著优势。土壤细菌所携带的intl1丰度如图1-C所示,丰度范围为(8.05×103)-(3.09×104)copies/g(P<0.05),比土壤噬菌体中intl1丰度高1-2个数量级。以上结果表明,土壤噬菌体中的ARGs总丰度和intl1丰度明显低于土壤细菌,而有机肥施用能显著提高土壤噬菌体中ARGs的总丰度,但对intl1丰度没有明显影响。
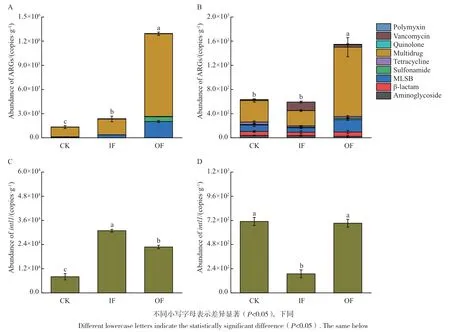
图1 不同施肥处理农田土壤细菌(A、C)和噬菌体(B、D)中ARGs与intl1丰度Fig. 1 Abundances of ARGs and intl1 in bacteria(A,C)and bacteriophages(B,D)in farmland soil under different fertilization treatments
对土壤噬菌体和细菌中不同ARGs亚型在不同施肥处理中的丰度进行对比,结果如图2所示。土壤噬菌体中多耐药类抗性基因mexF、emrD,MLSB类抗性基因oleC、mphA-01和β-内酰胺类抗性基因blaTEM的丰度处于较高水平。方差分析(ANOVA)表明,大部分噬菌体携带的ARGs亚型丰度在不同施肥处理中的差异不显著(P>0.05)。而MLSB类抗性基因mphA-01和多耐药类抗性基因mepA、mexF丰度在OF处理显著高于CK和IF(P<0.05);MLSB类抗性基因oleC丰度在CK和OF中显著高于IF(P<0.05);多耐药类抗性基因acrA-05、emrD和万古霉素类抗性基因vanA的丰度在不同施肥处理中均有显著性差异(P<0.05),其中acrA-05和emrD为 OF>CK>IF,vanA 为 IF>OF>CK。土壤细菌携带ARGs亚型丰度的差异整体上看与噬菌体的分布一致,其中多耐药类抗性基因mexF、emrD和MLSB类抗性基因oleC的丰度较高。与噬菌体中ARGs丰度分布不同的是,土壤细菌中绝大部分ARGs亚型在不同施肥处理中呈现出显著性差异(P<0.05)。
对比不同施肥处理下土壤噬菌体与细菌中各种ARGs亚型的丰度(图2),除β-内酰胺类抗性基因blaTEM外,噬菌体携带的其他ARGs亚型丰度均显著低于细菌(P<0.05),其中MLSB类抗性基因oleC和多耐药类抗性基因mexF基因丰度相差较大,达到3个数量级。
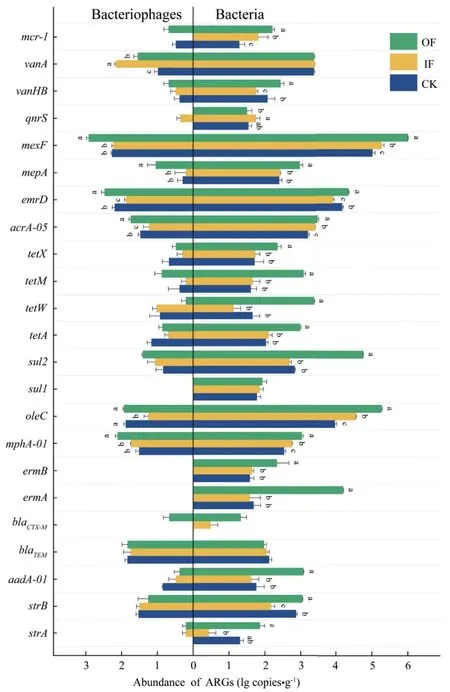
图2 土壤细菌和噬菌体中不同ARGs亚型的丰度Fig. 2 Abundances of ARG subtypes in the bacteria and bacteriophages in farmland soil under different fertilization treatments
2.3 土壤噬菌体与细菌携带ARGs的相关性
分别对不同施肥处理土壤噬菌体与细菌中ARGs亚型的丰度进行相关性分析(图3)。结果表明,在3个不同处理中,噬菌体与细菌携带的ARGs丰度之间均存在显著正相关性(P<0.001),其中CK中二者相关性最强(RCK2=0.665),其次为IF处理组(RIF2=0.612),而OF处理组中二者的相关系数最小(ROF2=0.409)。
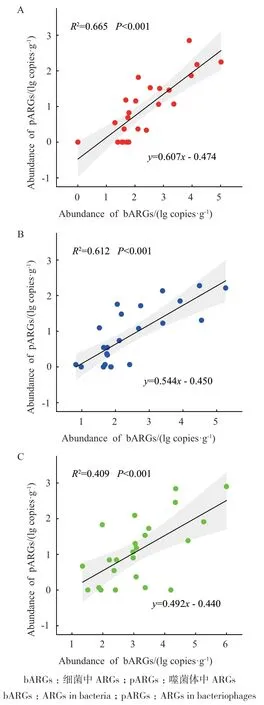
图3 不同施肥处理土壤噬菌体与细菌中ARGs丰度的相关性(A:CK;B:IF;C:OF)Fig. 3 Correlation of the abundances of ARGs between bacteria and bacteriophages fractions in farmland soil under different fertilization treatments(A:CK;B:IF;C:OF)
2.4 土壤理化性质与噬菌体和细菌携带ARGs的相关性
分别对不同施肥处理下噬菌体和细菌ARGs与土壤理化性质进行冗余分析(图4)。结果表明,ARGs在CK、IF、OF处理中与7种重金属元素(As、Hg、Cu、Cr、Cd、Pb、Zn)和5种其他理化因子(pH、OM、AP、AK、TN)显著相关(P < 0.05)。图4-A的结果显示,重金属元素对细菌ARGs分布的影响在第一轴和第二轴的解释量分别为74.87%和21.38%。pH和营养因子对细菌ARGs分布的影响在第一轴和第二轴的解释量分别为74.69%和21.26%(图4-B)。在所选择的因子中,Zn、Cr、Cu、OM、TN与细菌ARGs的相关性最大。其中Cu、Zn、OM、AP、AK、TN主要影响OF处理的土壤细菌ARGs分布;As、Hg、Cr、AP、AK主要影响IF处理的土壤细菌ARGs分布;pH主要影响CK的细菌ARGs分布。
重金属对噬菌体ARGs分布的影响在第一轴和第二轴的解释量分别为50.38%和24.32%(图4-C)。pH和营养因子对噬菌体ARGs分布的影响在第一轴和第二轴的解释量分别为49.58%和24.38%(图4-D)。在所选择的因子中,Zn、Cr、Cu、AP、OM与噬菌体ARGs的相关性最大。其中Cu、Zn、OM、AP、AK、TN主要影响OF处理的土壤噬菌体ARGs分布;As、Cr、Pb、AP、AK主要影响 IF处理的土壤噬菌体ARGs分布,pH主要影响CK中噬菌体ARGs分布。不同施肥处理中,所选择的因子对噬菌体ARGs分布的影响与对细菌的影响差异并不明显。
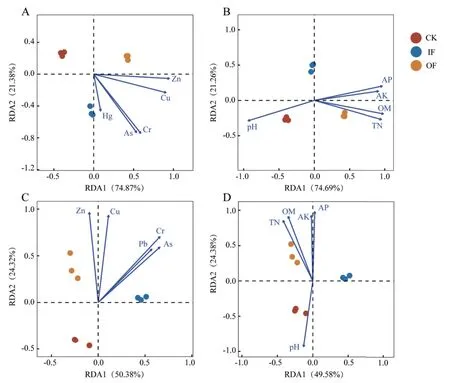
图4 细菌(A、B)与噬菌体(C、D)中ARGs与环境因子的冗余分析Fig.4 Redundancy analysis depicting the relationship between ARGs in bacteria(A,B)and bacteriophages(C,D)and environmental factors
综上所述,环境因子对施化肥和有机肥土壤中细菌及噬菌体携带ARGs的影响较大,对不施肥土壤的ARGs影响较小。因此,施肥可能通过改变土壤pH、重金属和营养因子水平来影响细菌和噬菌体ARGs的赋存特征。
3 讨论
3.1 农田土壤噬菌体与细菌中ARGs的分布特征
土壤是自然环境中ARGs的最大储存库和ARGs向食物链传递的媒介。研究到现阶段,已基本明确土壤ARGs整体上的种类和丰度,ARGs与土壤细菌之间的生物网络也在逐渐建立,然而对可移动ARGs(可移动抗性组)的认知还很有限,尤其是噬菌体及其介导的转导作用对土壤抗生素抗性的贡献还所知甚少[14]。有研究表明,环境中由噬菌体携带的那部分ARGs可能被低估[21-22]。本研究采用ddPCR技术对农田土壤中噬菌体和细菌携带的ARGs进行定量检测,以评估化肥和有机肥施用下土壤噬菌体和细菌携带ARGs的多样性与丰度。研究结果表明,不施肥及化肥和有机肥施用的农田土壤中噬菌体均携带多种 ARGs,其丰度达到(5.92×102)-(1.55×103)copies/g。有研究表明,不同作物农田土壤微生物总DNA 中 ARGs丰度范围在(6.47×109)-(1.41×1010)copies/g 之间[23]。
本研究结果表明,土壤细菌中ARGs的多样性高于噬菌体,总丰度和绝大多数ARGs亚型丰度也显著高于噬菌体。Sun 等[12]的研究表明,温室蔬菜土壤噬菌体中 ARGs 的丰度比细菌中的低约2个数量级。Wang等[13]检测到猪场粪便的细菌DNA中携带32种ARGs亚型,而噬菌体DNA中这些ARGs亚型的检出率仅为35.5%,且丰度显著低于细菌DNA中相应的ARGs,最大相差约3个数量级。尽管噬菌体中ARGs的多样性及丰度均低于细菌,但是噬菌体基因组中的ARGs可通过转导作用进行水平转移,因此其危害可能更大。此外,本研究还发现土壤噬菌体中一种β-内酰胺类抗性基因blaTEM丰度与细菌中该基因的丰度相当,需要引起重视。在土壤噬菌体中还检测到较高丰度的整合酶基因intl1[(1.89×102)-(7.09×102)copies/g]。农田土壤噬菌体中检测到ARGs和整合酶基因的存在,增加了土壤ARGs污染问题的复杂性,说明不仅要继续关注细菌所携带的ARGs[24-25],更要关注噬菌体所携带的ARGs及其介导的转导作用对ARGs水平转移的贡献[26-27]。
本研究在农田土壤噬菌体中检出丰度较高的为多耐药类抗性基因mexF、emrD,MLSB类抗性基因oleC、mphA-01和β-内酰胺类抗性基因blaTEM。Larrañaga等[11]在鲜切蔬菜和农田土壤噬菌体中检测到较高丰度的blaTEM和较低丰度的qnrA、qnrS。Sun等[12]在温室土壤噬菌体中检测到四环素类抗性基因tetM、tetX。Anand等[28]在不同动物养殖场的土壤噬菌体中检测到blaTEM、tetA、tetW,其中blaTEM丰度较高。以上不同类型的土壤样本研究结果与本文结果大致相同,但本研究发现土壤噬菌体携带高丰度的多耐药类抗性基因和MLSB类抗性基因,这在以往的研究并不多见,未来还需要对土壤噬菌体中这两类ARGs进行详细研究。
3.2 施肥对农田土壤噬菌体和细菌中ARGs的影响
Ross等[29]研究了施用牛粪的土壤中噬菌体携带 5种 ARGs(strA,strB,sul1,aadA,blaOXA-20)的丰度特征,发现ARGs丰度变化不大。而本文研究结果显示,施用有机肥会显著提高土壤噬菌体携带ARGs的总丰度。这可能是与前者检测的ARGs种类较少及施用的肥料不同有关。在其他环境样品噬菌体中也检测到ARGs的存在。Yang等[30]在鸡粪噬菌体中检测到blaCTX-M、ermB、mcr-1、qnrS、sul1、sul2、vanA、tetM和整合酶基因intl1,其中sul1和ermB丰度较高。Brown-Jaque等[31]从健康人群粪便样本分离出的噬菌体中检测到blaTEM、qnrA、qnrS、sul1,其中blaTEM丰度较高。Yang等[10]从四川某河水噬菌体中检测到了sul1、sul2、blaCTX-M和整合酶基因intl1,其中sul1检出率最高。Calero-Cáceres等[32]从未经处理的城市废水噬菌体中检测出blaTEM、blaCTX-M、sul1。以上研究结果中抗性基因sul1、qnrA和ermB的检出与本研究结果不一致,表明不同的环境介质可能对噬菌体携带的ARGs亚型有一定的影响。施用有机肥虽然能使土壤细菌和噬菌体中ARGs的总丰度都得到显著提高,但对不同ARGs亚型影响并不一致:有机肥施用能使土壤细菌中大多数ARGs亚型丰度显著提高,而土壤噬菌体中只有少数ARGs亚型的丰度显著提高。含有抗生素和耐药菌的有机肥长期施用,不但能向土壤输入外源的ARGs,而且抗生素的选择压会使土著菌的耐药性增加,引起土壤细菌中大多数ARGs亚型丰度提高[2]。而噬菌体是一种细菌病毒,可通过转导方式促进细菌间的基因水平转移。噬菌体在转导过程中,因为“错装”宿主细菌ARGs的频率通常很低(10-9-10-7)[14],所以有机肥施用对噬菌体基因组中ARGs的影响可能远低于细菌。
此外,对不同施肥处理中噬菌体与细菌携带ARGs亚型的丰度进行相关性分析发现,均存在显著正相关。Yang等[33]研究发现猪场污水处理厂中噬菌体和细菌所携带ARGs的丰度呈现显著正相关(P<0.000 1,R2=0.678 7),这与本文结果一致。对土壤理化性质与ARGs分布进行冗余分析发现,环境因子与施肥土壤中细菌和噬菌体ARGs赋存均有着密切关系。不施肥土壤细菌和噬菌体ARGs主要受pH影响;施化肥土壤细菌和噬菌体ARGs主要受As、Cr、AP、AK影响;施有机肥土壤细菌和噬菌体ARGs主要受Cu、Zn、OM、AP、AK、TN影响。Dickinson等[34]研究表明,重金属(Cu、Zn)污染与抗生素耐药性可能存在共选择关系。Zhao等[35]发现,重金属(Cu、Cr)和营养因子TN可能会驱动环境中ARGs的分布和传播。但是,土壤理化性质对噬菌体携带ARGs赋存特征的影响机制还需进一步研究。
由于细菌作为噬菌体的宿主,为噬菌体提供大量营养物质和遗传物质,因此噬菌体所携带的ARGs可能主要来源于细菌[7]。今后可采用宏基因组测序和生物信息学技术全面解析土壤噬菌体基因组中ARGs的多样性及其驱动机制,并与细菌基因组中ARGs的驱动机制进行对比分析,以扩展对土壤抗生素抗性的全面认知。
4 结论
农田土壤细菌和噬菌体携带的ARGs丰度具有显著正相关性,而且施用有机肥能同时显著提高土壤细菌基因组和土壤噬菌体基因组中ARGs的总丰度。施肥对土壤噬菌体携带的大多数ARGs亚型的丰度影响不大,但能显著提高土壤细菌中大多数ARGs亚型的丰度。
