南极菲尔德斯半岛潮间带沉积物细菌的多样性及分离菌产酶测定
2022-10-29王玉璟李胜男袁嘉琳
王玉璟, 李胜男, 袁嘉琳, 王 龙, 刘 杰
南极菲尔德斯半岛潮间带沉积物细菌的多样性及分离菌产酶测定
王玉璟, 李胜男, 袁嘉琳, 王 龙, 刘 杰
(青岛科技大学 海洋科学与生物工程学院, 山东 青岛 266042)
南极菲尔德斯半岛具有多种不同特征的生态地理微环境, 如长城科考站、企鹅岛、生物湾与黄金湾流域、半岛南部、碧玉滩等。这些区域因水文地质、动植物分布、人类活动程度等不同而具有明显的生态地理差异。本研究从这些微环境海岸潮间带采集了7份代表性沉积物样品, 采用16S rRNA基因高通量测序方法, 对其细菌类群的多样性以及环境理化因子的影响进行了比较分析; 同时采用常规可培养鉴定方法, 对样品的分离菌株产酶状况进行了初步测定。结果表明: 整体上看, 所有样品的细菌类群主要分布在45门、104纲、442属当中, 表现出比较高的多样性。其中, 优势菌门Proteobacteria主要分布在半岛南部、碧玉滩和生物湾潮间带沉积物中, 优势菌门Bacteroidetes主要分布在企鹅岛两侧和黄金湾潮间带沉积物, 长城站潮间带沉积物的优势门为Firmicutes。相似生态微环境的潮间带沉积物具有不同的优势菌门和纲; 而不同生态微环境潮间带沉积物却具相同的优势菌门和纲。有机氮(TON)、铵态氮(NH4+-N)、硝态氮(NO3–-N)、磷酸盐(PO43–-P)、亚硝态氮(NO2–-N)含量与企鹅岛、生物湾、黄金湾潮间带沉积物的菌群多样性具有相关性, 其中TON对生物湾样品的影响最大, 而有机碳(TOC)对所有样品的影响均较小。分离菌株的产酶实验表明: 在企鹅岛、生物湾、黄金湾等动物频繁出没的潮间带样品中, 蕴藏着一批产淀粉酶、酪蛋白酶、脂肪酶、七叶苷酶、过氧化氢酶能力较强菌株, 这为今后进一步的应用开发提供了宝贵的低温产酶菌株来源。
菲尔德斯半岛; 潮间带沉积物; 细菌类群多样性; 产酶菌株
两相地带中以陆、海交汇的潮间带最为典型, 因受潮涨、潮落影响, 其沉积物的微生物类群也具有不同于陆地与海洋的特征, 并引起国内外广泛关注。近些年, 研究人员对各类潮间带沉积物微生物的多样性[1-5]、抗性[6-7]、产酶[8-9]、污染指示等[10-11]方面进行了研究, 所采用的方法也多种多样。南极菲尔德斯半岛海岸线绵长, 溪流纵横, 具有南极无冰区典型的生态地理特征, 动植物资源丰富、人类干扰少, 微小特征生境众多。
本研究针对前期从菲尔德斯半岛的企鹅岛、生物湾、黄金湾、半岛南部碧玉滩、长城站等不同生态地理微环境采集的潮间带沉积物样品, 采用16S rRNA基因可变区高通量多样性测序法对所获得的细菌群落多样性组成、样品理化因子影响等结果、以及通过可培养法获得的分离菌株产酶情况进行全面比较分析[12]。以期比较全面地了解菲尔德斯半岛各种特征微生态环境潮间带沉积物的细菌多样性差异, 筛选产酶功能菌株, 为后续研究和应用奠定基础。
1 材料与方法
1.1 样品采集与处理
沉积物样品于2017年2月(中国第33次南极科学考察期间)采自菲尔德斯半岛(62°8.48′S~62°14.2′S, 58°53.40′W~59°1.50′W)周边沿海不同区域的潮间带(表1)。每个采样点相隔约10 m采集3份深度约5 cm的沉积物(去粗土壤)并均匀混合, 装入无菌袋中密封带回长城站实验室立即用2216E平板进行细菌分离、纯化; 同时用试剂盒提取样品总DNA保存于–80 ℃, 并与分离菌株、沉积物样品备份一起低温运回国内。

表1 样品采集信息
1.2 样品理化性质测定
样品理化性质测定包括有机碳(TOC)、有机氮(TON)、铵态氮(NH4+-N)、硝态氮(NO3–-N)、亚硝态氮(NO2–-N)、磷酸盐(PO43–-P)。样品去杂冻干处理后研成粉末。其中, TOC、TON取0.2 g, 加入5 mL 10%的HCl, 充分振荡过夜; 加入3 mL 10% 盐酸, 放置3 h后, 用超纯水清洗7次, 55 ℃烘干, 然后用EA3000元素分析仪(Euro Vector SpA)测定。其他4种营养盐的测定为: 称取处理样品2 g, 加入20 mL超纯水振荡1 min, 48 h内每隔4 h振荡1 min; 10 000 r/min离心10 min, 上清液0.4 μm膜过滤, 用微流分析仪(QuAAtro, SEAAnalytical GmbH)分析。
1.3 菌株分离及产酶菌株检测
样品的菌株分离培养, 采用2216E、R2A、M1海水培养基固体平板15 ℃培养, 划线分离[2]。
分离菌株的产酶检测, 采用在含相应底物的固体培养基上点接分离菌, 然后15 ℃培养7~15 d, 观察是否产水解透明圈或晕圈大小、显色深浅或产气泡多少等。其中产酪蛋白水解酶、几丁质水解酶、淀粉水解酶、纤维素水解酶、黄原胶水解酶、卡拉胶水解酶以菌落周围是否产生透明圈及大小来判断, 脂肪酶以产不透明晕圈来判断, 明胶酶以菌落周围是否出现液化圈来判断, 七叶苷酶以菌落周围有无黑褐色素产生来判断, 氧化酶以在菌落上滴加1%二甲基对苯二胺盐酸盐溶液显色法判断, 过氧化氢酶采用菌落覆盖5% H2O2是否产气泡判断[12]。
1.4 样品总DNA提取和16S rRNA基因高通量测序
沉积物样品总DNA提取使用土壤DNA提取试剂盒(E.Z.N.A产品), PCR扩增16S rRNA基因的V4-V5可变区。扩增引物为通用515F和907R。高通量测序由专业公司在IlluminaHiSeq2500(PE250)测序平台完成。
1.5 高通量多样性测序数据分析
PCR扩增序列去除Barcode及引物序列后拼接, 然后过滤低质量序列和嵌合体序列(最小得分25)[13-14], 得到有效序列用于后续分析。
以97%相似度进行有效序列的聚类及创建OTUs集, 选取每个OTU集中频数最高的代表序列进行比对和物种注释获得分类学信息。
采用Qiime计算α多样性指数, UPGMA分析β多样性, 蒙特卡洛、主坐标PCoA、冗余RDA等方法分析样品理化因子与细菌群落的相关性并绘制分析图。
2 结果与分析
2.1 测序结果及物种分析
本实验7份样品共鉴定出552 355个细菌序列, 其中每个样品有效序列平均为69 537条。按97%相似度聚类共得到5 494个OUTs集, 平均每个样品785个OUTs集[12]。
根据OTUs注释结果, 选取在门、纲水平相对丰富度排名前10、属水平排名前30的物种类群, 绘制物种相对丰度柱状图(图1、2、3)。
图1、2、3及其相关数据表明, 7个潮间带样品的细菌类群经注释主要分布在45个门、104个纲、442个属当中。
在门水平上(图1), 7个样品中均存在的门有Bacteroidetes、Proteobacteria、Planctomycetes和Actinobacteria, 且占比较大。其中02、05、07号样品的优势门为Bacteroidetes(分别占比42.5%、56.3%、40.1%), 01、04、06号样品的优势门为Proteobacteria(分别占比44.9%、51.1%、41.1%), 03号样品的优势门为Firmicutes (占比48.3%), 但在其他样品中较少; 另外, Planctomycetes在02号样品中的占比较高(14.2%), Actinobacteria以及Cyanobateria在06号样品中含量也较多。
在纲水平上(图2), 7个样品主要分布在104个纲中。其中01、02、04号样品的优势纲为Gammaproteobacteria(分别占比23.4%、21.5%、25.5%), 05、07号样品的优势纲为Flavobacteriia(占比42.1%、27.7%), 06号样品的优势纲为Betaproteobacteria (26.9%)。另外, Bacteroidia只在03样品中占比较大(约为20%), 而在其他样品中占比较少(少于3.2%), 且在01号样品中没有检测到; 同时, Bacilli在03、06号样品中有一定含量(18%、3.9%), 而在其他样品中的含量均低于0.06%。
在属水平上的聚类比较复杂(图3), 共聚类出442个属。其中01号样品的优势属为(11.84%), 02、07号样品的优势属为(8.9%和6.5%); 03号的优势属为(17.28%),和在该样品中的占比也较高(9.26%、3.53%), 而为该样品的特有属; 04样品的优势属为(4.1%), 而为该样品所特有属(2.66%); 05号的优势属为(5.98%); 06号的优势属为(14.69%), 同时属在该样品中占比也较高(12.11%), 而该属在其他样品中低于0.13%。另外, 7个样品中均有少量属。
2.2 样品内与样品间物种群落多样性分析
多样性分析结果表明(表2), 07号样品的香农指数(Shannon)和辛普森指数(Simpson)较高, 说明物种多样性及相对丰度比较高, 而01号样品相对较低。
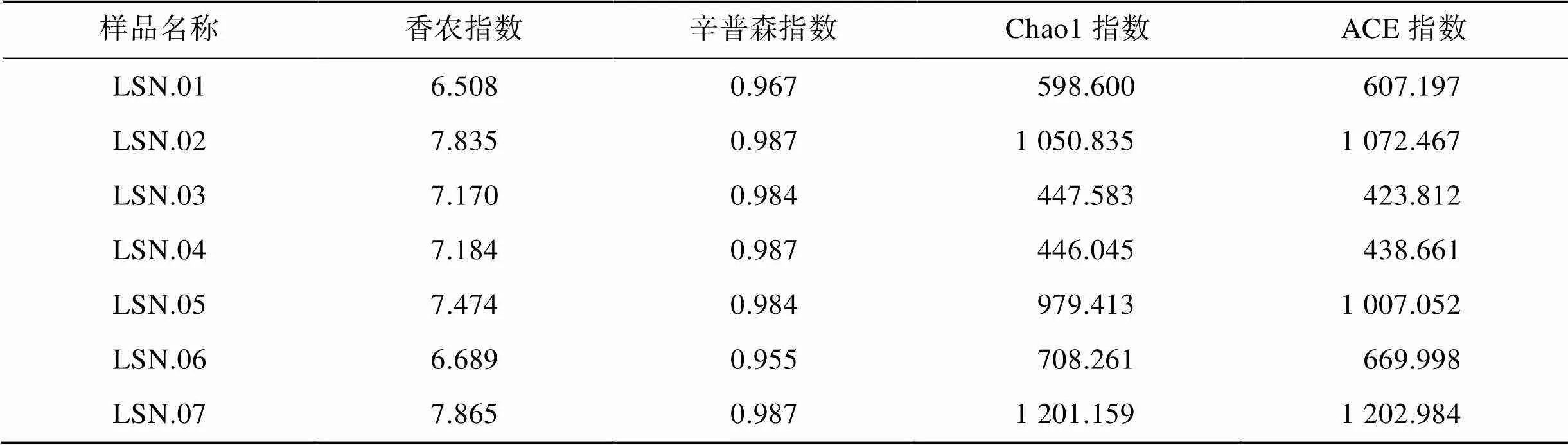
表2 α多样性指数
从多样性分析看出(图4), 02、07号样品的相似度最高(非加权Unifrac距离小于0.1), 其次为样品05样品; 而03、06号样品与其他样品的相似性均较低, 尤其是03号最低。反映出不同样本间微生物群落构成及其占比的差异。
基于样品间物种组成结构相似度的PCoA主坐标分析也表明(图5, 第一主坐标PC1), 02、05、07样品的距离十分接近(在同一个象限里), 尤其是02和07号, 说明它们之间的物种组成尤为相似; 虽然01和04号处在另一个象限, 但二者距离相对较远; 而03和06号样品分别位于两个不同的象限里, 且与其他5个样品相距均很远, 表明就细菌物种组成结构而言, 此两个样品特殊性非常明显。
2.3 样品理化因子与物种群落组成相关性分析
本研究对7份潮间带样品进行了包括有机碳(TOC)、有机氮(TON)、铵态氮、硝态氮、亚硝态氮磷酸盐等6项理化因子的测定(表3), 同时结合上述细菌群落数据进行了RDA冗余分析(图6), 以了解样品理化因子对造成样品细菌群落组成差异的影响关系。

表3 样品的理化性质
注: 表中数据为三次重复测试的平均值。
从表3的7个样品理化因子看到, 有机碳含量在0.06%~0.4%, 其中04样品含量最高(0.399%), 01样品含量最低(0.056%); 有机氮含量均较低且相差不大(0.02%~0.03%); 铵态氮相差比较大(0.5~9.1 μg/g), 其中05号样品(9.139 μg/g)远高于其他样品, 而03号含量最低(0.557 μg/g); 硝态氮相差也比较大(0.6~ 5.5 μg/g), 02号最高(5.486 μg/g), 05号最低(0.694 μg/g); 7个样品中亚硝态氮含量均不高(0.03~0.5 μg/g), 且相差不大; 而磷酸盐含量在7个样品中均较高(2.9~ 7.3 μg/g), 其中05号最高(7.286 μg/g), 03号最低(2.952 μg/g)。
从基于反映环境因子、样本、菌群之间关系的RDA冗余分析中可以看到(图6), 样品02、04、05、07主要分布在第一主成分的负方向上, 同时除TOC外的5种理化因子(TON、NH4+-N、NO3–-N、PO43–-P、NO2–-N)两两之间的夹角以及各个理化因子与第一主成分之间的夹角均为锐角, 表明这5种理化因子之间以及与这4个样品物种组成的相关性比较强。其中02、05、07这3个样品最为相似, 且与NH4+-N含量的相关性最强, 与TOC的相关性最小; 而04号样品则受TON含量的影响最大。另外从图6中还可以看到, 01、06、03号样品分散在第一主成分的正方向上, 相互之间相距较远, 且与上述6种理化因子含量之间没有明显的相关性。
2.4 可培养法与高通量法优势菌群的比较
为获得对菲尔德斯半岛潮间带沉积物细菌物种类群多样性更全面的了解, 我们在高通量多样性法测序分析的同时, 还采用常规可培养法对7个潮间带样品的细菌进行了分离纯化, 并进行了基于16S rDNA序列测定的系统发育鉴定分析[15], 然后就门、纲、属水平上的结果与高通量测序分析结果进行了优势菌群类别的比较(数据略)。整体上看, 两种分析方法结果中的优势菌群在门、纲水平上的重叠性较大; 而在属水平上却差异较大, 尤其是两种方法在优势属上的结果没有交叉重叠现象。
2.5 分离菌株的产酶情况
在可培养实验中, 我们针对从7个样品所分离的45株代表菌进行了淀粉酶、酪蛋白酶、脂肪酶、七叶苷酶、几丁质酶、明胶酶、纤维素酶、氧化酶、过氧化氢酶等9种酶的产生情况初步测试。
从表4中看到, 45株测试菌株中有6株产淀粉酶能力较强, 2株产酪蛋白酶较强, 14株产脂肪酶较强, 3株产七叶苷酶较强, 5株产过氧化氢酶较强, 但没发现产几丁质酶的菌株。这些低温下产酶能力较强的菌株主要集中在企鹅岛、生物湾、黄金湾等企鹅、海豹、鸟类频繁出没区域, 这为今后进一步的开发应用提供了难得的菌株来源。

表4 分离菌株产酶情况
注: “–”为无产酶活性, “+”为有产酶活性, “++”为产酶活性较强
3 讨论
特殊生境是获取功能微生物的良好环境。本研究样品主要来自南极菲尔德斯半岛周边特征微环境的潮间带沉积物。从高通量多样性测序结果看, 整体样品的细菌类群多样性程度较高(主要分布在45门、104纲、442属)。其中Proteobacteria、Bacteroidetes为多个微环境的优势菌门(如半岛南部、碧玉滩、生物湾、黄金湾、企鹅岛两侧等潮间带); 而长城站科考站潮间带的优势菌门为Firmicutes, 比较单一, 可能与人类和动物的频繁活动有关。从生态地理上看, 相似生态地理微环境的潮间带沉积物具有不同优势菌门、纲或属(例如: 人和动物稀少的半岛南部与碧玉滩, 半岛西侧的生物湾与黄金湾); 而企鹅岛两侧的生态地理环境差异较大, 尤其是企鹅、鸟类数量和阳光照射时长具有明显差异, 但它们却有相似的优势菌门、纲, 这种情况也得到了可培养法鉴定结果的印证。总之, 对于整个菲尔德斯半岛潮间带而言, 其细菌类群组成具有比较高的多样性, 但其中也表现出一些超乎我们预测的现象, 值得进一步综合研究。
土壤的理化性质可直接影响土壤细菌群落结构[16-19]。近年来, 对南、北极土壤细菌多样性主控理化因素影响的研究也较多[20-22]。本研究表明, 从沉积物样品理化因子与菌群组成关系看, 生态地理环境相似的潮间带沉积物菌群组成(例如半岛南部与碧玉滩, 生物湾与黄金湾等)与有机氮、铵态氮、硝态氮、亚硝态氮、磷酸盐的相关性均比较强(其中生物湾样品的菌群组成受有机氮的影响最大)。另外, 有机碳对所有样品菌群组成的影响均较小。上述样品间菌群多样性的差异是否是由此所引起的, 还有待进一步深入探讨。
本研究还对可培养分离菌进行过16S rDNA系统发育鉴定分析(数据略)。但从鉴定结果看, 与上述高通量测序分析相比, 优势菌群在门、纲水平上具有较大重叠; 而在属水平上却差异很大, 基本没有交叉重叠。出现这种现象的原因很多, 首先是2种方法对物种比对注释的灵敏度和精度范围不同[23]。高通量测序法是采用16S短序列引物针对整个宏基因组进行的物种多样性注释分析, 而可培养分离菌的16S rDNA序列测定则是采用16S长序列引物对单个菌株的序列测定和比对分析。因此前者在门、纲水平上信息量更大, 而在属水平上的精度要远低于后者。其次是可培养分离方法的限制。尽管本实验采用了2216E、R2A、M1三种高、寡营养培养基进行菌株分离, 但分离效果与实际情况还是相差甚远, 这也是令环境微生物学者十分头疼症结所在。但无论怎样, 可培养分离鉴定是研究、应用实体微生物基本途径, 将传统可培养法与现代高通量测序法结合起来, 将会对环境微生物的多样性做出更加客观全面的注释。
另外, 本研究在可培养实验中, 从企鹅岛、生物湾、黄金湾等动物频繁出没的微环境潮间带沉积物中发现了一批产淀粉酶、酪蛋白酶、脂肪酶、过氧化氢酶能力较强的菌株, 这些菌株均为低温培养菌(15 ℃培养), 其所产上述酶是否为低温酶、以及产酶条件和酶学性质尚需进一步研究, 但无疑它们极具应用开发价值。
本研究可为全面了解菲尔德斯半岛或其他类似极端环境的细菌多样性及产酶功能菌提供有价值的参考。
[1] Lasher C, Dyszynski G, Eerett K, et al. The diverse bacterial community in intertidal Anaerobic Sediments at Sapelo Island, Georgia[J]. Microbial Ecology, 2009, 58: 244-261.
[2] 刘杰, 卜蒙蒙, 孙景, 等. 俄罗斯库页岛潮间带沉积物可培养细菌的系统发育多样性[J]. 海洋科学, 2015, 39(9): 69-76.
Liu Jie, Bu Mengmeng, Sun Jing, et al. Phylogeny diversity of culturable bacteria from intertidalsediments samples of Russian Sakhalin Island[J]. Marine Sciences, 2015, 39(9): 69-76.
[3] 王雅苹, 史晓翀, 余少兰, 等. 青岛潮间带沉积物可培养厌氧细菌多样性的研究[J]. 海洋科学, 2015, 39(3): 92-99.
Wang Yaping, Shi Xiaochong, Yu Shaolan, et al. Diversity of culturable anaerobic bacteria isolated from intertidal sediments of Qingdao[J]. Marine Sciences, 2015, 39(3): 92-99.
[4] 王凯, 邹立, 高冬梅, 等. 黄河口潮滩春季细菌群落的分布特征及其影响因素研究[J]. 中国海洋大学学报(自然科学版), 2016, 46(1): 108-115.
Wang Kai, Zou Li, Gao Dongmei, et al. Characteristics of bacterial community and associated environmental impacts in intertidal mudflat of Yellow River Estuary, China[J]. Periodical of Ocean University of China, 2016, 46(1): 108-115.
[5] 李佳霖, 汪光义, 秦松. 秦皇岛近海养殖对潮间带微生物群落多样性的影响[J]. 生态环境学报, 2011, 20(5): 920-926.
Li Jialin, Wang Guangyi, Qin Song. Microbial communities of sediments influenced by mariculture from the coast of Qinhuangdao[J]. Ecology and Environmental Sciences, 2011, 20(5): 920-926.
[6] Hatha A A M, Neethu C S, Nikhil S M, et al. Relatively high antibiotic resistance among heterotrophic bacteria from arctic fjord sediments than water- Evidence towards better selection pressure in the fjord sediments[J]. Polar Science, 2015, 9(4): 382-388.
[7] 马悦欣, 于作镇, 于书渤, 等. 大连海区潮间带海藻附生细菌的抗真菌活性[J]. 大连水产学院学报, 2005, 20(2): 87-90.
Ma Yuexin, Yu Zuozhen, Yu Shubo, et al. Antifungal activities of epiphytic bacteria from the surfaces of seaweeds in intertidal zone in Dalian[J]. Journal of Dalian Fisheries University, 2005, 20(2): 87-90.
[8] 薛珊珊, 刘颖文, 王洁, 等. 湛江沿海潮间带产胞外抗菌活性海洋细菌的分离鉴定及多样性分析[J]. 基因组学与应用生物学, 2017, 36(5): 2005-2014.
Xue Shanshan, Liu Yingwen, Wang Jie, et al. Isolation, identification and biodiversity analysis of the marine bacteria producing extracellular antimicrobial activity from the intertidal zone along Zhanjiang coast[J]. Genomics and Applied Biology, 2017, 36(5): 2005-2014.
[9] 王洁, 崔丽娇, 兰柳波, 等. 中国南海硇洲岛潮间带产胞外蛋白酶的可培养海洋真菌多样性[J]. 微生物学通报, 2015, 42(2): 238-253.
Wang Jie, Cui Lijiao, Lan Liubo, et al. Diversity of culturable extracellular proteases producing marinefungi isolated from the intertidal zone of Naozhou Island in South China Sea[J]. Microbiology China, 2015, 42(2): 238-253.
[10] Paerl H W, Dyble J, Moisander P H, et al. Microbial indicators of aquatic ecosystem change: current applications to eutrophication studies[J]. FEMS Microbial Ecology, 2003, 46: 233-246.
[11] 王丽萍, 刘录三, 隋晓斌. 多环芳烃对渤海湾潮间带沉积物微生物群落结构的影响[J]. 海洋环境科学, 2013, 32(6): 856-859.
Wang Liping, Liu Lusan, Sui Xiaobin. Effects of PAHs to microbial community structure in Bohai tidal sediment[J]. Marine Environmental Science, 2013, 32(6): 856-859.
[12] 李胜男. 南极菲尔德斯半岛潮间带沉积物细菌的多样性研究[D]. 青岛: 青岛科技大学, 2019.
Li Shengnan. Study on the diversities of bacteria in intertidal sediments of the Fields Peninsula, Antarctica[D]. Qingdao: Qingdao University of Science and Technology, 2019.
[13] Bokulich N A, Subramanian S, Faith J J, et al. Quality-filtering vastly improves diversity estimates from Illumina amplicon sequencing[J]. Nature Methods, 2013, 10(1): 57-59.
[14] Caporaso J G, Kuczynski J, Stombaugh J, et al. Qiime allows analysis of high-throughput community sequencing data[J]. Nature Methods, 2010, 7: 335-336.
[15] Jie L, Sun Y w, Zhang D d, et al.sp. nov., isolated from a deep-sea seamount[J]. International Journal of Systematic and Evolutionary Microbiology, 2017, 67(6): 1996-2000.
[16] Neufeld J D, Mohn W W. Fluorophore-labeled primers improve the sensitivity, versatility, and normalization of denaturing gradient gel electrophoresis[J]. Applied & Environmental Microbiology, 2005, 71: 4893- 4901.
[17] Newsham K K. The biology and ecology of the liverwort cephaloziellavarians in Antarctica[J]. Antarctic Science, 2010, 22: 131-143.
[18] Stomeo F, Makhalanyane T P, Valverde A, et al. A biotic factors influence microbial diversity in permanently cold soil horizons of a maritime-associated antarctic dry valley[J]. FEMS Microbiology Ecology, 2012, 82: 326-340.
[19] Wallenstein M D, Mcmahon S, Schimel J. Bacterial and fungal community structure in arctic tundra tussock and shrub soils[J]. FEMS Microbiology Ecology, 2007, 59: 428-435.
[20] Chong C W, Dunn M J, Convey P, et al. Environmental influences on bacterial diversity of soils on signy island, maritime Antarctic[J]. Polar Biology, 2009, 32: 1571-1582.
[21] Chong C W, Pearce D A, Convey P, et al. Patterns in the distribution of soil bacterial 16S rRNA gene sequences from different regions of Antarctica[J]. The Global Journal of Soil Science, 2012, 181/182: 45-55.
[22] Chu H, FiererN, Lauber C L, et al. Soil bacterial diversity in the arctic is not fundamentally different from that found in other biomes[J]. Environmental Microbiology, 2010, 12: 2998-3006.
[23] Janssen P H. Identifying the dominant soil bacterial taxa in libraries of 16S rRNA and 16S rRNA genes[J]. Applied & Environmental Microbiology, 2006, 72(3): 1719-1728.
Bacterial diversity and enzyme production by the bacteria isolated from the intertidal regions of the Fildes Peninsula, Antarctica
Wang Yu-jing, Li Sheng-nan, Yuan Jia-lin, Wang Long, Liu Jie
(College of Marine Sciences and Biological Engineering, Qingdao University of Science and Technology, Qingdao 266042, China)
The Fildes Peninsula in Antarctica has diverse ecogeographical microenvironments possessing different characteristics, such as Great Wall Station, Penguin Island, Bio-bay, Golden Bay, south of the peninsula, and Biyu Beach. These regions have certain ecological and geographical differences owing to their distinct hydrogeology, flora and fauna distributions, and human activities. In this study, seven representative sediment samples were collected from coastal intertidal zones of these microenvironments, and the diversity of bacterial communities and the effects of environmental physicochemical factors were analyzed using the 16S rRNA gene high throughput sequencing method. Furthermore, the enzyme production status of culturable isolated strains was preliminarily determined. The results revealed that the bacterial groups in all samples were distributed throughout 45 phyla, 104 classes, and 442 genera, indicating high bacterial diversity. Proteobacteria were primarily distributed in the southern region of the Peninsula, Biyu Beach, and Bio-bay intertidal zones, while Bacteroidetes were mainly distributed in the intertidal zones of Penguin Island and Golden Bay; Firmicutes was the dominant phylum in the intertidal regions of the Great Wall Station. Remarkably, intertidal regions with similar ecological microenvironments had different dominant phyla and classes. However, the intertidal regions from dissimilar ecological microenvironments had the same dominant phyla and classes. The organic nitrogen (TON), NH4+-N, NO3–-N, PO43–-P, and NO2–-N contents were correlated with the bacterial group diversities at the Penguin Island, Biota Bay, and Golden Bay intertidalites. TON had the greatest impact on the Bio-bay intertidalite samples, while organic carbon had little effect on any of the samples. The enzyme producing analysis of the culturable isolated strains revealed a number of strains with a strong ability to produce amylase, casease, lipase, aesculin hydrolase, and catalase in the sediment samples of Penguin Island, Bio-bay, and Golden Bay. Thus, these isolates are valuable sources of low temperature, enzyme producing strains for further research.
Fields peninsula; intertidalite; bacterial group diversity; enzyme producing strains
Jan. 27, 2022
Q346
A
1000-3096(2022)09-0046-09
10.11759/hykx20220127001
2021-01-27;
2022-02-20
国家公共科研院所基本科学基金项目(GY0219Q10)
[Basic Science Fund project of National Public Research Institutes, No. GY0219Q10]
王玉璟(1995—), 男, 汉族, 山东省烟台市莱阳人, 硕士生, 研究方向: 极端环境微生物资源与应用, E-mail: wangyjdy123@163.com; 刘杰(1963—),通信作者, 硕士生导师, 研究方向: 环境微生物资源、分类与应用, E-mail: jieliu1301@sina.com.cn
(本文编辑: 赵卫红)
