基于卵形鲳鲹基因组重测序的InDel标记挖掘与耐低氧性状关联分析
2022-10-25伞利择刘宝锁郭华阳朱克诚张殿昌
伞利择 ,刘宝锁, ,张 楠, ,郭 梁, ,郭华阳, ,朱克诚, ,张殿昌,
1. 河北农业大学 海洋学院,河北 秦皇岛 066003
2. 中国水产科学研究院南海水产研究所/农业农村部南海渔业资源开发利用重点实验室,广东 广州 510300
3. 广东省海洋生物种业工程技术研究中心,广东 广州 510300
卵形鲳鲹 (Trachinotus ovatus) 具有生长快、肉质鲜美、经济价值高等优点,是我国南部沿海地区重要的海水养殖品种之一。其生存和生长容易受到各种环境因素的影响,其中水体中的溶解氧含量是关键因素之一。在自然条件下,海水通过风和浪的物理搅动使大量的氧气溶解在水中。水生生物光合作用产生的氧气也是水中溶解氧的重要来源。但当海水交换不及时,或赤潮爆发后,大量死亡的藻类在分解过程中消耗大量氧气[1],导致卵形鲳鲹缺氧。一些寄生虫例如刺激隐核虫 (Cryptocaryon irritans)[2]和眼点淀粉卵涡鞭虫 (Amylocdinium ocellatum)[3]的寄生会破坏鳃组织,使鱼无法进行气体交换,导致卵形鲳鲹缺氧死亡。
溶解氧的减少会对鱼类产生诸多影响,包括食物摄入量减少、生长速度减慢、游泳能力下降和影响鱼类的形态学特征和生存策略[4]。研究发现,低氧环境会导致尼罗罗非鱼 (Oreochromis niloticus) 的食欲下降并影响其生长速度[5];相比正常溶解氧的生存环境,虹鳟 (Oncorhynchus mykiss) 在低氧条件下游得更慢[6];长期缺氧会降低大西洋鲑 (Salmo salar) 卵的孵化率,甚至导致胚胎发育畸形或死亡[7]。一些鱼类为适应低氧环境,鳃组织的形态结构发生了变化。例如,鲫 (Carassius carassius)[8]、金鱼 (C. auratus)[9]和团头鲂 (Megalobrama amblycephalala)[10]通过重建鳃组织、暴露鳃板增加鳃的表面积以应对缺氧胁迫。
DNA标记是一种非常可靠的分子标记,随机扩增多态性DNA (Random Amplified Polymorphic DNA, RAPD)[11]、扩增片段长度多态性 (Amplified Fragment Length Polymorphism, AFLP)[12-13]、简单序列重复 (Simple Sequence Repeat, SSR)[14]、单核苷酸多态性 (Single Nucleotide Polymorphism, SNP)[15-17]和插入-缺失 (InDel)[18-19]等分子标记已广泛应用于遗传育种研究。在不同的个体中,由于基因的插入或缺失,相应的基因和基因组区域会有不同的序列长度。这些突变被称为InDel,可以通过插入转座元件、相似重复副本之间的不相等交叉事件或简单序列复制的滑移形成[20],并可能表现为功能丧失或无意义突变[21]。InDel和SNP是动物基因组中最丰富和分布最广泛的变异性来源,均非常适合于构建遗传连锁图谱、全基因组关联分析和遗传多样性分析等其他遗传学研究。然而,在分子标记辅助育种的过程中,InDel比SNP更方便可取,因为In-Del的多态性更易通过设计特异性引物、PCR扩增和琼脂糖凝胶电泳来显示。以往有多项畜牧和农作物研究使用了InDel标记[22-23],但在水产动物中使用InDel标记的研究仅占少数[18-19,24]。因此,本研究利用全基因组重测序技术挖掘卵形鲳鲹与耐低氧性状显著关联的InDel标记,所筛选到的InDel标记可作为分子标记辅助育种,并有助于鉴定与卵形鲳鲹耐缺氧性状相关的候选基因,为进一步研究卵形鲳鲹耐缺氧性状的分子机制提供依据。
1 材料与方法
1.1 实验材料
在海南省陵水县中国水产科学研究院南海水产研究所热带水产研究开发中心随机抽取90对卵形鲳鲹亲本。人工催产孵化后,对幼鱼进行标准化养殖管理。随机选取500尾3月龄的幼鱼 [体质量 (22±2.9) g] 运至车间,以流水的养殖方式暂养2周,水体溶解氧水平维持在 (8.8±0.2) mg·L-1,水体pH为8.1。在暂养期间,每天投喂2次商品配合饲料,饲料投喂量为鱼体体质量的5%,实验开始前24 h停止投喂。
1.2 低氧处理
将500尾卵形鲳鲹转移到体积为3 m3的养殖水桶中。在实验过程中,溶解氧含量由溶解氧测定仪 (WTW multi3620,德国) 监测。实验开始时溶解氧质量浓度为8.8 mg·L-1,实验开始后,关闭流水和氧气,以向水中注入氮气的方式降低溶解氧水平,1 h内将溶解氧水平降至窒息点 (1.3 mg·L-1),以1.3 mg·L-1水平一直维持到实验结束。当卵形鲳鲹在第一次触底后不能继续游超过10 s时,定义为死亡。快速捞起死鱼,记录死亡时间。收集鳍条保存在乙醇中,每个鳍条样品与死亡时间相对应。按照卵形鲳鲹的死亡时间分为2组,死亡顺序的前10%为缺氧敏感组,后10%为缺氧耐受组。对收集的共100尾卵形鲳鲹进行全基因组重测序。
1.3 DNA提取及测序
收集的鳍条组织使用MagPure tissue DNA试剂盒 (Magen,广州)并按照说明书步骤提取DNA。用1.0%琼脂糖凝胶电泳 (Biowest agrose, 西班牙)和 NanoDrop 2000 (Thermo Scientific, 美国) 分别测定提取的DNA的质量和浓度。检测合格的DNA送到天津诺禾致源生物公司进行全基因组重测序。
1.4 基因组重测序
基因组DNA被随机剪切成350 bp的片段。经过末端修复、磷酸化和添加poly-A尾构建DNA文库,DNA文库在Illumina HiSeq PE150平台进行重测序。100尾卵形鲳鲹重测序获得的原始数据使用软件fastq 0.23[25]进行质量过滤。过滤条件为:1) 删除包含接头 (adapter) 的reads;2) 删除N含量超过5%的reads;3) 删除低质量reads (Qphred≤20) 的碱基数占整个reads长度的50%以上的reads。
1.5 InDel的检测与注释
利用BWA 0.7.17软件将过滤后的100尾卵形鲳鲹的reads与参考基因组进行比对定位,并输出BAM文件。使用gatk 4.2.6.0软件[26]检测每个样品的InDel并统计数目,检测得到包含每个个体InDel的VCF文件和gff3格式的基因组注释文件。通过snpEff 5.1和ANNOVAR软件[27-28]获得InDel在基因组的位置以及变异位点为同义突变或者为非同义突变的信息。
1.6 变异基因功能注释
比较低氧敏感组和耐受组两组的差异InDel,筛选低氧耐受组所特有的InDel位点,并定位到变异的基因。通过ClusterProfiler对变异基因进行GO、KEGG富集分析,筛选与耐低氧相关的通路。通过KEGG注释筛选耐低氧相关的候选基因,进行基因功能分析。
1.7 InDel与耐低氧性状的关联分析
利用PLINK 1.90b6.21软件[29]对100尾卵形鲳鲹InDel数据进行质控,去除次要等位基因频率小于5%和个体变异缺失率大于5%的位点,并进行主成分分析 (Principal component analysis, PCA),并使用ggplot2软件包可视化PCA图。使用GCTA 1.26.0软件[30]评估个体间的亲属关系系数。最后,利用GEMMA 0.98.3软件[31]的线性混合模型 (Linear mixed model, LMM) 将InDel位点和表型数据结合起来进行全基因组关联分析。计算模型为:
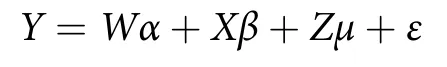
式中:Y为个体在缺氧胁迫下的生存时间;W为协方差矩阵;α为群体均值和显著主成分的协变量;X为固定效应矩阵;β为每个InDel位点的效应值;Z为基于InDel的亲缘关系矩阵;μ为加性遗传效应;ε为残差向量。关联分析结果利用ggplot2包绘制曼哈顿图和QQ (Quantile-Quantile) 图。
在显著相关InDel标记上下游50 kb的区域内筛选候选基因。利用BLAST在非冗余核酸数据库中进行序列比对,确定基因。根据基因的位置和功能,初步确定候选基因。
2 结果
2.1 测序结果质量分析
通过使用Illumina平台对100尾卵形鲳鲹进行PE150测序,生成了693.48 Gb原始序列数据,并保存在NCBI数据库 (PRJNA658481)。统计100尾卵形鲳鲹的测序数据,测序读段在每个位置的GC含量约为41%,且在整个测序过程基本稳定不变,呈水平线,无偏好性。前几位碱基在核苷酸组成上有一定偏好性,产生波动是由于测序读段连接的接头所引起 (图1-a)。碱基的测序质量Q30平均为90.8% (Q30即碱基正确识别率为99.9%),表明碱基测序的出错率很低,保证了后续分析的准确性 (图1-b)。根据PE150测序技术的特点,测序片段末端的碱基质量一般会比前端低。
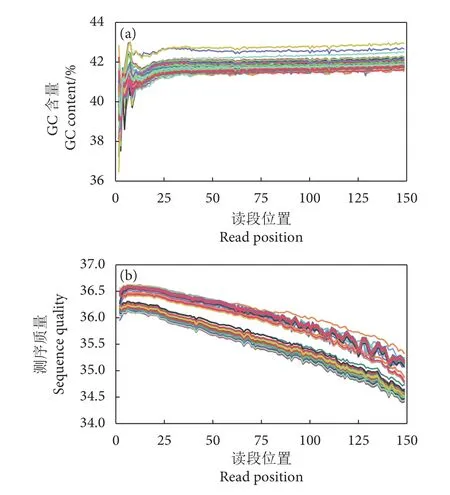
图1 卵形鲳鲹基因组 GC含量和测序质量分布图Fig. 1 GC content and sequence quality in T.ovatus
2.2 InDel序列长度及分布分析
通过GATK 4.2.6.0分析共检测到2 574 178个InDel位点。其中插入和缺失的个数分别为1103610和1 470 568个,缺失个数多于插入个数。大部分插入或缺失的片段大小为1~5 bp,分别占各自总数的75.8%和76.6%。1个碱基的插入和缺失的数量最多,分别为496 002和532 256个 (表1)。通过对InDel在基因组上的位置分析,发现有34.51%和35.64%的InDel位于基因间和内含子上,占据了InDel总数的一半以上,只有极少数 (0.68%) 的InDel位于外显子上 (图2)。本研究中采用了重测序的方法对InDel筛选,获得的InDel平均密度为372.1 InDel·Mb-1,24条染色体中,密度最大的是17号染色体 (4 886.6 InDel·Mb-1),密度最小的是4号染色体 (3 400.9 InDel·Mb-1),表明17号染色体发生最多的变异 (图3)。
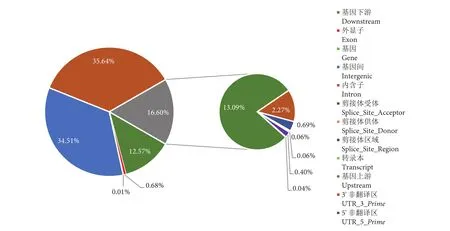
图2 InDel在基因组上的分布位置Fig. 2 Region of InDel on genome
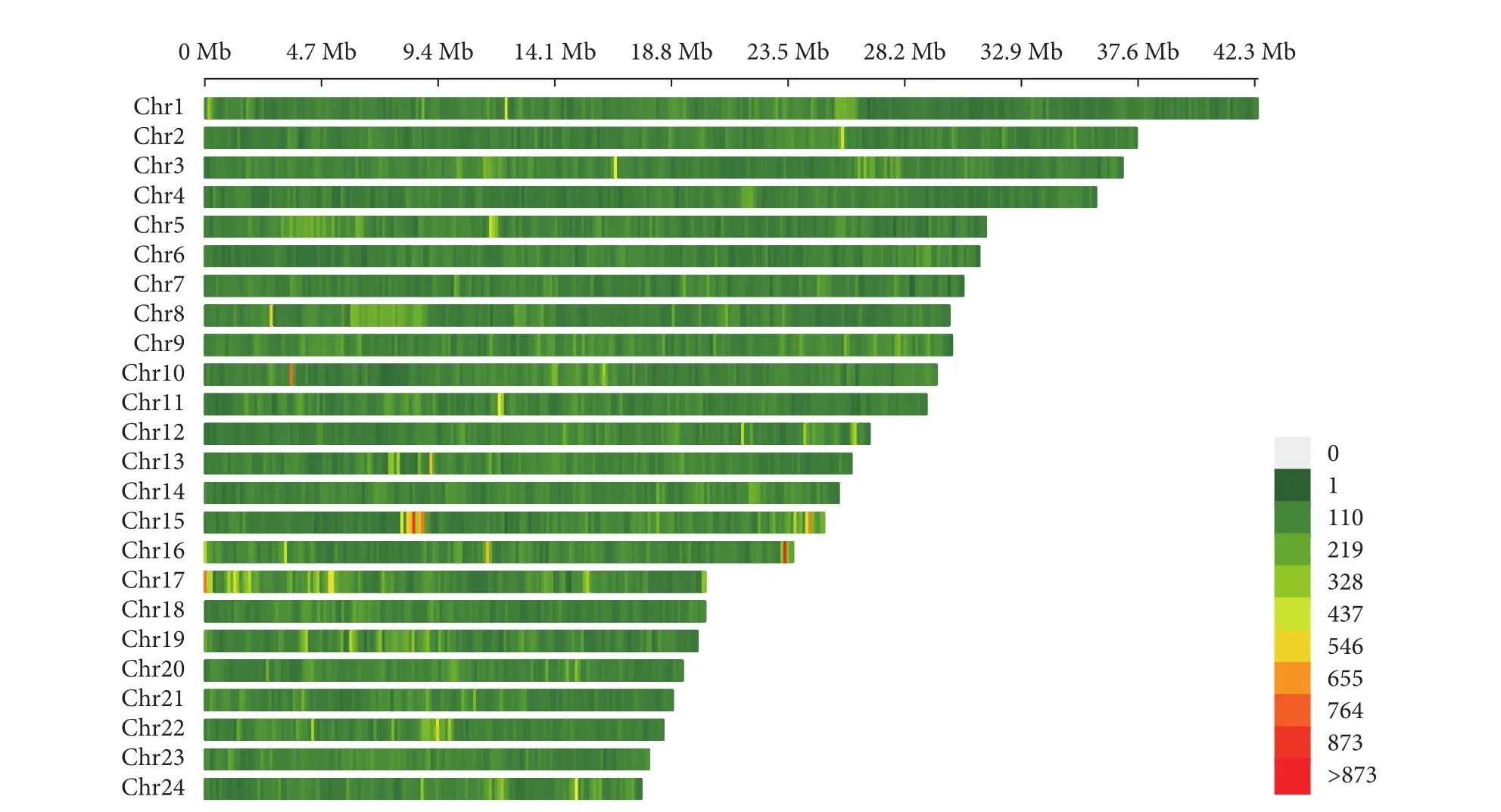
图3 InDel在基因组上的分布密度Fig. 3 Distribution density of InDel on genome
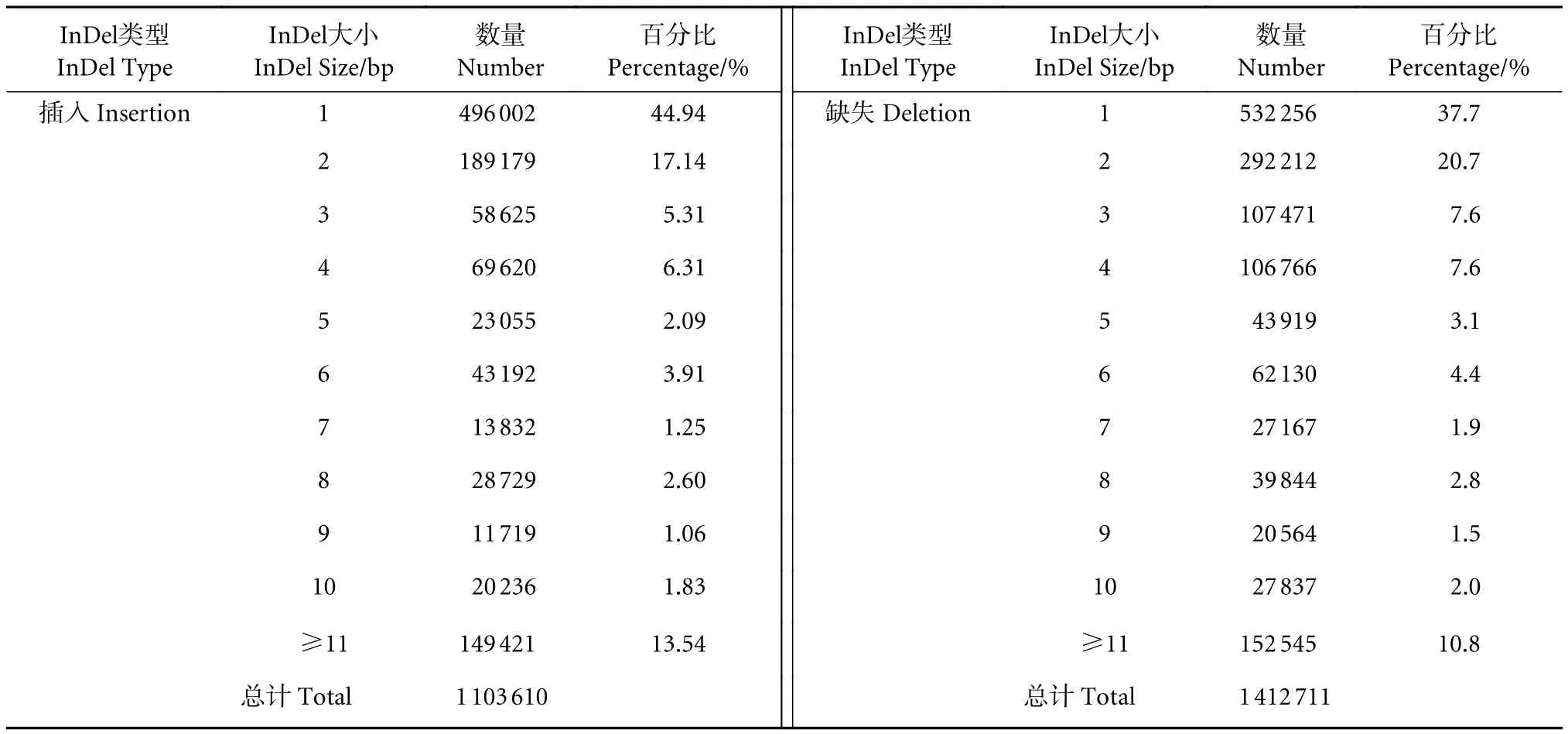
表1 鉴定到的不同长度InDel的数量Table 1 Number of InDels of different sizes identified
2.3 耐受组特有InDel分析
分别对敏感组和耐受组进行InDel筛选,敏感组和耐受组分别筛选到1 723 005和1 720 945个InDel,其中耐受组所特有的InDel 249 395个。使用snpEff 5.1和ANNOVAR软件对耐受组所特有的InDel进行基因分析,249 395个InDel中有2209个位于外显子上,涉及543个基因。通过GO注释分析,多数变异基因主要富集在染色体和顶体泡的组成、核酸结合和GTP结合的分子功能和细胞对DNA损伤刺激的反应,以及Wnt信号通路上 (表2)。通过KEGG富集分析,这些基因主要富集在核苷酸切除修复信号通路和细胞黏着分子上 (图4)。
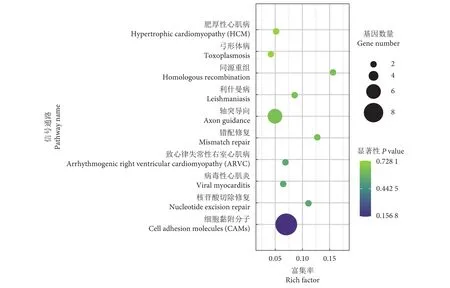
图4 KEGG富集分析Fig. 4 KEGG enrichment scatter plot

表2 变异基因GO功能分类注释Table 2 GO classification of mutated genes
2.4 InDel与耐低氧性状关联分析
利用InDel位点对实验群体进行主成分分析和亲缘关系分析。亲缘关系分析发现,大多数个体间的亲缘关系介于-0.1~0.3 (图5-a)。在主成分分析中,主成分因子1和2将实验样本分成5组 (图5-b)。采用GEMMA 0.98.3软件的线性混合模型对耐低氧性状进行全基因组关联分析,观察到的InDel位点的P值大于其预期值 (图5-c),表明线性混合模型处理样本数据是合理的,所得结果是可靠的。根据Bonferroni校正,阈值设置为P= (0.05/N),其中N为突变位点的数量。在本研究中,通过PLINK 1.90b6.21软件质控后,共有336 391个InDel位点被用于卵形鲳鲹耐低氧性状的关联分析。因此,本研究中的阈值设置为-lg (0.05/336 391)=6.83。由于Bonferroni校正过于严格会导致假阴性,所以需要根据曼哈顿图调整阈值[32-33]。阈值设置为-lg (1/336 391)=5.53作为建议显著性阈值,且达到该阈值的InDel位点可能与耐低氧性状关联。336 391个InDel位点中无InDel位点达到显著性阈值,包括调整后的阈值。但3个InDel位点的P值接近建议性显著性阈值。其中2个InDel位点位于3号染色体,根据InDel在染色体上的位置对其编号为In-Del22883061和InDel24919481,对应的-lgP分别为5.26和5.11。另外一个InDel位于19号染色体,将其命名为InDel14451779,对应的-lgP为5.25 (图 5-d)。
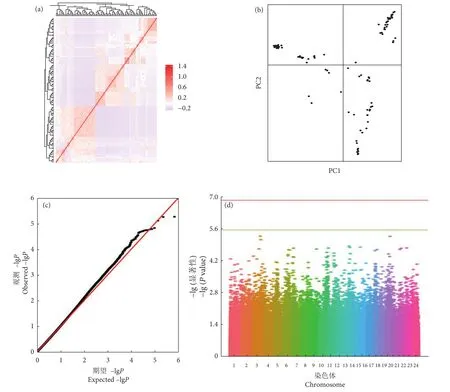
图5 100尾卵形鲳鲹亲缘关系热图 (a)、主成分分析 (b)、耐低氧性状全基因组关联分析的QQ图 (c) 和曼哈顿图 (d)Fig. 5 Heat map of genetic relationship (a), principal component analysis (b), Q-Q plot (c) and Manhattan plot (d) of genome-wide association analysis for low oxygen tolerance of 100 individuals of T. ovatus
2.5 显著InDel基因注释
对重要的3个InDel位点上下游50 kb的基因组序列进行注释后,探索可能与卵形鲳鲹耐缺氧相关的基因。3个位点共注释到9个耐低氧性状相关的候选基因。位于3号染色体上的2个InDel位点共注释到3个基因 (gpr153、acot7和lrfn2b)。另一位于19号染色体的InDel位点共注释到6个基因(tmem237b、mpp4、als2、LOC111232287、LOC111232276和LOC120797970)。
3 讨论
InDel是广泛分布于基因组的结构变异的主要来源之一,是对其他基于序列的遗传标记 (如SSRs和SNPs) 的有价值补充,被公认为遗传分析的有效标记系统,且InDel标记具有密度大、可快速和经济有效地进行基因分型等优点[34]。以往有关鱼类耐低氧性状分子标记筛选研究大多集中在SNP上,很少有关于筛选InDel标记的报道[32-33]。本研究利用重测序技术对100尾卵形鲳鲹进行测序,获得了693.48 Gb的测序数据,其中Q30 (正确识别率大于99.9%的碱基占总体碱基的百分比)平均值为90.8%,测序数据的质量主要分布在Q30(≥80%),这样能够保证后续分析的正常进行。在此基础上,计算了InDel位点在基因组上的平均分布密度为3 972.1 InDel·Mb-1,高密度的InDel位点为挖掘与耐低氧性状关联的位点提供了基础。通过对InDel在基因组位置的注释发现,大部分In-Del位点位于基因间和内含子上 (70.5%),只有极少的InDel位点位于外显子上 (0.7%),证明编码区的InDel位点数量较少,这与之前SNP在基因组上的分布情况一致[35]。这种分布模式不仅存在于卵形鲳鲹中,在狮头鹅 (Shitou goose)、陆地棉(Gossypium hirsutum) 等生物中也表现出相同的分布模式[36-37],位于非编码区的InDel位点多于位于编码区的数量,这一结果符合生物学特性。
通过对敏感组和耐受组的InDel位点进行分析,获得了249 395个耐受组特有的InDel位点,其中2 209个位于外显子上,涉及543个基因。在此基础上对这些基因进行功能注释,发现核苷酸切除修复信号通路和细胞黏着分子中均有InDel变异。核苷酸切除修复是4条DNA损伤修复的基本途径之一。本研究中发现rpa1、ercc3和pold3这3个基因富集到这条通路上,rpa1基因编码异三聚体复制蛋白A复合物的最大亚基,它与单链DNA结合,形成一个核蛋白复合物,在DNA代谢中发挥重要作用,参与DNA复制、修复[38]。pold3基因编码DNA聚合酶delta的66-kD亚基,DNA聚合酶delta具有聚合酶和3'—5'外切酶活性,在DNA复制和修复中起着关键作用[39]。位于这些在DNA复制和修复过程中发挥重要作用的基因上的InDel位点,可能影响了卵形鲳鲹的耐低氧能力。另一较多基因富集的信号通路细胞黏着分子是一种表达于细胞表面的蛋白,参与细胞的活化和信号转导等生物过程[40],这些信号中的变异基因可能影响了低氧环境下卵形鲳鲹信号转导的能力。
采用与敏感组比较、对耐受组所特有的位于外显子上的InDel进行基因注释的方法,获得的位点信息多,注释的基因数量大,仅依靠基因通路和基因功能等信息难以获得可靠的InDel位点用于分子标记的开发。InDel虽然在基因组上分布广泛,但在鱼类分子标记的开发中主要集中在SNP上,对InDel的关注很少,但也有利用InDel位点对斑节对虾 (Penaeus monodon) 早期性别鉴定的应用[41]。虽然在鱼类中仍无利用线性混合模型筛选与性状显著关联的InDel的研究,但已有在芝麻 (Sesamum indicum)、山羊 (Capra hircus) 等动植物中[42-43]的研究,利用全基因组关联分析 (Genome wide association study, GWAS) 的线性混合模型将InDel位点与表型信息关联,挖掘与表型信息显著关联的In-Del位点,Yang等[44]也利用全基因组关联分析的方法筛选到了南丹瑶鸡与鸡冠性状显著相关的In-Del位点。证明利用线性混合模型将InDel位点与性状关联的可行性。本研究为了筛选与耐低氧性状显著相关的InDel位点,首先利用InDel位点进行了主成分分析和亲缘关系分析。通过主成分分析,主成分因子1和2大致将实验群体分为5个群体,亲缘关系分析显示实验群体的亲缘关系大部分介于-0.1~0.3,这与San等[35]利用SNP分析的结果一致,保证了后续关联分析的准确性。在关联分析的结果中,InDel位点的P观察值和期望值相同,说明分析模型是合理的。但所有的P观测值均未明显超过期望值,说明分析结果未找到与耐低氧性状显著关联的位点,这可能是因为耐低氧性状是由微效多基因控制的,单个变异位点效应对性状的影响弱,不能达到显著性阈值;也可能是因为群体样本量小,本研究采用重测序的方法对InDel分型,成本虽然高但可以挖掘稀有变异,所以只对极端表型值的100尾个体进行了重测序,样本量偏小影响了检验效能。关联分析结果发现,没有位点达到显著性阈值,但有3个InDel位点接近显著性阈值,仍具有开发分子标记的价值。通过对显著性位点上下游50 kb的基因序列注释,共注释到9个候选基因,其中Acot7对中、长链脂酰辅酶a具有较高的酶活性。对小鼠 (Mus musculus) 的研究发现,Acot7具有保护神经元免受脂肪酸毒性的作用[45]。Lrfn2b编码一个突触黏附样分子,在小鼠中Lrfn2b可能通过对n-甲基-d-天冬氨酸 (N-methyl-D- aspartate, NMDA) 受体的调节作用来调控红细胞生成[46]。由此推测这2个基因可能在卵形鲳鲹抵抗低氧环境的过程中发挥作用。Mpp4编码的蛋白是鸟苷酸激酶家族中的一种视网膜特异性支架蛋白,参与组织光感受器带状突触中的突触前蛋白复合物[47]。Gpr135编码一个完整的膜蛋白,属于G蛋白偶联受体的A类视紫红质超家族,在大鼠 (Rattus norvegicus) 中敲除后其食物摄入量受到影响[48]。Als2编码的蛋白可以激活GTPases的Ras超家族成员,研究发现als2基因被敲除的斑马鱼(Danio rerio) 出现了严重的发育异常、游泳障碍和运动神经元紊乱[49]。Tmem237b编码一种四跨膜蛋白,影响斑马鱼的胚胎发育[50]。这4个候选基因在低氧胁迫下发挥的功能仍需进一步研究。
综上所述,卵形鲳鲹在低氧环境下,核苷酸切除修复信号通路和细胞黏着分子可能发挥了重要作用。通过InDel位点与耐低氧性状的关联分析,挖掘到了3个与选耐低氧能力相关的InDel位点,并对筛选到的InDel位点进行基因注释获得了9个候选基因。筛选到的InDel位点对后期分子标记选择育种的选择和鉴定有重要价值,注释到的候选基因为卵形鲳鲹耐低氧的机理研究提供了依据。
