基于SNP 分子标记的宁夏4 个野生枣种群遗传多样性对比分析
2022-10-25严亮亮宋丽华
王 博,严亮亮,张 悦,宋丽华
(宁夏大学农学院,银川 750021)
野生酸枣(Ziziphus jujubavar. spinosa)是一种在经济、生态等方面具有重要价值的野生品种。首先,与栽培枣树相比,野生酸枣树对逆境胁迫具有更强的耐受性,对脆弱与严峻的生态环境具有更广泛的适应性[1];其次,与其他大多数果树的野生种不同,野生酸枣在等位基因多样性、表型性状和采后品质方面差异较大[2];此外,野生酸枣树与栽培枣树品种容易杂交,为培育枣树新品种和改良枣树品质性状提供了大量新的遗传变异[3-4]。然而,作为一种具有极高开发和利用价值的乡土资源,人们对野生酸枣资源缺乏深远的认识,严重影响到该种质资源的保护与遗传改良。
分子鉴定是现在最受欢迎的以量化形式来分析种质资源变化一种方式[5]。张鹏飞[6]、殷晓[7]等利用9 对SSR 引物,对我国北方地区43 个酸枣古树资源进行了分析评价,建立了酸枣古树资源的DNA指纹图谱。张建英等[8]用RAMP 技术对采自河北省、山西省以及辽宁省的31 份酸枣样品进行遗传多样性分析将其进行分类,旨在阐明该树种不同区域生长群体间存在的遗传差异,保护该树种基因资源多样性,同时为酸枣新品种培育工作的种源选择提供理论依据。
目前生产中栽培的枣树(尽管现有品种很多)只捕获了野生酸枣种群中遗传多样性的很小一部分。这种“驯化综合征”导致了栽培枣的遗传背景狭窄与枣树生产的脆弱性,有必要从天然野生酸枣种质中导入抗性基因来改变栽培枣。本研究在前人应用新型的SNP(单核苷酸多态性)分子标记技术的基础上,以分布于宁夏不同生态立地的野生酸枣种群为研究对象,分析野生酸枣的遗传多样性差异与分布状态,了解宁夏野生酸枣的功能基因多态性并开发有关SNP 位点,从分子水平上揭示宁夏野生酸枣不同生态种群的起源、进化关系,拓宽野生酸枣树种质资源的遗传背景,以期为宁夏野生酸枣资源可持续原位保存及创新利用提供理论基础和依据。
1 材料与方法
1.1 试验区概况
本研究在实地勘察的基础上,选取了宁夏吴忠红寺堡区、贺兰山滚钟口、灵武白芨滩以及中卫日照山4 个野生酸枣种群,4 个种群地理分布与信息见图1 和表1。统计每个种群所在地的气候因子与立地条件,中卫日照山立地为荒漠与沙漠过渡带,土壤类型为灰漠土,灵武白芨滩土壤类型为荒漠土和干旱土,贺兰山滚钟口土壤类型为灰棕漠土,吴忠红寺堡土壤类型为棕漠土。由表1 可以看出,4 个种群所在生境之间的差异具有一定的规律性,即4 个种群从南向北年均温度与海拔呈上升趋势,年均蒸发量除吴忠红寺堡种群外均呈由南向北上升的趋势。

表1 各试验区地理概况与基本信息Table 1 Geographical overview and basic information of each tested area

图1 宁夏4 个野生酸枣种群调查分布图Figure 1 Distribution map of four wild jujube populations in Ningxia
1.2 野生酸枣SNP 标记的挖掘
利用 36 个红枣基因型(SRR3095649 —SRR3095689 、 SRR3310162 — SRR3310166 、SRR5041640 、 SRR5041641 、 SRR5041644 和SRR5041645)的序列数据,以及保存于NCBI 序列读库(SRA)中的相关种即台湾青枣 (SRR6267272)和Ziziphusspina-christi (SRR6277366))的序列数据进行SNP 数据挖掘。这些SRA 从数据库中读取下载和映射的枣参考基因组使用BWA 程序。基因组分析工具包(GATK) v3.5 使用带有默认参数的单倍型等位基因进行SNP 调用。然后对低质量等位基因进行筛选(参数:QD< 2.0||FS> 60.0||MQ< 40.0||MQRankSum<-12.5||ReadPosRankSum<-8.0)。同时下载了4 个用于枣树基因组组装的测序数据(SRR3081153、SRR3081197、SRR3081340 和SRR3081342),并包含在GATK SNP 调用步骤中。这些数据被用来作为内部参考,以纠正在枣基因组组装中错误或含糊的序列。在36 个枣树基因型中,包含4 个以上但小于34个等位基因的位点被认为是候选的SNP 位点。为了选择高质量的SNP 进行实验验证,排除了上游80 bp或下游80 bp 的SNP 位点附近可能存在其他SNP 位点的SNP。从发现的假设SNP 中,选择288 个假设SNP 的子集,使用nanofluidic 阵列基因分型系统(Fluidigm Co, South San Francisco, CA)进行验证测试。选用288 个SNP 引物,采用Fluidigm 设计引物,应用于野生枣种群的验证。
1.3 样品的采集和DNA 提取
按照统计学要求,每个种群随机标记30 棵长势基本一致健康的植株,两两之间距离间隔10 m 以上,采样时按照东西南北方向采集每棵树枣吊基部或者顶端健康无病虫害的完整幼嫩叶,采集后立刻放入装有变色硅胶干燥剂(天津市大茂化学试剂厂)的自封袋(10 cm × 15 cm)中干燥保存,用于送检提取DNA。
4 个野生酸枣种群样品编号依次为:101—130为吴忠红寺堡种群;201—230 为贺兰山滚钟口种群;301—320 为灵武白芨滩种群;401—430 为中卫日照山种群。由于灵武白芨滩种群酸枣植株整体较少,适宜采取样本的植株有限,为保证检测结果的准确性,故只选取20 株长势条件较为良好的酸枣植株进行采样,即采集20 袋叶片,其余3 个种群各采集30 袋叶片,4 个种群共计110 袋叶片。
取100 mg 干燥的叶片组织,采用CTAB(十六烷基三甲基溴化铵)法提取DNA。CTAB 提取缓冲液组分为:2.5% CTAB,1.5 mol·L-1NaCl, 0.025 mol·L-1EDTA, 0.1 mol·L-1Tris-HCl (pH=8.0), 0.5%β-mercaptoethanol, 4% PVP-10。采用超微量紫外分光光度计 (Thermo Scientific, Wilmington, DE, USA)测定DNA 在260、280 nm 处的吸收值,通过OD260/OD280检测纯度。DNA 样品浓度(µg·mL-1)计算: OD260×50 µg·mL-1×稀释倍数。提取的DNA 样本随后送往美国农业部贝尔茨维尔农业研究中心实验室用于进一步分析。
1.4 分子标记与基因分型
选取酸枣染色体上分布均匀的124 个SNP 位点进行检测和分析,采用纳米流控基因分型系统对野生酸枣种群中可能存在的SNP 标记进行了评价,124个SNP 位点的选择是基于多态性水平和两两连锁关系。当两个SNP 标记物表现出显著的连锁不平衡时,剔除信息指数值较低的那一个。所选124 个标记物被用于组装SNPtype 基因分型板(Fluidigm Corp)。
采用Fluidigm 96.96 动态ArrayTM (Fluidigm,San Francisco, CA) SNP 方法对野生酸枣进行基因分型。Fluidigm 96.96 动态数组同时运行96 个样本和96 个SNPs,生成9 216 个数据点。该基因分型试验包括一个特定的靶向扩增(STA)步骤,丰富了基因组DNA 模板,增强了单个集成流体电路(IFC)反应。使用Fluidigm EP1TM 成像仪拍摄的端点荧光图像。数据使用 Fluidigm 基因分型分析软件(Fluidigm, San Francisco, CA)进行分析。
1.5 遗传多样性分析方法
124 个SNP 位点的统计数据包括Shannon 信息指数(I)、观测杂合度(Ho)、期望杂合度(He)和近交系数(FIS)。本研究利用预期杂合度来估计遗传多样性。首先使用GenAlEx 6.5 程序[13-14],然后对每一个确定的自然种群进行分析。采用HW 检验用于检验哈迪-温伯格均衡的偏离,由Genepop 在Web 程序上执行(http://genepop.curtin.edu.au/)。
采用GenAlEx 6.5[14]中的距离法计算分析样本的两两遗传距离。利用相同的程序,根据两两距离矩阵结果进行主坐标分析(PCoA)。
基于贝叶斯聚类分析方法,利用软件结构v2.3.4[15]确定野生枣样本的种群结构。分析包括从4 个自然种群中抽取的全部140 棵枣树。聚类的数量(k 值)是在变量1 到10 之间建立的,并在不假设任何关于每个样本的遗传组或地理起源的先验信息的情况下进行分析。对每个已建立的集群(K)进行10次独立运行分析,每个集群的烧坏周期为100 000次,然后重复200 000 次MCMC。ΔK 值与目标检测最可能的计算数量的集群[16]使用结构程序。用计算机程序Clupp v1.1.2[17]和DISTRUCT v1.1[18]分别对排列结果进行汇总,并将估计的隶属度系数可视化。在任何集群中具有≥75%的成员(q 值)的加入都被视为该集群的正式成员,在任何集群中成员少于75%的加入都被认为是其贡献集群的混合。
采用分子方差分析(AMOVA),利用GenAlex 6.5 程序[14]分析样本的群体间分化。总分子方差被描述为来源区域之间和内部的组成部分,以及已确定种群之间和内部的组成部分。统计(类似于莱特的统计量)使用999 个随机排列进行统计显著性检测。此外,使用GenAlEx 6.5 程序[13-14]计算每对自然种群的固定指数(Fst)。
综上所述,为比较宁夏野生酸枣种群遗传多样性,利用AMOVA 分析了野生枣种群的总分子变异量,统计测试使用999 个随机排列。
2 结果与分析
2.1 基于SNP 分子标记的野生酸枣种群遗传特性分析
2.1.1 SLAF 标签和 SNP 分子标记的鉴定 通过序列分析,共获得了742 386 个SLAF 标签,平均测序深度为12.68,达到了SLAF 试验预期。从这些SLAF标签中共鉴定到多态性SLAF标签425 436个。本研究中酸枣的SNP 标记的开发是以枣基因组作为参考基因组,利用BWA[19]将测序reads 比对到参考基因组上,并使用GATK[20]和samtools[21]两种方法得到的SNP 标记交集作为最终可靠的SNP 标记数据集,共得到2 589 414 个群体SNP。这些SNP 的平均完整度(即SNP 所在位点在全部样品中的信息完整度)介于0.17% ~ 50.68%之间。此外,从分析所鉴定到的SNP 可以看到,不同样品的SNP 杂合率存在较大差异,多数样品具有较低的杂合率,在0.20% ~13.13%之间,表明其基因组的纯合度很高。
2.1.2 遗传结构分析 遗传多样性最直接的表现是遗传变异水平的高低,但是对于个体来说受到外界因子影响较大而且个体的寿命有限,由个体组成的种群是连续不断的,所以种群才是遗传进化的单位。这些种群在自然界中具有特定的分布模式,即种群的遗传结构。因此,研究种群的遗传多样性比研究个体的遗传多样性更具有实际意义。
贝叶斯聚类分析表明,110 棵野生酸枣分为两个基因集群,等位变异频率特征类型数K= 2 时,出现拐点。统计结果[22]表明,宁夏中卫日照山、灵武白芨滩、贺兰山滚钟口种群在地理结构上呈中北部分区,而宁夏吴忠红寺堡种群则形成了不同的类群(图2 和图3)。

图2 K 值与DeltaK 之间的变化趋势Figure 2 The change trend between K value and DeltaK
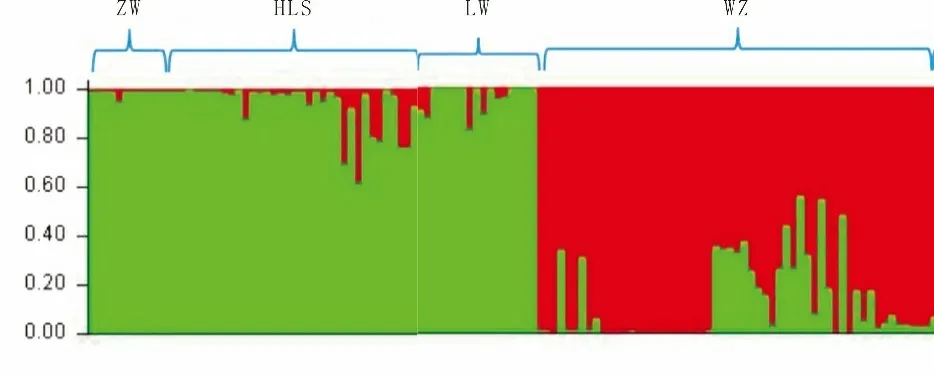
图3 4 个野生酸枣种群的群体遗传结构分析Figure 3 Analysis of population genetic structure of 4 wild jujube populations
2.1.3 遗传分化分析 根据两两Fst值确认种群分化(表2,平均Fst= 0.144)。其中分化程度最高的是宁夏中卫日照山种群与灵武白芨滩种群,其Fst值为0.191,属较大分化。分化程度最低的是宁夏贺兰山滚钟口种群与灵武白芨滩种群,其Fst为0.084,属中等分化程度,表明这两个种群之间可以聚为一簇,两个种群间亲缘关系较近,这一结果与贝叶斯聚类分析结果一致。分化程度越高则遗传多样性越高。
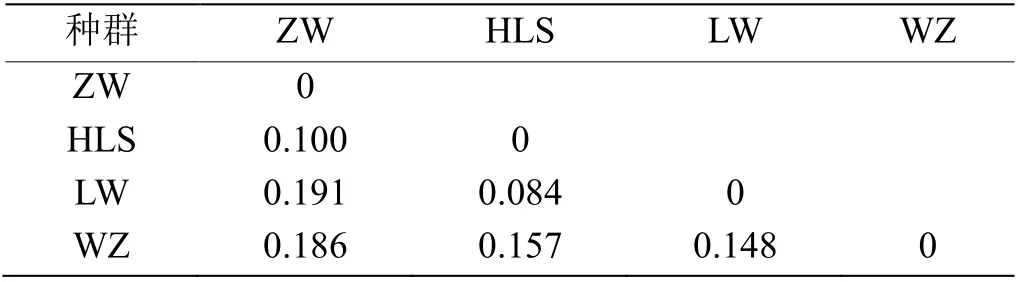
表2 宁夏4 个野生酸枣种群的Fst 值分析Table 2 Fst values of 4 wild jujube populations in Ningxia
该结果得到了分子方差分析(AMOVA)的验证,其中方差被划分为区域间和区域内、种群间和种群内的分量。在两个区域(北部和中部)中,总遗传变异的17%被分割,其中的31%被分割在不同的种群中,52.0%的总遗传变异被分割在种群内。这一结果可能是因为4 个种群之间地理位置相隔较远,所以种群之间产生基因交流的概率比较低导致的。
主坐标分析进一步说明了野生枣种群中北部分区的地理结构(图4)。3 个主要的PCoA 轴(占总变异量的49.4%)将110 棵枣树分成两个主簇,代表4 个地理位置的4 个自然种群。该分组模式与贝叶斯聚类分析结果一致。

图4 主坐标分析Figure 4 Principal coordinates analysis
2.1.4 亲缘关系分析 聚类分析进一步说明了4 个野生种群之间的关系(图5),在代表北部和中部地区的两个集群之间有明显的分隔。此外,贺兰山滚钟口种群(HLS)与灵武白芨滩种群(LW)更接近,而中卫日照山种群(ZW)和吴忠红寺堡种群(WZ)各自单独成一类。根据地理分布来看,贺兰山滚钟口种群与灵武白芨滩种群属北部,吴忠红寺堡种群属中北部地区而中卫日照山种群属中部地区。聚类分析结果和各种群地理分布一致,这也与对表型性状、生理指标与品质指标的聚类结果一致。

图5 宁夏野生酸枣种群 (及其同义类群)之间亲缘关系Figure 5 Relationship between wild jujube populations(and their synonymous groups)in Ningxia
2.2 基于期望杂合度下各种群遗传多样性分析
计算了包括110 棵野生酸枣树在内的4 个种群的总统计量,结果(表3)显示,4 个群体的观察杂合度(Ho)平均值为0.342,范围为0.292 ~ 0.385。平均基因多样性(期望杂合度)低于观察到的杂合度,范围为0.192 ~ 0.311,平均为0.253。4 个群体的近交系数(FIS)均为负值,均值为-0.309,范围为-0.471 ~-0.123。所有群体H-W 平衡均因杂合子过多而显著偏离(P< 0.001)。用期望杂合度(He)来评价遗传多样性大小依次为:中卫日照山种群 >贺兰山滚钟口种群 > 灵武白芨滩种群 > 吴忠红寺堡种群。
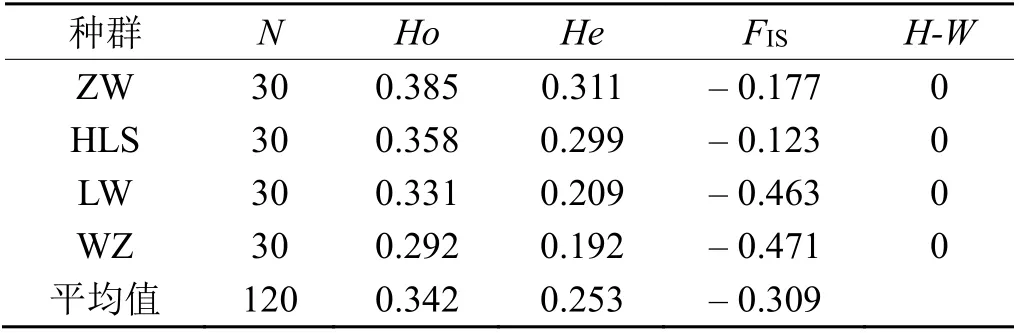
表3 在124 个SNP 位点上对每个群体样本数(N)、观察杂合度(HO)、期望杂合度(He)、近交系数(FIS)和哈迪-温伯格平衡检验Table 3 Observed heterozygosity (HO) expected heterozygosity(He) inbreeding lines (FIS) and hardy-weinberg equilibrium test observed for each population sample number (N)at 124 SNPs
3 讨论与结论
宁夏野生酸枣群体的基因多样性水平较高(He=0.253),这种中等水平的基因多样性可以解释为较长的寿命,重叠的世代和外交交配系统,这是由配子自交不亲和所控制[23-24]。与之前报道的基于微卫星的野生酸枣研究[6]相比,本研究的基因多样性要小得多。这种差异可能是由于SNP 标记是双等位基因,因此相对于SSRs,通常产生较低的基因多样性。
各种群观测杂合度均大于期望杂合度,而且近交系数为负。说明在种群中以杂合子为主,纯合子缺失,4 个群体的HW 平衡有显著偏离(P< 0.001)。这种显著的负FIS可能是由于样本量小的抽样偏差造成。这也可能是由于自交不亲和与部分无性繁殖在小群体中导致了显著的负FIS,如Stoeckel[25]和Navascués[26]等的解释。
通过排列检验,所有两两种群的Fst值均显著,各对种群的平均Fst为0.144,属于中等遗传分化,远大于张春梅等[27]采自覆盖中国北方的样本基于SSR 标记的研究结果(平均Fst= 0.091)。在宁夏,野生酸枣种群主要分布在空间隔离的生境中,在干旱或半干旱环境中形成的小斑块。多年来,生境的破碎化、种子的短距离散布和种群间有限的基因流动,均可加快种群间分化的速度。此外,当小种群在干旱地区分布时,有限的数量可能会产生始祖效应,这也可能导致较大的种群间分化。因此,宁夏独特的立地差异特征导致了宁夏野生酸枣的较大分化。
模型聚类分析和基于距离的聚类分析(PCoA、Structure 和NJ-tree)表明,从4 个种群中抽取的110棵野生酸枣树可以分为两个不同的遗传组(图2、图3 和图5)。第一组来自宁夏北部地区,第二组来自宁夏中部地区。巧合的是,这两个地区也是根据宁夏的农业生态因素划分的[28]。例如,中部地区的年降水量明显高于北部地区。因此,中部地区被划分为半干旱区,而北部地区被划分为干旱区。这两个生态区不同的环境因子可能影响北部和中部两个遗传类群的适应和进化。除占总遗传变异17%的显著区域间差异外,我们的研究还显示了较大的群体间差异。检测到的种群间差异为31%,而种群内差异占52%。通过排列检验,所有两两种群的Fst值均显著,各对种群的平均Fst为0.202,远远大于Zhang 等[2]采自覆盖中国北方的样本基于SSR 标记的研究结果(平均Fst= 0.091)。在宁夏,野生枣种群主要分布在空间隔离的生境中,在干旱或半干旱环境中形成的小斑块。多年来,生境的破碎化、种子的短距离散布和种群间有限的基因流动,均可加快种群间分化的速度。此外,当小种群在干旱地区分布时,有限的数量可能会产生始祖效应,这也可能导致较大的种群间分化。因此,宁夏独特的立地差异特征导致了宁夏野生枣的大分化。
综上所述,4 个野生酸枣种群地理分布上可分为两个主要地理类群(K= 2),分别代表宁夏中部和北部地区。中部地区包括吴忠红寺堡种群,北部地区包括中卫日照山、灵武白芨滩与贺兰山滚钟口种群;4 个野生酸枣种群之间的亲缘关系为贺兰山滚钟口种群与灵武白芨滩种群更接近,而中卫日照山种群(ZW)和吴忠红寺堡种群(WZ)各自单独成一类;4 个野生酸枣种群遗传分化表现为:中卫日照山种群与灵武白芨滩种群分化程度最高,其Fst值为0.191 属较大分化,分化程度最低的是宁夏贺兰山滚钟口种群与灵武白芨滩种群,其Fst为0.084属中等分化程度。
