APOBEC3B调控葡萄膜黑色素瘤复制应激的研究
2022-10-21宗春燕贾仁兵沈键锋
宗春燕,何 杰,张 哲,贾仁兵,沈键锋
上海交通大学医学院附属第九人民医院眼科,上海市眼眶病眼肿瘤重点实验室,上海200011
载脂蛋白B mRNA 编辑酶催化亚基3(apolipoprotein B mRNA editing catalytic subunit 3,APOBEC3) 蛋白家族拥有7 个成员,主要包括APOBEC3A (A3A) 、 APOBEC3B (A3B) 、APOBEC3C (A3C) 、 APOBEC3D (A3D) 、APOBEC3F (A3F) 、 APOBEC3G (A3G) 和APOBEC3H(A3H)[1],主要功能为催化单链DNA(single strand DNA,ssDNA)上的胞嘧啶脱氨形成尿嘧啶,引起突变。APOBEC 相关的突变可见于多种肿瘤,包括乳腺癌、肺癌、宫颈癌、膀胱癌和头颈癌等[2-3]。
研究[4-5]表明,APOBEC3B 在多种类型肿瘤的组织和细胞系中高表达,可能是多种肿瘤的主要诱变剂。APOBEC3B 会导致广泛的C→T 诱变和基因组尿嘧啶水平的升高,从而导致基因组尿嘧啶损伤[6-7],其所引起的突变特征在乳腺癌、宫颈癌、肺癌(包括腺癌和鳞状细胞癌)、头颈癌和膀胱癌中都有所表现[1-2]。APOBEC3B 这种诱变模式可能对癌变过程中体细胞突变起重要作用,并最终导致基因组不稳定[2]。一方面,APOBEC3B 的DNA 诱变能力可促进多种肿瘤发生发展,与其相关的突变可促进肿瘤亚克隆多样性,增强肿瘤耐药性[8]。另一方面,APOBEC3B 诱变导致的基因组不稳定为细胞毒性和免疫疗法提供了重要机会[9-10]。APOBEC3B 高表达与乳腺癌、肺癌、肾癌等多种肿瘤的不良预后(包括耐药性)相关[11-14]。APOBEC3B 高表达使透明细胞卵巢癌对一些基因毒性药物(如顺铂)治疗敏感[15]。APOBEC3B 所诱发的基因组不稳定甚至可能与常规化学治疗(化疗)药物(如铂类)造成的DNA 损伤一起形成新的合成致死组合,使肿瘤细胞对APOBEC3B损伤更敏感。
眼部黑色素瘤主要包括葡萄膜黑色素瘤(uveal melanoma,UM) 和结膜黑色素瘤(conjunctival melanoma,CM),是罕见的黑色素瘤类型,约占所有黑色素瘤病例的5%,但在眼部恶性肿瘤中比例较高[16-17]。其中,UM 是成人最常见的眼内恶性肿瘤,具有高度侵袭性,易复发、转移,一旦发生转移,死亡率极高[18]。目前针对UM 靶向治疗的研究有限,远远不能改善UM 患者预后不良的现状。本研究拟探索APOBEC3B 在UM 中的关键作用通路,确定其作用的关键下游基因,以期寻找UM治疗新的靶点。
1 材料与方法
1.1 实验材料
1.1.1 主要仪器 CO2培养箱(Thermo,美国),电泳装置(Bio-Rad,美国),离心机(Eppendorf,德国),Odyssey红外成像系统(LI-COR,美国),数字切片扫描仪(3DHIESTECH,匈牙利),HP Scanjet 5590 扫 描 仪(HP,美 国),LightCycler®480 Ⅱ(Roche,瑞士),激光扫共聚焦显微镜(Leica,德国)。
1.1.2 主要试剂 RPMI 1640 培养基、DMEM 培养基、胎牛血清、0.25%胰蛋白酶、青霉素-链霉素、嘌呤霉素(Gibco,美国),蛋白裂解液RIPA、氨苄青霉素钠、结晶紫染色液、蛋白酶抑制剂(Sangon Biotech,中国),DH5α 化学感受态细胞(Vazyme,中国)、PolyJet 转染试剂(SignaGen,美国),Trizol(Invitrogen,美国),限制性内切酶XhoⅠ、EcoRⅠ、BsmBⅠ(NEB,美国)、T4 连接酶(Vazyme,中国)、APOBEC3B 抗体(ab184990,Abcam,英国),β-actin 抗体(66009,Proteintech,美国),磷酸化复制蛋白A 32 kDa 亚基(replication protein A 32 kDa subunit,RPA32)抗体(Ser4/Ser8)(A300-245A-M,Bethyl,美国),荧光二抗(A21206,Invitrogen,美国),ProLong™Diamond 抗淬灭封片剂(Invitrogen,美国),细胞周期与细胞凋亡检测试剂盒(Beyotime,中国),细胞周期检测点激酶1(cell cycle checkpoint kinase 1, CHK1) 抑 制 剂 Rabusertib (S2626,Selleck,美国),Applied Biosystems™SYBR™Green预混液(Thermo Fisher Scientific,美国),硫酸羟脲(hydroxyurea,HU)(Selleck,美国)。
1.1.3 细胞和质粒 本研究所使用细胞系包括UM细 胞 系 (92-1、 OMM1、 OMM2.3、 MEL202、MEL270、MEL285、MEL290、MUM2B 和OCM1)、CM 细胞系(CRMM1、CRMM2 和CM2005.1)、皮肤黑色素瘤(skin cutaneous melanoma,SKCM)细胞系(A375、A2058)、视网膜色素上皮(retinal pigment epithelium,RPE)细胞,以及人肾上皮细胞293。UM 细 胞 系OMM1、OMM2.3、MEL285 和MEL290 由Martine J. Jager 教授(荷兰莱顿大学医学中心)馈赠,293 细胞系购自中国科学院典型培养物保藏委员会细胞库,其余细胞系均为上海市眼眶病眼肿瘤重点实验室库存。经鉴定,细胞系均无支原体污染。所有UM 细胞、CM 细胞和RPE 细胞均使用含10%胎牛血清的RPMI 1640培养基,A375、A2058和293 细胞使用含10%胎牛血清的DMEM 培养基,于37 °C 含5% CO2条件的培养箱中培养。载体质粒pGIPZ、lentiCRISPRv2 以及病毒包装质粒PMD2.G、PSPAX2均为上海市眼眶病眼肿瘤重点实验室库存。
1.2 实验方法
1.2.1 免疫组织化学染色 眼黑色素瘤患者组织和色素痣组织均来源于上海交通大学医学院附属第九人民医院,经上海交通大学医学院附属第九人民医院伦理委员会批准。组织载玻片通过乙醇进行一系列的脱蜡和再水化,然后用柠檬酸钠缓冲液进行抗原修复。肿瘤切片用含有0.1% Triton X-100 和3% H2O2的5%正常山羊血清在室温下封闭1 h,然后在4 ℃下与APOBEC3B 抗体(1∶100)孵育过夜。免疫组织化学(immunochemistry,IHC)染色完成后使用数字切片扫描仪进行扫描,使用CaseViewer 软件可视化观察扫描图片。
1.2.2 Western blotting 收获细胞并用PBS 冲洗3次,用蛋白裂解液RIPA于冰上裂解蛋白,并在4 ℃下离心收集蛋白质上清液。蛋白质样品定量后取20 μg 总蛋白上样至SDS-PAGE 电泳凝胶跑胶,待蛋白条带分离,完成电泳后将蛋白质转膜至PVDF 膜上。在室温下用5%牛奶封闭1 h 后,将膜与相应浓度的一抗在4 ℃下孵育过夜。第二日用TBST 溶液洗去多余的一抗后,将膜与荧光二抗于室温下共同孵育1 h。在使用TBST 溶液洗去多余的二抗后使用Odyssey 红外成像系统进行显像。使用Image J 软件对Western blotting条带灰度值进行统计。
1.2.3 稳转细胞系构建 ①APOBEC3B-shRNA 质粒构 建 。 靶 向APOBEC3B的 外 显 子 序 列(TTAAAGTTGAAAGTGAATGTGG) 设 计 合 成shRNA 编码序列,经退火后与载体质粒pGIPZ 一起使用限制性内切酶XhoⅠ和EcoRⅠ按照说明书进行酶切,酶切后使用T4 连接酶进行重组,经测序正确后使用。APOBEC3B-shRNA 质粒用于后续APOBEC3B敲低细胞系shAPOBEC3B构建,空载质粒用于对照细胞系shCtrl构建。②CRISPR-Cas9质粒APOBEC3BsgRNA 构 建。 靶 向APOBEC3B的 外 显 子 序 列(AAATCTCCTTTGGGACACAG) 设计合成sgRNA编码序列。使用BsmBⅠ核酸内切酶按照说明书对载体质粒lentiCRISPRv2 进行酶切,使用T4 连接酶将退火后的sgRNA 序列重组于lentiCRISPRv2 载体质粒中测序鉴定,测序结果证实插入片断与设计序列完全一致后使用。APOBEC3B-sgRNA 用于后续APOBEC3B敲除细胞系sgAPOBEC3B构建,空载质粒用于对照细胞系Vector 构建。③慢病毒包装。使用293 细胞进行慢病毒的包装,待细胞密度到达70%以上(10 cm皿)时进行。将60 μL PolyJet 转染试剂加入无血清DMEM 中配制500 μL PolyJet混悬液。配制含有目的质粒(5 μg)、PMD2.G(5 μg)和PSPAX2(10 μg)的质粒混悬液。室温静置5 min 后,将PolyJet混悬液加入质粒混悬液中,室温静置15 min后滴加至293细胞上清液中。培养6 h 后更换新鲜培养基,48 h 后收集病毒液,病毒经0.22 μm 滤头过滤后用于细胞感染。④靶细胞感染与筛选。将准备感染病毒的细胞以30%的密度接种至培养皿中,待其贴壁后开始病毒感染。更换新鲜培养基后以1∶1 的体积比例将病毒液缓慢滴加至培养基中,并以1∶1 000的比例加入聚凝胺(polybrene)。感染病毒48 h 后更换新鲜的细胞培养基,并使用相应的抗生素如嘌呤霉素(puromycin)进行筛选,使用嘌呤霉素筛选OMM2.3稳转细胞系时使用的浓度为2 μg/mL。筛选1周后使用Western blotting验证APOBEC3B敲低或敲除效率。另外,对APOBEC3B敲除的细胞系进行单克隆挑选,挑选出的单克隆细胞株分别通过Western blotting分析和基因组DNA 测序验证APOBEC3B敲除情况以及基因编辑情况以挑选出成功敲除APOBEC3B的单克隆细胞株。
1.2.4 克隆形成实验 将OMM2.3 sgAPOBEC3B和Vector 细胞按相应的密度接种至6 孔板,每组样本设置3个生物学重复。培养2~3周,每隔3 d换液。药物敏感性实验使用OMM2.3 shAPOBEC3B和shCtrl 细胞进行,使用CHK1 抑制剂Rabusertib 处理细胞,每隔3 d 进行加药处理。待长出肉眼可见的克隆后弃去培养液,用PBS 清洗后加入通用型组织固定液(4%PFA)固定15 min。弃去固定液后加入结晶紫染液常温染色20 min,用清水洗净后自然晾干,然后用HP Scanjet 5590 扫描仪进行扫描成像。使用Image J 软件对结果进行统计。
1.2.5 细胞周期分析 收集2×105个shAPOBEC3B及对照shCtrl OMM2.3 细胞,PBS 清洗细胞1 次后使用70%乙醇固定细胞,于4 ℃冰箱固定过夜。固定完成后用冰PBS 清洗,参照试剂盒说明书比例加入核糖核酸酶A(ribonuclease A,RNase A)以及碘化丙啶(propidium iodide,PI)染色30 min,流式细胞仪上机分析,得到原始数据后使用Flowjo 软件分析。
1.2.6 转录组测序 本研究中的转录组学测序(RNA sequencing,RNA-seq)由北京诺禾致源生物科技公司完成。每组共设置2 个生物学重复。使用Trizol 法从细胞中提取APOBEC3B敲低和对照OMM2.3 细胞总RNA,通过Agilent 2100 Bioanalyzer生物芯片分析系统检测RNA 完整性和总量,每个样品约需要1 μg 的总RNA。在转录组文库构建完成并质检合格后上机测序,对各组样本进行基因表达水平定量。直接从基因组网站下载参考基因组文件(http://hgdownload.cse.ucsc.edu/goldenpath/hg38/bigZips/hg38.fa.gz)和基因模型注释文件(https://ftp.ebi.ac.uk/pub/databases/gencode/Gencode_human/release_33/gencode.v33.primary_assembly.annotation.gtf.gz),计算每个基因的每千个碱基的转录每百万映射读取的片段数(fragments per kilobase of exon model per million mapped fragments,FPKM)值。对差异表达的基因进行基因集富集分析(gene set enrichment analysis,GSEA),使用基因本体(gene ontology,GO)、京都基因与基因组百科全书(kyoto encyclopedia of genes and genomes,KEGG)、Reactome、人类疾病分类(disease ontology,DO)、基因本体分析生物过程(gene ontology biological process,GOBP) 数据集,将基因按照在2 类样本中的差异表达程度排序,检测基因集合的表达变化。使用GraphPad Prism 7.0 软件绘制差异基因表达热图。
1.2.7 实时荧光定量PCR 使用Applied Biosystems™SYBR™Green 预混液配制10 μL PCR 反应体系,使用LightCycler®480 Ⅱ仪器进行实时荧光定量PCR(quantitative real-time PCR,qRT-PCR),使用配套软件进行CT值计算和可视化分析,以甘油醛-3-磷酸脱氢 酶 (glyceraldehyde-3-phosphate dehydrogenase,GAPDH)作为对照计算RNA 的表达量。用于qRTPCR的引物序列见表1。

表1 qRT-PCR 检测相关基因的引物序列Tab 1 Genes and primer sequences used for qRT-PCR analysis
1.2.8 免疫荧光染色 将不同处理组的OMM2.3细胞接种于爬片上,待贴壁完全后进行染色。HU 处理组使用2 mmol/L HU处理细胞4 h后进行染色。用PBS清洗细胞爬片后,用4%甲醛溶液固定爬片15 min,PBS清洗1 次,接着使用0.3%的Triton X-100 溶液对细胞进行通透。用5%BSA 溶液封闭爬片,待封闭完成后用100 μL 磷 酸 化RPA32 (phosphorylated RPA32,p-RPA32)抗体(1∶200 稀释)4 ℃孵育过夜。PBS洗3 次后加入荧光二抗,常温孵育1 h,PBS 洗3 次。在载玻片上滴1 滴含DAPI 的抗淬灭封片剂,然后将玻片盖在载玻片上。使用激光扫描共聚焦显微镜观察并拍摄照片,DAPI 核染色显示为蓝色,p-RPA32 显示为绿色。
1.2.9 数据库分析 使用GEPIA 网站(http://gepia.cancer-pku.cn)完成对癌症基因组图谱(The Cancer Genome Atlas,TCGA)数据库中UM 患者的预后分析及各类肿瘤中APOBEC3B基因表达情况分析。
1.3 统计学分析
用GraphPad Prism 7.0 软件进行统计学分析。定量资料采用表示。使用非配对t检验比较2组之间的差异。P<0.05表示差异具有统计学意义。
2 结果
2.1 APOBEC3B 在眼部黑色素瘤中的表达情况以及对患者生存的影响
为检测APOBEC3B 在眼部黑色素瘤中的表达情况,对眼部黑色素瘤组织进行了IHC 染色(图1A)。使用APOBEC3B 抗体对福尔马林固定石蜡包埋的良性痣组织和眼部黑色素瘤组织进行染色。首先,染色结果显示着色部位大多在细胞核内,这与APOBEC3 B 定位于核内相符合[19]。另外,良性色素痣组织中APOBEC3B 着色很弱,而在眼部黑色素瘤组织中,每个肿瘤组织切片的大部分细胞核中均可检测到强免疫阳性,说明相对于良性色素痣,眼部黑色素瘤组织中APOBEC3B表达水平更高。
另外,通过GEPIA网站对TCGA数据库中UM患者总生存时间以及无病生存时间的分析发现,APOBEC3B高表达与UM 患者总生存期缩短相关(P=0.032,图1B),且与UM 患者的无病生存期显著缩短相关(P=0.000,图1C)。对TCGA 数据库中肿瘤患者APOBEC3B基因表达情况进行分析,结果表明APOBEC3B在大多数的肿瘤组织中高表达(图1D)。
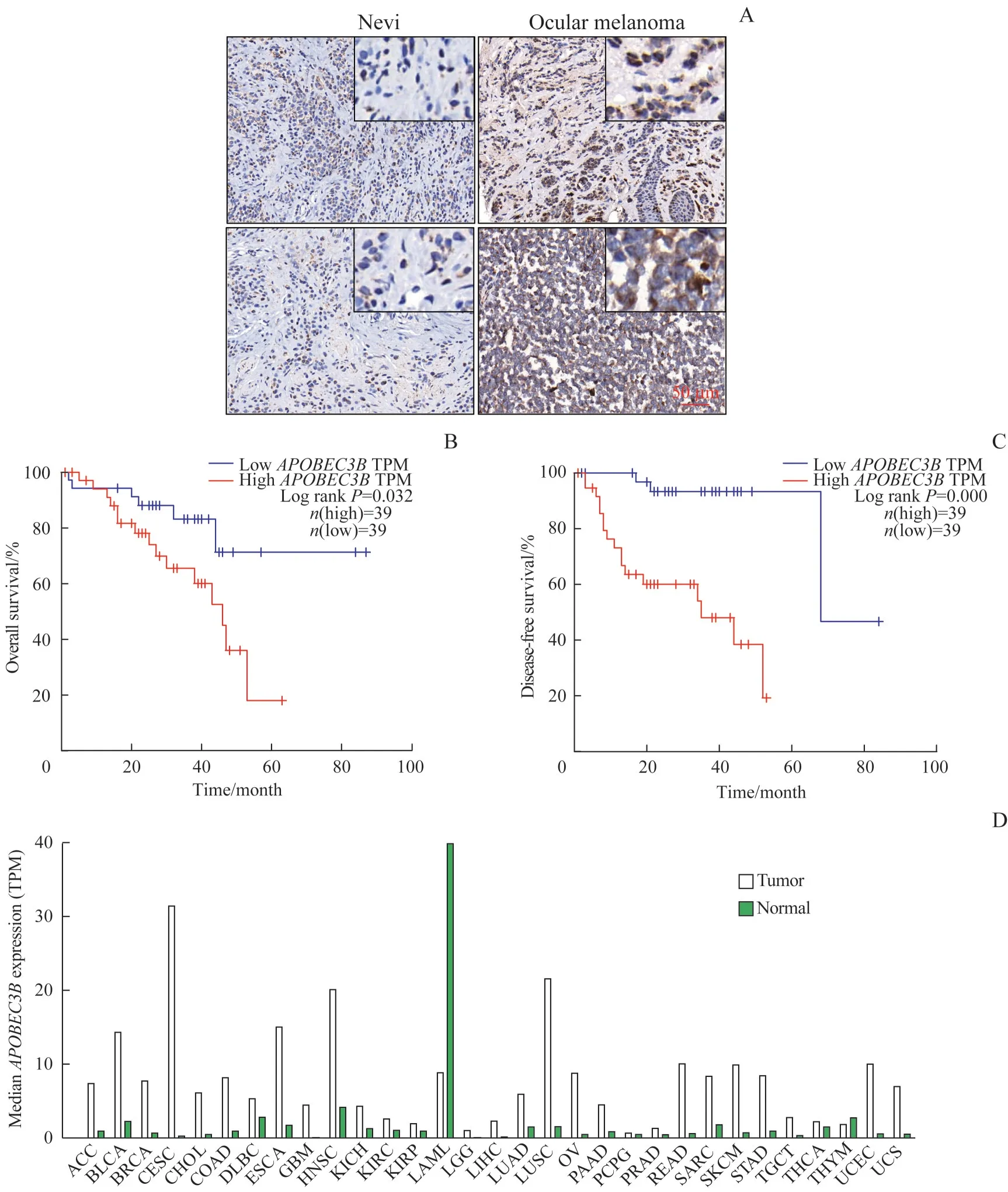
图1 APOBEC3B在肿瘤中的表达情况以及对UM患者生存的影响Fig 1 Expression of APOBEC3B in tumors and its effect on UM patient survival
2.2 APOBEC3B 敲低和敲除的OMM2.3细胞系的建立
以RPE细胞作为对照,应用Western blotting检测UM 细 胞 (92-1、 OMM1、 OMM2.3、 MEL202、MEL270、MEL285、MEL290、MUM2B 和OCM1)、CM 细 胞(CRMM1、 CRMM2 和CM2005.1) 和SKCM 细胞(A375 和A2058)总蛋白表达水平(图2A)。相较于RPE 细胞,大多数黑色素瘤的细胞系,尤 其 是UM 细 胞 系OMM2.3、 92-1、 MEL202、OMM1、MUM2B 中APOBEC3B 蛋白表达均显著升高。
为探索APOBEC3B 对UM 的作用机制,选取高表达APOBEC3B 蛋白的UM 细胞系OMM2.3 进行研究,并建立了APOBEC3B敲低和敲除的稳转细胞系。首先构建了APOBEC3B敲低的慢病毒质粒(shAPOBEC3B)和对照质粒(shCtrl),并将其分别转染至OMM2.3 细胞中,构建APOBEC3B敲低的OMM2.3 细胞系和对照细胞系;Western blotting 验证APOBEC3B 蛋白的敲低效率约为63.9%(图2B)。然后,通过CRISPR-Case9 技术,靶向APOBEC3B基因序列设计APOBEC3B敲除的APOBEC3B-sgRNA 慢病毒质粒。然后将构建好的APOBEC3B敲除质粒sgAPOBEC3B和对照质粒Vector包装成慢病毒,分别感染OMM2.3 细胞;经单克隆细胞株挑选后,使用Western blotting、基因组DNA 测序验证成功敲除APOBEC3B的单克隆细胞株(图2C、D)。结果显示,与对照未敲除APOBEC3B的OMM2.3 细胞相比,成功敲除APOBEC3B的细胞无对应蛋白表达条带,且在靶向序列附近检测到基因编辑。
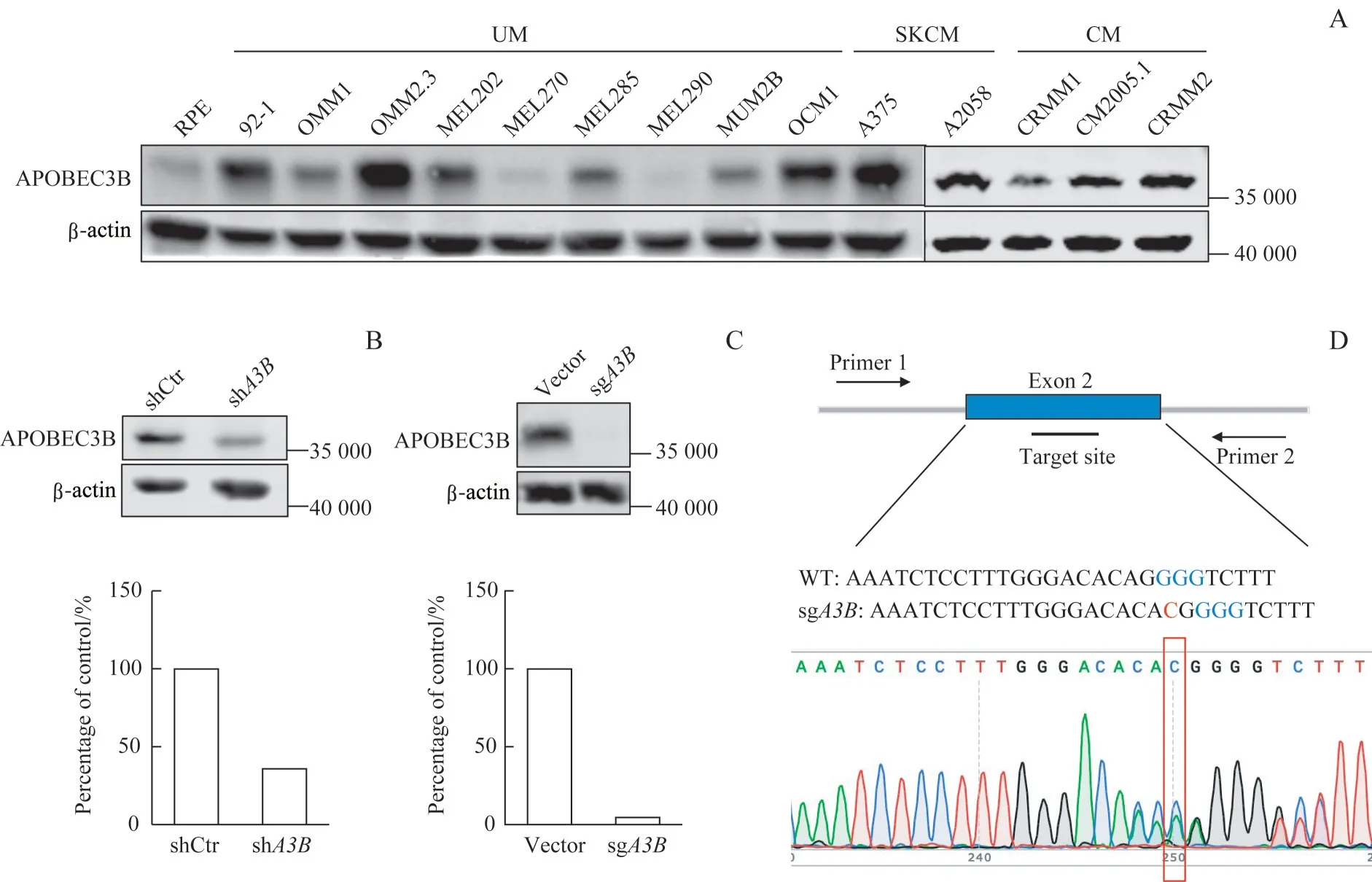
图2 APOBEC3B在肿瘤细胞中的表达情况和APOBEC3B敲低和敲除细胞系的构建Fig 2 Expression of APOBEC3B in tumor cells and construction of APOBEC3B knockdown and knockout cell lines
2.3 APOBEC3B的表达对OMM2.3的克隆形成能力和细胞周期的影响
为研究APOBEC3B 表达对OMM2.3 细胞的影响,对sgAPOBEC3B和Vector OMM2.3稳转细胞系进行克隆形成实验。在孔板中接种等量细胞数的sgAPOBEC 3B和Vector 细胞,待形成肉眼可见的克隆后进行染色,扫描成像(图3A)。结果表明,与对照未敲除APOBEC3B的OMM2.3 细 胞 相 比,APOBEC3B敲 除的细胞并未表现出克隆形成数量的显著改变(P>0.05,图3B)。
另外,通过使用流式细胞术,对shAPOBEC3B和shCtrl OMM2.3 细胞进行细胞周期检测(图3C)。经统计分析发现,APOBEC3B敲低并未导致细胞周期的显著改变(图3D)。这些结果表明,APOBEC3B在OMM2.3中的表达并不影响细胞的克隆形成和细胞周期。
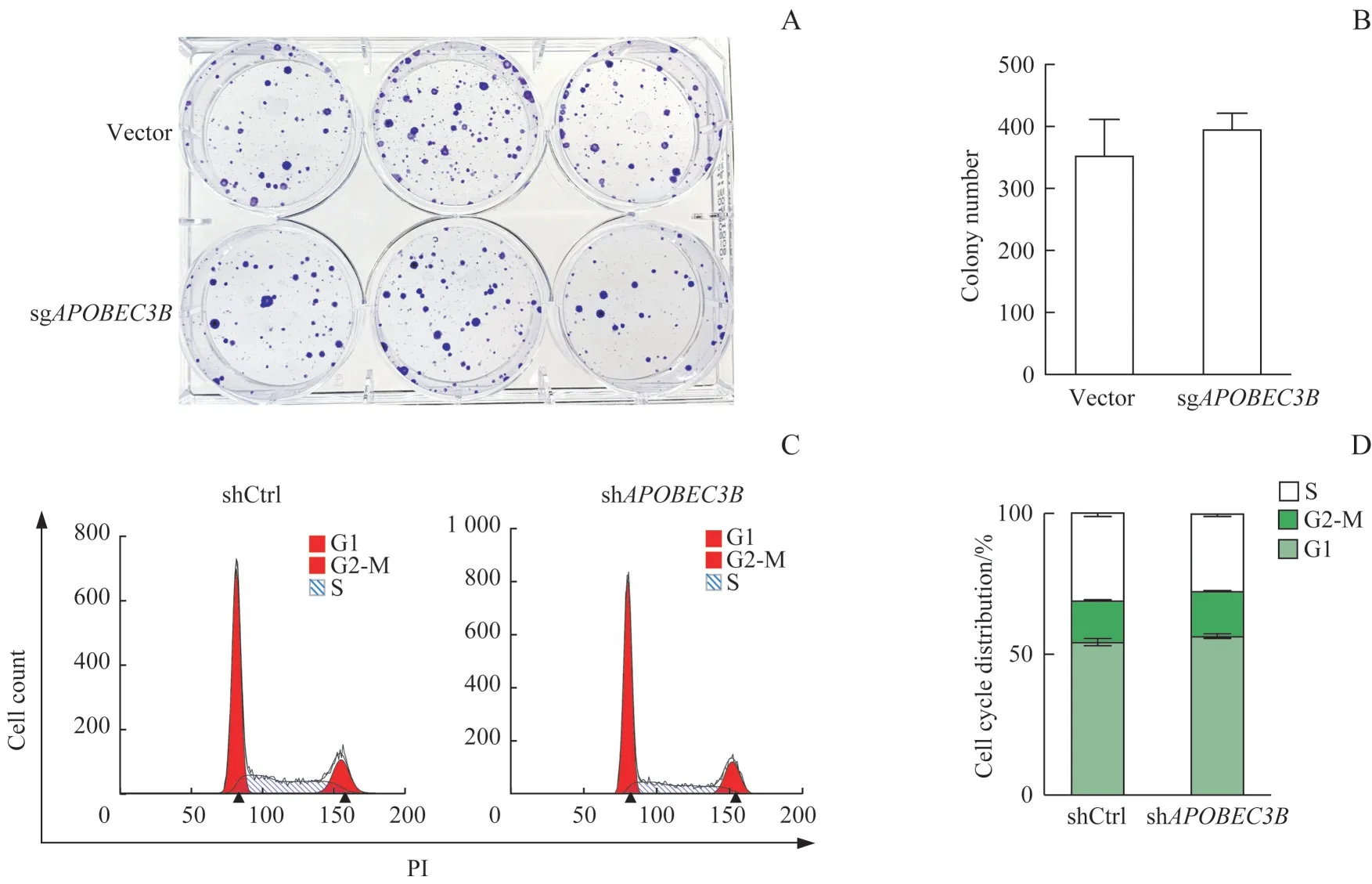
图3 APOBEC3B的表达对OMM2.3细胞的克隆形成能力和细胞周期的调控Fig 3 Clonogenic ability and cell cycle regulation of OMM2.3 cells by APOBEC3B expression
2.4 APOBEC3B对OMM2.3细胞复制应激相关通路的调控
为进一步探索APOBEC3B 在OMM2.3 细胞中的功能,识别APOBEC3B 下游效应子,对建立好的shAPOBEC3B细胞和对照shCtrl 细胞进行RNA-seq。经检测,shAPOBEC3B细胞和对照shCtrl 细胞中存在223个差异表达的基因,其中49个基因在APOBEC3B敲低后表达上调,174 个基因表达下调(图4A)。为研究这些基因的作用通路,通过GSEA 对这些差异基因进行了富集分析,结果表明,差异表达的基因富集到了一些DNA 复制通路(图4B)。对shAPOBEC3B细胞和对照shCtrl 细胞差异表达的基因进行热图分析,结果也表明,APOBEC3B敲低影响了一些复制应激相关通路基因,如增殖细胞核抗原基因(proliferating cell nuclear antigen,PCNA)等表达的改变(图4C)。同时,通过RT-qPCR对差异基因进行检测,验证了细胞周期蛋白H(cyclin H,CCNH)、黑色素瘤抗应激通路的介质多巴色素互变异构酶(dopachrome tautomerase,DCT)等下游基因表达改变(图4D)。因此推测,APOBEC3B 可能在DNA 复制过程中通过其催化突变的作用,参与OMM2.3细胞的复制应激过程,从而有可能使其对复制应激通路的靶向药物敏感。
在shAPOBEC3B细胞和对照shCtrl 细胞中,对复制应激通路关键靶标p-RPA32进行免疫荧光染色。结果表明APOBEC3B敲低后p-RPA32 荧光强度明显降低,并且在HU 诱导复制叉停滞、复制应激通路被激活时,这种现象更为明显(图4E)。为研究APOBEC3B 的表达是否会影响OMM2.3 细胞对复制应激通路靶向药物的敏感性,挑选CHK1 抑制剂对shAPOBEC3B和对照shCtrl OMM2.3 稳转细胞进行药物敏感性研究。将相同数量的APOBEC3B敲低及对照OMM2.3细胞系接种于孔板中,待细胞贴壁后对细胞进行CHK1 抑制剂药物处理,并设置不加药的DMSO组对照,待形成肉眼可见的细胞克隆后进行结晶紫染色,并扫描成像(图4F)。克隆形成实验结果表明,在1.25 μmol/L 的CHK1 抑制剂处理下,对照组的克隆形成明显减少(图4G)。而APOBEC3B敲低组的克隆形成数量虽然也有所减少,但是相较于对照组,克隆数量减少程度相对微弱。这表明与敲低APOBEC3B的OMM2.3 细 胞 相 比,APOBEC3B正 常表达的OMM2.3 细胞对CHK1 抑制剂的处理更为敏感。对照细胞系克隆形成数的减少程度明显大于APOBEC3B敲低组,证明APOBEC3B 表达使得OMM2.3细胞对CHK1抑制剂更为敏感。
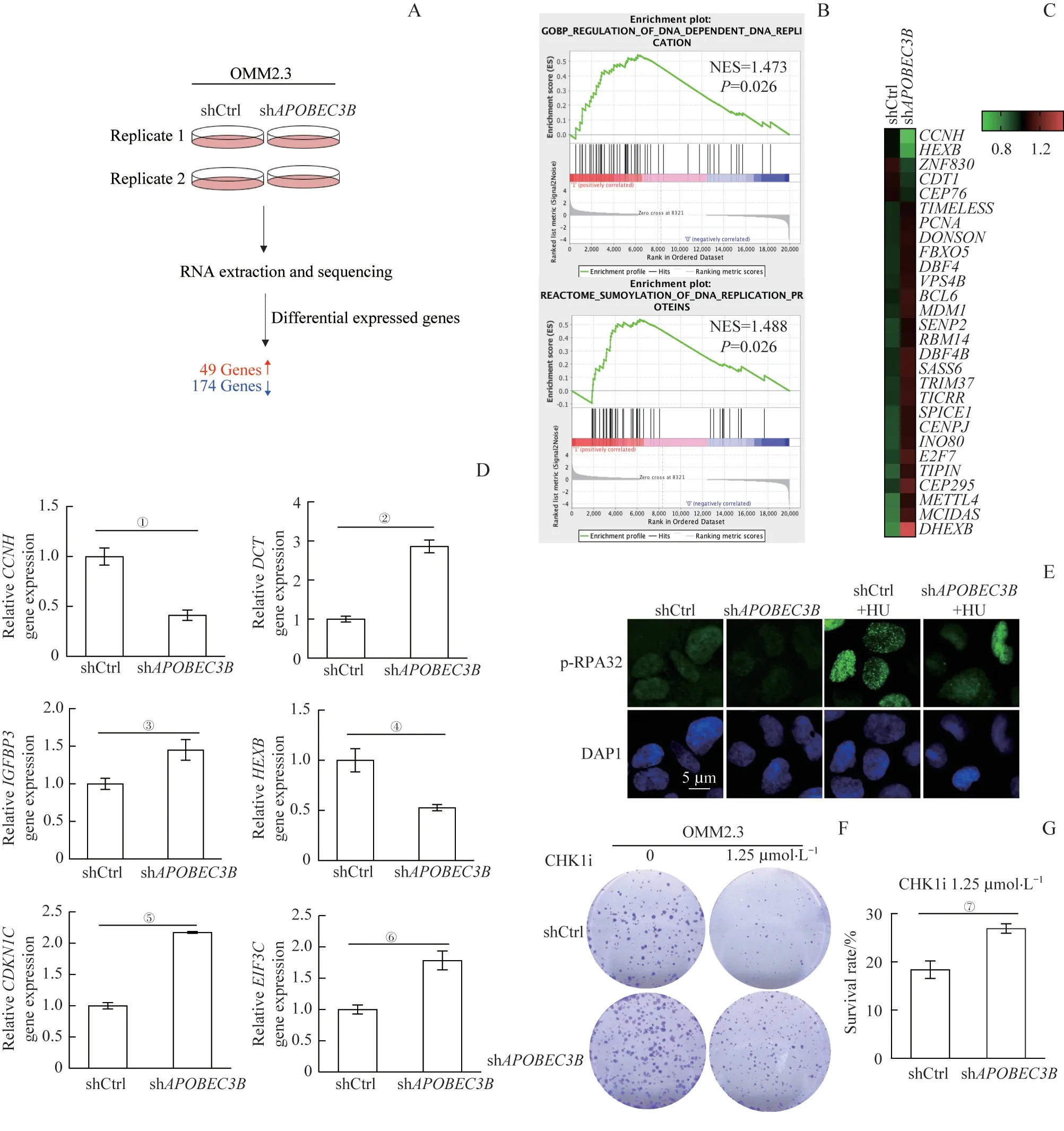
图4 OMM2.3细胞中APOBEC3B对复制应激相关通路的调控Fig 4 Regulation of replication stress-related pathways by APOBEC3B in OMM2.3 cells
3 讨论
由于肿瘤异质性,APOBEC3B 在不同的肿瘤中发挥不同作用。在乳腺癌中,APOBEC3B 高表达肿瘤的突变数量是低表达肿瘤的2 倍;内源性APOBEC3B 蛋白是乳腺癌细胞系提取物中唯一可检测到的具有将DNA 胞嘧啶突变至尿嘧啶的碱基编辑活性物质的来源[5]。研究[11,20]表明,APOBEC3B 是雌激素受体(estrogen receptor,ER)阳性的乳腺癌患者预后不良的生物标志物,且APOBEC3B 诱导的基因异常促进了乳腺癌进展。APOBEC3B 在透明细胞卵巢癌中高表达,且其高表达与预后改善相关[15]。APOBEC3B 表达使肿瘤细胞对顺铂产生的基因毒性作用更为敏感,因此APOBEC3B 可作为预测铂类化疗反应敏感性的候选生物学标志物。APOBEC3B 是胃癌预后独立危险因素,APOBEC3B 调控胃癌的肿瘤微环境,使表达效应分子和免疫检查点分子的肿瘤反应性CD8+T 细胞浸润减少[21]。另外,APOBEC3B还通过诱导S 期阻滞,调控肾上腺皮质癌细胞增殖[22]。而APOBEC3B 在眼部黑色素瘤中的作用尚未见文献报道。
在本研究中,对TCGA 数据库的分析结果显示APOBEC3B在UM 患者中高表达,且显著影响患者总生存期和无病生存期。在眼部黑色素瘤患者的组织样本以及细胞系中也分别验证了APOBEC3B 的高表达,因此推测APOBEC3B 在眼部黑色素发生发展中可能存在着重要功能。文献[23]报道APOBEC3B 在G2/M期显著高表达,可能参与调控细胞周期,然而敲除APOBEC3B并未导致OMM2.3 克隆形成能力和细胞周期的显著改变。APOBEC3B敲低和对照的OMM2.3细胞的RNA-seq 结果显示APOBEC3B敲低后的差异表达基因并不多,提示APOBEC3B 在UM 中作用机制可能处于相对下游的位置。进一步通过GSEA 分析差异基因发现,在OMM2.3 细胞中,APOBEC3B 参与调控DNA 复制通路。鉴于APOBEC3B 促进肿瘤突变负荷增加的功能[24],以及APOBEC3B 在乳腺癌中参与复制应激通路作用的相关报道[25],APOBEC3B可能与OMM2.3 细胞的复制应激过程相关。DNA 复制应激是肿瘤基因组不稳定性的主要来源[26]。APOBEC3B 可通过在DNA 复制叉处诱导无碱基位点,施加复制压力[27]。在APOBEC3B敲低的OMM2.3 细胞中检测到复制应激通路关键靶标p-RPA32水平的降低。在复制应激通路激活后,RPA32会被活化的共济失调毛细血管扩张Rad3 相关蛋白(ataxia telangiectasia and Rad3 related protein,ATR)和DNA 依赖性蛋白激酶(DNA-dependent protein kinase,DNA-PK)磷酸化,从而进一步导致CHK1激活和复制停滞[28]。因此,APOBEC3B 的表达可能会使细胞对一些靶向复制应激的药物敏感,可能作为UM治疗的靶标。
在本研究中,使用复制应激通路靶向药物CHK1抑制剂对APOBEC3B敲低和对照OMM2.3 细胞进行药物敏感性实验,结果发现,APOBEC3B 表达的OMM2.3 细胞对CHK1 抑制剂更敏感, 可见APOBEC3B有潜力作为UM 对CHK1抑制剂治疗敏感性的生物学标志物。然而,也有研究者认为APOBEC3B 活性只是影响复制应激通路靶向药物敏感性的一部分因素[27],其他因素如共济失调毛细血管扩张突变基因(ataxia telangiectasia-mutated gene,ATM)、p53基因丢失等也可能增加药物敏感性[29-31]。APOBEC3B 表达活性是否可以用于预测CHK1 靶向治疗敏感性,以及是否需要结合其他因素预测治疗敏感性,仍需要进一步研究。
总之,APOBEC3B参与OMM2.3细胞复制应激相关通路,有潜力作为评价CHK1抑制剂治疗敏感性的生物学标志物,为靶向治疗UM提供新的选择。
利益冲突声明/Conflict of Interests
所有作者声明不存在利益冲突。
All authors disclose no relevant conflict of interests.
伦理批准和知情同意/Ethics Approval and Patient Consent
本研究涉及的所有实验均已通过上海交通大学医学院附属第九人民医院研究伦理委员会批准(文件号SH9H-2019-T185-2)。所有实验过程均遵照《世界医学大会赫尔辛基宣言》的条例进行。受试对象或其亲属已经签署知情同意书。
All experimental protocols in this study were reviewed and approved by the Ethics Committee of Shanghai Ninth People's Hospital,Shanghai Jiao Tong University School of Medicine (approval letter No. SH9H-2019-T185-2),and all experimental protocols were carried out by following the guidelines of theWorld Medical Association Declaration of Helsinki. Consent letters have been signed by the research participants or their relatives.
作者贡献/Authors'Contributions
宗春燕、何杰参与了生物学实验;张哲参与了数据分析;沈键锋参与了实验设计;宗春燕、贾仁兵、沈键锋参与了论文的写作和修改。所有作者均阅读并同意了最终稿件的提交。
The biological experiment was completed by ZONG Chunyan and HE Jie. Data were analyzed by ZHANG Zhe. The study was designed by SHEN Jianfeng.The manuscript was drafted and revised by ZONG Chunyan,JIA Renbing and SHEN Jianfeng. All the authors have read the last version of paper and consented for submission.
·Received:2022-03-15
·Accepted:2022-06-25
·Published online:2022-07-25
参·考·文·献
[1] SWANTON C, MCGRANAHAN N, STARRETT G J, et al.APOBEC enzymes: mutagenic fuel for cancer evolution and heterogeneity[J]. Cancer Discov,2015,5(7):704-712.
[2] ROBERTS S A, LAWRENCE M S, KLIMCZAK L J, et al. An APOBEC cytidine deaminase mutagenesis pattern is widespread in human cancers[J]. Nat Genet,2013,45(9):970-976.
[3] ALEXANDROV L B, NIK-ZAINAL S, WEDGE D C, et al.Signatures of mutational processes in human cancer[J]. Nature,2013,500(7463):415-421.
[4] KUONG K J, LOEB L A.APOBEC3Bmutagenesis in cancer[J].Nat Genet,2013,45(9):964-965.
[5] BURNS M B, LACKEY L, CARPENTER M A, et al. APOBEC3B is an enzymatic source of mutation in breast cancer[J]. Nature,2013,494(7437):366-370.
[6] CESCON D W, HAIBE-KAINS B, MAK T W. APOBEC3B expression in breast cancer reflects cellular proliferation, while a deletion polymorphism is associated with immune activation[J].Proc Natl Acad Sci USA,2015,112(9):2841-2846.
[7] SOUSA M M L, KROKAN H E, SLUPPHAUG G. DNA-uracil and human pathology[J]. Mol Aspects Med,2007,28(3/4):276-306.
[8] VENKATESAN S, ROSENTHAL R, KANU N, et al. Perspective:APOBECmutagenesis in drug resistance and immune escape in HIV and cancer evolution[J]. Ann Oncol,2018,29(3):563-572.
[9] HOOPES J I, CORTEZ L M, MERTZ T M, et al. APOBEC3A and APOBEC3B preferentially deaminate the lagging strand template during DNA replication[J]. Cell Rep,2016,14(6):1273-1282.
[10] SEREBRENIK A A, STARRETT G J, LEENEN S, et al. The deaminase APOBEC3B triggers the death of cells lacking uracil DNA glycosylase[J]. Proc Natl Acad Sci USA, 2019, 116(44):22158-22163.
[11] SIEUWERTS A M, WILLIS S, BURNS M B, et al. Elevated APOBEC3B correlates with poor outcomes for estrogen-receptorpositive breast cancers[J]. Horm Cancer,2014,5(6):405-413.
[12] YAN S M,HE F,GAO B,et al. Increased APOBEC3B predicts worse outcomes in lung cancer: a comprehensive retrospective study[J].J Cancer,2016,7(6):618-625.
[13] LAW E K,SIEUWERTS A M,LAPARA K,et al. The DNA cytosine deaminase APOBEC3B promotes tamoxifen resistance in ERpositive breast cancer[J]. Sci Adv,2016,2(10):e1601737.
[14] XU L,CHANG Y,AN H M,et al. High APOBEC3B expression is a predictor of recurrence in patients with low-risk clear cell renal cell carcinoma[J]. Urol Oncol,2015,33(8):340.e1-340.e8.
[15] SEREBRENIK A A,ARGYRIS P P, JARVIS M C, et al. The DNA cytosine deaminase APOBEC3B is a molecular determinant of platinum responsiveness in clear cell ovarian cancer[J]. Clin Cancer Res,2020,26(13):3397-3407.
[16] MCLAUGHLIN C C, WU X C, JEMAL A, et al. Incidence of noncutaneous melanomas in the US[J]. Cancer, 2005, 103(5): 1000-1007.
[17] BLUM E S, YANG J, KOMATSUBARA K M, et al. Clinical management of uveal and conjunctival melanoma[J]. Oncology(Williston Park),2016,30(1):29-32,34-43,48.
[18] JAGER M J, SHIELDS C L, CEBULLA C M, et al. Uveal melanoma[J]. Nat Rev Dis Primers,2020,6(1):24.
[19] SALAMANGO D J, MCCANN J L, DEMIR Ö, et al. APOBEC3B nuclear localization requires two distinct N-terminal domain surfaces[J]. J Mol Biol,2018,430(17):2695-2708.
[20] TSUBOI M, YAMANE A, HORIGUCHI J, et al. APOBEC3B high expression status is associated with aggressive phenotype in Japanese breast cancers[J]. Breast Cancer,2016,23(5):780-788.
[21] XIA S Y, GU Y, ZHANG H J, et al. Immune inactivation by APOBEC3B enrichment predicts response to chemotherapy and survival in gastric cancer[J]. Oncoimmunology, 2021, 10(1):1975386.
[22] GARA S K, TYAGI M V, PATEL D T, et al. GATA3 and APOBEC3B are prognostic markers in adrenocortical carcinoma and APOBEC3B is directly transcriptionally regulated by GATA3[J].Oncotarget,2020,11(36):3354-3370.
[23] HIRABAYASHI S, SHIRAKAWA K, HORISAWA Y, et al.APOBEC3B is preferentially expressed at the G2/M phase of cell cycle[J]. Biochem Biophys Res Commun,2021,546:178-184.
[24] NIKKILÄ J, KUMAR R, CAMPBELL J, et al. Elevated APOBEC3B expression drives a kataegic-like mutation signature and replication stress-related therapeutic vulnerabilities in p53-defective cells[J]. Br J Cancer,2017,117(1):113-123.
[25] CESCON D W, HAIBE-KAINS B. DNA replication stress: a source of APOBEC3B expression in breast cancer[J]. Genome Biol, 2016,17(1):202.
[26] MACHERET M, HALAZONETIS T D. DNA replication stress as a hallmark of cancer[J]. Annu Rev Pathol,2015,10:425-448.
[27] BUISSON R, LAWRENCE M S, BENES C H, et al. APOBEC3A and APOBEC3B activities render cancer cells susceptible to ATR inhibition[J]. Cancer Res,2017,77(17):4567-4578.
[28] ASHLEY A K, SHRIVASTAV M, NIE J Y, et al. DNA-PK phosphorylation of RPA32 Ser4/Ser8 regulates replication stress checkpoint activation, fork restart, homologous recombination and mitotic catastrophe[J]. DNA Repair(Amst),2014,21:131-139.
[29] REAPER P M, GRIFFITHS M R, LONG J M, et al. Selective killing of ATM- or p53-deficient cancer cells through inhibition of ATR[J]. Nat Chem Biol,2011,7(7):428-430.
[30] TOLEDO L I, MURGA M, ZUR R, et al. A cell-based screen identifies ATR inhibitors with synthetic lethal properties for cancerassociated mutations[J]. Nat Struct Mol Biol,2011,18(6):721-727.
[31] KANU N, CERONE M A, GOH G, et al. DNA replication stress mediatesAPOBEC3family mutagenesis in breast cancer[J]. Genome Biol,2016,17:185.
