基于变分推断的磁共振图像群组配准
2022-09-09王远军
周 勤,王远军
基于变分推断的磁共振图像群组配准
周 勤,王远军*
上海理工大学 医学影像技术研究所,上海 200093
为解决基于深度学习的成对配准方法精度低和传统配准算法耗时长的问题,本文提出一种基于变分推断的无监督端到端的群组配准以及基于局部归一化互相关(NCC)和先验的配准框架,该框架能够将多个图像配准到公共空间并有效地控制变形场的正则化,且不需要真实的变形场和参考图像.该方法得到的预估变形场可建模为概率生成模型,使用变分推断的方法求解;然后借助空间转换网络和损失函数来实现无监督方式训练.对于公开数据集LPBA40的3D脑磁共振图像配准任务,测试结果表明:本文所提出的方法与基线方法相比,具有较好的Dice得分、运行时间少且产生更好的微分同胚域,同时对噪声具有鲁棒性.
深度学习;群组配准;变分推断;可变形配准
引 言
各类脑成像中,磁共振成像(magnetic resonance imaging,MRI)由于可以显示血液和其状态的微小变化、无损伤定位大脑的功能活动,帮助病患发现早期脑部疾病,已经成为使用最广泛的脑功能研究手段,并为许多脑部疾病的检测与治疗带来福音.图像配准因具有信息匹配、信息融合的功能而成为在疾病分析中不可或缺的一部分.图像配准是MRI分析中的一项常见任务,也是许多领域中的一个活跃研究课题.它将图像空间对齐到一个共同的解剖空间[1],多幅图像的配准可以作为成对(pairwise,PW)配准或群组(groupwise,GW)配准问题来处理[2].如下图1所示,PW配准指定某一图像作为模板,其他图像与模板配准;GW配准中包括一个联合优化问题,用整个序列的信息创建模板,以避免在后续研究中引入偏差.

图1 (a)成对配准(PW)和(b)群组配准(GW)图像配准示意图.(a)中M代表浮动图像,F为一组图像中任意一幅图像作为模板,f为变形场,(b)中模板由数据集中N幅图像信息联合构建
过去几十年,图像配准算法得到长足的发展.经典的配准算法和基于学习的方法受到很大关注,比较有代表性的算法有elastic模型[3]、B样条[4]和Demons[5].几何变换的微分同胚性是当前配准领域非常看重的优点,比较有代表性的算法有LDDMM[6]、DARTEL[7]和Syn[2].对于以上方法,大多数配准的模板都是任意指定的.但在图像集中随机选择的作为模板的图像往往不能代表图像集的结构变形和复杂性,并可能导致偏差和误导性分析,所以研究GW图像配准具有很重要的意义.近年来GW配准在配准领域越来越受欢迎,是因为它能提供更多有用的信息.Guimond等[8]提出一种建立平均解剖模型的方法,该方法在单个图像(模板图像)中提供平均强度和平均形状,以平均的方式消除了脑形状和强度变化.类似于文献[8]的方法,Seghers等[9]通过选择每个图像作为模板来对齐所有图像,并且使图像与平均变形场非刚性对齐.Wu等[10]提出使用自适应加权策略的SharpMean配准方法,进一步构建基于特征的GW配准方法,该方法在配准过程中实现解剖学上合理的对应.以上提出的几种GW配准算法都是将图像配准到它们的相似图像,其中相似图像称为中间模板[11,12]. Wang等[13]提出一种类似金字塔式的配准框架,该框架可以有效地配准大的图像数据集,配准性能相对较好.随着深度学习技术的兴起,许多研究者也在探索深度神经网络在GW图像配准中的应用.Che等[14,15]提出一个由主成分分析构建的模板图像引导的无偏差的深度GW配准框架,适用于多光谱图像.类似于文献[14,15]的方法,Haase等[16]使用了鲁棒的主成成分分析方法.不同的是,在变分正则化的基础上,他们提出基于一阶原始对偶优化的多级方案来解决由此产生的非参数配准的问题.由于单个模板可能无法捕捉数据集的可变形,Dalca等[17,18]提出一种用于产生条件模板的配准框架和学习策略.类似于文献[17,18]使用可变模板的范例做法,Siebert等[19]提出使用自动编码器实现GW配准的方法,以无监督学习方式学习图像的形状和外观.遵循可变形模板的范例,将其应用到图像集上对齐.最近,He等[20]提出一种无监督的端到端GW框架,该框架具有多步机制来逐步优化输出的变形场,而无需模板.这项工作主要用于二维医学图像配准任务,且预估的变形场不是微分同胚域.Balakrishnan等[21]提出一种基于卷积神经网络(convolutional neural networks,CNN)的无监督PW配准方法,他们使用了一种类似于U-Net的架构,并将其命名为voxelmorph.后来,他们扩展了该方法,提出一个基于无监督学习的推理算法,并将其命名为voxelmorph-diff[22].实验结果表明,他们提出的算法有助于提高Dice系数,性能与ANT和NifTYG相当,但是计算效率是ANT的150倍,是NifTYG的40倍.voxelmorph-diff和voxelmorph是近年来基于深度学习的PW配准领域比较经典的方法,计算效率也高于现有的一般方法.
本文提出一种基于变分推断的无监督端到端的GW图像配准方法.首先,使用平均输入图像的方式构建模板,将输入图像和模板图像在通道处堆叠并送入CNN学习;然后基于变分推断对变形场进行预估,得到的变形场可实现将训练图像变换到模板的公共空间.该方法在训练过程中不需要参考图像和真实的变形场.这种学习策略类似传统的优化迭代方法,但模型和参数由神经网络和其权重代替,可以利用随机梯度下降对模型进行优化.同以往的方法相比,主要创新性如下:(1)不同于传统的GW配准,提出了一个以无监督深度学习方式优化的GW配准框架,它可以同时直接输出所有输入3D图像的位移场,不需要输入参考图像,而且该框架不需要对齐的图像对.(2)为增强所提出框架对噪声和强度变化的鲁棒性,我们使用局部归一化互相关(normalized cross correlation,NCC)来衡量模板和扭曲输入图像之间的相似性损失.(3)与其它基于学习的配准方法相比,所提出的的方法具有网络设计简单,配准精度与Syn算法相当,且比voxelmorph-diff,voxelmorph算法速度更快等特点,并对噪声具有鲁棒性.
1 基于变分推断的无监督配准方法
1.1 相关理论
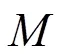
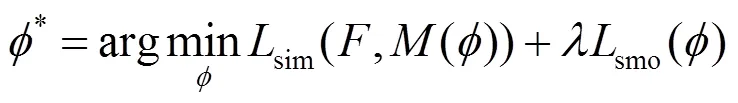
1.2 方法流程
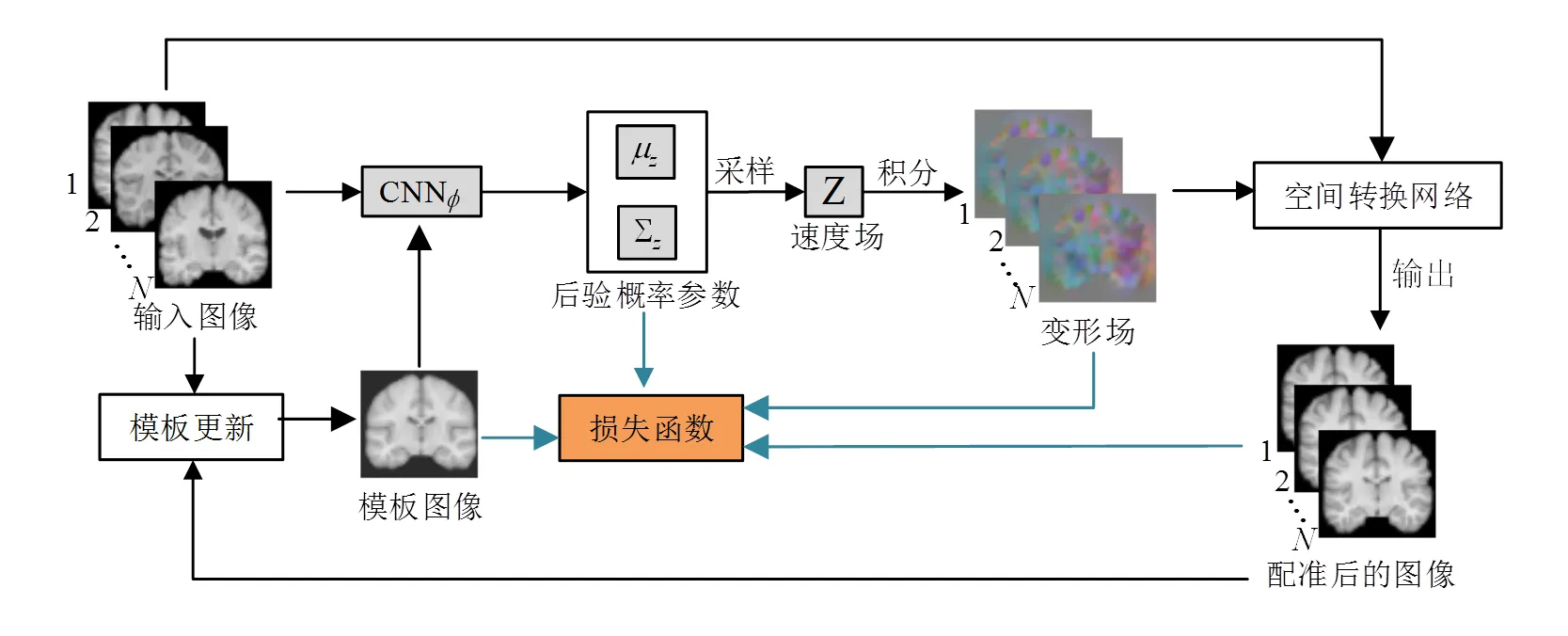
图2 基于变分推断的磁共振图像群组配准框架
1.3 模板更新
GW配准的一个关键点是对组均值图像进行鲁棒和精确的估计.基于微分同胚[2],我们使用如下步骤构建模板.
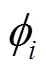
1.4 概率生成模型
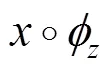
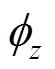
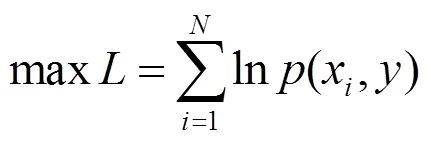
根据文献[22],对于每一个样本可知:
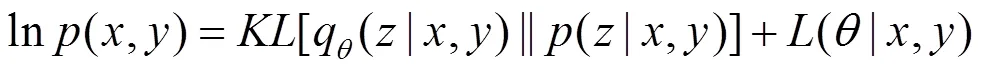
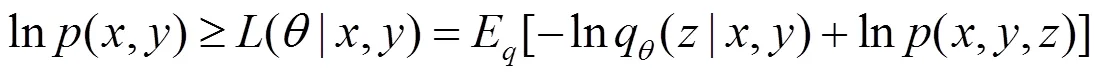
则有:
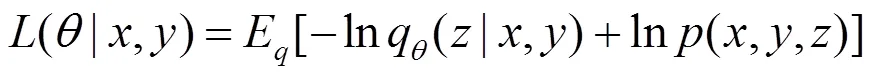
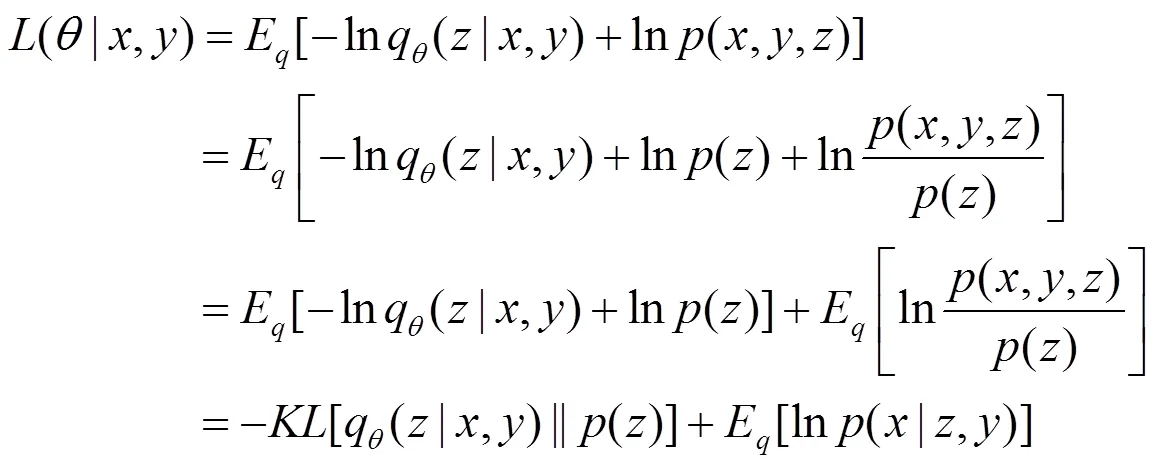

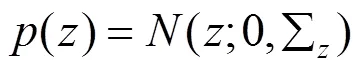
本文研究单模态变形配准,使用来测量衡量模板和扭曲输入图像之间的相似性损失,以增强其对噪声和强度变化的鲁棒性.任意两幅图像的局部归一化互相关可由以下公式计算:
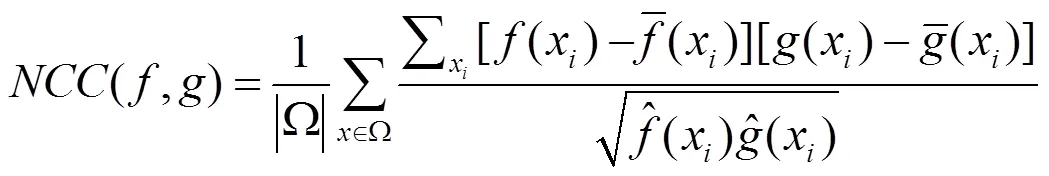

1.5 神经网络模型
GW方法旨在找到GW图像到模板空间的最佳变换,以及模板图像的计算.本文的网络架构包括解码网络、CNN、空间转换器和损失函数.在文献[22]中,Balakrishnan等明确的选择模板图像和运动图像来进行PW配准.相比之下,在我们的方法中,在输入到CNN网络之前,先将输入图像和生成的模板图像在通道堆叠并送入CNN网络,学习群组图像到模板图像的公共空间变换.
CNN模型由卷积、缩小/放大和跳跃连接组成,详细结构如图4所示.该架构由编码器和解码器两部分组成.网络采用通过将和连接成2通道3D图像而形成单个输入.在本文实验中,输入大小为160*160*192*2.编码阶段和解码阶段均使用内核大小为3的3D卷积,步长为2.在每个卷积操作后紧跟一个参数为0.2的LeakyReLU激励函数,增加网络的非线性,且步长均为1. 编码阶段使用4个步长为2、大小为3*3*3的卷积核进行下采样(最大值池化),使空间维度减半.编码阶段最终输出一个10*10*12*32的特征图.解码阶段使用4个上采样层和1*1*1卷积核.最终输出两个160*160*192*3大小的特征图分别对应近似后验概率的均值和协方差.由于上采样过程易造成信息丢失,我们在编码阶段和解码阶段之间加入跳跃连接(skip connection).其中,跳跃连接层可以将编码阶段阶段学到的信息对应传到解码阶段阶段,起到了提供更精细空间尺度的作用,从而补充信息,使得精确配准成为可能.
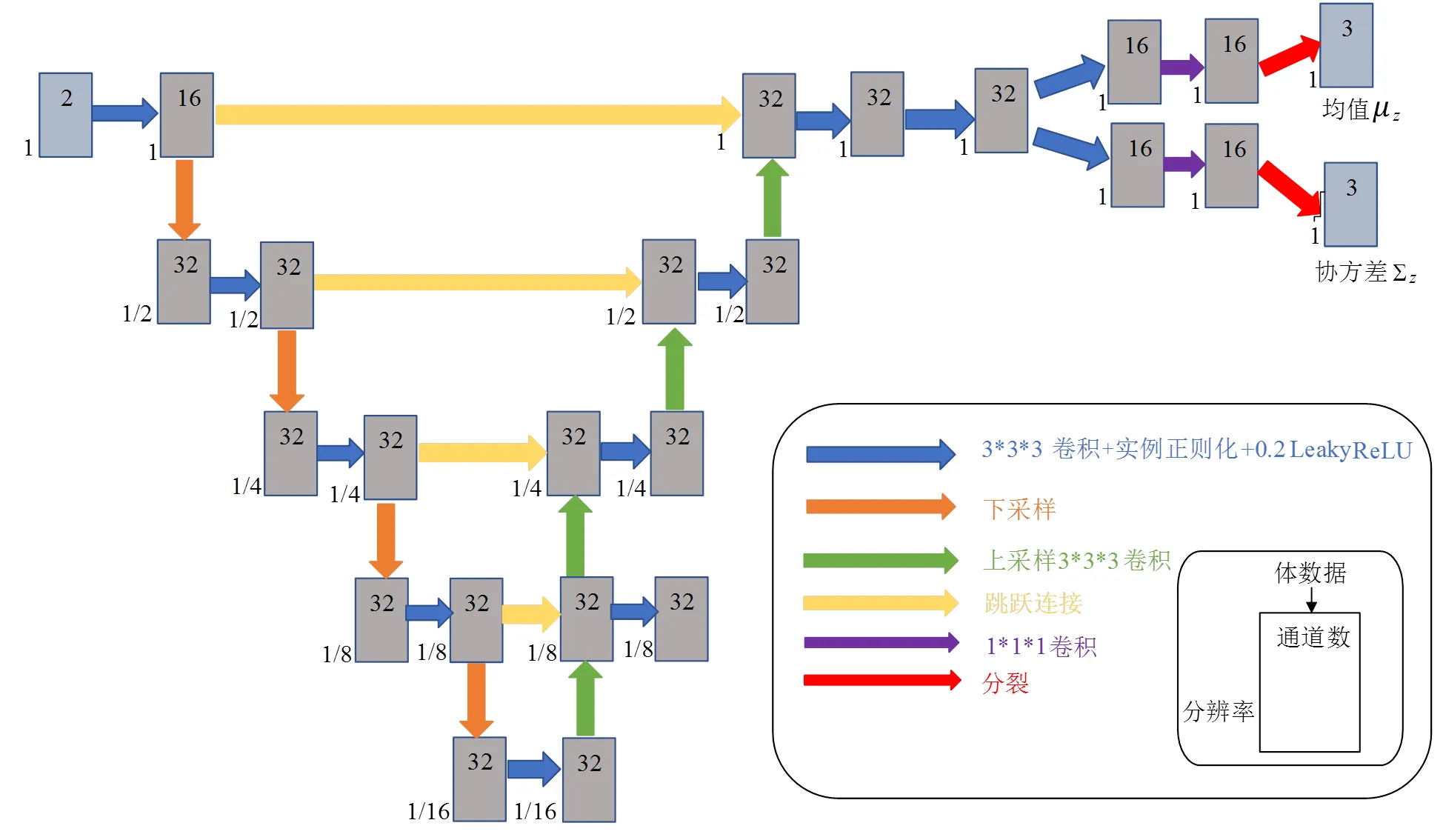
图4 本文使用的CNN网络框架
总体结构与大多数医学图像配准网络使用的U-net相同.但是,为满足GW配准的需要,做了以下几处改变:
(1)在原始的U-net中,下尺度层和上尺度层是通过最大池化和转置卷积实现的,取而代之的是一个更为简单的群组网格.
(2)批次数量将始终为一,因为在优化过程中,只有一组图像被送入网络.因此,批处理规范化被实例规范化所取代.
(3)卷积-归一化-激活操作的两个连续集合被减少到一个.这一变化提高了效率,但不影响性能.使用泄漏整流激活层(LeakyReLU)来代替原始整流线性激活(ReLU).
(4)由于内存大小限制,输入图像在输入CNN之前被缩小到原始分辨率的一半,然后将输出位移场提升到原始分辨率,用以变换输入图像.
1.6 损失函数
制定的损失函数旨在优化模型和减少公共空间生成的偏差.通过不断优化损失函数从而指导模型的学习.本文使用的损失函数为
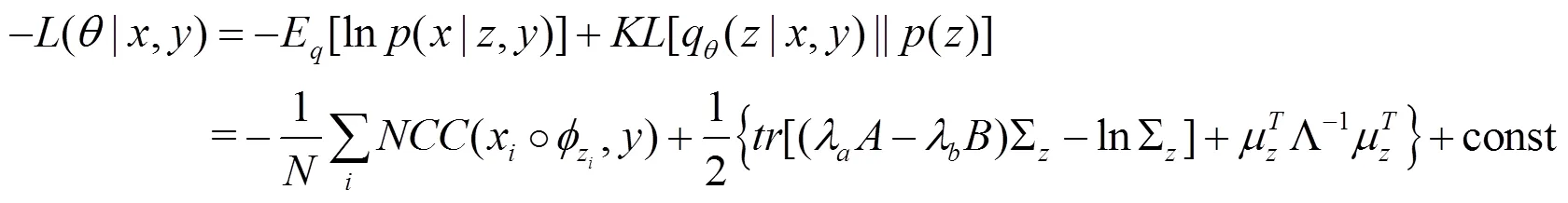
2 实验部分
2.1 实验描述
为验证本文提出算法的性能,使用公开数据集LONI LPBA40[23]进行实验.对数据集的所有3D磁共振图像都使用FSL和FreeSurfer软件进行偏移场矫正、大脑提取及线性配准、体素重采样至 1 mm*1 mm*1 mm和尺寸裁剪为160*160*192等预处理.并将含有40个3D磁共振图像的LPBA40数据集划分为训练集(30)、测试集(10).每个3D图像均有对应的标签(54个脑区标记),这些标签可用于评价配准的精度.
2.2 深度学习模型训练与测试
本文构建的深度模型使用无监督的学习方式进行训练.将初始模板和训练数据在通道维度上堆叠,然后送到CNN网络学习得到变形场,将得到的变形场作用于训练图像,输出配准后的图像,随后,更新模板直到所有图像都与最新的模板图像对齐.同训练一样,测试时将测试样本的总和取平均输入到训练好的模型进行多次迭代,计算迭代后的平均变形场.当迭代的变形场接近正交网格时,迭代终止,得到的最新模板作为测试时的模板图像.将得到的模板图像和测试图像输入到训练好的模型,将得到的变形场作用于测试图像标签,得到形变后的标签图,通过这种方式计算不同标签的Dice值.
我们使用Tensorflow在6-core Intel i7-8700K CPU和6 GB NVIDIA GeForce RTX 2060 GPU机器上实现模型的训练与测试.实验环境为DUDA10.0并行计算架构,操作系统为Win10,软件为PyCharm.并设计了以下两个不同的实验:(1)LPBA40数据集配准实验,以评估算法的配准精度;(2)在LPBA40数据集上叠加不同参数的噪声,以分析算法对噪声的鲁棒性.
本文还选取配准精度较高的Syn、voxelmorph-diff和voxelmorph三种算法进行了对比,其中后面两种方法是基于深度学习的经典的可变形PW配准算法.Syn使用ANTsPy实现,使用互相关(cross correlation,CC)系数作为其度量标准.voxelmorph-diff和voxelmorph两种方法使用与文献[22]相同的网络参数进行训练.
2.3 评估方法

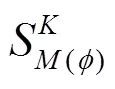
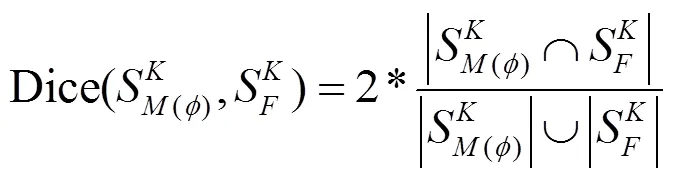
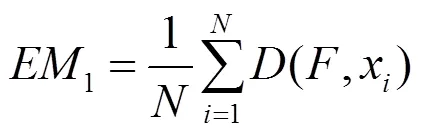
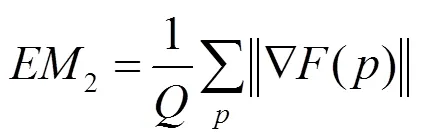
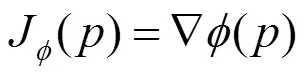
3 结果与讨论
3.1 LPBA40数据集配准实验
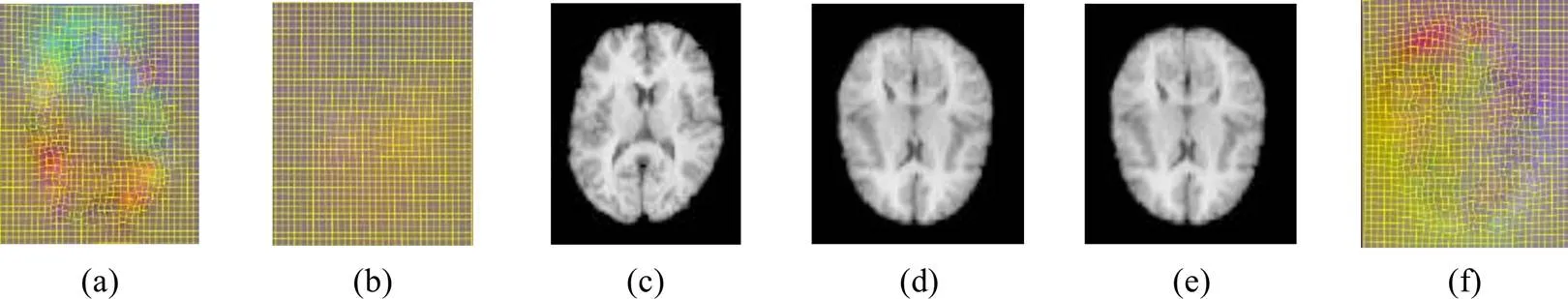
图5 测试结果中心切片:(a)第一迭代的变形场;(b)第二次迭代的变形场;(c)浮动图像;(d)模板图像;(e)配准后的图像;(f)变形场
表1 不同算法在LPBA40测试集上配准结果的多指标分析
*:括号内指方差. Avg. Dice为54个脑区配准后Dice的平均值.
GW配准的重点是将每幅图像变形到的公共空间,因此获得一幅具有代表性的模板图像是组配准的关键.其核心是找到一个无偏的模板图像,将其他图像配准到图像的公共空间.但很难验证模板的无偏向,我们取相关算法配准后的组均值图像,如图7所示,结果表明,我们的方法生成的模板图像比voxelmorph和voxelmorph-diff方法更清晰,其结构与Syn方法的结果大致一致,这意味着我们的方法至少与基线一样无偏.
3.2 本文方法对噪声的鲁棒性分析
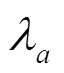
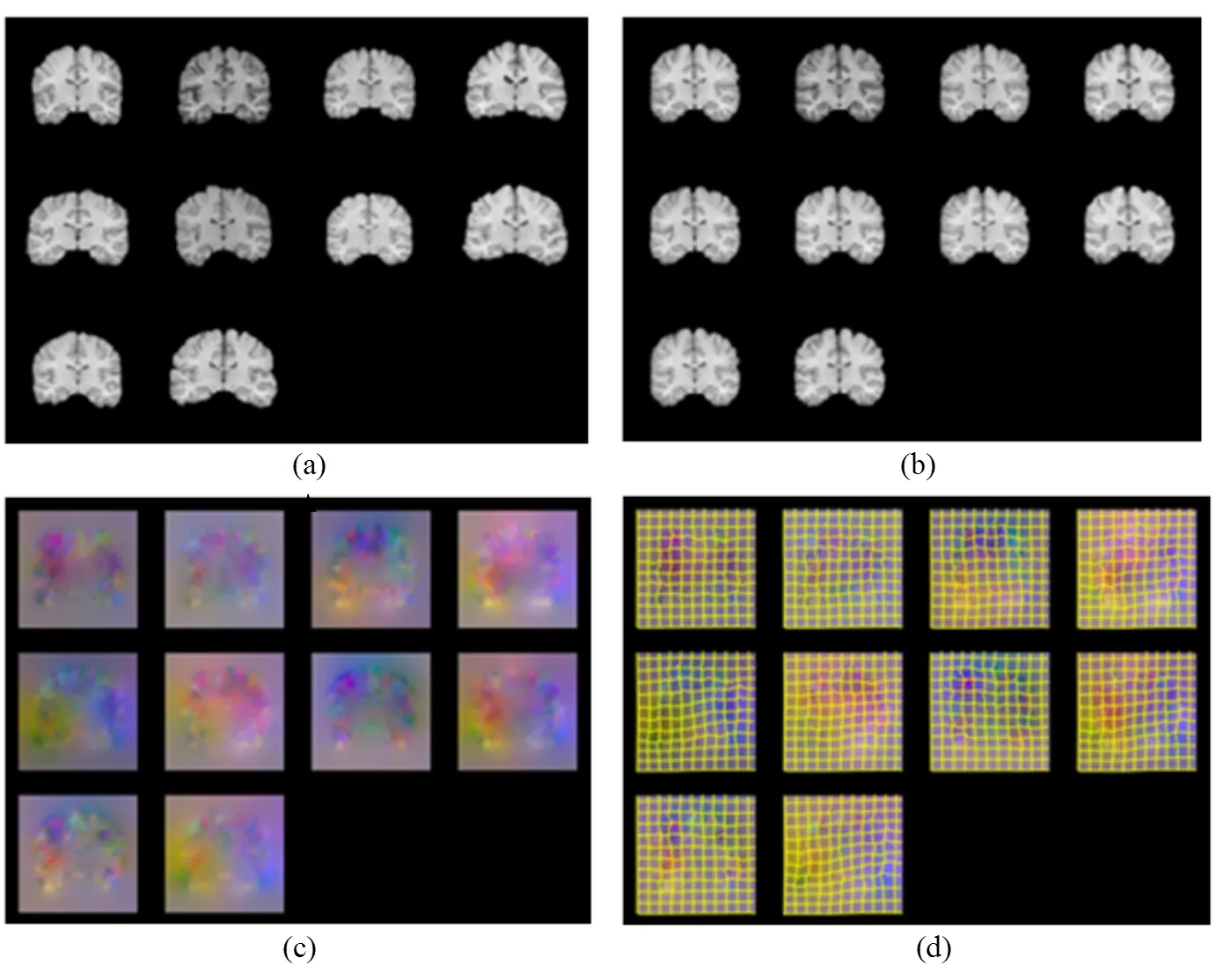
图6 基于本文算法得到的10幅脑磁共振图像冠状位中心切片测试集群组配准结果.(a)原图像;(b)配准后的图像;(c)变形场的彩色图像;(d)变形场的网格图像
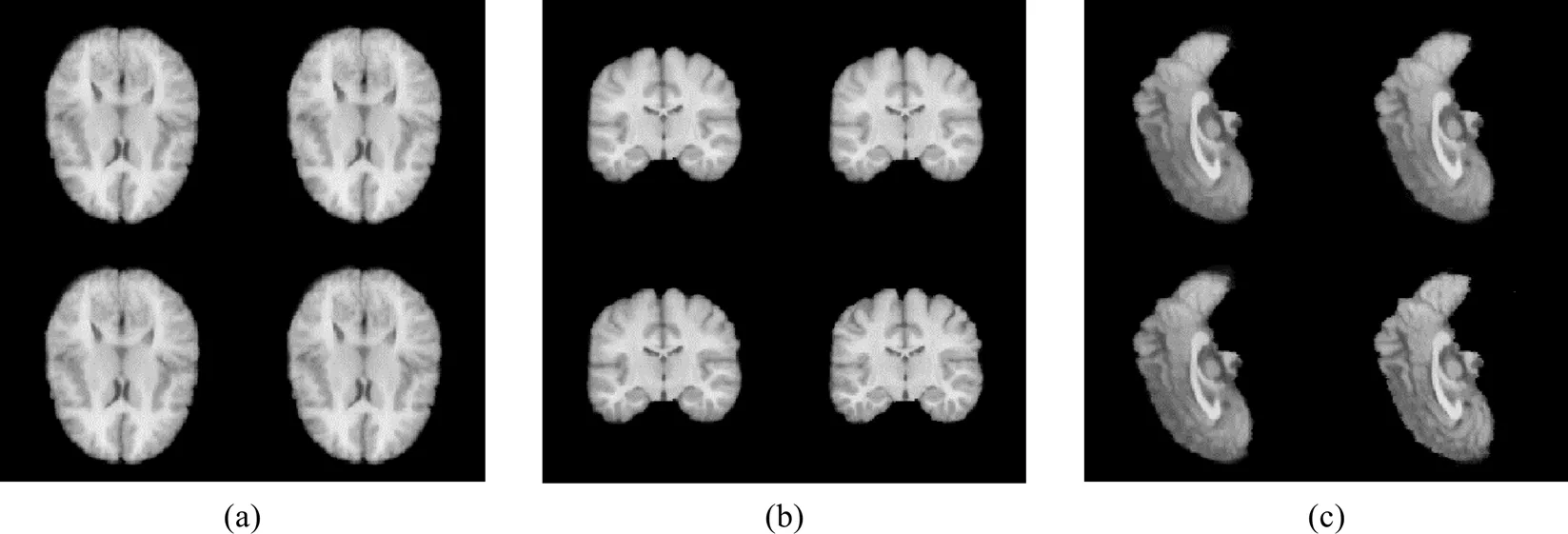
图7 不同算法得到的扭曲图像的均值图像.(a)矢状位中心切片;(b)冠状位中心切片;(c)水平位中心切片.每幅小图中第一行从左到右依次为voxelmorph算法的均值图像,voxelmorph-diff算法的均值图像.第二行从左到右依次为Syn算法的均值图像和本文方法的均值图像
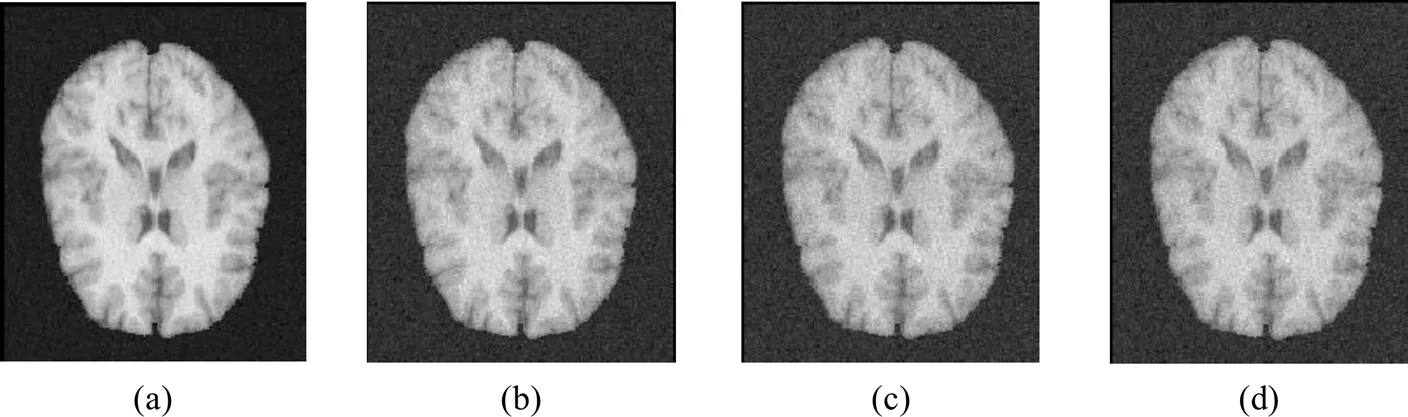
图8 不同噪声强度的浮动图像:(a)~(d)噪声均值均为0,方差分别为0.001、0.002、0.003、0.004
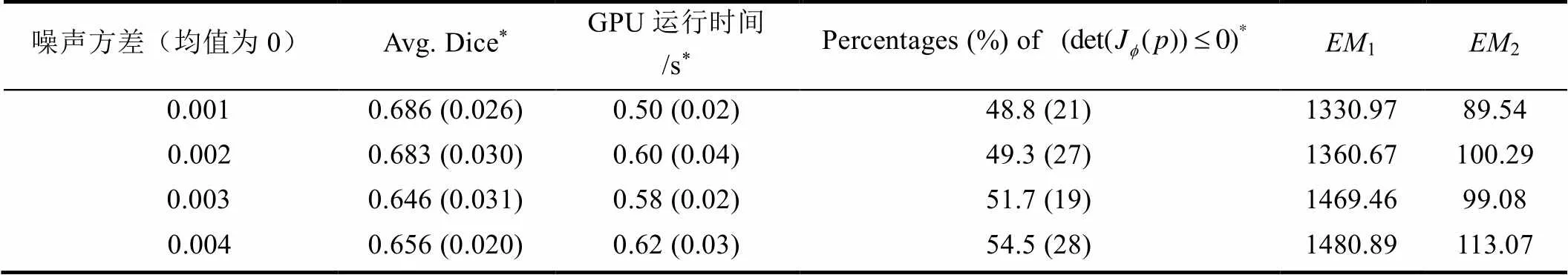
表2 使用本文方法对包含不同强度噪声的LPBA40测试集进行配准的多指标分析
*:括号内指方差. Avg. Dice为54个脑区配准后Dice的平均值.
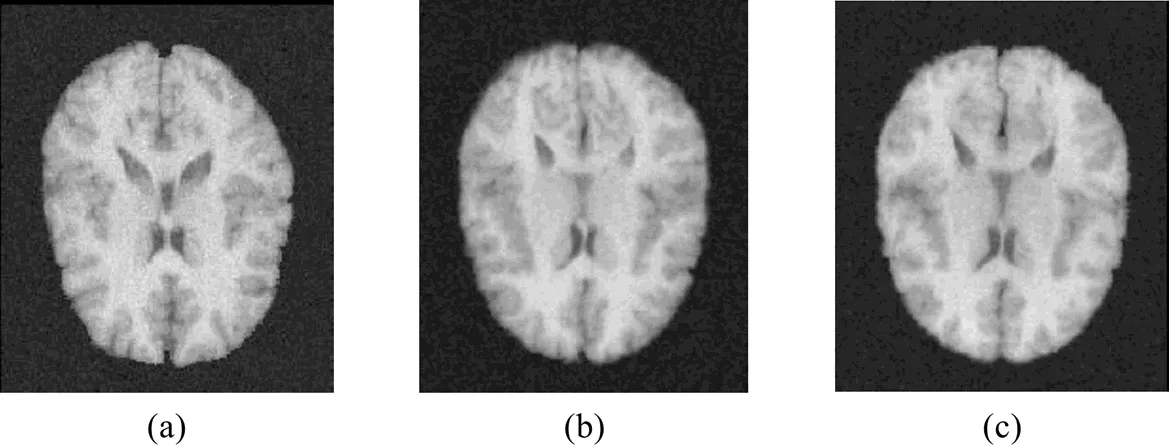
图9 使用本文算法对含均值为0、方差为0.002的噪声图像进行配准的结果:(a)浮动的图像;(b)构建的模板;(c)配准后的图像
4 结论
为解决传统配准算法配准时间长和精度低的问题,本文提出了一种基于变分推断的无监督GW配准方法.该方法将整个序列的信息融入到配准过程中,对预估的变形场使用概率生成模型进行建模,并用变分推断的方法求解.此外,我们设计一种基于NCC和先验的配准方法,以有效地控制变形场的正则化.利用LPBA40数据集配准实验以及方法相对噪声的鲁棒性分析实验,验证本文配准方法的性能.实验结果表明,我们的方法在多数评价指标上表现良好且具对噪声具有一定的鲁棒性.而且我们的GW配准模型可以同时配准多幅图像,其性能类似于Syn和基于学习的voxelmorph-diff,接近实时的GW图像配准.不足的是,尽管使用了微分同胚变换,但雅克比行列式也有负值且损失函数使用了过多的平滑项和正则化项,这可能增加近似误差的累积,并导致配准的速度下降.同时,本文对算法鲁棒性实验设计简单,后续可以考虑使用多线圈图像进行测试,并考虑将算法应用于多模态图像间的配准.
无
[1] Wang Y, Jiang F, Liu Y. Reference-free brain template construction with population symmetric registration[J]. Med Biol Eng Comput, 2020, 58(9):2083-2093.
[2] Martín-González E, Sevilla T, Revilla-Orodea A, et al. Groupwise non-rigid registration with deep learning: an affordable solution applied to 2D cardiac cine MRI reconstruction[J]. Entropy, 2020, 22(6):687.
[3] Gee J C, Reivich M, Bilaniuk L, et al. Evaluation of multiresolution elastic matching using mri data[J]. Proc Spie, 1991, 1445:226-234
[4] Rueckert D, Sonoda L I. Nonrigid registration using free-form deformations: application to breast mr images[J]. IEEE T Med Imaging, 1999, 18(8):712-721.
[5] Thirion J P. Image matching as a diffusion process: an analogy with Maxwell's demons[J]. Med Image Anal, 2011, 2(3):243-260.
[6] Zhong J, Phua D, Qiu A. Quantitative evaluation of lddmm, freesurfer, and caret for cortical surface mapping[J]. Neuroimage, 2010, 52(1):131-141.
[7] Asami T, Bouix S, Whitford T J, et al. Longitudinal loss of gray matter volume in patients with first-episode schizophrenia: dartel automated analysis and roi validation[J]. Neuroimage, 2012, 59(2):986-996.
[8] Guimond A, Meunier J, Thirion J P. Average brain models: a convergence study[J]. Comput Vis Image Und, 2000, 77(2):192-210.
[9] Seghers D, D'Agostino E, Maes F, et al. Construction of a brain template from mr images using state-of-the-art registration and segmentation techniques[C]// Medical Image Computing and Computer-Assisted Intervention--MICCAI 2004, 7th International Conference Saint-Malo, France, September 26-29, 2004, Proceedings, Part I. 2004.
[10] Wu G, Jia H, Wang Q, et al. Sharpmean: groupwise registration guided by sharp mean image and tree-based registration[J]. Neuroimage, 2011, 56(4):1968-1981.
[11] Wu G, Wang Q, Jia H, et al. Feature-based groupwise registration by hierarchical anatomical correspondence detection[J]. Hum Brain Mapp, 2012, 33(2):253-271
[12] Yanovsky I, Thompson P M, Osher S, et al. Topology preserving log-unbiased nonlinear image registration: theory and implementation[C]// IEEE Conference on Computer Vision & Pattern Recognition.Minneapolis, Minnesota, USA: IEEE, 2007.
[13] Wang Q, CHEN L Y yap p t, et al. Groupwise registration based on hierarchical image clustering and atlas synthesis[J]. Hum Brain Mapp, 2010, 31:1128-1140
[14] Che T, Zheng Y, Cong J, et al. Deep group-wise registration for multi-spectural images from fundus images[J]. IEEE Access, 2019, 7: 27650-27661.
[15] Che T, Zheng Y, Sui X,et al. Dgr-net: deep groupwise registration of multispectral images[C]// Information Processing in Medical Imaging - 26th International Conference , Hong Kong, china:IPMI, 2019:706-717.
[16] Haase R, Heldmann S, Lellmann J. Deformable groupwise image registration using low-rank and sparse decomposition[EB/OL]. [2020-06-10]. https://arxiv.org/abs/2001.03509.
[17] Dalca A V, Rakic M, Guttag J, et al. Learning conditional deformable templates with convolutional networks[C]//Neural Information Processing Systems 2019(NeurIPS 2019),Vancouver, BC, Canada, 2019:804-816.
[18] Yu E M,Dalca A V,Sabuncu M R. Learning conditional deformable shape templates for brain anatomy[M]. Lima: Machine Learning in Medical Imaging, 2020:353-352.
[19] Siebert H, Heinrich M P. Deep groupwise registration of mri using deforming autoencoders[M]. Berlin: Springer,2020:236-241.
[20] He Z Y, Chung A C S. Unsupervised end-to-end groupwise registration framework without generating templates[C]// 2020 IEEE International Conference on Image Processing (ICIP). IEEE, 2020:375-379.
[21] Dalca A V, Balakrishnan G, Guttag J, et al. Unsupervised learning of probabilistic diffeomorphic registration for images and surfaces[J]. Med Image Anal, 2019, 57:226-236.
[22] Balakrishnan G, Zhao A, Sabuncu M R, et al. Voxelmorph: a learning framework for deformable medical image registration[J]. IEEE T Med Imaging, 2019:1788-1800.
[23] Shattuck D W, Mirza M, Adisetiyo V, et al. Construction of a 3d probabilistic atlas of human cortical structures.[J]. Neuroimage, 2008, 39(3):1064-1080.
[24] WANG Y J, LIU Y.Group registration method based on topological center of images[J]. Chinese J Magn Reson, 2018, 35(4):60-67.
王远军,刘玉.基于图像集拓扑中心的群体配准方法[J]. 波谱学杂志, 2018, 35(4):60-67.
[25] CAI W Q, WANG Y J.Advances in the construction of the human brain map based on magnetic resonance imaging[J]. Chinese J Magn Reson, 2020, 37(2): 241-253.
蔡文琴, 王远军. 基于磁共振成像的人脑图谱构建方法研究进展[J]. 波谱学杂志, 2020, 37(2): 241-253.
[26] LIU K W, LIU Z L, WANG X Y, et al.Prostate magnetic resonance image classification based on cascading convolutional neural networks[J]. Chinese J Magn Reso, 2020, 37(2): 152-161.
刘可文, 刘紫龙, 汪香玉, 等. 基于级联卷积神经网络的前列腺磁共振图像分类[J]. 波谱学杂志, 2020, 37(2): 152-161.
Groupwise Registration for Magnetic Resonance Image Based on Variational Inference
,*
Institute of Medical Imaging Engineering, University of Shanghai for Science and Technology, Shanghai 200093, China
To address the low precision of pairwise registration method based on the deep learning and the time-consuming nature of traditional registration algorithm, this paper presents a method of unsupervised end-to-end groupwise registration based on variational inference, as well as a registration framework based on normalized cross correlation (NCC) and prior knowledge. The framework can warp all images in the group into a common space and effectively control the deformation field of the regularization, and it doesn't need a real deformation field or a reference image. The estimation of deformation field by this method can be modeled as a probability generation model and solved by variational inference. Then unsupervised training is implemented with the help of spatial transformer network and loss function. The registration results of 3D brain magnetic resonance image from the public data set LPBA40 show that: compared with the baseline method, the proposed method has better Dice score, less running time, better diffeomorphisms domain, and is robust to noise.
deep learning,groupwise registration, variational inference, deformable registration
O482.53
A
10.11938/cjmr20212918
2021-05-13;
2021-08-27
国家自然科学基金资助项目(61201067);上海市自然科学基金资助项目(18ZR1426900).
* Tel: 13761603606, E-mail: yjusst@126.com.
